猪流行性腹泻病毒TaqMan检测方法的建立及基于S基因的遗传变异分析
2023-02-27顾文源范京惠左玉柱
翟 刚,顾文源,刘 涛,刘 莹,张 帅,范京惠*,左玉柱*
(1.河北农业大学动物医学院,保定 071001; 2.河北省兽医生物技术创新中心,保定 071001;3.河北省动物疫病预防控制中心,石家庄 050000; 4.瑞普(保定)生物药业有限公司,保定 071001)
猪流行性腹泻(porcine epidemic diarrhea, PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)引起的一种急性、传染性强的肠道疾病[1-2]。可导致猪产生腹泻、呕吐、厌食和脱水等临床症状,仔猪感染该病后的死亡率极高[3-4];此外,该病具有接触传播的特性,可以通过污染的水、饲料等途径进行传播,这就限制了猪以及副产品的进出口贸易[3,5-6]。因此,该病毒严重影响了养猪业的健康发展,研发快速、灵敏的检测技术,制定正确的防控策略是极为重要的。PED最早于1971年出现在英国,随后于1978年首次分离该病毒株,并命名为CV777[7-8]。此后在日本、韩国和泰国等国家相继发现PEDV,在2010年以前,PEDV呈零星分布。2010年至今,PEDV变异毒株呈现出致病性更强、流行范围更广的特点,这就给PEDV的防控带来了更大的压力[3,9-10]。
PEDV为带囊膜的单股正链RNA病毒,属于甲型冠状病毒属,其基因组全长约为28 kb,共含有7个开发阅读框,分别编码ORF1a、ORF1b、Spike(S)、ORF3、envelope(E)、membrane(M)、nucleoprotein(N)[9,11-13]。其中,S蛋白为PEDV的纤突蛋白,是病毒表面的最大结构蛋白,含有多个中和表位,是其主要的抗原蛋白,在病毒入侵宿主细胞和诱导机体产生中和抗体等方面发挥着重要的作用。编码S蛋白的S基因是PEDV的重要毒力基因,该基因的碱基突变、插入和缺失会造成PEDV毒力的增强或减弱。该基因还是PEDV分型的重要依据。因此,深入研究S基因的结构以及生物特性有助于掌握PEDV的遗传变异规律。目前有多种PEDV的检测方法,狄亚心等[14]建立了RT-LAMP检测方法,李飞等[15]建立了SYBR Green Ⅰ qPCR检测方法,但均存在耗时长、敏感性差等问题。因此,在PEDV的防控方面,建立一种特异性强、敏感性高、耗时短的TaqMan qPCR检测方法,优先对猪场进行PEDV的快速全面筛查,再对阳性样品进行S基因扩增分析,可以实现对猪场不同流行毒株的检测,在PEDV病原诊断和基因结构分析等方面具有更广阔的应用前景。
本研究根据GenBank登录的PEDV毒株AJ1102的M基因保守序列设计了一对荧光引物和探针,建立了一种敏感性高、特异性强、操作简便的TaqMan qPCR检测方法,并对检测为阳性的样品进行S基因扩增。本研究不仅可以实现对PEDV不同毒株的检测和病毒拷贝数准确定量、还为猪场预防和控制PED的发生及传播提供理论依据。
1 材料与方法
1.1 病料的采集与处理
2021—2022年,自河北省规模化养猪场采集疑似PED腹泻样品(粪便或小肠内容物)。猪圆环病毒2型(PCV2)、传染型胃肠炎病毒(TGEV)、猪繁殖与呼吸综合征病毒(PRRSV)、伪狂犬病毒(PRV)、猪瘟病毒(CSFV)以及猪丁型冠状病毒(PDCoV)均由河北农业大学动物医学院预防兽医学实验室保存。
1.2 主要试剂
反转录试剂盒购自北京聚合美生物科技有限公司;DH5α感受态细胞由本实验室制备;Fast TaqMan Mixture购自北京康为世纪生物科技有限公司;DNA纯化试剂盒、质粒小提试剂盒购自杭州倍沃医学科技有限公司;猪流行性腹泻病毒通用型实时荧光RT-PCR检测试剂盒购自国测生物;pMD19-T载体、DL2000 DNA Marker购自宝生物工程(大连)有限公司。
1.3 引物设计
参考GenBank上登录的PEDV毒株AJ1102的M基因保守序列应用Primer Premier 5.0软件设计特异性荧光引物PEDV-F/R和探针PEDV-P以及PEDVS全基因扩增引物PEDVS-F/R(表1),引物由生工生物工程(上海)股份有限公司合成。

表1 PEDV的引物及探针
1.4 qPCR标准品的制备及检测方法的建立
用PEDV引物PEDV-M-F和PEDV-M-R扩增从PEDV样品中提取RNA,并反转录成cDNA,PCR产物经琼脂糖凝胶电泳检测为阳性后,对其进行回收纯化,随后将产物与pMD19-T载体经连接转化、菌液PCR鉴定为阳性的样品送至上海生工生物技术有限公司进行测序鉴定。提取鉴定为PEDV阳性菌质粒作为qPCR标准品,通过Nanodrop 2000测定质粒浓度,换算为质粒拷贝数。
对反应条件的不同变量进行优化;将标准品进行10倍的倍比稀释,选取9个不同的浓度(1.09×109~1.09×101拷贝·μL-1)作为模板在最优反应条件下进行qPCR扩增,以Cq值为纵坐标,模板浓度的对数为横坐标建立标准曲线。对扩增结果利用Bio-Rad软件分析后建立标准曲线。
1.5 特异性试验
用DNA/RNA提取试剂盒提取PCV2、PRV样品的DNA,TGEV、PRRSV、CSFV及PDCoV样品提取RNA并反转录成cDNA作为模板,采用上述优化好的方法进行qPCR,检验该方法的特异性。
1.6 敏感性试验
将标准品进行10倍倍比稀释作为模板在最优反应条件下进行qPCR扩增,同时利用普通PCR方法进行扩增,对两种检测方法得到的结果进行对比来验证本方法的敏感性。
1.7 重复性试验
选取经10倍倍比稀释的标准品作为模板,应用建立的方法进行组内试验,每个浓度重复3次;在不同的时间对上述选取的质粒标准品进行组间试验,每个浓度重复3次,根据每个浓度的Cq值计算组内和组间的变异系数(CV)。
1.8 临床样品检测
将本实验室采集的54份仔猪腹泻疑似样品提取RNA,并反转录成cDNA,采用建立的qPCR检测方法进行扩增,同时采用猪流行性腹泻病毒通用型实时荧光RT-PCR检测试剂盒进行检验,比较二者的检测结果。
1.9 S全基因的扩增及序列分析
将通过qPCR检测为PEDV阳性的样品进行普通PCR扩增,PCR产物经琼脂糖凝胶电泳检测为阳性后,对其进行回收纯化,随后将产物与pMD19-T载体经连接转化、菌液PCR鉴定为阳性的样品送上海生工生物技术有限公司进行测序鉴定。
从NCBI GenBank上选取具有代表性的毒株35条,用MEGA7.0软件构建遗传进化树,用DNAStar7.0软件进行氨基酸序列分析。
2 结 果
2.1 PEDV标准品的构建及qPCR检测方法的建立
将构建的重组质粒pMD19T-PEDV作为模板,用合成的PEDV-F/PEDV-R进行普通PCR扩增,得到目的条带大小为124 bp,胶回收送生工生物工程(上海)股份有限公司测序,结果均与预期一致,表明正确构建了重组质粒pMD19T-PEDV标准品。通过Nanodrop 2000测定标准品的浓度,换算成拷贝数为1.09×1010拷贝·μL-1。
经过优化后最终确定qPCR反应体系为25 μL,含引物各1 μL,探针0.5 μL,2×Fast Probe Mixture 12.5 μL,RNase-Free ddH2O 7 μL及模板3 μL。反应程序:95 ℃ 30 s,95 ℃ 10 s;53 ℃ 25 s,40个循环,每个循环结束收集荧光信号。
采用优化后的qPCR方法对重组质粒pMD19T-PEDV标准品进行qPCR扩增,结果显示,标准品稀释度在1.09×109~1.09×101拷贝·μL-1时呈现良好的线性关系,回归方程式为y=-3.8x+40.23,扩增效率R2为99.8%,结果成立,可以对样品进行定量分析。
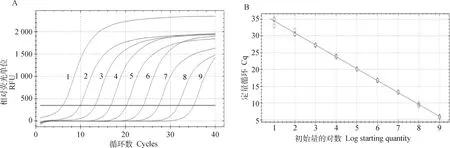
1~9.不同稀释度的重组质粒pMD19T-PEDV(1.09×109~1.09×101拷贝·μL-1)
2.2 特异性试验
利用qPCR方法对PEDV、PCV2、PCV3、TGEV、PRV、PRRSV、PDcoV及CSFV的核酸进行检测,同时设置阴阳对照。结果显示,仅PEDV为阳性,而其它病原均为阴性,表明本方法具有良好的特异性。
2.3 敏感性试验
利用qPCR方法对10倍倍比稀释的标准品进行检测,同时用普通PCR方法扩增检验其敏感性。结果显示,该方法对PEDV检测出的最低拷贝数为1.09×101拷贝·μL-1,而普通PCR方法的最低拷贝数为1.09×103拷贝·μL-1,表明本研究建立的TaqMan qPCR检测方法具有较高的敏感性。
2.4 重复性试验
将PEDV的标准品pMD19T-PEDV(1.09×107、1.09×104、1.09×102拷贝·μL-1)作为模板,采用建立的qPCR方法进行组内及组间的重复性试验,组内和组间的变异系数均小于1%,表明该方法具有良好的重复性。
2.5 临床样品检测
应用本研究建立的qPCR对来自河北省不同地区采集的54份疑似PED样品进行检测,结果显示,阳性率为27.8%(15/54),与猪流行性腹泻病毒通用型实时荧光RT-PCR检测试剂盒的检测结果一致,符合率为100%,并随机挑取3份阳性样品,经测序鉴定后,证实为PEDV,表明此次建立的方法可用于PEDV的流行病学调查。
2.6 PEDV S全基因扩增
将检测为PEDV的样品应用普通PCR方法对其进行S全基因的分段扩增,PCR产物经琼脂糖凝胶鉴定,结果显示,出现与预期片段大小符合的条带,随后测序及拼接。将获得的S基因上传至NCBI GenBank中,登录号为OL870433~OL870436、OL841533~OL841538。
2.7 PEDV S基因遗传进化分析及序列分析
为了解本研究的9株PEDV的遗传特性,利用MegAlign软件,将从NCBI公布的典型序列中选取35株PEDV毒株基因组序列与本研究得到的S基因进行比对。结果显示,本试验获取的9株毒株序列之间的核苷酸相似性为96.6%~99.1%,氨基酸的相似性为94.6%~98.7%;与参考株的核苷酸相似性为92.6%~99.3%,其中,与CHGD-01及BJ2011-1的亲缘关系较近,核苷酸相似性为98.3%~99.3%,氨基酸相似性为95.6%~98.9%。与CV777等经典毒株的亲缘关系较远,核苷酸相似性为92.6%~94.4%,氨基酸相似性为91.3%~93.8%;与近几年我国的流行毒株核苷酸的相似性为96.5%~99.3%。
应用Mega 7.0软件,将本研究分离得到的9条S全基因序列及从NCBI上参考的35条S全基因进行分析建立系统进化树,结果显示,进化树分为GⅠ和GⅡ大群,GⅠ和GⅡ分别分为GⅠa、GⅠb、GⅡa及GⅡb(图2)。结果显示,分离得到的9株PEDV毒株全部位于GⅡ群上。其中,CH/HB/BD01、CH/HB/BD02、CH/HB/BD03、CH/HB/CZ02、CH/HB/XT01、CH/HB/HD01、CH/HB/HD02与CHGDGZ和CHGD-01、GJL位于同一分支,属于GⅡa亚群;CH/HB/ZJK01和CH/HB/ZJK02分别与BJ2011-1、AH2012处于同一分支,属于GⅡb亚群。因此可以得出,本研究得到9条S基因的毒株为变异毒株,表明目前主要的流行毒株为变异毒株。

图2 PEDV S基因核苷酸序列遗传进化树
利用MegAlign软件,将得到的S基因与PEDV变异毒株BJ-2011-1进行序列比对,结果发现,9株S基因序列存在多处突变,处于GⅡb分支上的突变位点较多,为21~24处;分布于GⅡa分支上的毒株与CV777相似突变位点较多,共有11个。其中,CHHBBD02、CHHBBD03、CHHBCZ02、CHHBHD02、CHHBZJK01及CHHBZJK02存在1个氨基酸的插入。
3 讨 论
PED是养猪业常见的疾病之一,给养猪业造成了巨大的经济损失[16]。为了降低PED的发病率,人们通过接种疫苗的方法来防控PEDV,取得了一定的成效。然而,由于PEDV不断的变异,从而导致原有疫苗的保护力逐渐降低甚至失效,使得防控的难度逐渐加重[17-18]。在临床上,由于PEDV与PDCoV、TGEV等病毒引起的症状和病理变化相似性高,临床不能通过临床症状和病理变化作出准确的诊断。因此,建立一种快速、精准及敏感性高的诊断方法是极为重要的。目前,已有多种常规实验室诊断方法应用于PEDV的临床检测。但是,ELISA相较于TaqMan qPCR的方法具有灵敏度较低、成本高等缺点[19];普通PCR具有检测时间长、灵敏度相对较低的缺点[20];IFA、免疫组化及电镜等检测技术,对于仪器及人员的要求较高[21-22],且无法满足大批量的临床诊断。相较于以上几种方法,qPCR不仅具有操作简便、成本低的优点,而且检测时间短,可适用于基层的临床检测。在PEDV的分子研究机制上,监测PEDV毒力基因的变化对PED的防控可提供重要的理论依据。
本研究建立了一种快速、灵敏、准确的TaqMan qPCR检测方法。可实现在临床中对PEDV 的全面筛查。S蛋白是PEDV的关键毒力蛋白,其氨基酸的序列决定了蛋白的可塑性,该蛋白含有多个B细胞表位和抗体结合域,对病毒与抗体和免疫细胞的结合发挥着重要作用,其基因结构的改变可以对PEDV的毒力等生物特性产生巨大的影响[23-24]。依据本方法对本实验室从河北省在2021―2022年不同地区收集的腹泻样品进行筛查,将筛查出来的阳性样品进行S全基因的克隆,并进行S基因的遗传进化分析,从而了解当前流行毒株的基因结构,掌握其遗传变异规律。结果显示,克隆的9条S基因均位于GⅡ群的不同分支,与疫苗株CV777的亲缘关系较远。其中,CHHBBD01、CHHBBD02、CHHBBD03、CHHBHD01、CHHBHD02、CHHBCZ02和CHHBXT01与CHGD-01氨基酸序列的相似性最高为93.5%~97.6%,位于GⅡ-a分支;而CHHBZJK01和CHHBZJK02则与BJ-2011-1氨基酸的相似性最高为97.9%~98.9%,位于GⅡ-b分支。结果表明,目前,河北省PEDV的流行毒株与常规疫苗具有较大的差异。PEDV的S蛋白含有4个主要的中和表位区(499―639、748―755、764―771及1 368―1 374 aa),其中,499―639 aa为COE抗原区,该区的突变会对PEDV的毒力及抗原性产生很大的改变[25]。本研究通过对得到的9株PEDV S蛋白的氨基酸序列进行分析,结果显示,9株序列的氨基酸相似性为94.6%~98.7%,S蛋白氨基酸与国内流行毒株相比,均发生多处突变,并且部分突变与疫苗株CV777相同。此外,将9株S蛋白的氨基酸与GenBank登录的35条序列进行比对,结果发现,扩增的9株S蛋白均含有多处特异性突变,这些突变与PEDV的毒力的关系暂未研究。结果表明,在河北省部分地区毒株的抗原性等生物学功能可能已经产生变化。
4 结 论
建立了一种快速检测猪流行性腹泻病毒(PEDV)的通用型TaqMan qPCR检测方法,并对2021―2022年河北省不同地区采集的54份疑似PED样本进行检测,对检测出PEDV的样品进行S基因的扩增和测序,从序列的相似性分析和遗传进化研究两个方面得出河北省PEDV流行毒株分布在GⅡ群中的两个进化分支,且氨基酸序列发生多处特异性突变。
