核糖体小亚基V4和V9区引物扩增大亚湾真核浮游生物的比较研究
2023-02-22郭雨沛黄良民钟瑜陈靖夫谭烨辉陈绵润邱大俊
郭雨沛, 黄良民, 钟瑜, 陈靖夫, 谭烨辉, 陈绵润, 邱大俊,*
核糖体小亚基V4和V9区引物扩增大亚湾真核浮游生物的比较研究
郭雨沛1,2, 黄良民1,2, 钟瑜1, 陈靖夫1,2, 谭烨辉1,2, 陈绵润3, 邱大俊1,2,*
1. 中国科学院南海海洋研究所, 中科院热带生物资源与生态重点实验室, 广州 510301 2. 中国科学院大学, 北京 101408 3. 国家海洋局南海规划与环境研究院,广州 510300
对大亚湾水体的环境DNA分别进行18S rDNA的V4和V9区的引物扩增, 通过高通量测序技术进行测序, 并比较分析二者浮游真核生物基因多样性和相对丰度。18S rDNA V4区引物扩增共检测出浮游动物56 纲, 101 目, 浮游植物52 纲, 69 目; 18S rDNA V9区引物扩增共检测出浮游动物47 纲, 81目, 浮游植物56 纲, 101 目。两对引物对浮游真核生物扩增都具有较高覆盖度, 在纲级别上二者的结果相近: 颚足纲(Maxillopoda)是浮游动物优势类群; 甲藻纲(Dinophyceae)、圆筛藻纲(Coscinodiscophyceae)、小豆藻纲(Mamiellophyceae)是浮游植物优势类群, 其中甲藻纲多样性与丰度的结果相近, 而18S rDNA V9区引物扩增得到的圆筛藻纲丰度高于18S rDNA V4区引物。分析结果表明, 18S rDNA V4区引物扩增的浮游动物多样性比18S rDNA V9区引物高, 而18S rDNA V9区引物扩增的浮游植物多样性比18S rDNA V4区引物高。同时, 通过高通量测序技术首次确定大亚湾海区大量存在着寄生型甲藻(Syndiniales), 小豆藻目(Mamiellales)。
高通量测序; 真核浮游生物; 基因多样性; 浮游植物; 浮游动物; 大亚湾
0 前言
真核浮游生物是海洋生态系统的重要组成部分, 具有种类多样性高, 分布广等特征[1—2]。浮游植物和浮游动物是真核浮游生物的主要类群。浮游植物是海洋初级生产力的最主要的贡献者, 为维持生态系统的物质循环和能量传递提供了重要的基础[1]。浮游动物大多为初级消费者, 在物质循环、能量传递过程中起着重要的作用[3—4]。
近年来, 高通量测量技术大量应用到海洋真核浮游生物基因多样性的研究, 通过扩增环境样品中核糖体小亚基基因进行浮游生物多样性分析[5—8]。基于Illumina测样平台测序长度的限制, 分别选取核糖体小亚基不同的区域扩增测序[9], 其中碱基变异率较高的V4区和V9区应用最广, 这两个区域分别有一对引物最常使用[8—10], 但目前已开展不同引物扩增近海海域真核生物的比较研究甚少。目前已报道使用高通量测序研究真核浮游生物基因多样性的海域主要包括太平洋、大西洋(地中海、红海)、两极等诸多海域[8,11,12], 研究大多选取18S rDNA V4区和V9区引物。各海域真核生物呈现多样性高与高丰度等特征, 其中囊泡虫类(Alveolata)占比最多, 一般其序列的丰度占真核生物总丰度的25—40%[12—14], 有些研究区域高达50%, 如R. Piredda在地中海海域使用18S rDNA V4区引物研究中发现囊泡虫类占比高达56%, 其次为不等鞭毛藻类(Stramenopiles)为20%—25%[15]; 而Comeau等人使用18S rDNA V4区引物发现北冰洋附近海域的不等鞭毛藻占比较低, 在某些年份甚至低于5%[16]。常见的海洋囊泡虫类有顶复门(Apicomplexa)、纤毛虫(Ciliophora)以及甲藻门(Dinoflagellata)等。其中, 甲藻门占比最高, Wu等在太平洋西北沿岸海域研究显示其在微微型真核浮游生物中占比可接近60%[17]。Vargas等在Tara全球海洋考察中研究多个海域使用18S rDNA V4区引物和V9区引物发现, 大洋区域的甲藻门丰度占微微型真核浮游生物比例高于近岸海域。甲藻门的群落结构在不同海域分布有所差异, 近岸海域寄生型甲藻约占甲藻纲的一半, 而开阔大洋区域中寄生型甲藻比例有所提高[18]。Lin等人使用18S rDNA V4区引物在研究近岸、大陆架和外海中发现硅藻占比稳定, 约为40%[19]。韩郁烨等使用18S rDNA V4区引物在研究北欧海中发现, 微微型真核浮游生物群落的主要类群为囊泡虫类(54.61%),主要由甲藻纲(18.42%)和海洋囊泡虫新类群I(23.01%)组成;后鞭毛类占21.55%,其中真菌在各站中都有较高的相对丰度,为18.86%[20]。李冉等人通过18S rDNA V9区引物研究南海海域发现微型浮游植物主要以硅藻为主, 微微型浮游植物主要以绿藻为主[21]。但两对引物在近海与海湾中对于浮游动物与浮游植物的扩增结果有何差异的认识还不够系统深入, 因此有必要对此开展进一步研究。
大亚湾位于南海北部, 毗邻珠江河口, 受河口低盐水与外海高盐水共同影响。水体交换能力较弱, 受自然环境和人类活动的影响, 生态环境复杂多样, 是我国海洋生物多样性最高的区域之一[22—23]。大亚湾海域浮游生物使用传统的分类方法共鉴定出浮游植物8 门156 种[24—25], 浮游动物8 门275 种[26], 物种组成和丰度分布有明显的季节性和区域性差异: 夏季浮游生物多样性略高, 湾内多样性高于湾外[27—28]。目前, 大亚湾基于分子测序技术进行物种多样性的研究较少。Jiang等人使用rbcL区引物发现大亚湾浮游植物优势种为硅藻[29]。然而, 大亚湾海区还未系统开展高通量测序技术分析真核浮游生物基因的多样性及其群落结构及其不同引物的比较研究。
本文进行大亚湾水体环境DNA的18S rDNA V4和V9区引物扩增, 通过高通量测序技术进行测序, 分析大亚湾浮游真核生物的基因多样性及相对丰度, 并对两种引物扩增真核浮游生物的结果进行比较分析, 明确大亚湾真核浮游生物群落的组成, 评估两对引物在扩增海湾区域真核浮游动物与浮游植物的差异性和适用性。本文可为我国海湾与近海浮游真核生物基因多样性的研究提供方法参考与科学依据。
1 材料和方法
1.1 样品采集
2013年4月, 选取大亚湾常规调查站位S1、S9以及S10进行采集水样(图1)。使用5 L Niskin瓶采集表层水样, 水样通过3 µm滤膜过滤后, 再通过0.2 µm滤膜进行过滤, 分别收集3 µm和0.2 µm滤膜放入不同的1.5 mL离心管, 加入DNA lysis保护液进行常温保存[29]。回到实验室后, 转入-20 ℃冰箱中保存。

图1 大亚湾调查站位分布
Figure 1 Distribution of survey stations in Daya Bay
1.2 DNA提取与PCR扩增和测序
裂解缓冲液保存的样品加入蛋白酶K并置于55 ℃水浴24小时, 使用CTAB法提取DNA, DNA溶液保存在-20 ℃冰箱中[30]。
真核浮游生物的扩增分别选取目前最为常用的18S rDNA V4区和V9区的两对引物, 分别为V4F和V4R、V9F和V9R[8,16]。以1 μl提取的DNA作为模版, 采用25 μl反应体系(SuperStar Plus PCR MIX, Genestar)进行扩增。PCR退火温度分别为46 ℃(V4)和48 ℃(V9)。PCR扩增产物进行切胶纯化回收, 采用Illumina Novaseq技术测序平台(PE250)进行测序。
1.3 数据处理与分析
下机数据进行拼接、质控、过滤获得有效序列。通过降噪(Divisive Amplicon Denoising Algorithm)得到不含扩增与测序错误、不含嵌合体的生物学序列, 序列去重(Dereplication, 相当于以100%相似度聚类)获得单碱基精度的代表序列, 利用ASVs (Amplicon Sequence Variants)的概念将代表序列构建成的OTU(Operational Taxonomic Units)[27]。基于OTU数量通过 Vegan 包计算分析每个样品的稀释曲线[28]。分别筛选出每个样品浮游植物的 OTU 数据, 并与NCBI 数据库进行比对注释, 分析每个样品不同分类水平的OTU丰度与真核生物的群落结构组成(参数阈值: e value-5)。绘制不同分类水平真核生物群落结构组成图; 当物种数较多时, 选取序列条数大于全部样品测序总量0.01%的物种进行绘制。
2 结果和分析
大亚湾3 个站位水样通过18S rDNA V4区引物扩增得到486437 条序列, 单个样品扩增得到的序列条数为77719—84902 条序列, 共获得1519 条 OTU(表1)。18S rDNA V9 区引物扩增共得到446614 条序列, 单个样品扩增得到序列条数为63341—80664, 共获得1821 条OTU(表1)。18S rDNA V4区和V9区得到的OTU的稀释曲线均在测序sequence达到5 万条后逐渐平缓, 覆盖率达到99%, V4区和V9区引物扩增得到的序列能够覆盖并呈现出样品中真核浮游生物多样性(图2)。
2.1 两种引物扩增结果的比较
两对引物扩增得到的结果存在着明显的差异(图3)。18S rDNA V4区引物扩增出浮游生物6 类, 隶属于43 门, 94 纲, 184 目(表1)。18S rDNA V9区引物扩增出7 类 , 隶属于40 门, 85 纲, 171 目(表1)。在本研究中, V4区和V9区引物分别没有扩增得到古虫类和变形虫类(表2)。
18S rDNA V4区引物检测出浮游动物34 门, 56 纲, 101 目, 其中颚足纲占绝对优势, 同时检测到较高丰度的纤毛虫(图4e—h)。18S rDNA V9区鉴别出浮游动物31 门, 47 纲, 81 目, 其中颚足纲同样丰度最高, 但未检测到纤毛虫(图4e-h)。18S rDNA V4区引物扩增检测出浮游植物9 门, 52 纲, 69 目, V9区鉴别出9 门, 52 纲, 75 目(图5e-h)。18S rDNA V4区引物扩增结果检测得到甲藻门主要由寄生型甲藻、沟膝藻目和裸甲藻目组成, 其中寄生型甲藻占比最高; 硅藻门主要由海链藻目组成。18S rDNA V9区引物扩增结果检测得到甲藻门中膝沟藻目丰度最高, 硅藻门主要圆筛藻目组成, 绿藻门中小豆藻目占比最高(图5e-h)。两对引物对丰度较小的物种检测出的结果差距较大。

表1 大亚湾6个样品的序列与OTUs数量
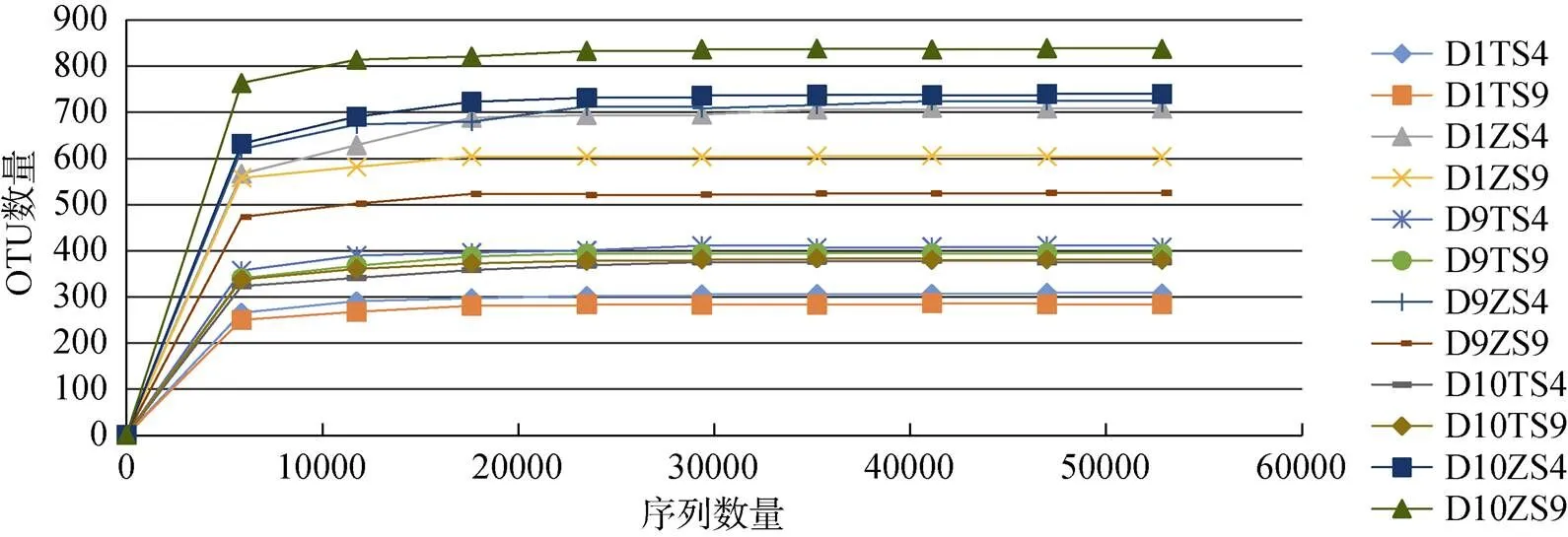
图2 样品稀释曲线
Figure 2 Rarefaction curve of each sample
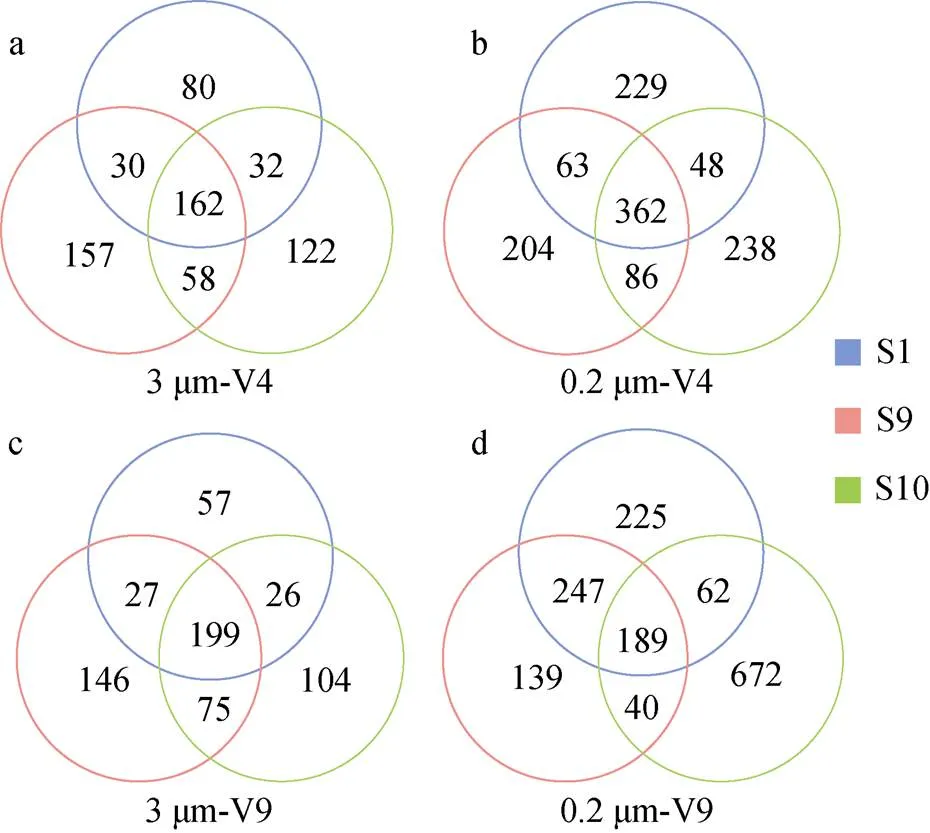
注: a. 三个站位小型和微型浮游生物V4区扩增得到OTUs数量Veen图; b. 三个站位微微型浮游生物V4区扩增得到OTUs数量Venn图; c. 三个站位小型和微型浮游生物V9区扩增结果得到OTUs数量Veen图; d. 三个站位微微型浮游生物V9区扩增得到OTUs数量Veen图。
Figure 3 Venn diagram of OTUs abundance using V4 and V9 primers amplification at three stations samples in Daya Bay
2.2 大亚湾真核浮游生物基因多样性
6 个样品共检测到真核浮游生物49 门, 107 纲, 415 目; 包括后鞭毛生物(Opisthokonta, 包括动物和真菌)、囊泡虫类、不等鞭毛藻、变形虫(Amoebozoa)、有孔虫(Rhizaria)、古虫(原Excata, 现Discoba)、绿色植物(Viridiplantae)等(表1)。18S rDNA V4 区引物共检测到小型和微型浮游生物(≥3 ) 有效序列611 条, 18S rDNA V9区引物共检测到有效序列1156 条, 鉴定出大亚湾小型和微型浮游生物6 类(没有检测到变形虫)(表2), 40 门, 81 纲, 168 目(表2); 其中占比最高的类群是后鞭毛生物, 其次为囊泡虫类和不等鞭毛虫; 优势门类为节肢动物门(Opisthokonta: Arthropoda), 甲藻门(Alveolata: Dinoflagellata), 硅藻门(Stramenopiles: Bacillariophyta) (表2)。18S rDNA V4区引物共检测到微微型浮游生物(0.2-3 µm)的有效序列1277 条, 18S rDNA V9 区引物共检测到有效序列1345 条, 检测出7 类微微型浮游生物, 49 门, 106 纲, 227 目; 其中占比最高的类别为囊泡虫类, 其次为不等鞭毛虫、浮游植物和未定地位的种类。优势门类依次为甲藻门, 节肢动物门, 绿藻门(Viridiplantae: Chlorophyta)。微微型浮游生物样品中未分类的OTU占比达到7%(图4a—d, 图5a—d)。

表2 大亚湾真核浮游生物大类丰度情况
2.2.1 大亚湾浮游动物基因多样性
三个站位共检测到小型和微型浮游动物24 门, 54 纲, 91 目。节肢动物门占绝对优势, 占比79.1%。其次为脊索动物门(Chordata), 占比6%。优势类群包括哲水蚤目(Calanoida)、剑水蚤目(Cyclopoida), 这两目占到全部种类的73%, 帘蛤目(Veneroida)等紧随其后(图4)。浮游动物丰度的优势种占据绝对地位。微微型浮游动物32门, 62纲, 92目。在浮游动物中节肢动物门仍然占优势, 但相对3 µm样品而言, 占比下降, 仅为28%。其次为环节动物门(Annelida)和纤毛门(Ciliophora)分别占14%和7%。优势类群包括叶须虫目(Phyllodocida)、环节哲水蚤目(Calanoida)、剑水蚤目(Cyclopoida), 这三目占比均匀, 占全部种类的45%, 与浮游植物类似, 一些无法准确鉴定的种类占到全部浮游动物的15%。
2.2.2 大亚湾浮游植物基因多样性
大亚湾小型和微型浮游植物10门, 23纲, 67目。甲藻门、硅藻门在浮游植物中占绝对优势, 甲藻门占比63%, 硅藻门30%。优势类群包括沟膝藻目(Gonyaulacales)、裸甲藻目(Gymnodiniales)、海链藻目(Thalassiosirales), 这三目占到全部种类的50%, 多甲藻目(Peridiniales)和硅藻目(Bacillariales)紧随其后, 相对丰度占比超过1%的均是甲藻门和硅藻门中的类别(图5)。
大亚湾微微型浮游植物9门, 27纲, 91目。甲藻门、绿藻门在浮游植物中占绝对优势, 甲藻门占比60%, 硅藻门17%, 其次为隐藻门(Cryptophyta)和硅藻门。优势类群包括寄生性甲藻目(Syndiniales)与小豆藻目(Mamiellales)这两目占到全部种类的43%, 另外, 未确定分类地位的类群中甲藻纲和绿藻纲占比也较高, 占浮游植物总序列的26%。
3 讨论
3.1 高通量测序测定真核浮游生物多样性的优缺点分析
基于Illumima MiSeq二代高通量测序平台的高通量测序技术, 较低的费用就能获得几十万到几百万条DNA分子序列, 检测出样品中不同的物种类群以及基因丰度比值[33,34]。本研究采用两组引物对大亚湾样品进行高通量测序分析, 鉴定样品的物种多样性。2013年4月大亚湾海域浮游植物中甲藻纲占比最高, 其次为圆筛藻纲或小豆藻纲, 浮游动物中则是颚足纲占比最高。高通量测序的结果与传统镜检对大亚湾调查结果不同, 大亚湾海区传统调查镜检发现在不同的季节, 各物种丰度有所变化, 但浮游植物最高丰度的类群都是硅藻门的圆筛藻纲, 其中以骨条藻占绝对优势, 其次为甲藻纲, 其中原甲藻目占比最高[35—38]。而浮游动物则以软体动物为主, 其次为节肢动物类。环境样品高通量测序得到真核浮游生物的相对丰度和物种多样性与常规镜检调查结果有所差异是正常现象[18]。基因多样性与形态多样性是两个不同的比较层级。即使是单个细胞中, 核糖体基因也可能存在多个拷贝, 不同种群生物之间的基因拷贝数差异很大。在一些物种中这种基因拷贝数差异可以高达3 个数量级, 因此基因扩增表达出的结果与不同物种的细胞丰度存在巨大差异[39,40]。同时这种多拷贝与生物体大小并无关系, 一些单细胞的甲藻和纤毛虫生物也被证实包含多个核糖体基因拷贝[41—42]。绿藻纲在高通量测序结果中占比比镜检数据高, 而传统镜检仅统计细胞较大绿藻的种类[35—38], 其原因是传统镜检方法很难对小体积高丰度的绿藻进行鉴定与计数。调查中微微型样品中绿藻的丰度远远高于微型样品, 这些小体积的绿藻无法通过镜检鉴定。研究中发现高通量测序得到的基因多样性高于常规镜检得到的物种数量, 常规镜检中很少观测到Chromerida、Mediophyceae等种类。高通量测序需要的采样体积需求量更少, 覆盖面更广, 获取的研究结果更为全面。其次, 由于物种基因多拷贝以及基因突变的存在, 目标基因片段存在多态性, 这也会造成基因多样性高估。以硅藻为例, 单个细胞的多态性可以达到0.5%—2%, 这种多态性也会高估其多样性[9,43]。V4区和V9区基因扩增均检测到样品中有大量的未命名序列, 这说明大亚湾存在许多小粒径的未鉴定藻类。浮游动物以及游泳动物迁移过程中, 停留在水体中的粪便与脱落的体表碎片同样会被高通量测序的检测到, 相对于传统的鉴定方法, 其更加灵敏。

注: 小型和微型浮游动物分别在门(a)、纲(e)、目(i)级别群落OTUs丰度图; 小型和微型浮游动物分别在门(b)、纲(f)、目(j)级别群落组成相对丰度图; 微微型浮游动物分别在门(c)、纲(g)、目(k)级别群落OTUs组成图; 微微型浮游动物分别在门(d)、纲(h)、目(i)级别群落组成相对丰度图。
Figure 4 Compared V4 and V9 marker absolute abundance and relative abundance of zooplankton at different taxonomic levels of three stations in Daya bay

注: 两种引物得到小型和微型浮游植物分别在门(a)、纲(e)、目(i)级别群落组成图; 两种引物得到小型和微型浮游植物分别在门(b)、纲(f)、目(j)级别群落组成相对丰度图; 两种引物得到微微型浮游植物分别在门(c)、纲(g)、目(k)级别群落组成图; 两种引物得到微微型浮游植物分别在门(d)、纲(h)、目(i)级别群落组成相对丰度图。
Figure 5 Compared V4 and V9 marker absolute abundance and relative abundance of phytoplankton at different taxonomic levels of three stations in Daya bay
大亚湾样品18S rDNA V4区与V9区引物扩增结果显示, 微型样品中浮游植物以甲藻、硅藻为主, 微微型样品以寄生性甲藻为主。北欧海区使用18S rDNA V4区引物研究微微型浮游植物中也发现寄生性甲藻为优势类群[20]。与rbcL引物主要针对自养藻类进行扩增[29], 而18S rDNA V4区和V9区引物能够扩增出异养的生物, 包括寄生性甲藻。
3.2 V4区引物与V9区引物扩增结果的比较
18S 区域变异相对保守, 是常见的真核浮游生物的目标区域[44], 目前使用较多的是18S rDNA V1—3, V4和V9区引物, V1—3(500bp左右)区域引物的目标条带相对保守, 只能鉴定到纲或门水平[43]。过去18S rDNA V4区和V9区引物的扩增的结果较少进行直接的对比分析。长片段序列在鉴定中结果更准确, 短片段序列在目以下的级别分类中, 不同的种属的序列特征常相同, 无法明确其在种属水平上的分类地位[18]。实验中18S rDNA V4区目标片段450bp, 用来比对的长度约为380bp, 而18S rDNA V9区引物目标片段250bp, 用来比对的长度约为170bp, 18S rDNA V9区引物由于扩增片段长度问题, 在较低级别上难以区分。本研究中使用的两对引物在鉴定物种的结果显示18S rDNA V9区比V4区引物分辨力更弱。
通过研究发现, 两对引物在浮游植物和浮游动物鉴定能力上有所区别, 18S rDNA V4引物扩增结果对浮游动物的覆盖度高于18S rDNA V9引物, 而18S rDNA V9区引物扩增结果能够更全面地覆盖浮游植物, 两对引物关于浮游植物鉴定的差距远低于浮游动物。张莉等在黄海区域中进行比较研究也发现基于18S r DNA V9扩增获得的浮游植物的多样性数目较于18S rDNA V4扩增获得的高[45]。以轮藻门为例, 两对引物的检测结果不仅在丰度上差异巨大, 多样性也难以覆盖。整体说来, 针对甲藻门和绿藻门浮游植物, 18S rDNA V9区的覆盖度高于18S rDNA V4区, 而两对引物对硅藻门以及其他藻类的的鉴定可以相互补充。两对引物都检测出大量未命名的甲藻门和绿藻门藻类, 其中18S rDNA V4区引物检测出的未命名藻类高于18S rDNA V9区引物的结果。18S rDNA V4与18S rDNA V9区变异程度不同, 两对引物在物种多样性以及丰度鉴定中存在着一定差异, 因此覆盖度也所差别[18]。18S rDNA V9区的短片段在低级别鉴定中无法区别序列相似物种导致低级别18S rDNA V9区多样性有所降低。除目标片段序列变异度的差异外, 18S rDNA V4 区引物三维立体结构更复杂[46], 在扩增以及测序过程中带来更多的少碱基变异产物, 因此在低分类水平上反而体现出物种多样性。这种由于结构复杂导致的突变同样可以解释18S rDNA V4区引物扩增产物在各物种中都存在比18S rDNA V9区引物更多的未命名物种藻类的现象。本次研究中发现18S rDNA V9区引物扩增出的硅藻类整体丰度较高, 但无法扩增纤毛虫类的生物。Stock等人指出18S rDNA V4 区引物能够扩增出一些特定的物种序列, 可能是因为18S rDNA V9 区引物更易扩增出其他物种, 削弱了其对一些物种的扩增[8], 凸显出18S rDNA V4区引物对一些种类的扩增能力。
4 结论
本次研究确定了18S rDNA V4区和18S rDNA V9区两对引物都能扩增出大亚湾浮游真核生物绝大多数类群, 但两对引物存在一定的差异。18S rDNA V4引物扩增结果中浮游动物的多样性高于18S rDNA V9引物的结果, 而18S rDNA V9区引物的结果浮游植物多样性高于18S rDNA V4区引物的结果, 同时明确18S rDNA V4区引物扩增序列的分辨率高于18S rDNA V9区引物扩增序列。明确了2013年4月大亚湾浮游真核生物具有很高的多样性, 共8 门, 22 纲, 51 目。小型和微型浮游真核生物中哲水蚤目种类的丰度最高, 浮游植物以甲藻门为主, 包括裸甲藻目以及膝沟藻目; 微微型浮游生物中浮游动物主要以叶须虫目为主, 浮游植物中甲藻为为优势类群, 确定了大量未定名种类的存在。首次确定了大亚湾海区存在着大量寄生性甲藻目与小豆藻目的藻类。
[1] HOPPENRATH M, M ELBRÄCHTER, DREBES G. Marine phytoplankton: selected microphytoplankton species from the North Sea around Helgoland and Sylt[M]. Journal of Phycology, 2010, 46.
[2] POMEROY L R. The ocean’s food web, a changing paradigm[J]. BioScience, 1974, 24(9): 499—504.
[3] 李少菁, 许振祖, 黄加祺, 等. 海洋浮游动物学研究[J]. 厦门大学学报(自然科学版), 2001, 40(2): 574—574.
[4] 刘建康. 高级水生生物学[M]. 北京: 科学出版社, 1999.
[5] SIERACKI C K, SIERACKI M E, YENTSCH C S. An imaging-in-flow system for automated analysis of marine microplankton[J]. Marine Ecology Progress Series, 2017, 168(1): 285—296.
[6] MASSANA R, PEDRÓS-ALIÓ C. Unveiling new microbial eukaryotes in the surface ocean[J]. Current Opinion in Microbiology, 2008, 11(3): 213-218.
[7] LAROCHE J, GEIDER R J, GRAZIANO L M, et al. Induction of specific proteins in eukaryotic algae grown under iron-deficient, phosphorus-deficient, or nitrogen- deficient conditions[J]. Journal of Phycology, 2010, 29(6): 767—777.
[8] STOECK T, BASS D, NEBEL M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water[J]. Molecular Ecology, 2010, 19: 21—31.
[9] HUGERTH L W, MULLER E, HU Y, et al. Systematic design of 18S rRNA gene primers for determining eukaryotic diversity in microbial consortia[J]. PLoS ONE, 2014, 9(4): e95567.
[10] ZHU F, RAMON M, FABRICE N, et al. Mapping of picoeucaryotes in marine ecosystems with quantitative PCR of the 18S rRNA gene[J]. FEMS Microbiology Ecology, 2010(1): 79—92.
[11] EGAS C, C HENRÍQUEZ-CASTILLO, DELHERBE N, et al. Short timescale dynamics of phytoplankton in Fildes Bay, Antarctica[J]. Antarctic Science, 2017: 217—228.
[12] ZHANG Wenjing, PAN Yongbo, YANG Jun, et al. The diversity and biogeography of abundant and rare intertidal marine microeukaryotes explained by environment and dispersal limitation[J]. Environmental Microbiology, 2018, 20(2): 462—476.
[13] LE GALL F, YOGEV T, BERMAN-FRANK I, et al. Diversity of active marine picoeukaryotes in the Eastern Mediterranean Sea unveiled using photosystem-II psbA transcripts[J]. Multidisciplinary Journal of Microbial Ecology, 2010, 4(8): 1044—1052.
[14] PERNICE M C, LOGARES R, GUILLOU L, et al. General Patterns of Diversity in Major Marine Microeukaryote Lineages[J]. PLoS ONE, 2013, 8(2): e57170
[15] PIREDDA R, TOMASINO M P, D’ERCHIA, A. M, et al. Diversity and temporal patterns ofplanktonic protist assemblages at a Mediterranean LTER site[J]. FEMS Microbiology Ecology, 2016, 93(1), doi:10.1093/femsec/fiw200.
[16] COMEAU ANDRÉ M, LI W K W, TREMBLAY JEAN-ÉRIC, et al. Arctic ocean microbial community structure before and after the 2007 record sea ice minimum[J]. PLoS ONE, 2011, 6(11): e27492.
[17] Wu W, Logares R, Huang B, et al. Abundant and rare picoeukaryotic sub-communities present contrasting patterns in the epipelagic waters of marginal seas in the northwestern Pacific3 / 3 Ocean[J]. Environmental Microbiology, 2016, doi:10.1111/1462-2920.13606.
[18] BESCOT N L, PROBERT I, CARMICHAEL M, et al. Eukaryotic plankton diversity in the sunlit ocean[J]. Science, 2015, 348(6237): 1261605.
[19] LIN Yajuan, NICOLAS C, ADRIAN M, et al. Specific eukaryotic plankton are good predictors of net community production in the Western Antarctic Peninsula[J]. Scientific Reports, 2017, 7(1): 14845.
[20] 韩郁烨, 郭萃, 段雪萍, 等. 北欧海微微型真核浮游生物的多样性和群落结构及其与环境因子的关系[J]. 海洋与湖沼, 2019, 50(6): 1263—1270.
[21] 李冉. 热带/亚热带海区原生生物分子多样性研究[D]. 厦门: 厦门大学, 2017.
[22] F徐恭昭. 大亚湾环境与资源[M]. 安徽: 安徽科学技术出版社, 1989.
[23] SONG Xingyu, HUANG Liangmin, ZHANG Jianlin, et al. Harmful algal blooms (HABs) in Daya Bay, China: Anstudy of primary production and environmental impacts[J]. Marine Pollution Bulletin, 2009, 58(9): 1310—1318.
[24] YANG Xi, TANG Yehui, LI Kaizhi, et al. Long-term changes in summer phytoplankton communities and their influencing factors in Daya Bay, China (1991–2017)[J]. Marine Pollution Bulletin, 2020(161): 111694
[25] 柯志新, 黄良民, 谭烨辉, 等. 2007年夏季南海北部浮游植物的物种组成及丰度分布[J]. 热带海洋学报, 2011(1): 133—145.
[26] 杜飞雁, 王雪辉, 贾晓平, 等. 大亚湾海域浮游动物种类组成和优势种的季节变化[J]. 水产学报, 2013, 37(8): 1213—1119.
[27] 廖秀丽, 陈丕茂, 马胜伟, 等. 大亚湾杨梅坑海域投礁前后浮游植物群落结构及其与环境因子的关系[J]. 南方水产科学, 2013, 9(5): 109—119.
[28] 唐森铭, 严岩, 陈彬. 春夏季大亚湾核电厂温排水对海洋浮游植物群落结构的影响[J]. 应用海洋学学报, 2013, 32(3): 373—382.
[29] JIANG Zhaoyu, WANG Youshao, CHENG Hao, et al. Spatial variation of phytoplankton community structure in Daya Bay, China[J]. Ecotoxicology, 2015, 24(7/8): 1450—1458.
[30] QIU Dajun, Huang Liangmin, LIU Sheng, et al. Nuclear, Mitochondrial and plastid gene phylogenies of dinophysis miles (Dinophyceae): evidence of variable types of chloroplasts[J]. PLoS ONE,2011, 6(12): e29398.
[31] BOKULICH N A, SUBRAMANIAN S, FAITH J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1): 57—59.
[32] CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. Multidisciplinary Journal of Microbial Ecology, 2012, 6(8): 1621—1624.
[33] TABERLET P, COISSAC E, POMPANON F, et al. Towards next‐generation biodiversity assessment using DNA metabarcoding[J]. Molecular Ecology, 2012, 21(8): 2045—2050.
[34] VALENTINI A, POMPANON F, TABERLET P. DNA barcoding for ecologists[J]. Trends in Ecology & Evolution, 2009, 24(2): 110—117.
[35] 粟丽, 黄梓荣, 罗艳. 大亚湾夏、冬浮游植物群落结构与环境因子[J]. 海洋科学进展, 2019, 37(2): 284—293.
[36] 姜歆, 柯志新, 向晨晖, 等. 黄良民. 大亚湾夏季和冬季超微型浮游生物的时空分布及环境调控[J]. 生态科学, 2018, 37(2): 1—10.
[37] 姜犁明, 赵明忠, 徐智焕, 等. 2011年秋季和2012年春季大亚湾海域浮游植物的分布及主要影响因素[J]. 海洋学研究, 2013, 31(2): 86—92.
[38] 魏雷. 大亚湾海域浮游植物群落结构调查——以2011—2012年为例[J]. 农业与技术, 2017, 37(16): 243—244.
[39] GONG Jun, DONG Jun, LIU Xihan, et al. Extremely high copy numbers and polymorphisms of the rDNA operon estimated from single cell analysis of oligotrich and peritrich ciliates[J]. Protist, 2013, 164(3): 369—379.
[40] P LÓPEZ-GARCÍA, F RODRÍGUEZ-VALERA, C PEDRÓS-ALIÓ, et al. Unexpected diversity of small eukaryotes in deep-sea Antarctic plankton[J]. Nature, 2001, 409(6820): 603—607.
[41] PAWLOWSKI J, JOSÉ FAHRNI, BOWSER S S. Phylogenetic analysis and genetic diversity of[J]. Journal of Foraminiferal Research, 2008, 32(2): 173—176.
[42] DIEZ B, PEDROS-ALIO C, MASSANA R. Study of genetic diversity of eukaryotic picoplankton in different oceanic regions by small-subunit rRNA gene cloning and sequencing[J]. Applied & Environmental Microbiology, 2001, 67(7), doi:10.1023/A:1022289509702.
[43] ALVERSON A, KOLNICK L. Intragenomic nucleotide polymorphism among small subunit (18S) rDNA paralogs in the diatom genus Skeletonema (Bacillariophyta)[J]. Journal of Phycology, 2010, 41(6): 1248—1257.
[44] BIK H M, PORAZINSKA D L, CREER S, et al. Sequencing our way towards understanding global eukaryotic biodiversity[J]. Trends in Ecology & Evolution, 2012, 27(4): 233—243.
[45] 张莉, 张远, 林佳宁, 等. 基于多个扩增子的DNA metabarcoding技术探究黄海微型真核浮游植物多样性[J]. 环境科学, 2019, 40(9): 206—214.
[46] WUYTS J. Comparative analysis of more than 3000 sequences reveals the existence of two pseudoknots in area V4 of eukaryotic small subunit ribosomal RNA[J]. Nucleic Acids Research, 2000, 28(23): 4698—708.
Comparison of eukaryotic plankton compositions using 18S rRNA gene V4 and V9 primers amplification in Daya Bay
GUO Yupei1,2, HUANG Liangmin1,2, ZHONG Yu1, CHEN Jingfu1,2, TAN Yehui1,2, CHEN Mianrun3,Qiu Dajun1,2,*
1. CAS Key Laboratory of Tropical Marine Bio-resources and Ecology, South China Sea Institute of OceanologyChinese Academy of Sciences, Guangzhou 510301, China 2. University of Chinese Academy of Sciences, Beijing 101408, China 3. South China Sea Institute of Planning and Environmental Research, SOA, Guangzhou 510300, China
We compared the genetic diversity and relative abundance of eukaryotic plankton in Daya Bay using two genetic markers(18S rRNA V4 and V9 regions) to amplify the eDNA and high-throughput sequencing technology. 56 classes, 101 orders of zooplankton and 52 classes, 69 orders of phytoplankton were detected using 18S rDNA V4 markers. And 47 classes, 81 orders of zooplankton and 56 classes, 101 orders of phytoplankton were detected using 18S rDNA V9 markers. Both 18S rDNA V4 and V9 regions resultsperformed well to coverage communities and provided similar eukaryotic plankton distribution patterns at class taxonomic level. Maxillopoda is the dominant group of zooplankton, while Dinophyceae, Coscinodiscophyceae and Mamiellophyceae are the dominant groups of phytoplankton. Especially, two genetic markers amplification results present the similar diversity and relative abundance of Dinophyceae at order taxonomic level. The results of 18S rDNA V9region presented higher abundance of Coscinodiscophyceae than that of V4. The results showed that the diversity of zooplankton amplification by 18S rDNA V4 primers was higher than that of 18S rDNA V9 primers, while that of phytoplankton amplification by 18S rDNA V9 primers was higher than that of 18S rDNA V4 primers. In this study, we first report high abundance parasitic Syndiniales and Mamiellales in Daya Bay.
High-throughput sequencing; eukaryotic plankton; gene diversity; phytoplankton; zooplankton; Daya Bay
郭雨沛, 黄良民, 钟瑜, 等. 核糖体小亚基V4和V9区引物扩增大亚湾真核浮游生物的比较研究[J]. 生态科学, 2023, 42(1): 242–251.
GUO Yupei, HUANG Liangmin, ZHONG Yu, et al. Comparison of eukaryotic plankton compositions using 18S rRNA gene V4 and V9 primers amplification in Daya Bay[J]. Ecological Science, 2023, 42(1): 242–251.
10.14108/j.cnki.1008-8873.2023.01.028
Q178.53;Q7
A
1008-8873(2023)01-242-10
2021-05-01;
2021-07-21
国家自然科学基金(42276165, 41776154); 广州海洋实验室2019年度人才团队引进项目(GML2019ZD0405)
郭雨沛(1995—), 女, 硕士, 从事浮游植物研究, E-mail: guoyp95@scsio.ac.cn
邱大俊, 男, 博士, 研究员, E-mail: djqiu@scsio.ac.cn
