SNP-array技术在流产组织遗传学分析中的价值
2023-02-13张卫云王玉敏李秀勤李凤启
张卫云,王玉敏,李秀勤,李凤启
1. 周口市妇幼保健院(市儿童医院)检验科(河南周口 466000)
2. 周口市儿科研究所(河南周口 466000)
稽留流产为临床常见不良妊娠结局,可表现为胚胎停育且滞留女性宫腔内,严重影响母体健康,甚至危及女性生命安全[1]。研究表明14%流产由染色体异常导致,因此对流产绒毛组织进行染色体分析,有助于明确流产原因且为再次妊娠提供科学指导[2-3]。目前常采用荧光原位杂交技术进行检测,可用于间期、中期核染色体检测,对于检测周期限制性较小,但是检测过程中染色体分析、细胞培养等操作耗时较久,临床应用时间利用率较低[4]。单核苷酸多态性微阵列(single nucleotide polymorphism microarrays,SNP-array)技术属于新型、方便、高效、快速分子核型分析技术,通过芯片平台可对DNA水平进行较好检测[5]。近年来SNP-array技术在发育迟缓患儿遗传学分析中应用广泛,在单亲二倍体、杂合性缺失方面检测效果较好[6]。因此,SNP-array技术应用于流产组织遗传学分析有望改善检测结果。
1 资料与方法
1.1 研究对象
选择2020年7月—2022年2月经周口市妇幼保健院确诊为稽留流产的50例患者为研究对象。纳入标准:①符合稽留流产诊断标准且孕周在7~12周[7];②沟通、理解、智力正常且依从性较好的患者;③单胎妊娠且接受相关检查。排除标准:①存在其他遗传疾病的患者;②合并子宫肌瘤、恶性肿瘤、子宫内膜异位症或其他影响胚胎着床疾病的患者;③由于男方原因导致流产的患者;④合并严重肝、肾、肺、心等功能异常的患者。本研究已获所有患者及家属知情同意。
1.2 检测方法
SNP-array技术步骤主要包括:①预处理:无菌条件下将绒毛组织离心、重悬;②提取DNA:按照Invitrogen Ambion的RNA试剂盒提取绒毛组织DNA,计算纯度、浓度;③杂交处理:按照Affymetrix Cytoscan 650K 芯片说明书进行纯化、扩增、片段化、连接、标记、酶切、孵育、杂交、染色、洗涤等操作;④数据处理:利用Affymetrix Cytoscan 2500Dx系统及CHAS软件对上述结果进行数据处理及分析。
荧光原位杂交技术检测由专业检验医师处理绒毛组织,具体步骤包括:①制片:剔除血块、蜕膜等杂质后采用海斯迪克HKQS-209不锈钢剪刀剪碎成绒毛枝,然后通过固定、消化等操作得到细胞悬浮液,最后进行滴片、固定、烘烤;②探针杂交:将玻片洗涤后给予消化、脱水、高温变性、取出等操作,最后在42℃下给予特异性探针进行杂交;③检测计数:由检验医师利用荧光显微镜(Leica,DM2500 LED)进行细胞计数。
全部患者染色体突变致病性均采用Characterization of Germline variants(CharGer)软件进行自动化解读,根据 The American College of Medical(ACMG)指南对突变致病性进行评判,数据库采用 Online Mendelian Inheritance in Man(OMIM)。
1.3 观察指标及评价标准
染色体异常:检测染色体异常数低于全部细胞计数的10%为阴性,提示样本正常;染色体异常数高于全部细胞计数的60%为阳性,提示样本异常;染色体异常介于全部细胞计数的10%~60%之间为嵌合体。
1.4 统计学分析
计量资料采用均数和标准差(±s)表示,采用t检验分析;计数资料采用例数和百分比(n,%)表示,利用卡方检验分析。本研究采用SPSS 25.0软件进行分析,以P<0.05表示差异有统计学意义。
2 结果
2.1 一般情况
研究共纳入50例患者,平均年龄为(36.25±4.17)岁,平均体重为(53.79±4.20)kg,以中专或高中学历为主(58%),患者的一般资料,详见表1。
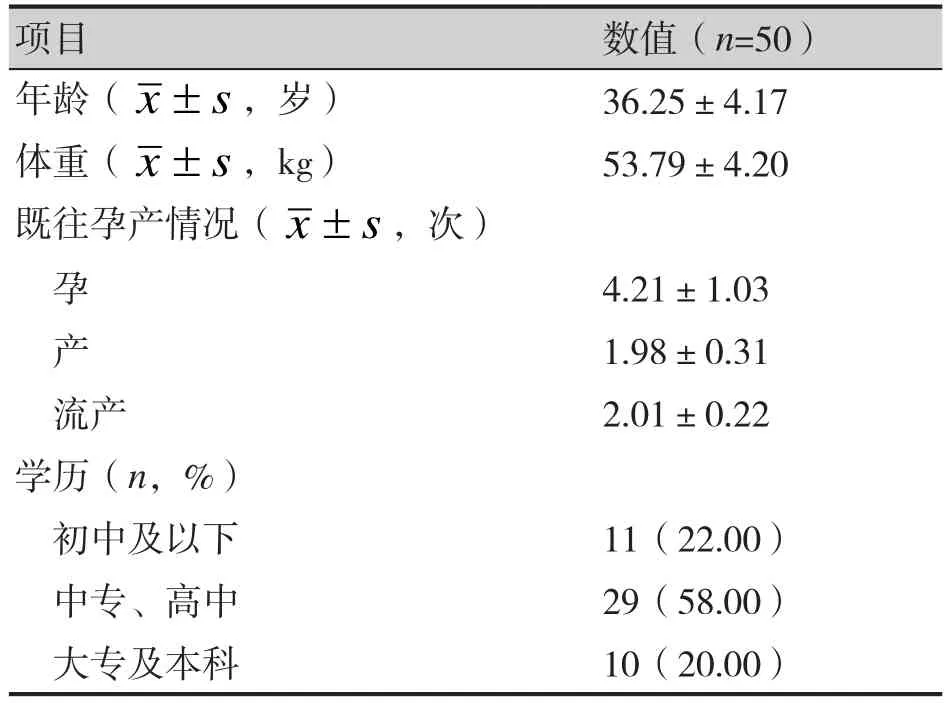
表1 患者一般资料Table 1. Basic information of included patients
2.2 染色体异常检出情况对比
SNP-array组染色体异常34例,其中整倍体异常22例,非整倍体异常12例;对照组中染色体异常24例,其中整倍体异常15例,非整倍体异常9例。SNP-array组染色体异常检出率为68.00%,高于对照组48.00%(χ2=4.105,P=0.043)。见表2。
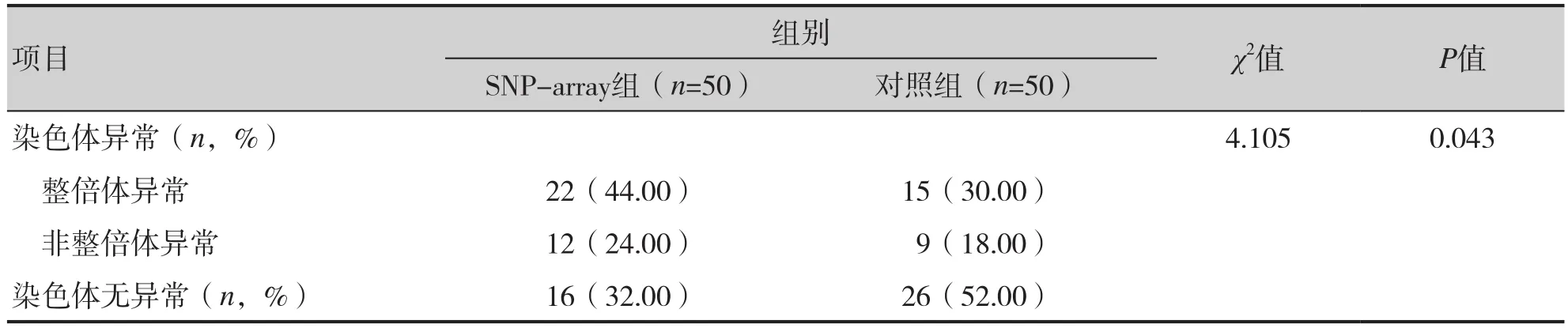
表2 两组染色体异常检出情况对比Table 2. Comparison of detection of chromosome abnormalities between two groups
2.3 SNP-array组染色体微重复微缺失具体情况
SNP-array技术分析按样本纳入顺序进行编号,其中染色体微重复微缺失9例,3例致病性不明确。33号为单一位点重复,7号为单一位点缺失,2号为两位点重复,41号为两位点缺失,其余5个均为2个位点及以上微重复微缺失,详见表3。
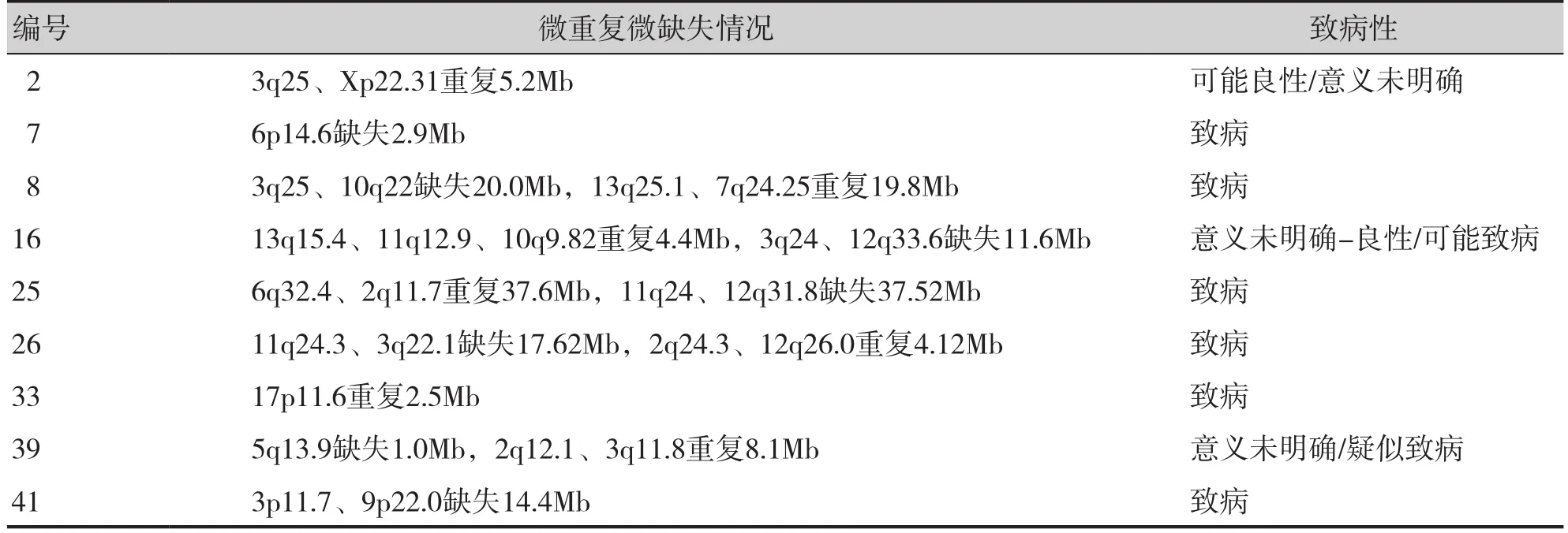
表3 SNP-array组染色体微重复微缺失具体情况(n,%)Table 3. Details of chromosome micro-replication and microdeletion in SNP-array group (n, %)
2.4 染色体非整倍体异常检出情况对比
SNP-array组非整倍体异常检出例数多于对照组,其中45、X染色体异常检出3例,16-三体异常检出2例,18-三体异常检出1例,详见表4。
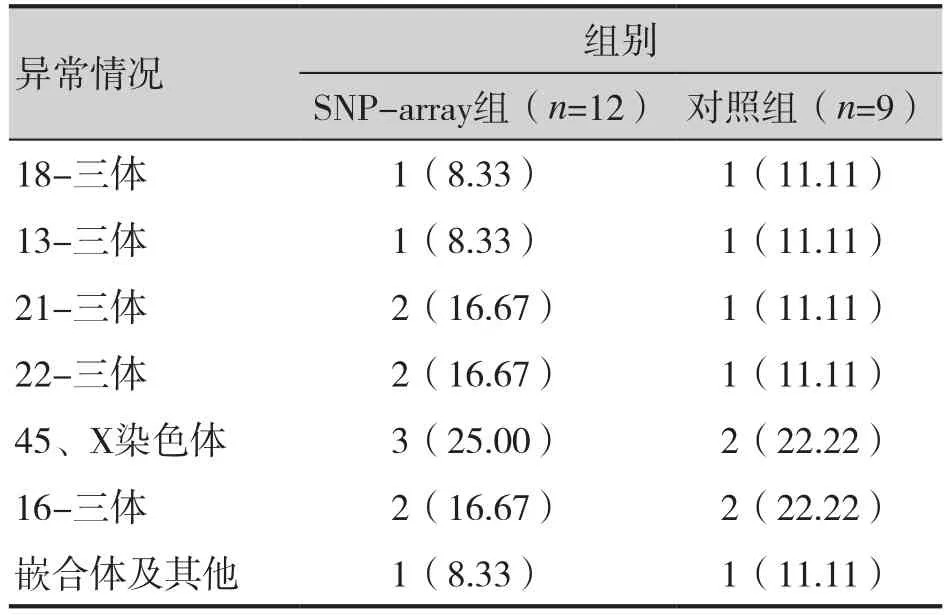
表4 两组染色体非整倍体异常检出情况对比(n,%)Table 4. Comparison of abnormal detection of chromosome aneuploidy between two groups(n, %)
3 讨论
稽留流产患者胚胎不能排出体外,滞留宫腔,严重影响女性患者生命健康与生活质量,还可导致紧张、焦虑、抑郁、恐慌等负性情绪[8]。引起稽留流产因素可包括内分泌、免疫、环境、父体、母体及遗传等情况,其中染色体异常属于常见病因[9]。目前常采用荧光原位杂交技术进行检测,明确病因并为下次妊娠提供科学指导,但荧光原位杂交技术检测范围有限,部分染色体不能覆盖,对于稽留流产病因检出效果欠佳[10]。SNP-array技术着重检测DNA CNVs,通过芯片识别染色体重排情况,同时有利于检出染色体存在的不平衡情况,明确基因组变异情况及变异数,应用于流产组织分析可有望改善异常染色体检出率[11]。
研究结果显示,SNP-array技术对染色体异常检出率为68.00%,高于对照组48.00%。说明相比于传统荧光原位杂交,SNP-array技术对流产组织染色体异常检出率较好。因传统荧光杂交检测法不能有效检测整个染色体组,检测范围局限于22、21、16、13、18染色体,其他倒位、重复、缺失、易位检测效果也较差[12]。SNP-array技术可检测所有染色体的丢失与获得,利用芯片及数据库识别染色体排列情况,最终得出致病、可能致病、不明确、可能良性、良性五种结果[13]。SNP-array技术对染色体的全覆盖对于染色体异常检测优势明显,有助于改善流产组织染色体异常检出率。本研究结果与谢晓蕊研究结果一致,SNP-array对流产绒毛染色体变异异常检出率较高[14]。
对照组中染色体异常24例,其中整倍体异常15例,非整倍体异常9例;SNP-array组中染色体异常34例,其中整倍体异常22例,非整倍体异常12例。说明SNP-array技术对于两种染色体异常均可检出,且相比于传统方法优势明显。原因在于SNP-array技术通过监测DNA到了拷贝数变异检测染色体重复及微缺失,检测过程中充分考虑拷贝数变异的多态性特征。朱丽芬等研究也表明SNP-array技术对于非整倍体异常检测效果良好[15]。SNP-array技术结合DECIPHER、DGV等多个数据库对结果进行分析,数据充分且结果可靠,对于非整倍体异常检测效果较好[16]。传统检测手段对于倒位携带、平衡易位等情况检测效果较差,且对于绒毛染色体显带效果欠佳,分辨率较低,而SNP-array技术分辨率可达到1Mb,可有效确定重复片段断裂情况及染色体缺失情况。
SNP-array组中有3例致病性不明确。一方面由于研究过程中突变结果解读为自动化解读,因此存在数据库资源丰度不足导致部分致病情况分析欠佳的情况;另一方面可能是由于检测来源为母亲单方,需要增加父亲染色体检测得出最终结果。
综上,SNP-array技术在流产组织遗传学分析中对异常染色体检出率较高,具有较好的临床意义,有望广泛应用。
