向日葵HaLACS9基因的克隆与功能分析
2023-02-10杨佳宝周至铭吕新华
杨佳宝 张 展 周至铭 吕新华 孙 黎,*
向日葵基因的克隆与功能分析
杨佳宝1张 展2周至铭1吕新华1孙 黎1,*
1石河子大学生命科学学院, 新疆石河子 832003;2兵团兴新职业技术学院, 新疆铁门关 841007
长链酰基CoA合成酶(long-chain acyl-CoA synthetase, LACS)催化游离脂肪酸合成酰基CoA, 在植物脂质代谢中发挥重要作用。本研究从向日葵中筛选并克隆得到1个LACS家族基因, 同源比对表明HaLACS9具有LACS酶的保守结构域, 与拟南芥()、莴苣()和刺菜蓟() LACS9蛋白具有较高的相似性。亚细胞定位预测HaLACS9定位于叶绿体。启动子区含有多种逆境及激素响应相关元件。qRT-PCR分析表明,在向日葵多个器官中表达, 在花后10 d的种子中优势表达。干旱、盐、外源脱落酸和赤霉素处理均能诱导基因在向日葵根、茎和叶中的表达。分析在向日葵种子发育不同时期的相对表达量发现,在向日葵种仁发育早期高水平表达, 与籽油的快速积累密切相关。随着种子发育和籽油积累速率的减慢,转录水平呈下降趋势。缺陷型酵母互补试验证明HaLACS9具有酰基CoA合成酶活性, 并且油酸是其偏好的催化底物。推测与向日葵种子发育过程中的油脂积累和非生物胁迫响应相关。
向日葵;; 表达模式; 酵母功能互补
脂肪酸是形成多种脂质的关键成分, 如磷脂及其他膜脂、三酰甘油(triacylglycerols, TAGs)、表皮角质层脂质和木栓质等, 因此脂肪酸在维持膜结构的完整性、代谢供能以及形成植物表面屏障以抵御生物和非生物胁迫等方面具有重要作用[1-7]。脂肪酸在各代谢途径之间的转运, 首先需要酰基CoA合成酶(acyl-CoA synthetases, ACSs)的活化, 将其形成高能量的CoA硫酯, 该过程主要包括2步反应, 首先游离的脂肪酸与ATP结合生成腺苷化的中间体, 然后中间体和CoA的硫脂键结合生成脂肪酰基CoA[4]。长链酰基CoA合成酶(long-chain acyl-CoA synthetases, LACSs)是ACS蛋白亚家族的成员, 主要以长链(C14~C20)或超长链(>C22)脂肪酸为底物[8-10]。其中拟南芥AtLACS1定位在内质网上, 偏好以C16:0和C20~C30脂肪酸为底物[9]。AtLACS2可以催化ω-羟基棕榈酸(ω-hydroxypalmitic acid)[11]。AtLACS6和AtLACS7可以催化大多数脂肪酸, 但对种子中的二十碳烯酸(C20:1)有较高的偏好性[12]。AtLACS9对于油酸(C18:1)具有很高的催化活性[8]。因此, LACS酶在植物脂质代谢过程中起关键作用。
真核生物LACS具有ACS蛋白家族的共同特征, 其一是都含有一个高度保守的AMP结合结构域(AMP-binding domain), 含有10~12个高度保守的氨基酸残基, 是ATP的结合位点[13]; 其次是都含有一个ACS信号序列, 由大约25个保守的氨基酸残基组成, 可能是ACS的激活位点和脂肪酸结合部位[14]。但与ACS蛋白不同的是, LACS蛋白具有一个大约由45个氨基酸残基组成的连接域(linker)[15], 该区域与LACS的活性密切相关。
高等植物LACS家族成员十分丰富, 各亚家族成员的功能研究在拟南芥中开展得较为深入。拟南芥中有9个LACS基因(~), 其中7个可以互补缺陷型酵母, 各成员在植物代谢中的作用也存在差异[8]。如和定位于内质网, 在多个器官中均有表达, 主要参与表皮蜡和角质的形成[2,9];和参与花粉外壁脂质的形成, 对于植物的生存至关重要[16];和定位于过氧化物酶体, 参与脂肪酸的β-氧化, 在拟南芥种子萌发的形态建成过程中起着重要作用[1,5];定位在质体膜上主要参与种子油脂的合成[17-19]。其他植物LACS家族成员也有研究报道, 如油菜()基因在油菜中的过表达可增加植株的含油量, 并且该基因偏好以硬脂酸(C18:0)为底物[20]; 苹果()基因在脂肪酸和表皮蜡合成中起关键作用, 并且参与干旱和盐胁迫的响应[21-22]; 棉花()基因在棉花花药发育早期起着重要的作用[23]等。但对向日葵中LACS家族成员的生物学功能研究还十分有限。
向日葵(L.)是世界重要的油料经济作物, 主要在我国华北和西北地区种植。葵花籽油中富含不饱和脂肪酸, 主要为油酸和亚油酸(C18:2), 约占总脂肪酸含量的87%, 是一种优质保健食用油[24]。而且向日葵抗逆性较强, 耐干旱耐瘠薄, 有较强的耐盐碱能力[25], 在地方经济的可持续发展和生态环境保护中具有不可替代的重要作用。LACS是调控油脂代谢的重要酶类, 同时在植物抵御非生物胁迫的耐受性方面也具有重要作用[22]。随着向日葵基因组测序工作的完成, 课题组前期已鉴定出13个向日葵LACS基因家族成员。本研究从向日葵‘T303’ (油用)品系中克隆的cDNA序列, 通过荧光定量PCR技术分析其组织表达及在干旱、盐胁迫、脱落酸和赤霉素处理下的表达模式, 探究向日葵种仁油脂合成与表达的关系, 并将转入LACS缺陷型酵母细胞YB525中进行功能互补试验, 探究HaLACS9的底物偏好性, 以期为揭示的功能奠定基础, 也为向日葵脂质代谢的基因调控和抗逆机制提供理论依据。
1 材料与方法
1.1 材料
1.1.1 试验材料及处理 供试材料为油用向日葵‘T303’ (种仁含油量约为50%), 于2020年春季种植于石河子大学农试场试验田。3周后采集根、茎、叶和子叶, 待油葵开花后采集花样品, 并在第0天进行人工自花授粉, 套袋做标记。在开花后10 d, 每隔7 d (即花后10、17、24、31和38 d)采集发育中的种仁, 液氮速冻后储存于-80℃冰箱备用。
选取饱满的向日葵‘T303’的种子, 种植于装有蛭石和营养土的花盆中, 置于光照培养箱中培养3周左右[(25±1)℃, 16 h光照/8 h黑暗], 选取长势一致的幼苗进行外源脱落酸(abscisic acid, ABA)、赤霉素(gibberellin, GA3)、干旱和盐胁迫处理。
植物激素处理: 制备新鲜的100 μmol L–1ABA和150 μmol L–1GA3溶液[21], 分别喷洒在盆栽的向日葵叶片上, 在0、1、6、12、24 h和0、1、3、6、12、24 h时取根、茎、叶, 于-80℃冰箱保存备用, 以0 h处理为对照。
干旱和盐胁迫处理: 将向日葵幼苗从花盆中取出, 清洗根部备用。分别将向日葵幼苗放入15% PEG-6000的营养液或NaCl (0、150和300 mmol L–1)溶液中进行干旱和盐胁迫处理[26]。干旱胁迫处理0、1、3、6和12 h时取根、茎、叶, 以0 h处理为对照; 盐胁迫处理24 h时取根、茎、叶, 以无菌水处理为对照。以上每组试验都设置3组重复, 经液氮速冻后于–80℃冰箱保存备用。
1.1.2 菌株与主要试剂 pMD19-T载体购自宝生物工程(大连)有限公司。大肠杆菌DH5α、酵母穿梭表达载体pYES2由本实验室保存。缺陷型酵母()YB525菌株(DD)由上海师范大学朱骏教授惠赠。
1.2 方法
1.2.1 向日葵基因克隆 以拟南芥AtLACS9蛋白序列作为查询序列, 利用NCBI blastp工具(https://blast.ncbi.nlm.nih.gov/)在向日葵基因组中进行同源性筛选, 得到候选序列(OTG15459), 设计引物9-F和9-R扩增基因的编码区(表1)。提取向日葵发育中的种仁RNA, 反转录成cDNA后进行PCR扩增, 产物纯化后与pMD19-T载体连接, 筛选阳性克隆送上海生工生物工程公司测序。
1.2.2 生物信息学分析 使用NCBI CDD (http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)软件对蛋白保守结构域进行预测; 使用ExPASy (https://web.expasy.org/protparam/)分析蛋白质理化性质; 使用TMHMM 2.0 (http://www.cbs.dtu.dk/ services/TMHMM/)预测蛋白序列跨膜区; 使用Predictprotein (https://predictprotein.org/)进行亚细胞定位分析; 利用SignalP 5.0 (https://services.healthtech.dtu.dk/service.php?SignalP-5.0)进行蛋白序列的信号肽分析; 使用MEGA 7.0软件中的邻近法(Neighbor- Joining)构建系统进化树; 使用Clustal W结合GeneDoc软件进行多重序列比对; 采用SOPMA数据库(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat. pl?page=npsa_sopma.html)和Phyre2 (http://www.sbg. bio.ic.ac.uk/phyre2/html/page.cgi?id=index)软件分别预测蛋白质的二级结构和三级结构; 选取基因起始密码子上游2000 bp的DNA序列, 利用PlantCARE (http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)和TBtools软件分析顺式作用元件。使用MEME 程序(http://meme-suite.org/tools/meme)分析蛋白序列中的保守结构域。
1.2.3基因的表达分析 利用Primer Premier 5.0软件设计基因的实时荧光定量PCR (qRT-PCR)引物, 以向日葵(AF107577)为内参基因(表1)。使用ChamQUniversal SYBR qPCR Master Mix (诺唯赞生物科技有限公司, 南京)进行qRT-PCR反应, 分析基因在向日葵不同组织、ABA及盐旱胁迫下的表达特征。每个样品设3次生物学重复, 采用2–∆∆Ct法计算差异表达[27]。采用SPSS 26.0中单因素方差法对结果进行分析(one-way ANOVA), Duncan’s法进行多重比较, 设定分析显著差异水平为<0.05。所有数据均用3次生物学重复的平均值±标准差()表示, 并使用GraphPad Prism 8.0.2绘图。
1.2.4 向日葵种子油脂提取 采集不同发育阶段(花后10、17、24、31和38 d)的油用向日葵‘T303’的籽粒, 去壳取种仁, 采用索氏抽提法提取总油脂[28],每个样品重复3次。

表1 本研究的引物及序列
下画线表示酶切位点。The underlines indicate restriction sites.
1.2.5在酵母突变体YB525中的表达 酿酒酵母()突变菌株YB525同时缺失和, 分别编码LACS蛋白Faa1p和Faa4p, 因此, 该菌株缺乏90%的LACS酶活性, 无法在以脂肪酸为唯一碳源的培养基上生长, 常用于外源LACS活性分析[29-31]。使用引物p-F和p-R (表1)扩增的编码区序列, 构建酵母表达载体pYES2-。采用LiAc转化法[32]转化至缺陷型酵母YB525感受态细胞中, 同时转pYES2空载体作为对照, 通过缺省尿嘧啶固体培养基(Yeast synthetic drop-out agar medium without uracil, SCU)筛选阳性转化克隆。阳性转化子在含2% (w/v)半乳糖的SCU液体培养基上进行诱导表达。
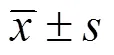
2 结果与分析
2.1 HaLACS9基因的克隆及序列分析
以向日葵cDNA为模板, 通过RT-PCR得到2091 bp的完整开放阅读框(图1-A)。染色体定位分析发现其位于9号染色体上的153,400,893~ 153,406,613基因座, 编码696个氨基酸, 具有11个外显子和10个内含子(图1-B)。同源比对发现, 向日葵HaLACS9蛋白与拟南芥AtLACS9、莴苣() LsLACS9和刺菜蓟() CcLACS9蛋白的同源性较高, 分别为74.4%、82.1%和86.7% (图2)。
利用NCBI数据库的CCD分析其保守结构域发现, HaLACS9含有一个高度保守的AMP结合结构域(AMP-binding domain)和一个ACS信号结构域(ACS signature domain), 表明HaLACS9属于典型的植物ACS基因家族成员。此外, 介于AMP结合结构域和ACS信号结构域之间, 还存在一个LACSs亚家族特异的linker连接域(LACS-specific linker domain), 该连接域在不同植物与不同成员的序列相似性差异较大。进一步使用MEME网站搜索7个植物中的LACS9蛋白保守基序(图3)发现, 除莴苣() LsLACS9缺少motif 5外, 其他6种植物的LACS9蛋白具有相似的保守基序, 因此可以确认HaLACS9为向日葵所含有的LACS亚家族成员之一。
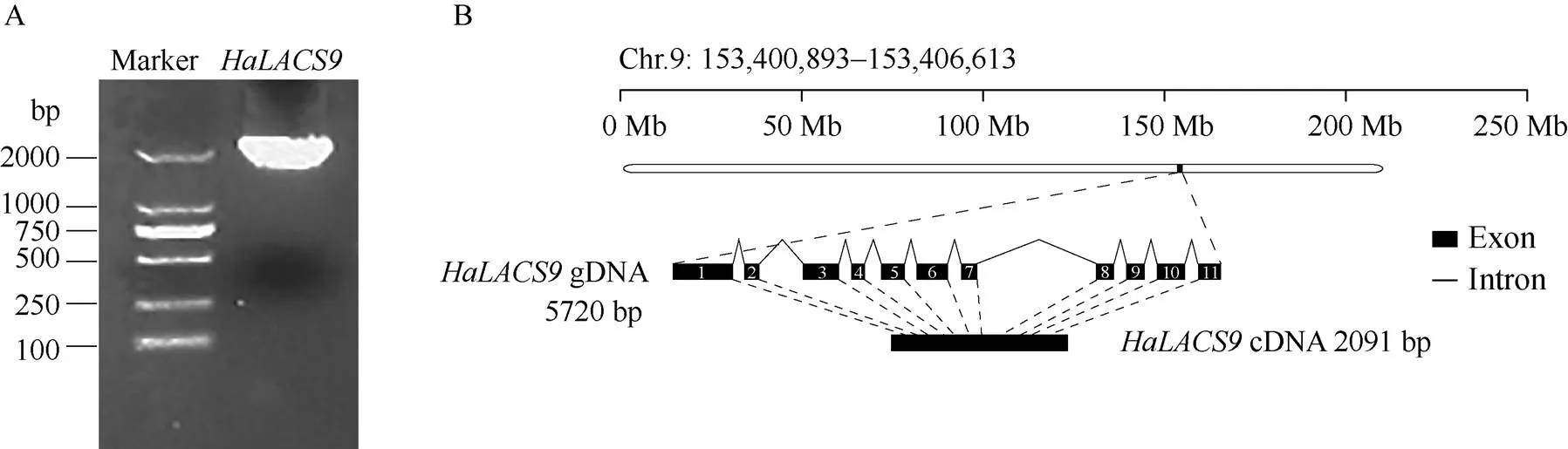
图1 向日葵HaLACS9的克隆(A)及染色体定位分析(B)

(图2)
At: 拟南芥; Ha: 向日葵; Cc: 刺菜蓟; Ps: 山杏; Os: 水稻; Ta: 小麦; Ls: 莴苣。
At:; Ha:L.; Cc:; Ps:; Os:; Ta:; Ls:.
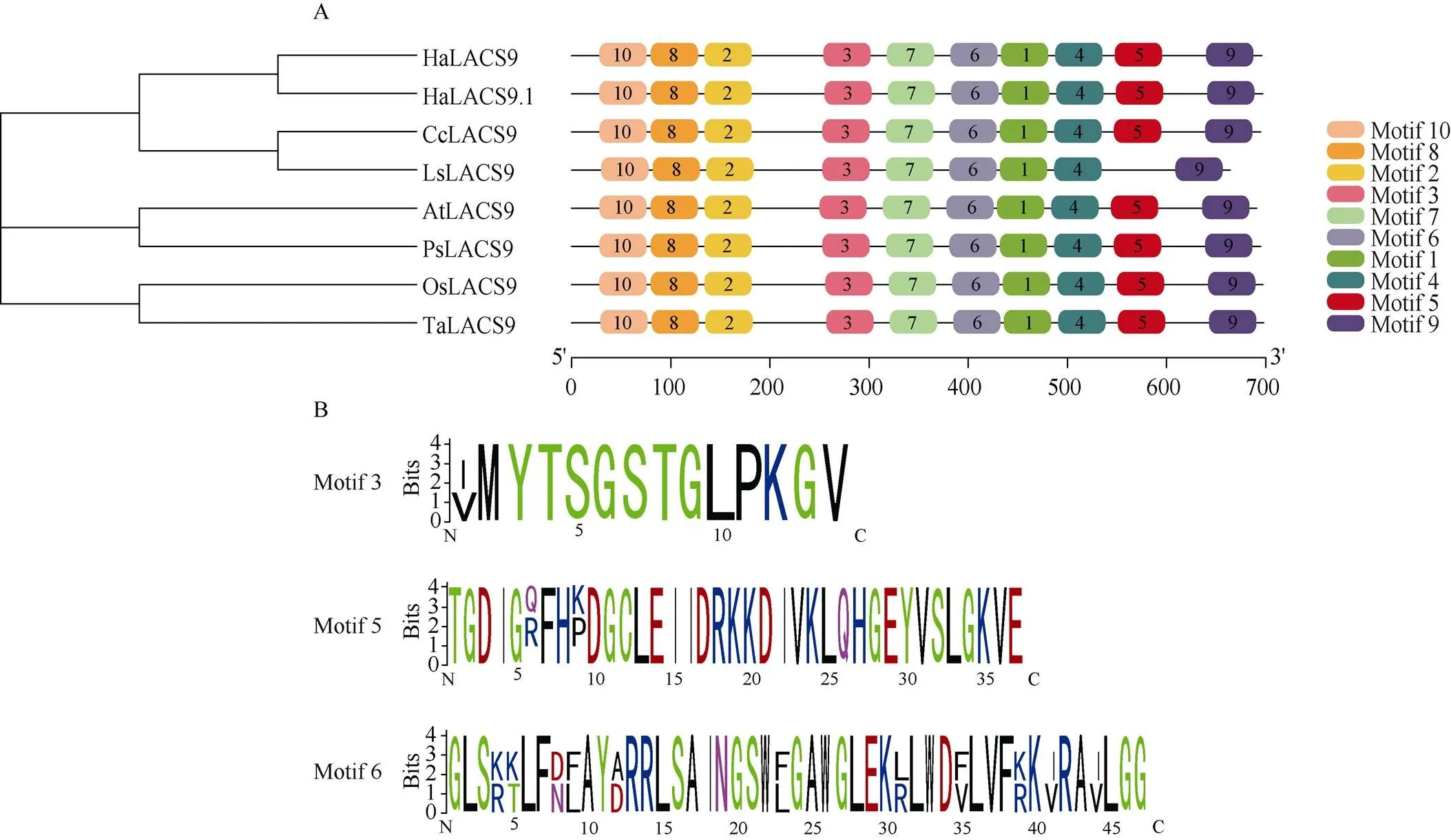
图3 不同植物LACS9序列的保守基序分布(A)和保守基序分析(B)
At: 拟南芥; Ha: 向日葵; Cc: 刺菜蓟; Ps: 山杏; Os: 水稻; Ta: 小麦; Ls: 莴苣。
At:; Ha:L.; Cc:; Ps:; Os:; Ta:; Ls:.
采用SOPMA数据库对HaLACS9蛋白的二级结构进行分析发现, 其包含α-螺旋、β-折叠、无规则卷曲和延伸链, 含量分别为37.79%、6.75%、38.07%和17.93%。通过Phyre2软件预测HaLACS9的三级结构(图4-A)发现, HaLACS9具有ACS家族共有的AMP结合结构域。HaLACS9蛋白的分子量为76.23 kD, 等电点为6.10, 平均亲水系数为-0.033, 为亲水性蛋白。不稳定指数31.81, 属于稳定性蛋白。跨膜结构预测发现HaLACS9蛋白没有跨膜结构。信号肽预测数据显示, HaLACS9的切割位点在N末端第17位点和第18位点之间(图4-B), Predictprotein在线网站进行亚细胞定位预测分析表明该蛋白定位于叶绿体, 暗示其可能与拟南芥AtLACS9具有相似的功能, 执行着将合成后的脂肪酸转运出质体的任务, 并且参与向日葵种仁中油脂的合成调控。
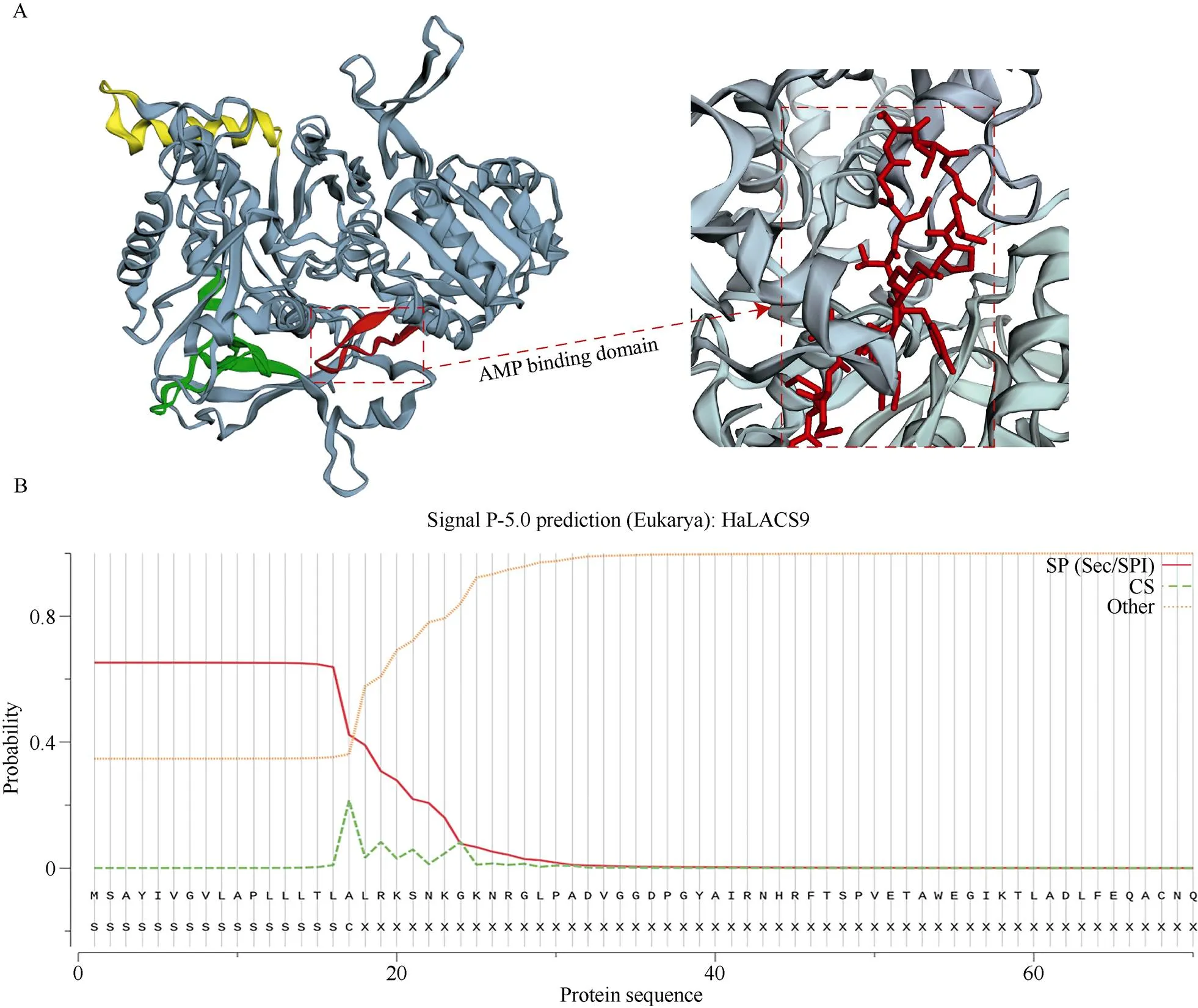
图4 HaLACS9的结构分析
A: HaLACS9的三级结构预测; AMP结合结构域用红色方框表示。B: HaLACS9的信号肽预测。
A: the tertiary structure prediction of HaLACS9 and the AMP binding domain are shown in red box. B: the signal peptide prediction of HaLACS9.
2.2 HaLACS9系统进化分析
HaLACS9与已报道的HaLACS9.1同源性为89.5%, 与刺菜蓟()、莴苣()、芝麻()、蓖麻()、山杏()和拟南芥中LACS9同源基因序列相似性分别为86.7%、82.1%、78.3%、76.4%、76.9%和74.4%, 表明在植物进化中LACS9较为保守, 可能在不同植物中具有相同或相似的功能。LACS系统进化树分析显示(图5), HaLACS9与同为菊科的刺菜蓟CcLACS9以及莴苣LsLACS9的亲缘关系最近, 其次是芝麻、蓖麻、拟南芥等其他双子叶植物, 与油棕、水稻、玉米等单子叶植物的LACS9亲缘关系较远, 该结果与向日葵在陆生植物中的进化地位相符。
2.3 HaLACS9启动子顺式作用元件分析
利用PlantCARE在线软件, 对起始密码子上游2000 bp的序列进行顺式作用元件分析。结果表明(图6),启动子区含有TATA-box (RNA聚合酶II结合位点, 可以保证转录精确起始)等基本启动子元件。同时,还拥有响应非生物胁迫以及激素的顺式作用元件, 包括LTR (参与低温响应)、DRE (参与脱水响应)、STRE (参与胁迫响应)、W-box (参与损伤响应)、AuxRR (参与生长素响应)、MeJA (参与茉莉酸甲酯响应)和GARE-motif (参与赤霉素响应)等。说明可能在向日葵生长发育过程中发挥重要调控作用, 且受外界环境如温度、水分及多种激素的影响。
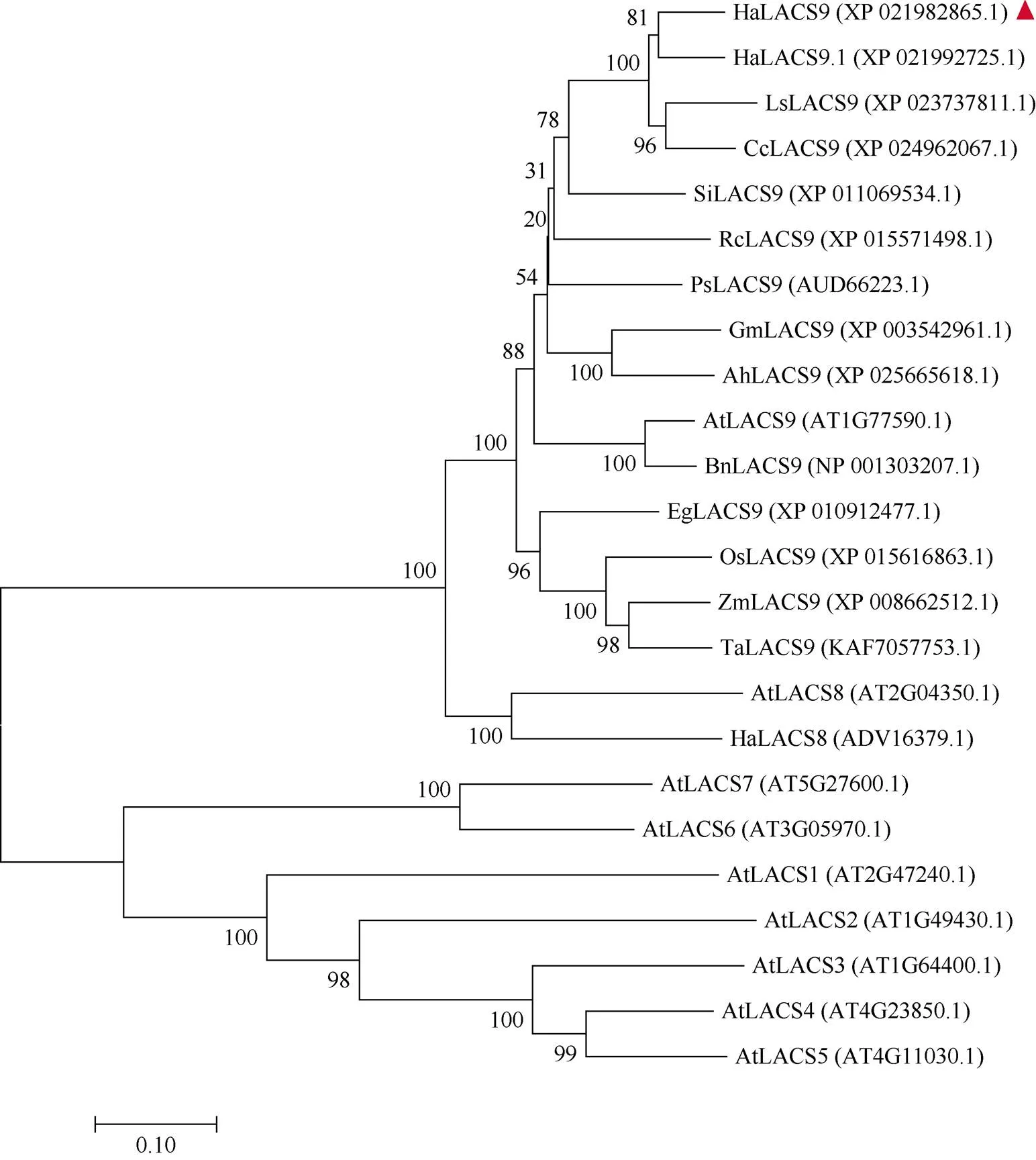
图5 HaLACS9与同源蛋白的系统进化树
At: 拟南芥; Ha: 向日葵; Ls: 莴苣; Cc: 刺菜蓟; Si: 芝麻; Rc: 蓖麻; Ps: 山杏; Gm: 大豆; Ah: 花生; Eg: 油棕; Bn: 甘蓝型油菜; Zm: 玉米; Os: 水稻; Ta: 小麦。红三角标记为目的蛋白。
At:; Ha:L.; Ls:; Cc:; Si:; Rc:; Ps:; Gm:; Ah:; Eg:; Bn:; Zm:; Os:; Ta:. Red triangle indicates the target protein.

图6 HaLACS9启动子顺式作用元件分析
2.4 HaLACS9基因在不同器官中的表达分析
qRT-PCR分析表明(图7),在向日葵根、茎、叶、子叶、花和发育中的种子(花后10 d)中均有表达, 在种子中的表达量最高, 是根中表达量的17倍, 在茎、子叶和花中的表达量普遍较低。
2.5 HaLACS9在盐、旱胁迫下的表达分析
为探讨在盐、旱胁迫响应中可能的分子机制, 使用qRT-PCR分析在干旱和NaCl胁迫下的表达水平(图8)。在根中15% PEG- 6000模拟干旱处理6 h后,的表达量与对照相比上调, 但是误差线较大, 可能是因为其中一个样本的数值偏高所致; 在茎中,在3 h表达量最高, 约为0 h的3倍; 在叶中,的表达量在1 h显著上调, 随着胁迫处理时间的延长, 表达量显著下调(图8-A)。
NaCl处理24 h结果(图8-B)显示,在150 mmol L–1NaCl处理的茎中相对表达量最高; 在根中,的表达量与对照相比无显著差异; 在叶中,的表达量在300 mmol L–1NaCl处理24 h后显著下调。
2.6 HaLACS9对外源ABA和GA3的响应
为探究对外源激素的响应, 利用qRT- PCR方法检测外源ABA和GA3处理下在根、茎、叶器官中的表达水平(图9)。在根中100 μmol L–1ABA后, 随着处理时间的延长,的表达量与对照相比呈下调趋势; 在茎中,的表达量显著上调, 在24 h表达量达到最高; 在叶中,的表达量呈现“先升后降”的趋势, 在6 h时达到最高(图9-A)。
在根中150 μmol L-1GA3处理后,的表达量在3 h显著上调, 是对照的44倍, 之后开始下降; 在茎中,的表达量在1 h时显著上调, 约是对照的14倍; 在叶中,的表达量在GA3处理后的3、6、12 h处显著降低(图9-B)。
2.7 向日葵种仁不同发育时期油脂含量与HaLACS9表达量的相关分析
为探究向日葵种仁发育过程中油脂含量的变化与表达的关系, 本研究检测了向日葵种仁5个不同发育阶段(花后10、17、24、31和38 d)的含油量, 并以根中的表达量(图7)为对照分析了在种仁不同发育阶段的表达量(图10)。结果表明,在种仁发育前期(10 d)表达量较高, 随着种子的发育成熟,的表达量逐渐降低; 对应地, 本研究通过检测向日葵种子发育5个时期的种仁含油量发现, 随着种子个体不断发育膨大, 种仁含油量(出油率)逐渐升高; 在种子发育中期(17~24 d)油脂含量呈快速增长趋势, 随着种子逐渐发育至成熟阶段, 在发育后期(31~38 d)种仁中油脂含量增长缓慢。
2.8 酵母功能互补试验和底物偏好性分析
为探究编码的酰基CoA合成酶是否具有活化长链脂肪酸的能力, 构建了pYES2-重组质粒, 转化酿酒酵母突变株YB525进行功能互补试验和底物偏好性分析(图11)。本研究在培养基中分别添加不同链长的脂肪酸作为底物, 根据酵母细胞的生长状况确定是否具有酰基CoA合成酶活性, 并探究的底物偏好性。结果表明, 转化了pYES2-重组质粒的缺陷型酵母可以正常生长, 而转化了pYES2空载体的酵母不能生长, 说明HaLACS9可以弥补YB525酵母基因的缺陷, 证明其具有LACS酶活性。此外, 酵母细胞生长状况显示, 细胞在含有油酸(C18:1)的培养基上生长速率最快, 而在含有月桂酸(C12:0)的培养基中生长速率最慢, 说明HaLACS9可以活化多种脂肪酸, 并且具有底物偏好性, 优先利用C18:1等长链脂肪酸作为底物。

图7 HaLACS9的组织表达分析
不同小写字母表示数值有显著差异(0.05)。
Different lowercase letters indicate there are significantly different at< 0.05.
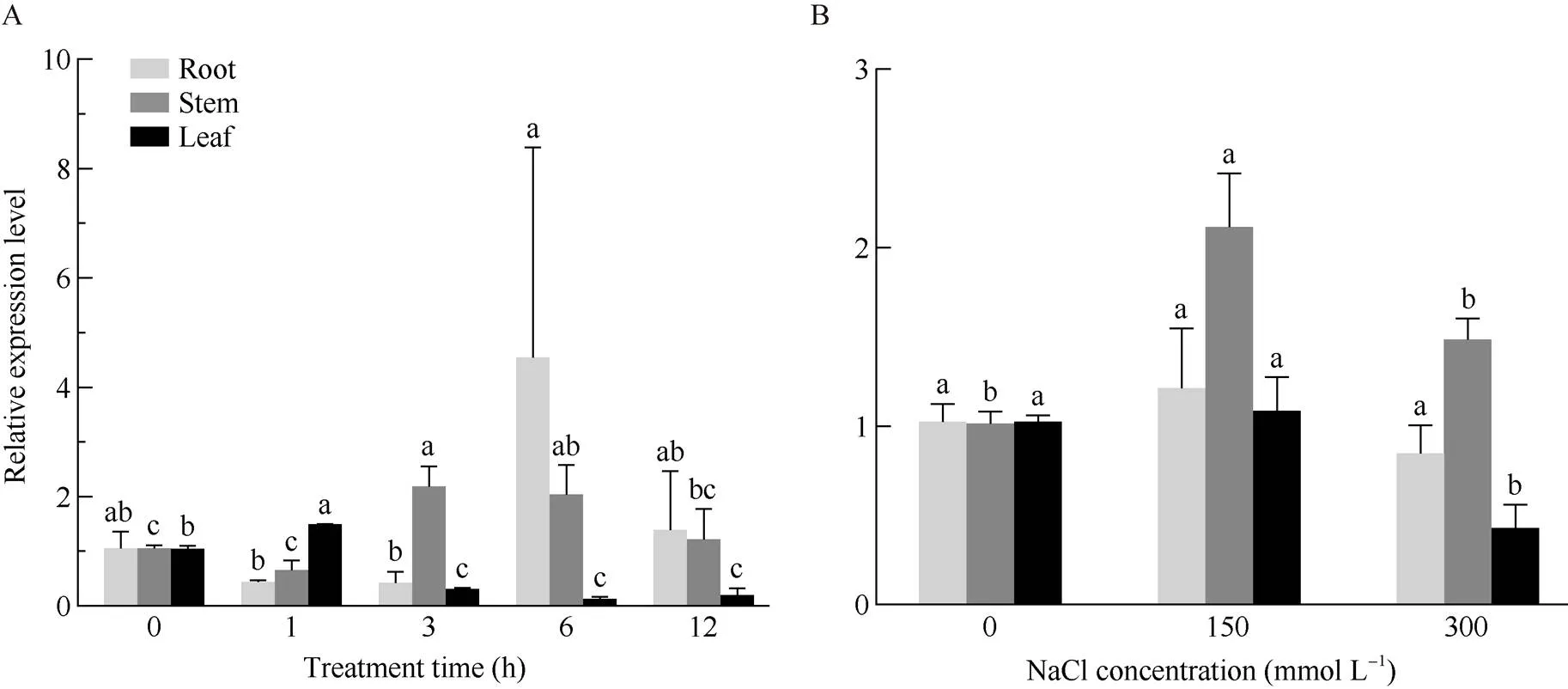
图8 PEG (A)和NaCl (B)胁迫对HaLACS9相对表达量的影响
不同小写字母表示数值有显著差异(0.05)。
Different lowercase letters indicate there are significantly different at< 0.05.
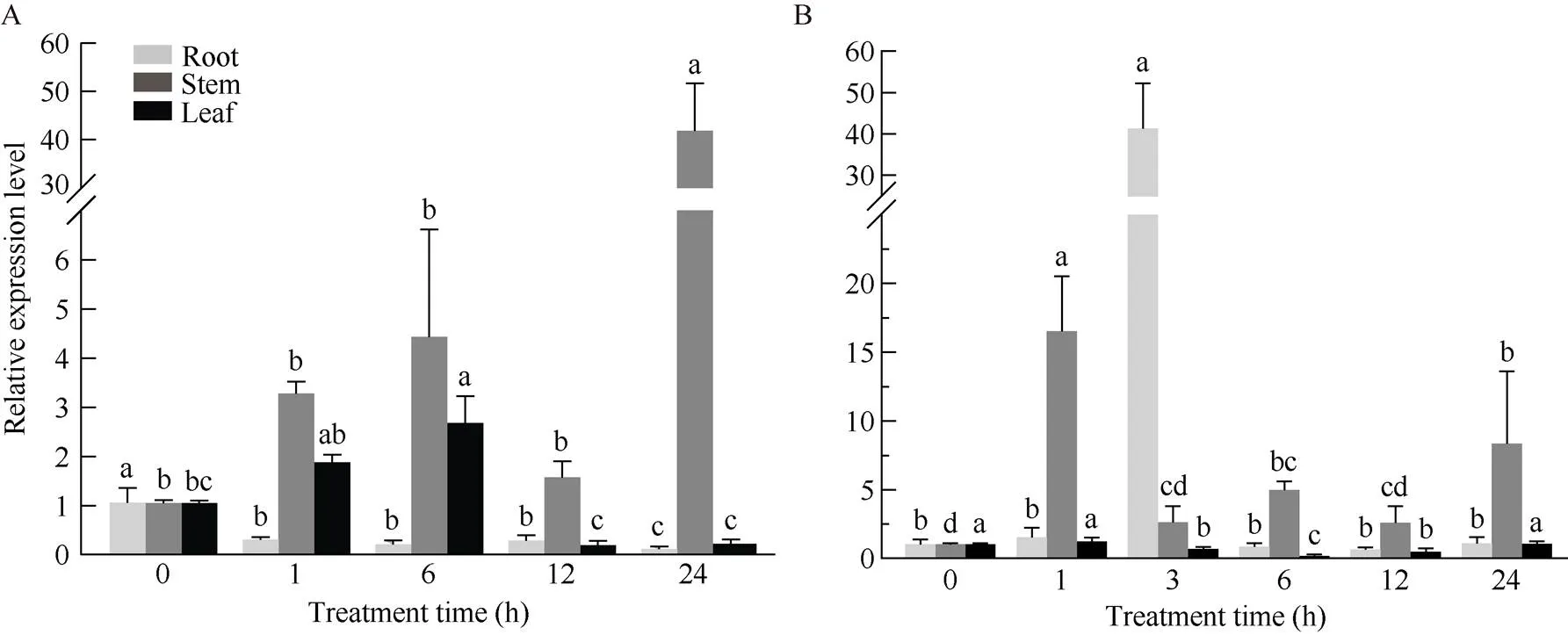
图9 外源ABA (A)和GA3 (B)处理对HaLACS9相对表达量的影响
不同小写字母表示数值有显著差异(0.05)。
Different lowercase letters indicate there are significantly different at< 0.05.
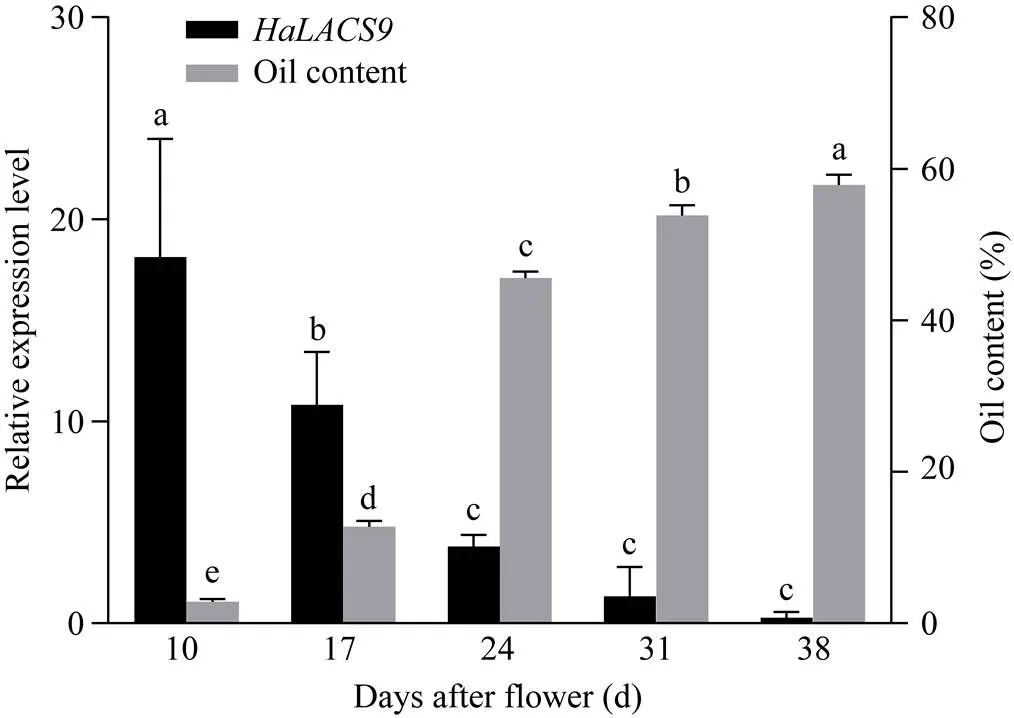
图10 向日葵种仁不同发育时期HaLACS9相对表达水平和含油量
不同小写字母表示数值有显著差异(0.05)。
Different lowercase letters indicate there are significantly different at< 0.05.
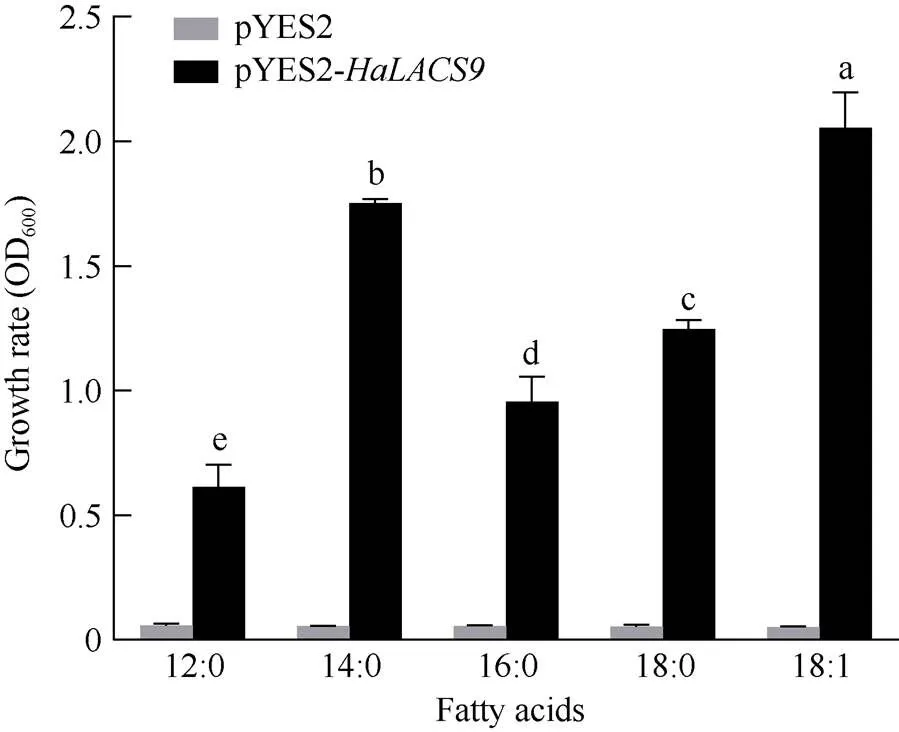
图11 酵母生长状况
不同小写字母表示数值有显著差异(<0.05)。
Different lowercase letters indicate there are significantly different at< 0.05.
3 讨论
越来越多的证据表明,在植物脂质代谢中的重要性, 该家族基因参与多个脂肪酸相关的代谢途径, 包括磷脂代谢、甘油脂代谢、茉莉酸生物合成以及脂肪酸的β-氧化。许多高等植物的基因已经被克隆和鉴定, 但目前为止对向日葵的研究报道较少。本研究的目的是鉴定向日葵中的基因并研究其在脂质代谢和非生物胁迫中的功能。通过搜索向日葵基因组数据库和我们前期构建的转录组数据, 本研究从向日葵中克隆鉴定了一个的cDNA。序列比对发现HaLACS9与拟南芥、莴苣、刺菜蓟等植物LACS9蛋白的相似性较高(74.4%、82.1%和86.7%), 与已报道的向日葵HaLACS1[34]的相似性为89.5%。HaLACS1与拟南芥质体型AtLACS9的同源性较高(74.2%), 在同一个进化分支上, 隶属于LACS9亚家族成员[34], 因此本研究将其命名为HaLACS9.1。本研究通过分析蛋白的保守结构域发现, HaLACS9与HaLACS9.1在ACS信号结构域和LACS特异linker连接域的保守氨基酸上存在差异(图2), 暗示它们在功能上出现了分化。前人研究表明在向日葵发育的种子中呈现持续高表达状态(花后12~30 d)[34]。本研究克隆的主要在向日葵种子发育早期(花后10~17 d)高表达, 此时正是向日葵种子的快速膨胀期和籽油快速积累期, 推测其在向日葵籽油早期合成和累积过程中具有重要作用。
酵母功能互补试验证明编码了有活性的LACS酶, 能够互补酵母突变株YB525的缺陷表型, 这与拟南芥AtLACS9的研究结果一致。AtLACS9对油酸(C18:1)的偏好性要显著高于C16:0、C16:1、C18:0、C18:2和C20:1[8]。大豆GmLACS能够优先利用长链饱和脂肪酸作为底物, 尤其是C16:0、C18:0和C18:1脂肪酸[35]。基因转入YB525不仅提供了一个检测该酶活性的有效方法, 而且提供了一个分析HaLACS的底物特异性的有效方法。当将转基因酵母置于以不同脂肪酸(从长链到短链脂肪酸)为唯一碳源的SCU培养基时, 发现携带的转基因酵母更加倾向于利用长链脂肪酸(C14~C18)为底物, 而不是短链脂肪酸(C12), 表明HaLACS9具有较高的活性催化长链脂肪酸转化为酰基CoA。在供试的4种脂肪酸底物(C14:0、C16:0、C18:0和C18:1)中, HaLACS9对油酸的底物偏好性最高(图11), 这与向日葵种子中含有大量油酸相一致。
植物LACS蛋白在不同组织中亚细胞定位的差异与其功能密切相关[23]。AtLACS9是拟南芥唯一定位于质体中的LACS, 拟南芥双突变体叶片中的油酸和亚油酸相较于野生型出现了不同程度的积累, 并且内质网衍生的脂质前体的合成明显减少, 说明与在脂质前体的合成中起重要作用[5]。植物细胞器之间的脂肪酸运输和脂质加工也离不开LACS酶[3], 如油棕EgLACS9参与脂肪酸在质体和内质网之间的运输[36]。向日葵HaLACS9.1蛋白定位在质体和内质网2种细胞器中, 可能参与内质网–质体膜之间的脂质运输[34]。最近研究发现, 植物LACS可能具有多种生物学功能, 如水稻功能缺失突变体导致水稻的节间变短、矮化、叶绿体淀粉颗粒积累, 说明可以抑制淀粉的降解[37]。OsLACS9定位在叶绿体膜上, 其N末端30个氨基酸的信号序列对于该蛋白靶向运输到质体至关重要。本研究利用生物信息学软件预测到向日葵HaLACS9是无跨膜结构域的亲水性蛋白, 具有信号肽, 预测定位在质体中, 暗示它可能是分泌蛋白而不是膜蛋白。与拟南芥相似性较高, 都在发育的种子中优势表达, 而且其编码蛋白均定位于叶绿体中, 暗示可能是的直系同源基因在向日葵籽油合成与累积过程中起重要作用, 其是否具有将“活化”的脂肪酸转入/转出质体的功能, 还需要进一步的试验验证。
顺式作用元件在基因转录起始调控中起着至关重要的作用。基因启动子区含有多个与激素响应和逆境胁迫相关的调控元件, 推测其可能参与向日葵多种生物和非生物胁迫的响应。脂肪酸是甘油脂的重要组成部分, 也是合成角质和表皮蜡的前体物质。角质和表皮蜡是植物与环境之间的一道天然屏障, 具有防止植物非气孔性失水、抵御病虫害和非生物逆境等多种功能[38]。研究表明拟南芥和参与表皮蜡质合成[2]。苹果的过表达增强了苹果愈伤组织对PEG、ABA和NaCl的耐受性, 表明是响应非生物胁迫的重要调节因子[21]。苹果和在拟南芥中表达, 降低了拟南芥表皮细胞的渗透性, 减少了水分散失, 提高了转基因拟南芥对干旱和盐胁迫的耐受性[22]。本研究证实, 外源ABA、GA3、PEG和NaCl胁迫均能诱导在向日葵根、茎或叶中上调表达, 推测可能在向日葵的生长发育以及逆境胁迫响应方面具有重要作用。后续将通过转基因拟南芥等方法分析该基因的下游调控网络, 观察植株在非生物胁迫下的抗性表现, 探索在调控植物逆境胁迫中的具体机制。
目前基因在拟南芥等模式植物上的研究已取得重大进展, 本研究通过克隆得到向日葵家族成员中的一个基因, 初步明确了在参与植物脂质合成与逆境应答过程中可能具有重要作用, 但其功能和作用机制仍需要深入研究, 从而为后续进一步运用基因工程方法改善向日葵等油料作物的品质和抗逆性提供候选基因资源。
4 结论
本研究从向日葵中克隆得到基因, 该基因cDNA全长为2091 bp, 编码696个氨基酸, 相对分子量为76.23 kD, 等电点为6.10, 无跨膜结构, 有信号肽, 预测定位于叶绿体。HaLACS9能够互补酵母突变株YB525的缺陷表型, 具有酰基CoA合成酶活性, 对长链脂肪酸底物具有偏好性, 尤其偏好油酸。在向日葵种仁发育早期高表达, 并且响应干旱、高盐、ABA和GA3的调控, 可能在向日葵种仁油脂积累及应答外界环境信号过程中发挥重要作用。
[1] Fulda M, Schnurr J, Abbadi A,Heinz E, Browse J. Peroxisomal acyl-CoA synthetase activity is essential for seedling development in., 2004, 16: 394–405.
[2] Judy S, Jay S, John B. The acyl-CoA synthetase encoded byis essential for normal cuticle development in., 2004, 16: 629–642.
[3] Li-Beisson Y, Shorrosh B, Beisson F, Andersson M X, Arondel V, Bates P D, Baud S, Bird D, Debono A, Durrett T P, Franke R B, Graham I A, Katayama K, Kelly A A, Larson T, Markham J E, Miquel M, Molina I, Nishida I, Rowland O, Samuels L, Schmid K M, Wada H, Welti R, Xu C C, Zallot R, Ohlrogge J. Acyl-lipid metabolism., 2013, 11: e0161.
[4] Grevengoed T J, Klett E L, Coleman R A. Acyl-CoA metabolism and partitioning., 2014, 34: 1–30.
[5] Jessen D, Roth C, Wiermer M, Fulda M. Two activities of long-chain acyl-coenzyme a synthetase are involved in lipid trafficking between the endoplasmic reticulum and the plastid in., 2015, 167: 351–366.
[6] Fich E A, Segerson N A, Rose J K C. The plant polyester cutin: biosynthesis, structure, and biological roles., 2016, 67: 207–233.
[7] Ingram G, Nawrath C. The roles of the cuticle in plant development: organ adhesions and beyond., 2017, 68: 5307–5321.
[8] Shockey J M, Fulda M S, Browse J A.contains nine long-chain acyl-coenzyme a synthetase genes that participate in fatty acid and glycerolipid metabolism., 2002, 129: 1710–1722.
[9] Lyu S Y, Song T, Kosma D K, Parsons E P, Rowland O, Jenks M A.encodes long-chain acyl-CoA synthetase 1 (LACS1) that has overlapping functions with LACS2 in plant wax and cutin synthesis., 2009, 59: 553–564.
[10] Shockey J, Browse J. Genome-level and biochemical diversity of the acyl-activating enzyme superfamily in plants., 2011, 66: 143–160.
[11] Schnurr J, Shockey J, Browse J. The acyl-CoA synthetase encoded by LACS2 is essential for normal cuticle development in., 2004, 16: 629–642.
[12] Fulda M, Shockey J, Werber M, Wolter F P, Heinz E. Two long-chain acyl-CoA synthetases frominvolved in peroxisomal fatty acid beta-oxidation., 2002, 32: 93–103.
[13] Babbitt P C. Ancestry of the 4-chlorobenzoate dehalogenase: analysis of amino acid sequence identities among families of acyl: adenyl ligases, enoyl-CoA hydratases/isomerases, and acyl-CoA thioesterases., 1992, 31: 5594–5604.
[14] Iijima H, Fujino T, Minekura H, Suzuki H, Kang M J, Yamamoto T. Biochemical studies of two rat acyl-CoA synthetases, ACS1 and ACS2., 1996, 242: 186–190.
[15] Steinberg S J, Morgenthaler J, Heinzer A K. Very long-chain acyl-CoA synthetases. Human “bubblegum” represents a new family of proteins capable of activating very long-chain fatty acids., 2000, 275: 35162–35169.
[16] Jessen D, Olbrich A, Knufer J, Kruger A, Hoppert M, Polle A, Fulda M. Combined activity of LACS1 and LACS4 is required for proper pollen coat formation in., 2011, 68: 715–726.
[17] Schnurr J A, Shockey J M, de Boer G J, Browse1 J A. Fatty acid export from the chloroplast. Molecular characterization of a major plastidial acyl-coenzyme a synthetase from., 2002, 129: 1700–1709.
[18] Zhao L F, Katavic V, Li F L, Haughn G W, Kunst L. Insertional mutant analysis reveals that long-chain acyl-CoA synthetase 1 (LACS1), but not LACS8, functionally overlaps with LACS9 inseed oil biosynthesis., 2010, 64: 1048–1058.
[19] Zhao L F, Haslam T M, Sonntag A, Molina I, Kunst L. Functional overlap of long-chain acyl-CoA synthetases in., 2019, 60: 1041–1054.
[20] Ding L N, Gu S L, Zhu F G, Ma Z Y, Li J, Li M, Wang Z, Tan X L. Long-chain acyl-CoA synthetase 2 is involved in seed oil production in., 2020, 20: 21.
[21] Zhang C L, Mao K, Zhou L J, Wang G L, Zhang Y L, Li Y Y, Hao Y J. Genome-wide identification and characterization of apple long-chain acyl-CoA synthetases and expression analysis under different stresses., 2018, 132: 320–332.
[22] Zhang C L, Zhang Y L, Hu X, Xiao X, Wang G L, You C X, Li Y Y, Hao Y J. An apple long-chain acyl-CoA synthetase, MdLACS4, induces early flowering and enhances abiotic stress resistance in., 2020, 297: 110529.
[23] Wang X L, Li X B. Thegene encodes an acyl-CoA synthetase which is essential for normal microsporogenesis in early anther development of cotton., 2009, 57: 473–486.
[24] Dyer J M, Stymne S, Green A G, Carlsson A S. High-value oils from plants., 2008, 54: 640–655.
[25] Hryvusevich P, Navaselsky I, Talkachova Y, Straltsova D, Keisham M , Viatoshkin A , Samokhina V, Smolich I, Sokolik A, Huang X, Yu M, Bhatla S C, Demidchik V. Sodium influx and potassium efflux currents in sunflower root cells under high salinity., 2020, 11: 613936.
[26] Li J J, Liu H, Yang C, Wang J, Yan G J, Si P, Bai Q J, Lu Z Y, Zhou W J, Xu L. Genome-wide identification ofgenes and expression analysis under different biotic and abiotic stresses inL., 2020, 143: 111924.
[27] Livak Kenneth J A, Schmittgen Thomas D B. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCTmethod., 2001, 25: 402–408.
[28] Harwood H J. Oleochemicals as a fuel: mechanical and economic feasibility., 1984, 61: 315–324.
[29] Knoll L J. Complementation ofstrains containing fatty acid activation gene (FAA) deletion with a mammalian acyl-CoA synthetase., 1995, 270: 10861–10867.
[30] Færgeman N J, Black P N, Zhao X D. The acyl-CoA synthetases encoded withinandinfunction as components of the fatty acid transport system linking import, activation, and intracellular utilization., 2001, 276: 37051–37059.
[31] Li H, Melton E M, Quackenbush S, DiRusso C C, Black P N. Mechanistic studies of the long chain acyl-CoA synthetase Faa1p from., 2007, 1771: 1246–1253.
[32] Gietz R D, Schiestl R H. Frozen competent yeast cells that can be transformed with high efficiency using the LiAc/SS carrier DNA/PEG method., 2007, 2: 1–4.
[33] 宋燕子, 贾彬, 林柏成, 胡章立, 黄瑛. 莱茵衣藻酰基辅酶A合成酶cDNA克隆及其酵母表达. 生物技术通报, 2015, 31: 119–124.
Song Y Z, Jia B, Lin B C, Hu Z L, Huang Y. cDNA cloning and yeast expression of acyl-CoA synthetase of., 2015, 31: 119–124 (in Chinese with English abstract).
[34] Aznar-Moreno J A, Venegas Calerón M, Martínez-Force E, Garcés R, Mullen R, Gidda S K, Salas J J. Sunflower () long-chain acyl-coenzyme a synthetases expressed at high levels in developing seeds., 2014, 150: 363–373.
[35] 于莉莉, 谭小力, 侯文胜. 大豆长链脂酰辅酶A合成酶基因在酵母中的表达. 大豆科学, 2011, 30: 719–722.
Yu L L, Tan X L, Hou W S. Enzyme activity analysis ofin., 2011, 30: 719–722 (in Chinese with English abstract).
[36] Chlo G, Thierry J, Julien S, Philippe L, Virginie V, MawussD T A, Thierry B, Dany S, Philippe A, James T, Tristan D G, Fabienne M, Stphane D. Gene coexpression network analysis of oil biosynthesis in an interspecific backcross of oil palm., 2016, 87: 423–441.
[37] Kitajima-Koga A, Baslam M, Hamada Y, Ito N, Taniuchi T, Takamatsu T, Oikawa K, Kaneko K, Mitsui T. Functional analysis of rice long-chain acyl-CoA synthetase 9 (OsLACS9) in the chloroplast envelope membrane., 2020, 21: 2223.
[38] McFarlane H E, Watanabe Y, Yang W, Huang Y, Ohlrogge J, Samuels A L. Golgi- and trans-Golgi network-mediated vesicle trafficking is required for wax secretion from epidermal cells., 2014, 164: 1250–1260.
Cloning and function analysis of agene inL.
YANG Jia-Bao1, ZHANG Zhan2, ZHOU Zhi-Ming1, LYU Xin-Hua1, and SUN Li1,*
1College of Life Sciences, Shihezi University, Shihezi 832003, Xinjiang, China;2Bingtuan Xingxin Vocational and Technical College, Tiemenguan 841007, Xinjiang, China
Long-chain acyl-coenzyme A (CoA) synthetase (LACS) catalyzes the formation of acyl-CoAs from free fatty acids, which plays important roles in lipid metabolism in plant. Here, a LACS family gene namedwas screened and cloned from sunflower (L.). Homologous protein comparison showed that HaLACS9 had conserved LACS domains and had higher homology with,andLACS9 proteins. Subcellular localization prediction indicated that HaLACS9 was localized in the chloroplast. The promoter region ofgene contained a large number of hormone and stress responses related elements. The qRT-PCR revealed thatgene was ubiquitously expressed in sunflower organs, and the highest expressed in seeds at 10 days after flowering. The transcription levels ofwere induced by drought, salt, ABA, and GA3treatments in sunflower roots, stems, and leaves. The relative expression pattern ofat seed developmental stages in sunflower was higher at the early seed developmental stages, related to sunflower seed oil rapid accumulation periods. The relative expression ofwas gradually decreased at late stages of seed development and the slow accumulation rate of oil. HaLACS9 was proved to have the synthetase activity of acyl-CoA in complementation test of deficient yeast mutant and prefers to activate oleic acid substrate. It was speculated thatparticipated in the regulation of sunflower seed lipid biosynthesis and abiotic stress responses.
sunflower;; expression patterns; yeast complementation
10.3724/SP.J.1006.2023.14251
本研究由国家自然科学基金项目(31760064, 31360052)资助。
This study was supported by the National Natural Science Foundation of China (31760064, 31360052).
孙黎, E-mail: sunlishz@126.com
E-mail: 2516040371@qq.com
2021-12-29;
2022-06-07;
2022-07-07.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220706.1530.008.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
