钙调素基因(HcCaM7)及其蛋白乙酰化修饰参与红麻响应非生物胁迫的作用
2023-02-10吴启境陈灿妮胡亚丽罗登杰廖长君
黄 震 吴启境 陈灿妮 吴 霞 曹 珊 张 辉 岳 娇 胡亚丽 罗登杰 李 赟 廖长君 李 茹 陈 鹏,*
钙调素基因()及其蛋白乙酰化修饰参与红麻响应非生物胁迫的作用
黄 震1吴启境1陈灿妮1吴 霞1曹 珊1张 辉1岳 娇1胡亚丽1罗登杰1李 赟1廖长君3李 茹2陈 鹏1,*
1广西大学农学院 / 广西高校植物遗传育种重点实验室, 广西南宁 530004;2广西大学生命科学与技术学院, 广西南宁 530004;3广西博世科环保科技股份有限公司, 广西南宁 530007
钙调素是一类钙依赖性调节蛋白, 参与植物的生长发育、抗逆胁迫等多种生物学过程。本课题组前期通过蛋白质乙酰化修饰组学研究发现, 红麻钙调素蛋白7的乙酰化修饰参与了红麻花粉的发育调控。为研究其参与抗逆性的机制, 本研究以红麻保持系P3B双核期的花药为材料, 使用PCR法克隆了钙调素基因, 最大开放阅读框(open reading frame, ORF)为450 bp, 其由149个氨基酸组成, 编码相对分子质量16.85 kD的蛋白; 亚细胞定位结果显示, HcCaM7蛋白的表达主要定位在细胞质和细胞膜中; 利用病毒诱导的基因沉默技术沉默基因, 导致红麻沉默植株的生长受到抑制; 进一步在体外采用基因密码子扩展技术对发生乙酰化修饰氨基酸位点进行突变, 成功获得具有体外乙酰化修饰位点的蛋白HcCaM7mut, 并成功诱导表达了无乙酰化修饰的蛋白HcCaM7, 结果表明HcCaM7蛋白发生乙酰化修饰后可以显著促进NADK (NAD激酶)活性; 用点板法检测含有HcCaM7蛋白和HcCaM7mut蛋白的重组菌在盐(400 mmol L–1和500 mmol L–1NaCl)、干旱(400 mmol L–1和600 mmol L–1甘露醇)、重金属(30 mmol L–1和50 μmol L–1CdCl2)及低温胁迫后(利用液氮反复冻融模拟)在LB固体培养基上的存活率发现, 含有HcCaM7蛋白的重组菌存活率显著高于空载对照菌, 而含有乙酰化修饰的HcCaM7mut蛋白的重组菌存活率进一步提升, 表明HcCaM7蛋白能够提高大肠杆菌对非生物胁迫的耐受性, 并且乙酰化修饰后效果更佳。因此,基因可以调控红麻生长发育和响应非生物胁迫, 乙酰化修饰可以促进HcCaM7蛋白发挥作用。
红麻; 钙调素蛋白7; 蛋白乙酰化修饰; 病毒诱导的基因沉默; 基因密码子扩展技术; 非生物胁迫
红麻(L.)是锦葵科木槿属一年生韧皮纤维作物, 以采收茎秆和韧皮部纤维为主要栽培目的, 主要用途包括纸制品、建筑材料、吸附剂、牲畜饲料、汽车内饰以及生物质能源等[1-2]。红麻具有耐盐、耐旱和耐重金属等特性, 因此被视为环境治理应用方面的潜力作物[3-4]。
钙调素(CaM)是一种广泛存在于生物体内且自身没有酶活性的钙依赖性调节蛋白[5-6], 属三大钙感受器蛋白(CaM/CML、CDPK和CBL蛋白)之一[5-7]。钙调素经过Ca2+激活与其靶蛋白结合, 将钙信号向下游传递, 并与下游响应因子结合, 从而调控植物细胞分裂、生长发育、抗逆等生物学过程[8-9]。植物中, 已有较多有关CaM蛋白对增强植物抗逆性的研究报道: 在拟南芥中, 过表达或基因都能够显著提高拟南芥的抗盐能力[10], 过表达基因能够显著提高拟南芥抗高温能力[11], 过表达基因能够显著提高拟南芥的抗冷胁迫能力[12], 过表达基因增强拟南芥盐胁迫耐受性[13],在拟南芥中异位超表达能显著提高拟南芥的耐热性[14]; 在水稻中, OsCaM1-1蛋白能够提高水稻的耐盐能力和促进侧根的生长[15]; 在烟草中, 异源超表达一种南极抗冻鱼()的CaM基因, 能够增强其耐冷能力[16-17]。相关试验研究果表明, CaM蛋白在植物抗逆方面的作用十分丰富, 甚至一种蛋白具有多种抗逆境作用, 但是在红麻中, 有关CaM7功能的报道甚少, 研究其功能对于红麻抗逆研究具有重要意义。
蛋白质的翻译后修饰(post-translational modifications, PTMs)在很大程度上决定了蛋白质的活性、功能发挥、细胞定位、稳定性及蛋白质之间的相互作用等生物学功能, 对蛋白质功能起着精细的调节作用[18]。细胞内存在多种蛋白质翻译后调控方式, 如泛素化、磷酸化、乙酰化等, 它们都对细胞蛋白质发挥正常的生物学功能有着至关重要的作用[19]。近年来, 赖氨酸乙酰化作为一种动态可逆的翻译后修饰, 引起了广泛的关注, 它主要包括组蛋白乙酰化和非组蛋白乙酰化[20-22]。在植物中, 乙酰化修饰在抗逆性作用过程中扮演重要角色, 蛋白质去乙酰化可调控生物逆境与非生物逆境相关基因的表达来适应环境, 如是最早被研究并被证明和植物抗逆相关的去乙酰化酶基因[23], 转基因拟南芥植物中,基因的过表达显著增强了拟南芥对盐和干旱胁迫的耐受性[24]。有研究表明, 组蛋白的乙酰化修饰可以调控茉莉酸的合成和信号途径, 从而提高多种植物的抗旱能力[25]。当水稻去乙酰化酶基因缺失以后, 水稻的抗胁迫能力增强[26]; 当番茄受到盐胁迫以后,、、和表达上调,和表达下调[27], 从而增强了番茄的耐盐能力。HcCaM7蛋白在P3A中的乙酰化水平显著低于P3B, 因此推测CaM7蛋白及其乙酰化修饰参与调控红麻细胞质雄性不育的发生以及红麻非生物胁迫抗性[1]。
近几年来, 非天然氨基酸(unnatural amino acid, ncAAs)的基因编码技术成为了研究蛋白质功能的有效手段, 基因密码子扩展技术(genetic code expansion strategy)的原理是使用一对正交的氨基酰基-tRNA合成酶(aaRS)和tRNA来指导非天然氨基酸与一个新的功能群结合, 以响应蛋白质中精确控制位置的特定密码子[28]。此项技术主要使用aaRS (aminoacyl-tRNA synthetase)/tRNA正交系统在发生无义突变的位点通读突变密码子, 达到翻译全长蛋白的目的[29-30], 因此, 基因密码子扩展技术成为了研究蛋白质翻译后修饰的一种重要手段。
HcCaM7蛋白是红麻CaM家族中的一员, 目前还没有HcCaM7蛋白及其乙酰化修饰参与红麻生长发育及逆境胁迫调控作用的相关报道。本研究克隆了基因, 对其进行生物信息学分析和亚细胞定位; 采用VIGS基因沉默技术研究了其在红麻自然生长条件下的功能; 并进一步利用基因密码子扩展技术和体外表达分析了乙酰化修饰对HcCaM7蛋白功能发挥的作用。该研究结果为明确基因及其蛋白乙酰化修饰在调控红麻生长、发育及逆境胁迫响应等功能方面提供理论依据。
1 材料与方法
1.1 材料
本试验所用红麻材料为不育系P3A和保持系P3B, 红麻植株种植于广西大学试验田(22°48'N, 108°22'E), 种植期间进行常规的田间管理。菌种GV3101、DH5α、BL21(DE3)等均由本实验室保存。
1.2 红麻总RNA提取与第1链cDNA的合成
使用RNA提取试剂盒(Vazyme, RC101)提取P3B双核期花药的总RNA。用1.0%琼脂糖凝胶电泳检测 RNA 的完整性, 并用NanoDrop ND-100检测RNA的浓度和纯度。使用诺唯赞反转录试剂盒(R312-01)合成第一链cDNA, 并置于–20℃保存备用。
1.3 HcCaM7基因的克隆、表达分析
根据红麻基因组的序列, 利用Primer Premier 5.0软件设计扩增HcCaM7 (基因号为Hc.16G007630.t1)全长的特异性引物HcCaM7 (表1), 以P3B花药cDNA为模板, 采用高保真Mix (Vazyme, P520)进行扩增。扩增产物电泳后切胶回收并连接到克隆载体pEASY-Blunt, 转入大肠杆菌DH5α, 挑选阳性克隆进行测序。分别以红麻不育系P3A和保持系P3B的根、茎、叶的cDNA为模板, 以红麻为内参基因, 通过qRT-PCR研究基因在P3A和P3B不同组织中的表达差异。
1.4 HcCaM7基因的生物信息学分析
分别在NCBI网站(https://www.ncbi.nlm.nih. gov/Structure/cdd/wrpsb.cgi)和DNAMAN软件中对进行基因结构预测和序列比对; 利用MEGA7软件的Neighbor Joining (NJ)构建进化树。用expasys WISS网站(https://www.swissmodel. expasy.org/)预测蛋白质的二级和三级结构。
1.5 HcCaM7蛋白亚细胞定位分析
设计HcCaM7-GFP引物(表1), 扩增目的基因编码区序列。利用重组法(Vazyme, C112)将目的基因与带有GFP标签的pRTVcGFP载体进行连接, 将融合表达载体转入大肠杆菌DH5α感受态, 挑选测序正确的克隆子提取质粒。通过原生质体瞬时转染技术将-GFP重组质粒转化到拟南芥原生质体, 避光和室温条件下孵育12 h。用剪口枪头向载玻片中加入少量原生质体溶液, 将盖玻片从一端轻轻盖在载玻片上, 尽量避免出现气泡, 激光共聚焦显微镜下观察绿色荧光以研究目的蛋白的亚细胞定位情况, 并照相记录。
1.6 病毒诱导的基因沉默(virus-induced gene silencing, VIGS)分析
根据编码序列设计带有pTRV2载体序列信息引物HcCaM7-pTRV2 (表1), 扩增262 bp片段, 连接到pTRV2载体, 测序成功后提取质粒转化到农杆菌GV3101中。选择pTRV1、pTRV2空载体和重组质粒-pTRV2单克隆农杆菌转化子, 在含50 mg L–1卡那霉素和50 mg L–1利福平的LB液体培养基中, 28℃ 220转 min–1振荡过夜, 使OD600值到1.0左右, 7000´离心5 min收集菌液, 用重悬液(10 mmol L–12-甲基乙酸磺酸盐、200 μmol L–1乙酰丁香酮、10 mmol L–1MgCl2)重悬菌体至OD600= 0.8~1.0, 之后将pTRV2分别与pTRV2空载体和- pTRV2按体积比1∶1混合, 室温下暗置2~3 h。随后用1 mL注射器注射红麻叶片背面, 注射面积达到90%, 24℃暗培养24 h, 转入正常光培养14 d左右, 取单株叶片, 提取RNA并进行荧光定量PCR法检测相对表达量。以注射了pTRV2菌液的植株为对照, 筛选出表达量降低的沉默植株, 并进行表型观察。
1.7 HcCaM7基因和HcCaM7蛋白在红麻P3A和P3B花药中表达差异分析
为判断基因与HcCaM7蛋白在红麻P3A和P3B花药中表达量差异是否导致HcCaM7蛋白乙酰化修饰水平的差异, 根据的全长序列设计引物荧光定量PCR引物HcCaM7-qPCR (表1),以红麻为内参基因, 红麻不育系P3A为对照, 利用2–ΔΔCT法[31]计算目的基因的相对表达量。
将纯化的HcCaM7蛋白制备Rabbit anti- HcCaM7抗体(抗体由南京金斯瑞公司制备)。将Rabbit anti-HcCaM7作为一抗, HRP-conjugated Goat Anti-Rabbit IgG (Sangon Biotech)作为二抗, Western-blot检测HcCaM7蛋白在红麻P3A和P3B中的表达量。
1.8 基于基因密码子扩展技术的乙酰化位点定点突变
本研究通过运用基因密码子扩展技术进行体外诱导具有乙酰化修饰的HcCaM7mut蛋白, 研究乙酰化修饰对HcCaM7蛋白功能的影响。本研究中所用的pTECH-MbAcK3RS (IPYE)质粒是编码MbAcK3RS (IPYE)和pylT的表达质粒[32-34], 运用该质粒就能够识别终止密码子TAG并将乙酰赖氨酸(AcK)携带到TAG处, 进行翻译。根据课题组前期的蛋白组数据, 找出HcCaM7的乙酰化修饰位点, 将其突变成为终止密码子TAG, 之后运用基因密码子扩展技术[35], 将乙酰化赖氨酸引入到TAG位点上。具体操作如下: 利用CE design设计点突变引物HcCaM7mut (表1), 乙酰化修饰位点来自课题组前期研究[1], 以1.3中的-pEASY-Blunt质粒为模板进行PCR扩增, 将扩增产物经I消化处理后进行胶回收, 将回收产物用重组酶连接成环, 转入大肠杆菌DH5α感受态, 提取质粒测序。最终HcCaM7编码赖氨酸的密码子AAG成功被突变成TAG, 提取重组质粒HcCaM7mut-pEASY-Blunt。
1.9 蛋白表达及纯化
设计引物HcCaM7-PET32α (表1), 分别以HcCaM7-pEASY-Blunt和HcCaM7mut-pEASY-Blunt为模板进行扩增, 利用无缝克隆的方法将它们连接到pET32α。BL21 (DE3)菌株的蛋白乙酰化水平显著低于大肠杆菌K12衍生菌株, 故将其作为表达宿主[36], 将重组质粒HcCaM7-PET32α和HcCaM7mut- pET32α分别与含有ACK结合系统的pTECH质粒一起转入大肠杆菌BL21 (DE3)中。选择HcCaM7和HcCaM7mut单克隆菌落接种于含有氨苄青霉素和氯霉素的LB培养基中, 置于37℃、220转min–1摇床中摇菌培养过夜。次日, 将菌液接种到100 mL含有氨苄青霉素和氯霉素的LB培养基中, 接种比例为1∶100。将培养液置于37℃、220转min-1的摇床上, 摇至OD600达到0.6~0.8。取菌液冰浴30 min后, 加入终浓度为20 mmol L–1的NAM(烟酰胺, 去乙酰化酶抑制剂)、10 mmol L–1的ACK (乙酰赖氨酸)和0.4 mmol L–1的IPTG, 在28℃条件下诱导蛋白表达。重组蛋白用Ni-IDA 6 FF Sefinose Resin Kit (Sangon Biotech)纯化, 并将含有融合蛋白的上清样品加入镍柱中, 镍柱用结合缓冲液平衡。收集流出来的液体, 上柱重复1~2次。用含60 mmol L–1咪唑的洗涤液以至少5~10倍的柱床体积冲洗杂蛋白。然后用0.5倍柱床体积、含有250 mmol L–1咪唑的蛋白缓冲液连续洗脱融合蛋白6次。
1.10 Western-blot检测
取纯化后的蛋白, 经裂解变性后进行SDS- PAGE电泳, 考马斯亮蓝染色和Western-blot分析。用一抗Anti-6×His Tag mouse monoclonal antibody (Sangon Biotech)和二抗AP-conjugated Goat Anti- mouse IgG (Sangon Biotech)检测是否含有His标签; 用一抗Ac-lysine Antibody (Santa Cruz Biotechnology)和二抗AP-conjugated Goat Anti-mouse IgG (Sangon Biotech)检测乙酰化修饰水平。剩余的蛋白用液氮冷冻, 放在-80℃冰箱下保存, 备用。
1.11 HcCaM7蛋白的乙酰化修饰对红麻NADK酶活的影响
用磷酸盐缓冲溶液分别将无乙酰化修饰的蛋白HcCaM7和发生了乙酰化修饰的蛋白HcCaM7mut配制为蛋白浓度为0.4 mg mL–1的溶液(pH 5.5)。用NAD激酶试剂盒(苏州科铭生物技术有限公司)提取红麻NADK酶液, 蛋白溶液按照体积比1∶10添加到红麻NADK酶液中, 以红麻NADK酶液为空白组,按照NAD激酶试剂盒说明书检测NADK酶活, 并将其NADK酶活力定义为100%, 评价HcCaM7和HcCaM7mut对红麻NADK活力的影响, 从而分析乙酰化修饰对HcCaM7的影响。
1.12 CaM7乙酰化修饰对大肠杆菌响应非生物胁迫的影响
对照菌pET32α以及1.7中的含有HcCaM7蛋白、HcCaM7mut蛋白的重组菌, 当OD600达到0.6, 加入终浓度为 0.4 mmol L–1的IPTG, 28℃、200转 min–16 h诱导培养, 将3种菌稀释至相同浓度(OD600=1.2), 进行不同非生物胁迫抗性检测存活率, 试验重复3次。
氯化钠、甘露醇、重金属Cd抗性检测: 通过预试验确定氯化钠的处理浓度为400 mmol L–1和500 mmol L–1, 甘露醇的处理浓度为400 mmol L–1和600 mmol L–1, CdCl2的处理浓度为30 mmol L–1和50 μmol L–1, 取6 μL 3种经梯度稀释后的菌液分别垂直加到LB固体培养基(含有0.4 mmol L–1的IPTG和NaCl/甘露醇/CdCl2)上, 37℃, 倒置培养12 h, 统计存活率。
低温抗性检测: 取1 mL的3种菌液于液氮中反复冻融6次, 然后取经梯度稀释后取6 μL分别垂直加到LB固体培养基(含有0.4 mmol L–1IPTG)上, 37℃, 倒置培养12 h, 统计存活率。
2 结果与分析
2.1 HcCaM7基因的克隆及在不同组织表达分析
以红麻花药RNA反转录获得cDNA为模板, 进行PCR扩增, ORF长度为450 bp (图1-A), 经序列比对, 测序结果为目的序列。通过qRT-PCR研究发现,基因在P3A根部的表达量显著高于P3B, 在茎中两者表达量几乎一致, 在P3B的叶片中,基因表达量显著高于P3A (图1-B)。
2.2 HcCaM7的生物信息学分析
基因ORF编码149个氨基酸残基的蛋白。理化性质分析表明, HcCaM7蛋白的相对分子量为16.85 kD, 理论等电点为4.11。蛋白功能域预测结果显示(图2-A), HcCaM7蛋白是PTZ00184 super family成员, 其三级结构为EF结构(图2-B)。从HcCaM7与其他物种的氨基酸序列进行进化树构建可知, 红麻的CaM7蛋白和木槿的CaM7蛋白亲缘关系最近, 其次为陆地棉CaM7蛋白(图2-C)。

表1 研究所用引物列表
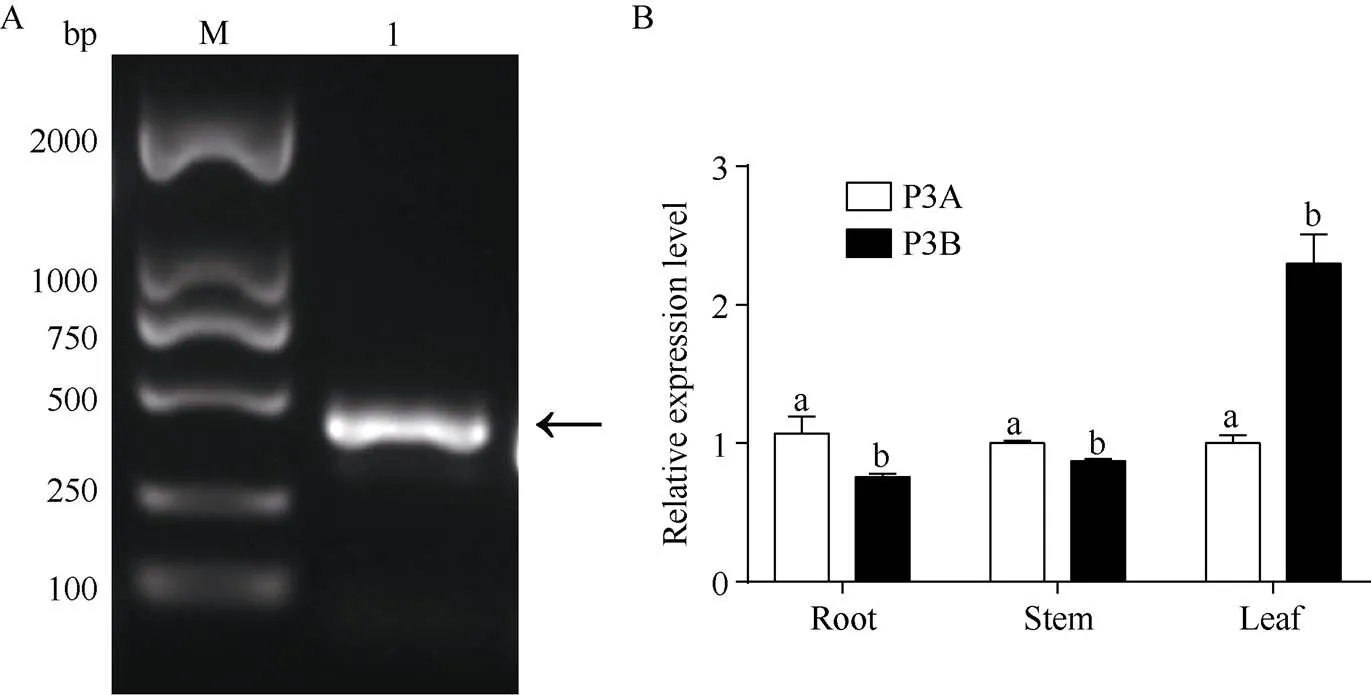
图1 HcCaM7基因的克隆及表达分析
A:的PCR扩增产物; M: DL2000 marker; 1:ORF扩增。B:基因在红麻P3A和P3B不同组织中表达分析。数据为3个生物学重复±标准差。柱上不同小写字母表示差异显著(< 0.05)。
A: PCR amplification product of; M: DL2000 marker; 1:ORF amplification product. B: expression analysis ofgene in different tissues of kenaf P3A and P3B. The error bar represents ± SD of three biological replication. Different lowercase letters are significantly different at the 0.05 probability level.
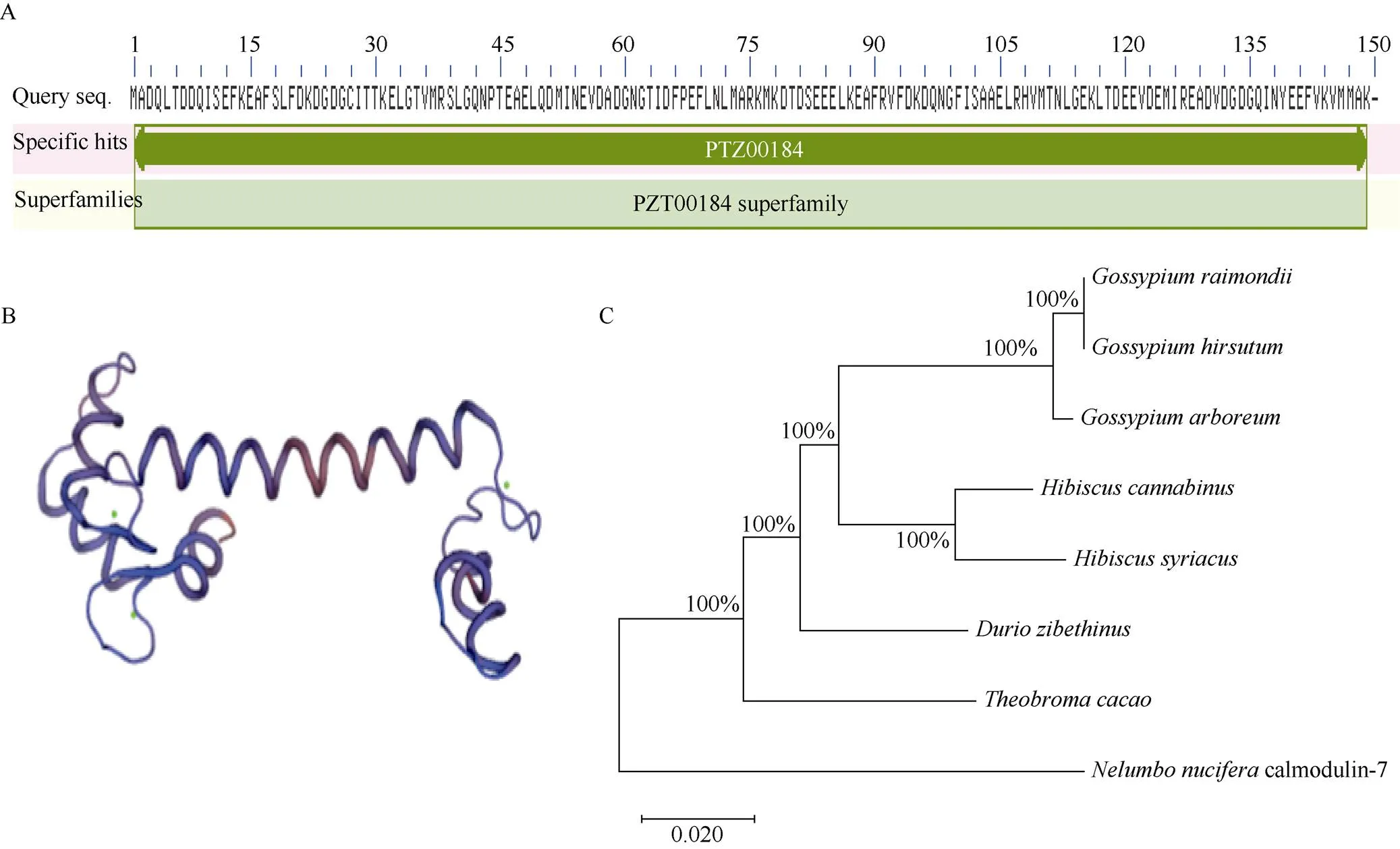
图2 HcCaM7的生物信息学分析
A: HcCaM7蛋白保守结构域的预测; B: HcCaM7蛋白三级结构预测结果; C: HcCaM7蛋白的系统发育树分析。
A: the prediction of conserved domains of HcCaM7; B: the predicted 3D structure of HcCaM7 protein; C: the phylogenetic tree of HcCaM7 proteins.
2.3 HcCaM7蛋白亚细胞定位
观察转入HcCaM7pRTVcGFP融合表达载体的原生质体发现, HcCaM7pRTVcGFP在细胞质和细胞膜发出明显绿色荧光(图3), 表明HcCaM7蛋白定位在细胞质和细胞膜上, 这与网站在线预测的结果相一致。
2.4 HcCaM7基因VIGS沉默分析
研究表明, 当植物中的基因通过VIGS沉默后表达水平降低到40%以下时, 植株表型一般会有显著差异[31]。本研究用qPCR检测到了9株注射-pTRV2载体的植株, 其表达量均低于注射pTRV2空载体(对照)的植株的40%以上(图4-A)。结果显示, 与空白对照CK和空载对照pTRV2植株相比, HcCaM7-pTRV2植株的生长受到明显的抑制, 其中鲜重和株高分别比空载对照pTRV2植株显著下降31.1%和18.4% (图4-B, C)。沉默HcC后, 红麻植株的生长整体受到了显著抑制作用, 表明在红麻正常生长发育过程中发挥着十分重要的作用。
2.5 HcCaM7基因及其蛋白相对表达量分析
对红麻不育系P3A和保持系中的P3B双核期花药中基因的相对表达量分析(图5-A)表明,在两者花药中表达量相近, 没有显著差异; 在红麻P3A和P3B花药中, HcCaM7蛋白表达量也几乎一致(图5-B)。表明在红麻P3A和P3B中CaM7蛋白的乙酰化修饰差异不是由基因表达量差异导致的。

图3 HcCaM7蛋白亚细胞定位
A: Mcherry通道; B: GFP通道; C: 明场; D: 融合场。
A: Mcherry channel; B: GFP channel; C: bright channel; D: merge channel.
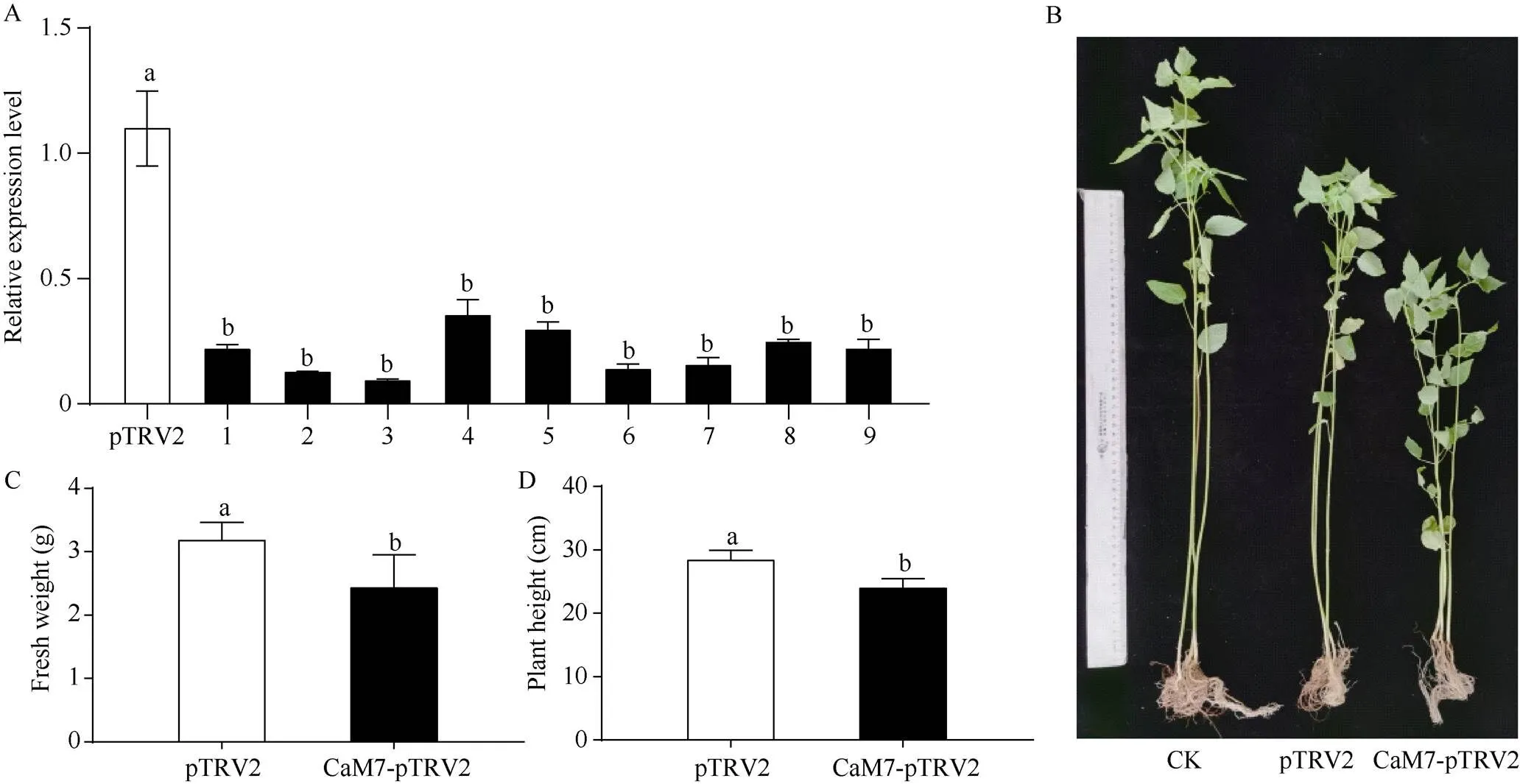
图4 HcCaM7基因VIGS沉默分析
A: 转基因植株的qRT-PCR分析, pTRV2空载为对照, 1~9代表基因沉默植株; B: 沉默植株的表型分析; C: CaM7-pTRV2红麻植株鲜重; D: CaM7-pTRV2红麻植株株高。数据为3个生物学重复±标准差; 不同小写字母表示之间差异显著(< 0.05)。
A: the qRT-PCR of transgenic plants. The control plant of empty vector is pTRV2. 1–9 represents gene silencing plants of. B: phenotypic characters ofsilencing plants; C: the fresh weight of kenaf CaM7-pTRV2; D: plant height of kenaf CaM7-pTRV2. Values are means ± SDs of three biological replication. Different lowercase letters are significantly different at the 0.05 probability level.
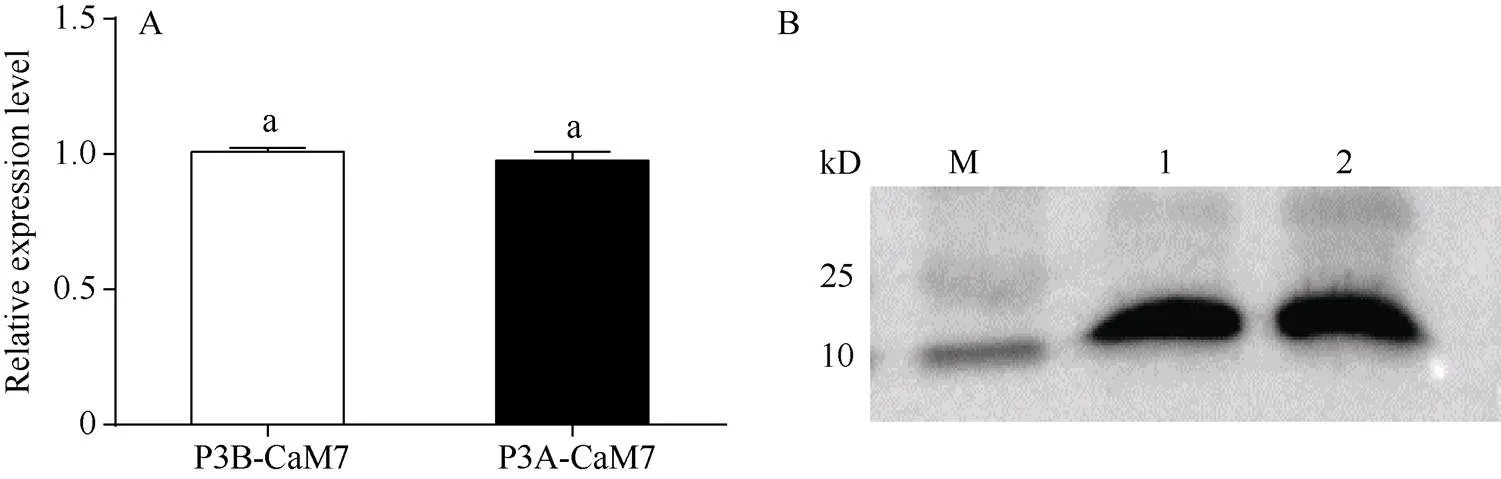
图5 红麻P3A和P3B花药中HcCaM7基因和其蛋白在表达水平定量分析
A:在红麻P3A和P3B花药中相对表达量; B: HcCaM7蛋白在红麻P3A和P3B花药中表达水平, 1: P3A的花药, 2: P3B的花药。数据为3个生物学重复±标准差; 不同小写字母表示之间差异显著(< 0.05)。
A: the relative expression levels ofgenes in kenaf P3A and P3B anthers; B: the relative expression level of HcCaM7 protein in the anthers of P3A and P3B of kenaf, 1 and 2 represent the anther of P3A and P3B, respectively. Values are means ± SDs with three biological replication. Different lowercase letters are significantly different at the 0.05 probability level.
2.6 HcCaM7基因定点突变
由图5可知, 在红麻P3A和P3B中, HcCaM7蛋白乙酰化修饰水平差异不是由基因表达量和蛋白表达量差异造成, 因此, 运用基因密码子扩展技术进行体外诱导具有乙酰化修饰的HcCaM7mut蛋白,究乙酰化修饰对HcCaM7蛋白功能的影响。
根据课题组前期的蛋白组数据发现, HcCaM7蛋白的乙酰化修饰位点只有一个, 在第59位置的赖氨酸残基上, 命名其为K59。对K59进行定点突变, 将测序结果和原序列比对, 发现K59的AAG成功被突变为终止密码子TAG (图6)。将突变后的HcCaM7mut连接到pET-32α载体后, 与pTECH质粒共转化大肠杆菌BL21 (DE3), 诱导具有乙酰化修饰的HcCaM7mut蛋白。

图6 HcCaM7蛋白乙酰化修饰位点定点突变
2.7 HcCaM7蛋白及其点突变蛋白诱导纯化及Western-blot检测分析
诱导蛋白发现, 在28℃、6 h的条件下, 可以得到较多的可溶性蛋白。上清液经Ni-IDA亲和层析纯化分析, 洗脱可以获得较高浓度的蛋白带。pET-32α空载体能诱导出20 kD大小的蛋白, 因此, 目标蛋白大小为36.8 kD (图7-A, B)。Western-blot检测发现, HcCaM7蛋白和HcCaM7mut蛋白都有His标签, 可以确定为重组蛋白(图7-C)。进一步的经乙酰化抗体孵育结果显示, HcCaM7蛋白不存在乙酰化修饰, 而HcCaM7mut蛋白存在较高水平的乙酰化修饰(图7-D)。
2.8 HcCaM7蛋白乙酰化修饰促进了NADK活性
CaM可以与NADK结合并促进NADK的活性; 将无乙酰化修饰的蛋白和有乙酰化修饰的蛋白分别加入到红麻NADK酶液中, 通过分析NADK酶活的变化来看探讨乙酰化对HcCaM7功能的影响。结果显示, NADK酶液中加入了HcCaM7蛋白之后, 其活性显著提高; 加入具有乙酰化修饰的HcCaM7mut蛋白后活性进一步提高, 比加入HcCaM7蛋白高了11.37% (图8)。表明HcCaM7蛋白能够提高红麻NADK的活性, 推测乙酰化修饰HcCaM7蛋白后可以进一步促进HcCaM7蛋白发挥作用。
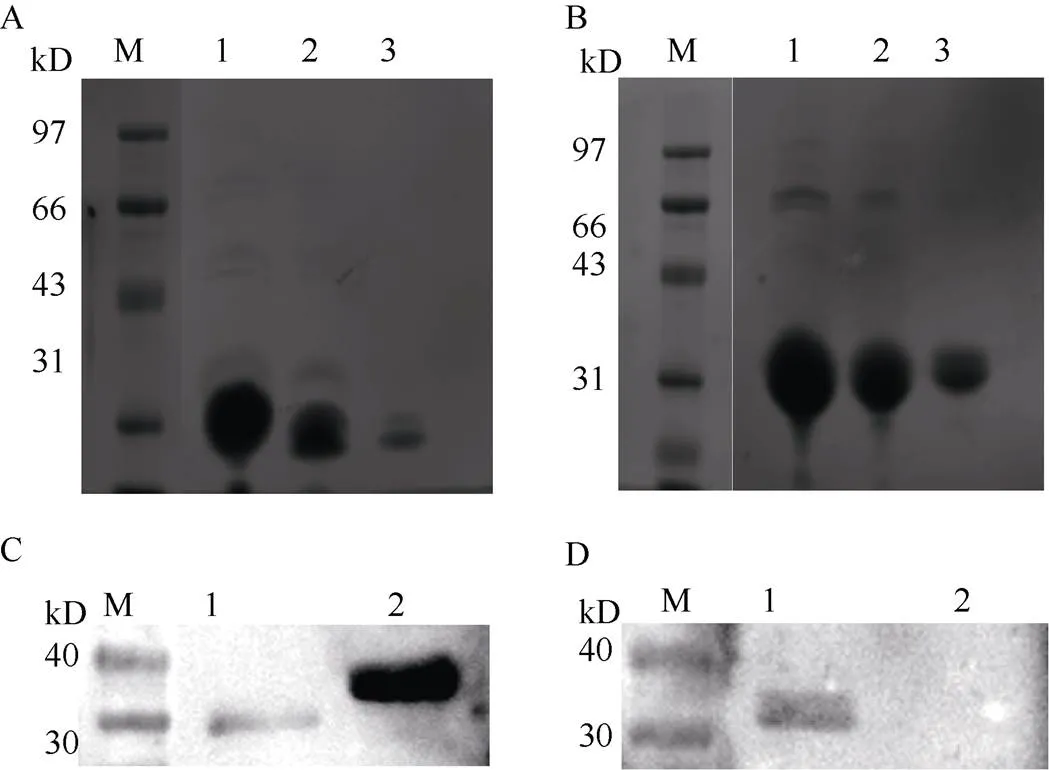
图7 HcCaM7蛋白及其点突变蛋白纯化结果及His标签和乙酰化修饰检测
A: HcCaM7mut蛋白纯化结果, 1、2、3为纯化结果; B: HcCaM7蛋白纯化结果, 1、2、3为纯化结果; C: His-Tag Western-blot检测结果, 1是HcCaM7mut蛋白, 2是HcCaM7蛋白; D: 蛋白乙酰化修饰Western blot检测结果, 1是HcCaM7mut蛋白, 2是HcCaM7蛋白。
A: the purification results of HcCaM7mut protein; B: the purification results of HcCaM7 protein; C: His-Tag Western-blot detection results, 1 is HcCaM7mut protein, 2 is HcCaM7 protein; D is the acetylation modification test result, 1 is HcCaM7mut protein and 2 is HcCaM7 protein.
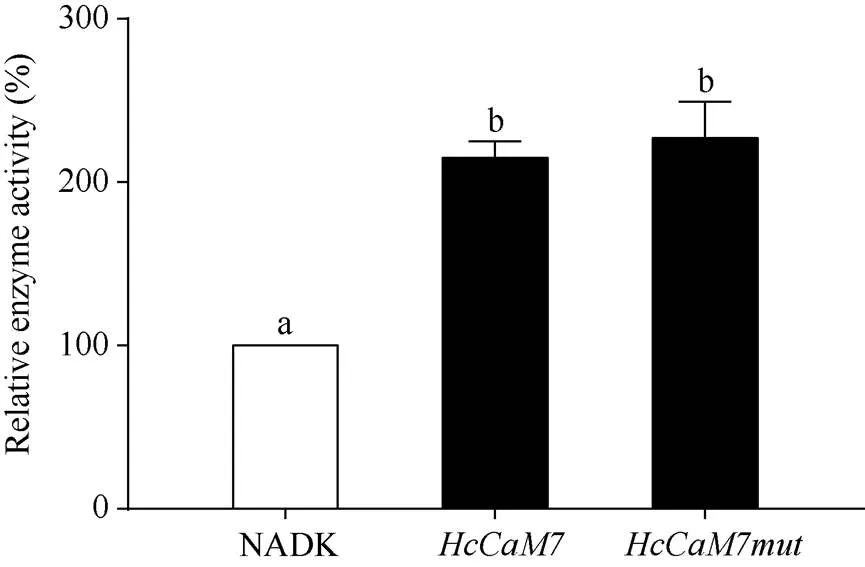
图8 HcCaM7乙酰化修饰对NADK酶活的影响
数据为3个生物学重复±标准差。不同小写字母表示之间差异显著(< 0.05)。
Values are means ± SDs with three biological replication. Different lowercase letters are significantly different at the 0.05 probability level.
2.9 HcCaM7蛋白及其乙酰化修饰在大肠杆菌相应非生物胁迫中的作用
采用点板法对转HcCaM7蛋白和乙酰化修饰的HcCaM7mut蛋白重组菌和空载体对照菌进行了NaCl 抗性、甘露醇抗性、重金属镉抗性和冷胁迫抗性检测, 统计稀释了104倍的菌落数, 结果表明, 对照菌和重组菌在空白LB基本固体培养基上生长良好, 没有显著差异; 但在400 mmol L–1和500 mmol L–1NaCl (图9-A, a)、400 mmol L–1和600 mmol L–1的甘露醇(图9-B, b)、30 mmol L–1和50 μmol L–1的CdCl2(图9-C, c)、液氮反复冻融6次(图9-D, d)处理后, 含有HcCaM7蛋白的重组菌存活率显著高于空载对照菌, 且含有HcCaM7mut的重组菌存活率比含有HcCaM7蛋白的重组菌显著提高, 表明HcCaM7蛋白使大肠杆菌对盐胁迫、甘露醇胁迫、重金属镉胁迫以及冷胁迫的抗性增强, HcCaM7mut蛋白能够使大肠杆菌对这些非生物胁迫的抗性进一步增强。
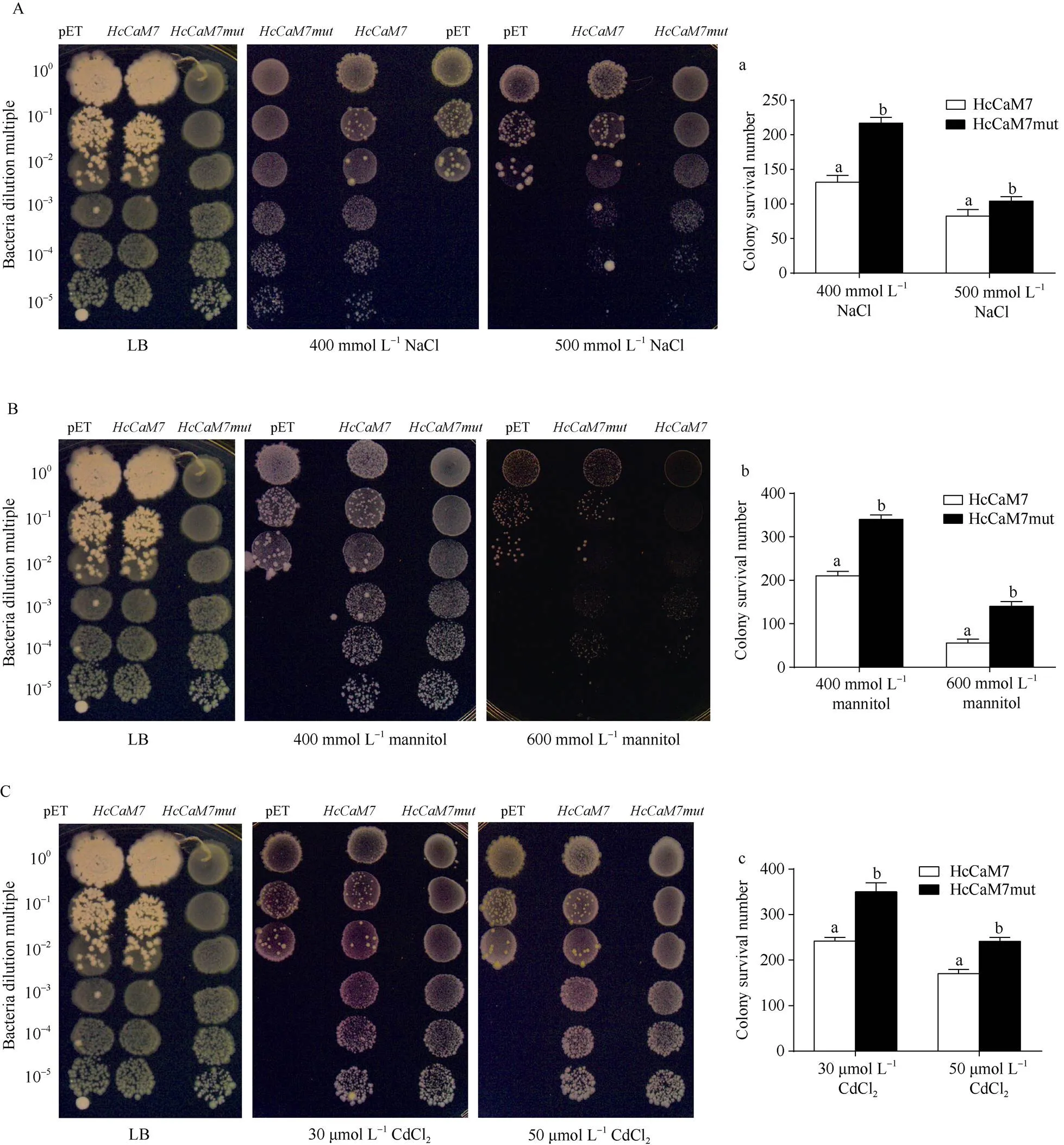
(图9)
A: 盐胁迫; B: 甘露醇胁迫; C: CdCl2胁迫; D: 冷胁迫; a, b, c, d: 胁迫后稀释了104倍的菌落存活数。数据为3个生物学重复±标准差。不同小写字母表示之间差异显著(< 0.05)。
A: salt stress; B: mannitol stress; C: CdCl2stress; D: cold stress; a, b, c, d: the colony survival number diluted 104times after cold stress. Values are means ± SDs with three biological replication. Different lowercase letters are significantly different at the 0.05 probability level.
3 讨论
3.1 HcCaM7基因及其蛋白乙酰化修饰参与调控了红麻生长发育
Ca2+作为植物体内的一种信号载体, 是调节植物发育进程的第二信使, 植物体内通过Ca2+浓度变化来传递信号从而进行调节和控制着一系列细胞和生理反应[37-38]。目前, 植物CaM是被研究的最清楚的一类钙感受器蛋白, 在植物生长发育过程中的参与的多种调控路径及发挥的作用也已经陆续被报导[39]。有研究者认为, NAD激酶、NADP磷酸酶与植物的种子萌发、抗逆性以及育性等多种生理过程密切相关[40]。因此, CaM可以通过调节与其相互作用的酶等来影响植物体生长发育、育性以及抗逆[41]。本研究基于实验室前期蛋白质组学研究结果, 成功从红麻中克隆得到了基因, 得知其编码149个氨基酸, 它是CaM家族中的一员。亚细胞定位结果显示,定位在细胞膜和细胞质上, 因而猜想, 当受到外界刺激的时候, 能够很快接触到信号并做出反应。通过VIGS技术发现, 当红麻幼苗的基因被沉默以后, 红麻幼苗生长显著受到了抑制, 具体表现在株高、鲜重方面, 因此,在红麻生长方面具有十分重要的作用。
乙酰化修饰有时会对蛋白产生巨大的影响, 甚至使酶活发生显著提升或者下降[20], 因而研究乙酰化修饰对于研究蛋白功能十分重要。将具有乙酰化修饰的HcCaM7mut蛋白和无乙酰化修饰的HcCaM7蛋白与红麻NADK酶液混合后发现, NADK的活性均提高了, 表明HcCaM7蛋白对NADK活性具有促进作用; 之后发现具有乙酰化修饰的HcCaM7mut蛋白对酶活的提高大于无乙酰化修饰的HcCaM7蛋白, 证明了乙酰化修饰能够促进HcCaM7发挥作用, 对HcCaM7具有正调控作用。据报道, 经过活化的CaM可以通过直接与NADK结合来发挥作用[5], 因此推测乙酰化修饰可能通过改变了HcCaM7蛋白的构象来促进其与NADK结合, 从而增加了NADK的活性。在红麻P3A和P3B的双核期花药中,的表达量几乎一致, 但是P3B花药中HcCaM7蛋白乙酰化水平高于P3A, 由此推测, HcCaM7蛋白的乙酰化修饰差异导致对红麻花药的生命活动如能量代谢等产生了影响, 从而影响红麻的育性。
3.2 乙酰化修饰增强HcCaM7蛋白应对逆境胁迫能力
目前, 已经有上百种非天然氨基酸可以经过基因密码子扩展技术在翻译过程中被特异的整合到蛋白质特定位置[42], 从而得到含有非天然氨基酸的蛋白质。该技术已经在不少研究中被使用过, 已经比较成熟[34,43], 基因密码子扩展技术还使位点特异性标记的细胞蛋白的活细胞和超分辨率成像成为可能[44]。赖氨酸残基可逆乙酰化是一种广泛存在于自然界的翻译后修饰, 如细胞信号转导和能量代谢过程中, 乙酰化修饰可以通过调节相关酶的活性、蛋白质的结构等来调节生命活动。乙酰辅酶a合成酶1 (Acetyl-coA synase 1, ACSS1)和乙酰辅酶a合成酶2 (ACSS2)分别位于细胞质基质和线粒体中, 乙酰化修饰抑制了ACSS1和ACSS2的活性, 而SIRT1和SIRT3介导的去乙酰化恢复了ACSS1和ACSS2的活性[45]。本研究中, 基因密码子扩展技术利用改造后的合成酶[32]和优化后的同源tRNA pyl[28], 将乙酰赖氨酸引入到经过定点突变的HcCaM7基因中的TAG终止密码子上, 从而在K59上产生乙酰化修饰的HcCaM7mut突变体。运用基因密码子扩展技术和原核表达, 成功得到了具有乙酰化修饰HcCaM7mut蛋白和无乙酰化修饰的HcCaM7蛋白。本研究通过非生物胁迫试验发现, HcCaM7蛋白提高了大肠杆菌的抗甘露醇、盐、重金属Cd以及冷胁迫的能力, 当HcCaM7蛋白经过乙酰化修饰以后, 使大肠杆菌对非生物胁迫的抗性得到进一步的加强。我们推测, 乙酰化修饰能够增强HcCaM7蛋白的活性或者使HcCaM7蛋白的构象发生改变, 更利于其与抗逆境相关的酶、物质结合, 促进相关代谢, 从而增强植株抗逆性。
4 结论
本研究克隆了红麻基因, 其表达主要于细胞质和细胞膜中, 参与了红麻的生长调控。HcCaM7蛋白乙酰化修饰差异不是由其本身基因及其蛋白表达水平的差异导致的。利用密码子扩展技术研究表明体外表达的HcCaM7蛋白可以增强大肠杆菌的非生物胁迫的抗性, 发生乙酰化修饰后, 这种作用更加明显。因此, HcCaM7蛋白可能参与红麻生长发育和在逆境胁迫发挥重要作用, 乙酰化修饰能够对HcCaM7蛋白的作用进行正调控, 促进其进一步发挥功能。
[1] Chen P, Wei F, Li R, Li Z Q, Kashif M H, Zhou R Y. Comparative acetylomic analysis reveals differentially acetylated proteins regulating anther and pollen development in kenaf cytoplasmic male sterility line., 2019, 166: 960–978.
[2] Chen P, Li Z Q, Luo D J, Jia R X, Lu H, Tang M Q, Hu Y L, Yue J, Huang Z. Comparative transcriptomic analysis reveals key genes and pathways in two different cadmium tolerance kenaf (L.) cultivars., 2021, 263: 12.
[3] Tang M Q, Yue J, Huang Z, Hu Y L, Li Z Q, Luo D J, Cao S, Zhang H, Pan J, Wu X, Wu Q J, Chen P. Physiological and DNA methylation analysis provides epigenetic insights into chromium tolerance in kenaf., 2022, 194: 11.
[4] Kashif M H, Wei F, Tang D F, Tang M Q, Luo D J, Hai L, Li R, Chen P. iTRAQ-based comparative proteomic response analysis reveals regulatory pathways and divergent protein targets associated with salt-stress tolerance in kenaf (L.)., 2020, 153: 13.
[5] 林亚, 李世国, 谢莉萍, 张荣庆. 栉孔扇贝钙调素类似蛋白基因的克隆及其与钙调素基因表达特征的比较分析. 水产科学, 2014, 33: 692–701.
Lin Y, Li S G, Xie L P, Zhang R Q. Cloning of calmodulin-like protein gene from Scallop chlamys farreri and comparison of its expression characteristics with calmodulin gene., 2014, 33: 692–701(in Chinese).
[6] 顾采琴. Ca2+、CaM及其目标酶与乙烯诱导番茄和草莓果实成熟衰老关系的研究. 浙江大学博士学位论文, 浙江杭州, 2003.
Gu C Q. Studies of Relationship between Ca2+, CaM, Its Target Enzymes and Ethylene Inducing Maturation, Ripening and Senescence of Tomato and Strawberry Fruits. PhD Dissertation of Zhejiang University, Hangzhou, Zhejiang, China, 2003 (in Chinese with English abstract).
[7] 马力耕, 崔素娟, 徐小冬, 孙大业. G蛋白在细胞外钙调素启动花粉萌发和花粉管伸长中的作用. 自然科学进展, 1997, (6): 149–116.
Ma L G, Cui S J, Xu X D, Sun D Y. Role of G protein in extracellular calmodulin-activated pollen germination and pollen tube elongation., 1997, (6): 149–116 (in Chinese).
[8] Hrabak E M, Chan C W M, Gribskov M, Harper J F, Choi J H, Halford N, Kudla J, Luan S, Nimmo H G, Sussman M R, Thomas M, Walker-Simmons K, Zhu J K, Harmon A C. TheCDPK-SnRK superfamily of protein kinases., 2003, 132: 666–680.
[9] Kim K N, Lee J S, Han H, Choi S A, Go S J, Yoon I S. Isolation and characterization of a novel rice Ca2+-regulated protein kinase gene involved in responses to diverse signals including cold, light, cytokinins, sugars and salts., 2003, 52: 1191–1202.
[10] Zhou S, Jia L, Chu H, Wu D, Peng X, Liu X, Zhang J, Zhao J, Chen K, Zhao L.CaM1 and CaM4 promote nitric oxide production and salt resistance by inhibiting-nitrosoglutathionereductase via direct binding., 2016, 12: 28.
[11] Xuan Y, Zhou S, Wang L, Cheng Y, Zhao L. Nitric oxide functions as a signal and acts upstream ofin thermotolerance inseedlings., 2010, 153: 1895–1906.
[12] Cha J Y, Su'udi M, Kim W Y, Kim D R, Kwak Y S, Son D. Functional characterization of orchardgrass cytosolic Hsp70 (DgHsp70) and the negative regulation by Ca2+/binding., 2012, 58: 29–36.
[13] Yoo J H, Park C Y, Kim J C, Heo W D, Cheong M S, Park H C, Kim M C, Moon B C, Choi M S, Kang Y H, Lee J H, Kim H S, Lee S M, Yoon H W, Lim C O, Yun D J, Lee S Y, Chung W S, Cho M J. Direct interaction of a divergent CaM isoform and the transcription factor, MYB2, enhances salt tolerance in., 2005, 280: 3697–3706.
[14] Wu H C, Luo D L, Vignols F, Jinn T L. Heat shock-induced biphasic Ca2+signature and OsCaM1-1 nuclear localization mediate downstream signalling in acquisition of thermotolerance in rice (L.)., 2012, 35: 1543–1557.
[15] Yang J, Ji L, Liu S, Jing P, Xie G. The CaM1-Associated CCaMK-MKK1/6 cascade positively affects the lateral root growth through auxin signaling under salt stress in rice., 2021, 72: 6611–6627.
[16] Reddy A S N, Ali G S, Celesnik H, Day I S. Coping with stresses: roles of calcium and calcium/calmodulin-regulated gene expression., 2011, 23: 2010–2032.
[17] Yang N, Peng C, Cheng D, Huang Q, Xu G, Gao F, Chen L. The over-expression of calmodulin from Antarctic notothenioid fish increases cold tolerance in tobacco., 2013, 521: 32–37.
[18] Mann M, Jensen O N. Proteomic analysis of post-translational modifications., 2003, 21: 255–261.
[19] Kahn P. Molecular biology: from genome to proteome: looking at a cell’s proteins., 1995, 369–370.
[20] Rardin M J, Newman J C, Held J M, Cusack M P, Sorensen D J, Li B A, Schilling B, Mooney S D, Kahn C R, Verdin E, Gibson B W. Label-free quantitative proteomics of the lysine acetylome in mitochondria identifies substrates of SIRT3 in metabolic pathways., 2013, 110: 6601–6606.
[21] Choudhary C, Kumar C, Gnad F, Nielsen M L, Rehman M, Walther T C, Olsen J V. Mann M. Lysine acetylation targets protein complexes and co-regulates major cellular functions., 2009, 325: 834–840.
[22] Wang Q J, Zhang Y K, Yang C, Xiong H, Lin Y, Yao J, Li H, Xie L, Zhao W, Yao Y F, Ning Z B, Zeng R, Xiong Y, Guan K L, Zhao S M, Zhao G P. Acetylation of metabolic enzymes coordinates carbon source utilization and metabolic flux., 2010, 327: 1004–1007.
[23] Zhou C, Lin Z, Duan J, Miki B, Wu K.HISTONE DEACETYLASEis involved in jasmonic acid and ethylene signaling of pathogen response in arabidopsis., 2005, 17: 1196–1204.
[24] Sridha S, Wu K. Identification of AtHD2C as a novel regulator of abscisic acid responses in., 2006, 46: 124–133.
[25] Kim J M, To T K, Matsui A, Tanoi K, Kobayashi N I, Matsuda F, Habu Y, Ogawa D, Sakamoto T, Matsunaga S. Acetate-mediated novel survival strategy against drought in plants., 2017, 3: 1.
[26] Zhao J, Zhang J, Wei Z, Wu K, Duan J. Expression and functional analysis of the plant-specific histone deacetylase HDT701 in rice., 2014, 5: 8.
[27] Li T, Su H, Li Z, Xu X, Wang H, Li Y, Li Z. Genome-wide identification and expression analysis in biotic and abiotic stress ofgene family in tomato., 2015, 36: 1994–2001.
[28] Fan C, Xiong H, Reynolds N M, Soell D. Rationally evolving tRNA(Pyl) for efficient incorporation of noncanonical amino acids., 2015, 43: 10230–10235.
[29] Shi N N, Yang Q, Zhang H R, Lu J Q, Lin H S, Yang X, Abulimiti A, Cheng J L, Wang Y, Tong L, Wang T C, Zhang X D, Chen H M, Xia Q. Restoration of dystrophin expression in mice by suppressing a nonsense mutation through the incorporation of unnatural amino acids., 2021, 6: 195–206.
[30] Nikic-Spiegel I. Expanding the genetic code for neuronal studies., 2020, 21: 3169–3179.
[31] Li Z Q, Hu Y L, Chang M M, Kashif M H, Tang M Q, Luo D J, Cao S, Lu H, Zhang W, Huang Z, Yue J, Chen P. 5-azacytidine pre-treatment alters DNA methylation levels and induces genes responsive to salt stress in kenaf (L.)., 2021, 271: 10.
[32] Bryson D I, Fan C, Guo L T, Miller C, Soll D, Liu D R. Continuous directed evolution of aminoacyl-tRNA synthetases., 2017, 13: 1253–1260.
[33] Wang L, Brock A, Herberich B, Schultz P G. Expanding the genetic code of., 2001, 292: 498–500.
[34] Tharp J M, Liu W R. Genetic code expansion: Synthetases pick up the PACE., 2017, 13: 1205–1206.
[35] Venkat S, Chen H, Stahman A, Hudson D, McGuire P, Gan Q, Fan C. Characterizing lysine acetylation of isocitrate dehydrogenase in, 2018, 430: 1901–1911.
[36] Weinert B T, Iesmantavicius V, Wagner S A, Scholz C, Gummesson B, Beli P, Nystrom T, Choudhary C. Acetyl-phosphate is a critical determinant of lysine acetylation in., 2013, 51: 265–272.
[37] Berridge M J, Bootman M D, Li P. Calcium: a life and death signal., 1998, 395: 645–648.
[38] Kong D, Ju C, Parihar A, Kim S, Cho D, Kwak J M.glutamate receptor homolog3.5 modulates cytosolic Ca2+level to counteract effect of abscisic acid in seed germination., 2015, 167: 1630–1642.
[39] Cheval C, Aldon D, Galaud J P, Ranty B. Calcium/calmodulin- mediated regulation of plant immunity., 2013, 1833: 1766–1771.
[40] Stephan C, Laval-Martin D L. Changes in NAD(+) kinase activity during germination ofand., and effects of drought stress., 2000, 157: 65–73.
[41] Jiang X, Gao Y, Zhou H, Chen J, Wu J, Zhang S. Apoplastic calmodulin promotes self-incompatibility pollen tube growth by enhancing calcium influx and reactive oxygen species concentration in., 2014, 33: 255–263.
[42] Chung H H, Benson D R, Schultz P G. Probing the structure and mechanism of Ras protein with an expanded genetic code., 1993, 259: 806–9.
[43] Tang H, Zhang P, Luo X. Recent technologies for genetic code expansion and their implications on synthetic biology applications., 2021, 434: 167382–167382.
[44] Uttamapinant C, Howe J D, Lang K, Beranek V, Davis L, Mahesh M, Barry N P, Chin J W. Genetic code expansion enables live-cell and super resolution imaging of site-specifically labeled cellular proteins., 2018, 140: 13986–13986.
[45] Hallows W C, Lee S, Denu J M. Sirtuins deacetylate and activate mammalian acetyl-CoA synthetases., 2006, 103: 10230–10235.
Role of calmodulin gene () and its protein acetylation is involved in kenaf response to abiotic stress
HUANG Zhen1, WU Qi-Jing1, CHEN Can-Ni1, WU Xia1, CAO Shan1, ZHANG Hui1, YUE Jiao1, HU Ya-Li1, LUO Deng-Jie1, LI Yun1, LIAO Chang-Jun3, LI Ru2, and CHEN Peng1,*
1College of Agriculture, Guangxi University / Key Laboratory of Plant Genetics and Breeding, Nanning 530004, Guangxi, China;2College of Life Science and Technology, Guangxi University, Nanning 530004, Guangxi, China;3Guangxi Bossco Environmental Protection Technology, Nanning 530007, Guangxi, China
Calmodulin (CaM) is a kind of calcium-dependent regulatory proteins involved in plant growth and development, stress tolerance, and other biological processes. In the previous study in kenaf acetylome, our team found that the protein acetylation modification calmodulin protein7 (CaM7) was involved in the regulation pollen development in kenaf. In order to explore its specific mechanism, we cloned the calmodulin genefrom kenaf P3B binuclear anther by using the PCR cloning way. Its maximum open reading frame (ORF) was 450 bp, encoding a protein containing 149 amino acids with a molecular weight of 16.85 kD. Subcellular localization revealed thatwas mainly located in cytoplasm and cell membrane. Silencingby virus induced gene silencing technique caused growth inhibition in kenaf. Furthermore, the protein HcCaM7mut with acetylation modification site was successfully obtainedand the expression of HcCaM7 without acetylation modification was successfully induced. Acetylation of HcCaM7 protein significantly promoted NADK (NAD kinase) activity, indicating HcCaM7 acetylation involved in its functional regulation. The recombinant bacteria containing HcCaM7 protein and HcCaM7mut protein were detected in NaCl (400 mmol L–1and 500 mmol L–1NaCl), drought (400 mmol L–1and 600 mmol L-1mannitol) and heavy metals (30 μmol L–1and 50 μmol L–1) by dot plate method. The results showed that the survival rate of recombinant bacteria containing HcCaM7 protein was significantly higher than that of empty control bacteria, in addition, the survival rate of recombinant bacteria containing acetylated HcCaM7mut protein was further improved. The results indicated that HcCaM7 protein could enhance the abiotic stress resistance of, and the effect of acetylation modification was better. Therefore,gene regulated the growth and development of kenaf and abiotic stress resistance, and acetylation modification could promote the role of HcCaM7 protein.
kenaf; calmodulin protein 7; protein acetylation modification; virus induced gene silencing; gene codon extension technology; abiotic stress
10.3724/SP.J.1006.2023.24031
本研究由国家自然科学基金项目(31960368), 财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-16-E14)和广西博世科环保科技股份有限公司合作项目(GXU-BFY-2020-015)资助。
This study was supported by the National Natural Science Foundation of China (31960368), the China Agriculture Research System of MOF and MARA (CARS-16-E14), and the Guangxi Bossco Environmental Protection Technology Co., Ltd. (GXU-BFY-2020-015).
陈鹏, E-mail: hustwell@gmail.com
E-mail: 549935163@qq.com
2022-01-25;
2022-06-07;
2022-07-04.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220701.1635.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
