利用CRISPR/Cas9探究水稻OsPIN5c基因功能
2023-02-10杨晓祎王慧慧张艳雯侯典云张红晓康国章胥华伟
杨晓祎 王慧慧 张艳雯 侯典云 张红晓 康国章 胥华伟,*
利用CRISPR/Cas9探究水稻基因功能
杨晓祎1王慧慧1张艳雯1侯典云1张红晓1康国章2胥华伟1,*
1河南科技大学农学院, 河南洛阳 471000;2河南农业大学省部共建小麦玉米作物学国家重点实验室, 河南郑州 450046
生长素极性运输在植物生长发育中发挥着重要作用, 生长素输出载体蛋白(PIN-FORMED, PIN)是控制生长素极性运输的关键蛋白。虽然部分水稻基因功能已有报道, 但水稻的生物学功能仍有待研究。本研究在第1外显子处设计靶点并构建CRISPR/Cas9载体, 通过转化粳稻品种日本晴获得24株转基因植株, PCR产物直接测序分析表明其中15株发生突变, 突变率为62.5%, 突变类型为双等位突变; 进一步从T1代突变体中筛选获得3种不同的纯合突变体, 将其命名为、和。序列比对分析表明, 这3种类型的突变均造成移码突变和蛋白翻译提前终止, 由原来的398个氨基酸分别缩短为109、106和250个氨基酸; 跨膜螺旋结构域分析表明, 3种突变体中OsPIN5c蛋白的跨膜结构完全消失; 蛋白结构预测表明3种突变体中OsPIN5c蛋白螺旋结构明显减少。突变体的苗高、根长和不定根数均显著低于野生型植株。组织特异性表达表明主要在根中表达。突变体根中和表达量提高; 生长素合成关键基因、、和的表达量也显著提高。突变体根的向重性受到部分抑制。本研究利用CRISPR/Cas9基因编辑技术获得纯合突变体并探讨了基因功能, 为利用基因进行作物遗传改良提供了潜在的基因资源。
; CRISPR/Cas9; 基因功能; 水稻
生长素是最早发现的植物激素, 参与调控几乎所有生长发育过程中的细胞分裂和伸长过程[1]。不同组织部位中生长素的含量和分布与生长素极性运输密切相关[2], 而PIN-FORMED (PIN)蛋白是调控生长素极性运输的重要蛋白家族[3]。水稻(L.)是重要的粮食作物[4], 深入研究基因的功能, 可为调控水稻株型、提高品质及产量等提供潜在的基因资源。
生长素主要在地上部幼嫩组织如叶原基及幼叶中合成[5-6], 然后通过生长素极性运输系统向下运输到成熟器官或组织中发挥作用, 其中至少包括生长素输入载体AUXIN RESISTANT/LIKE RESISTANT (AUX1/LAX)和生长素输入/输入载体多重抗药性/磷酸糖蛋白家族MDR/PGP/ABCB (multidrug resistance-like/P-glycoproteins)以及PIN蛋白家族[7], 其中PIN基因家族在极性运输中起着关键作用[3]。最早对基因的研究来源于双子叶模式植物拟南芥()[8]。拟南芥中共有8个基因,基因家族的每个成员的表达模式均不大相同[8]。基因在拟南芥的生长发育中发挥着重要作用, 如缺失使植物出现异常的针状花序[9], 缺失会使根向重性丧失[10-11], 缺失会使下胚轴向性生长受到抑制等[12]。与拟南芥相似, 水稻也是多基因家族, 水稻中有12个基因, 分别为4个()、1个、3个()、1个、1个和2个(), 其中、和为单子叶植物特有的基因[13-14]。不同基因的组织表达特异性不同[13-14]。的4个基因在根、茎、根茎结合部均能检测到表达, 但以根中表达量最高[15]。其中和共同参与调控根系的发育, 影响水稻的株高、分蘖角度等重要农艺性状;和则共同参与调控稻穗的正常发育[15]。主要在根和根茎结合部表达[13]。过表达会使植株矮化、分蘖数增多等[16];也通过介导生长素的分布参与调控根系的向性反应[17]和铝胁迫[18-19]。定位于质膜上, 主要在维管组织中表达, 参与调控水稻的抗旱性, 敲除会影响水稻根系的正常发育[20]。主要在叶鞘、茎、叶、根(主要是成熟区和伸长区)中表达, 通过介导生长素的稳态、运输及分布从而改变植株的构型和产量; 过表达会使水稻株高、穗长、结实率等重要农艺性状参数等降低, 而敲除则反之[21]。定位于质膜, 主要在根茎结合部的维管组织和分蘖芽中表达, 参与调控水稻的分蘖数以及产量等[22]。综上,基因在调控水稻生长发育中发挥着重要作用, 因此深入研究基因功能对发掘优质基因进行水稻遗传改良具有重要意义。
如上所述, 虽然部分基因功能已有报道, 但的基因功能仍不明确。随着CRISPR/ Cas9基因编辑技术的逐步完善, 近年来利用CRISPR/Cas9技术研究基因功能成为一条高效、快速的途径, CRISPR/Cas9是公认的最简易高效的基因编辑技术[23]。通过CRISPR/Cas9技术突变基因, 获得突变体, 为研究基因功能奠定基础。本研究以水稻日本晴(Nipponbare)为材料, 利用CRISPR/Cas9基因编辑技术获得纯合突变体。7 d突变体幼苗的苗高、根长和不定根数与野生型无显著差异, 但14 d幼苗的苗高、根长和不定根数均显著低于野生型植株, 表明在调控水稻株型中发挥着重要作用。突变体根中和的表达量显著上调; 生长素合成关键基因和基因的表达也受到显著诱导。此外,突变体根的向重性受到部分影响。突变体的获得为进一步发掘的基因功能及利用创制新的种质资源奠定基础。
1 材料与方法
1.1 试验材料
于2018—2021年在河南科技大学农学院进行。大肠杆菌DH10B感受态细胞及农杆菌() EHA105感受态细胞由河南科技大学农学院资源植物评价与创新利用实验室自制。基因编辑载体CRISPR-RICE由中国科学院上海植物逆境生物学研究中心朱健康研究员提供。限制性内切酶I购自NEB (北京)有限公司; T4连接酶及高保真酶PrimeSTAR等购自宝日医生物技术(北京)有限公司; 质粒提取试剂盒、琼脂糖凝胶回收试剂盒等由OMEGA Bio-Tek公司提供; 引物及测序由生工生物工程(上海)股份有限公司完成。
1.2 基因编辑载体的构建
通过华南农业大学亚热带农业生物资源保护与利用国家重点实验室刘耀光院士团队开发的基因编辑工具包CRISPR-GE (http://skl.scau.edu.cn/)进行靶位点的设计[24]。选择靠近5'编码区的序列作为靶位点, 通过水稻全基因组BLAST比对分析验证靶位点的特异性。在上下游引物的5'端分别添加接头序列TGTGT和AAAC, 用于将目的片段连入CRISPR- RICE载体。基因编辑载体的构建参考吴世洋等[25], 通过冻融法将重组CRISPR-OsPIN5c基因编辑载体转入农杆菌EHA105感受态细胞。通过农杆菌介导法转化水稻粳稻品种日本晴, 获得抗性植株。
1.3 基因编辑株系的筛选
采用CTAB法提取T0代水稻叶片基因组DNA[26], 通过PCR扩增片段获得转基因阳性植株。参考基因组序列, 在靶位点两侧设计引物PIN5c-Assay-F和PIN5c-Assay-R, 通过PCR扩增基因组片段并送至生工生物工程(上海)股份有限公司测序, 测序结果通过CRISPR-GE分析测序结果的突变类型[27]。为筛选获得无转基因成分的纯合突变体, 首先以PCR扩增片段的方法进行筛选, 随后以植株叶片为材料进一步以潮霉素溶液浸泡法进行验证[28-29]。无转基因成分纯合突变体载体构建及突变体筛选引物见表1。
1.4 生物信息学分析
以MEGA7进行蛋白序列比对[30], 比对结果进一步通过Jalview进行修饰[31]。通过在线网站(TMHMM Server v. 2.0: http://www.cbs.dtu.dk/services/ TMHMM/)分析蛋白的跨膜螺旋结构域[32]。通过SWISS MODEL网站(https://swissmodel.expasy.org/)预测蛋白三级结构[33]。
1.5 ospin5c突变体表型分析
稻种浸种2 d后移入铺有浸湿滤纸的培养皿中, 30℃暗培养催芽。露白后将稻种转移到96孔板中, 以木村B营养液继续培养, 分别在第7天和第14天统计苗高、根长和不定根数。试验重复3次, 以GraphPad Prism 8.0通过单因素方差分析及Tukey’s法多重比较进行数据分析及作图。
1.6 基因表达分析
以野生型日本晴幼苗为材料, 通过qRT-PCR分析的组织表达特异性, 将根中的表达量定为1。以根为材料分析突变对其他基因及基因表达的影响, 以(Os03g0718100)作为内参, 以2–ΔΔCT法计算基因的相对表达量, qRT-PCR实验方法参考吴世洋等[25]。试验进行至少3次生物学重复, 每次3个技术重复。所用引物见表1。
1.7 ospin5c突变体根向重性分析
根的向重性分析参考Lino等[34]的方法。将0.7%灭菌琼脂倒入4.5 mL四面透光比色皿(上部内径11 mm × 11 mm, 高度43 mm, Thermo Fisher)冷却后备用。稻种灭菌后胚朝上将其1/2插入琼脂中, 置于黑色培养盒中, 培养盒底部及内壁铺上浸湿纸巾, 顶部以保鲜膜封住以保持湿度进行暗培养。根长约1 cm时将其水平放置开始向重性处理, 拍照并统计0、3、6、9、12和24 h的根部弯曲角度。

表1 引物及序列
2 结果与分析
2.1 sgRNA的设计以及基因编辑表达载体的构建
参考基因组(LOC_Os09g32770)序列, 通过在线分析工具包CRISPR-GE在第1外显子处设计20 bp的靶位点序列, 靶位点位于CDS的第289~308 bp处(图1-A)。引物退火形成Oligo二聚体, 与CRISPR-RICE载体进行连接, 通过引物M13-F和PIN5c-CRISPR-R进行阳性克隆的筛选, 结果表明可以扩增得到一段约290 bp的DNA条带(图1-B), 分子量大小与预期吻合。测序确认获得重组CRISPR/Cas9载体, 转化至农杆菌感受态细胞EHA105。
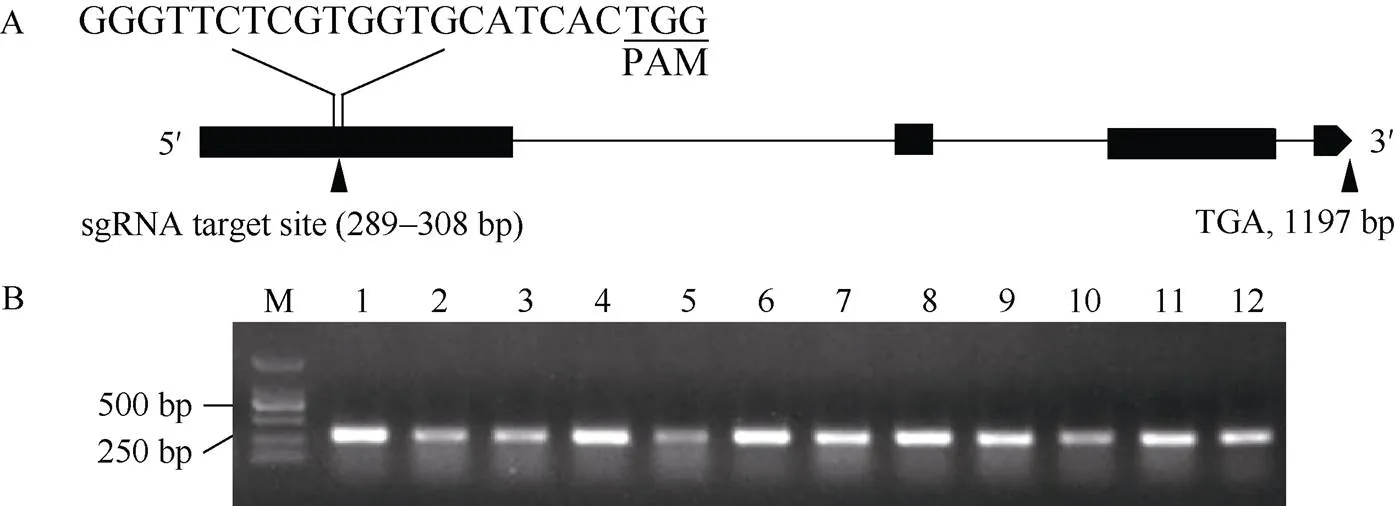
图1 OsPIN5c gRNA靶位点示意图及阳性克隆筛选
A:靶位点示意图; PAM: 前间隔序列邻近基序; 黑色下画线表示PAM序列。B: PCR筛选阳性克隆, M: DL2000 DNA marker; 1~12: PCR产物。
A:target site in schematic diagram; PAM: protospacer adjacent motif; black line indicates PAM sequences. B: positive clones by PCR. M: DL2000 DNA marker; 1–12: PCR products.
2.2 转基因株系的突变类型分析
通过农杆菌介导法获得T0代转化植株, PCR检测表明外源片段成功整合到水稻基因组(结果未显示), 最终共筛选到24株阳性植株。扩增含有靶位点的基因组序列并测序分析突变类型(图2-A)。结果表明, 在24株转基因植株中, 共有15株发生了突变, 突变率为62.5%, 突变类型为双等位突变。共检测到2种主要类型的突变, 一种为删除10个碱基和1个碱基, 一种为删除2个碱基和插入1个碱基, 且发生编辑的位置不同(图2-B)。
将T0代筛选获得的突变植株继续种植收获种子,通过PCR扩增结合测序分析从T1代植株中筛选纯合突变体。最终获得删除1、2和10个碱基的3种不同类型纯合突变体, 将其分别命名为、和(图3)。
Cas9蛋白的存在可能会造成潜在的脱靶效应, 为排除Cas9的潜在干扰, 进一步通过PCR扩增片段结合叶片潮霉素浸泡法筛选获得无转基因成分的突变体。结果如图4所示, 从25株纯合株系中共筛选到6株无转基因成分的突变体(图4-A), 潮霉素叶片浸泡实验进一步确认了PCR结果的准确性 (图4-B)。
2.3 OsPIN5c突变蛋白的生物信息学分析
为确认蛋白水平是否发生了有效突变, 分析了OsPIN5c突变蛋白在氨基酸序列、蛋白跨膜结构域以及三级结构方面的变化。蛋白序列比对结果表明, 3种突变都导致移码突变并使转录提前终止, 编码氨基酸数由野生型的398个缩短为109、106和250个(图5-A)。以MEGA7比对野生型和突变体OsPIN5c蛋白氨基酸序列, 结果表明OsPIN5c-2蛋白的前99个氨基酸残基与野生型一致, 而OsPIN5c-1和OsPIN5c-3蛋白的前101个氨基酸残基与野生型一致, 剩余氨基酸的相似性都较低(图5-B)。
PIN蛋白功能与跨膜结构密切相关[35]。跨膜螺旋结构域分析表明, 野生型OsPIN5c蛋白含有7个跨膜螺旋结构域, 其中前5个位于中央亲水环的上游, 后2个位于中央亲水环的下游(图6)。虽然蛋白序列比对分析表明OsPIN5c突变蛋白N段有约100个氨基酸残基和野生型完全一致(图5), 但跨膜螺旋结构域分析表明3种突变体中OsPIN5c蛋白的跨膜螺旋结构域全部消失(图6-A)。蛋白质三级结构预测表明, OsPIN5c突变蛋白的三级结构更加简单, 与野生型的完全不同, 仅剩下一个较明显的螺旋结构(图6-B)。综上, 一系列分析表明OsPIN5c在核酸及蛋白水平都发生了突变, 表明基因功能可能受到影响。由于3个突变体中都发生了有效突变, 后续主要以和进行表型分析等实验。
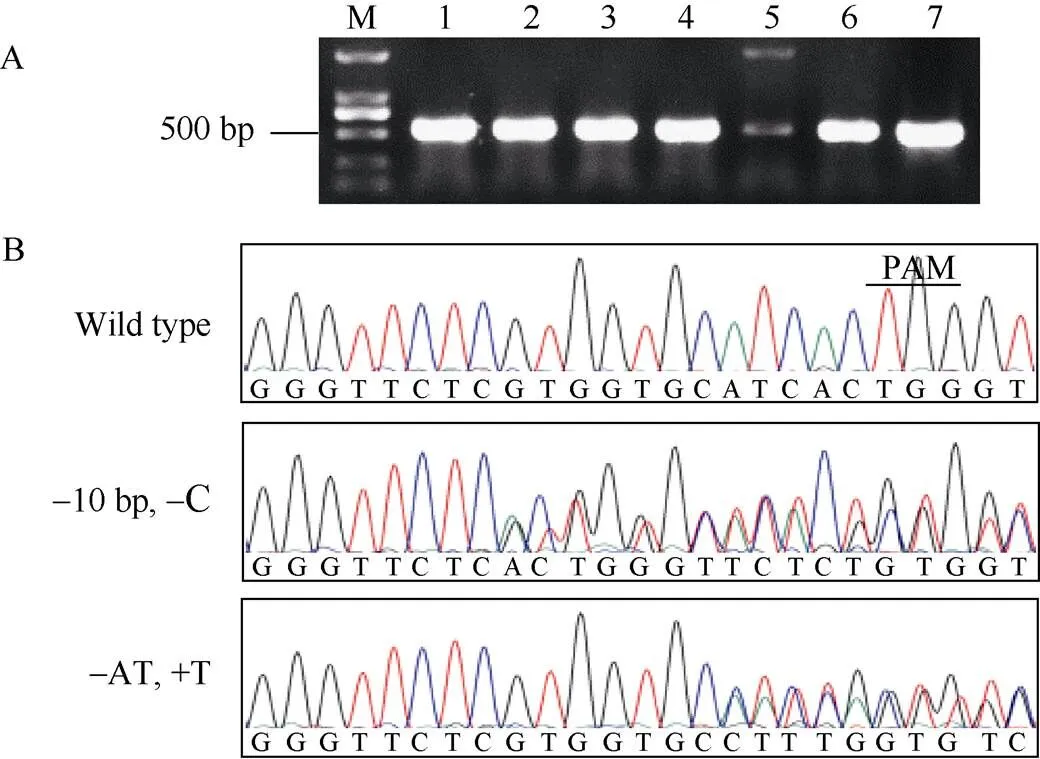
图2 T0代突变植株的筛选及分析
A: PCR扩增基因组片段; M: DL2000 DNA marker; 1~7: PCR产物。B: 突变类型分析, PAM: 前间隔序列邻近基序; 黑色下画线表示PAM序列。
A: PCR amplification ofgenomic fragments; M: DL2000 DNA marker; 1–7: PCR products. B: mutation type analysis. PAM: protospacer adjacent motif; black line indicates PAM sequence.
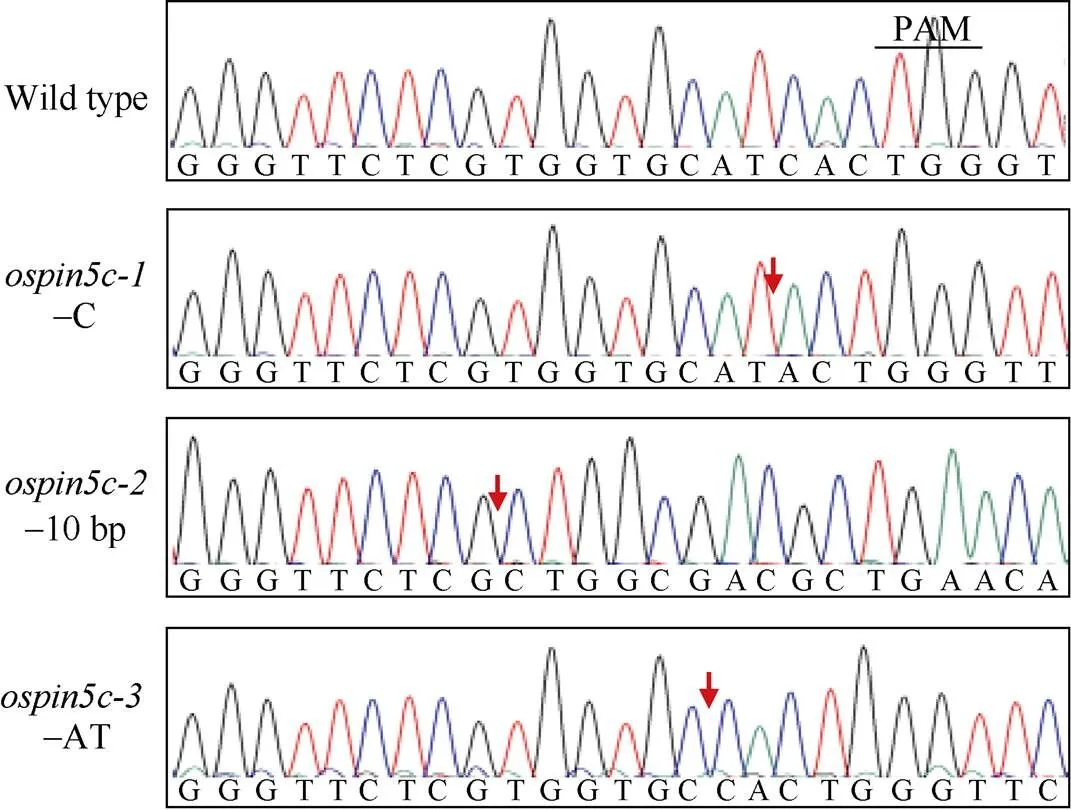
图3 T1代纯合ospin5c突变体的筛选
PAM:前间隔序列邻近基序; 黑色下画线表示PAM序列; 红色箭头表示突变位点。
PAM: protospacer adjacent motif; black line indicates PAM sequence; red arrows show the mutation sites.
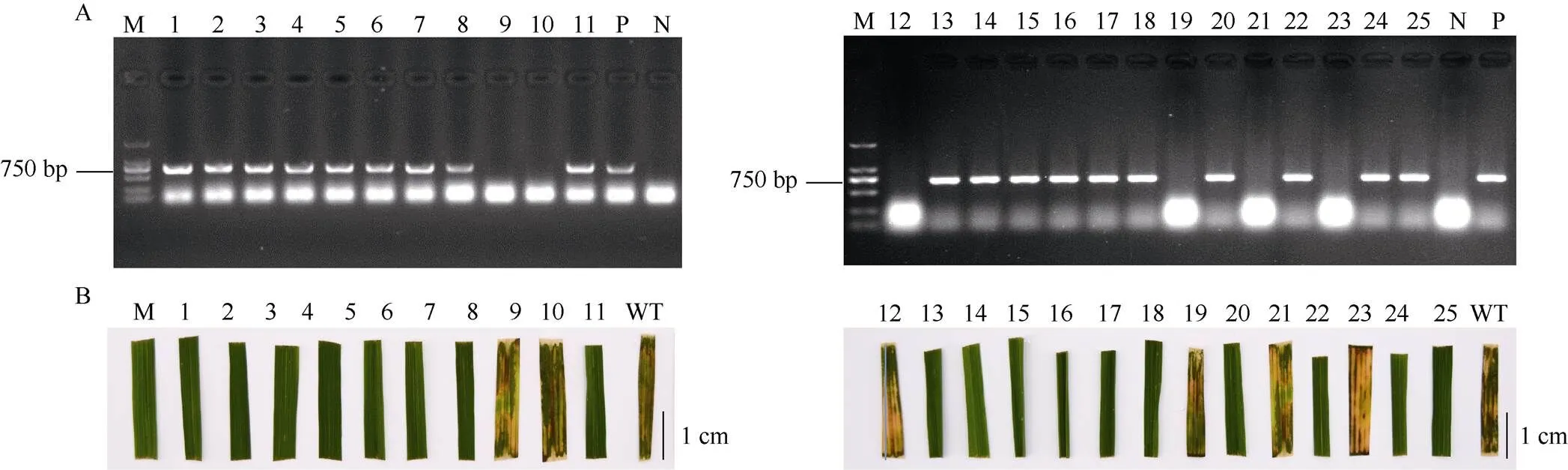
图4 无转基因成分植株筛选
A: PCR筛选结果; M: DL2000 DNA marker; 1~25:PCR扩增结果; P: 阳性对照(质粒); N: 阴性对照(WT)。B: 叶片潮霉素抗性筛选结果, 1~25:离体叶片潮霉素筛选。
A: PCR screening results; M: DL2000 DNA marker; 1–25:PCR amplification results; P: positive control; N: negative control. B: hygromycin sresistance assay of detached leaves. 1–25: hygromycin sresistance assay ofdetached leaves.

(图5)
A: 靶位点氨基酸变化; PAM: 前间隔序列邻近基序; 下画线对应的氨基酸表示突变氨基酸; 星号表示翻译的终止。B: 突变蛋白比对分析。
A: amino acid change of the target sites; PAM: protospacer adjacent motif; underlined amino acids indicate mutant amino acids; asterisk indicates the end of translation. B: blast analysis of the mutant proteins.
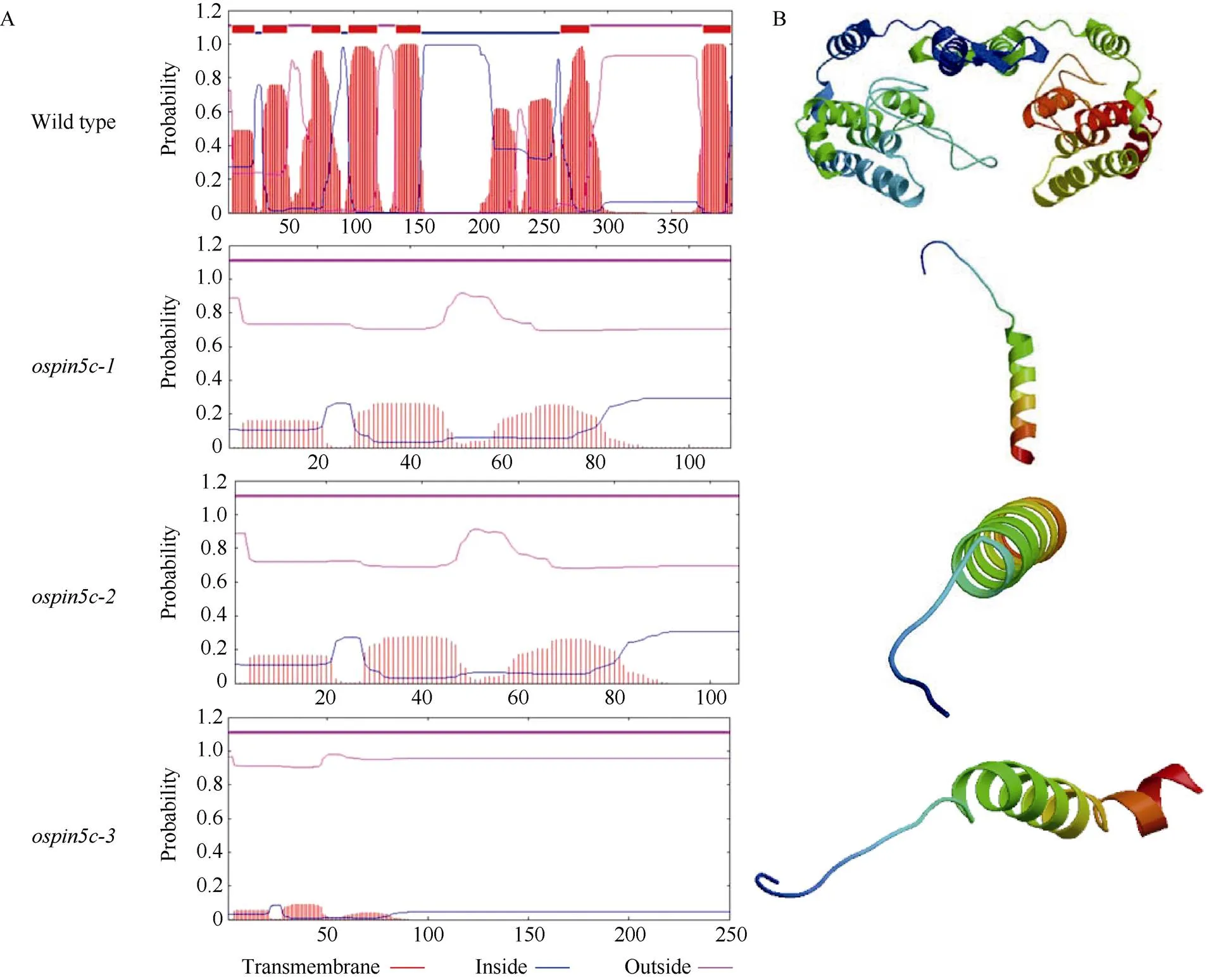
图6 OsPIN5c突变蛋白的跨膜螺旋结构域及三级结构分析
A: 跨膜螺旋结构域分析; B: 三级结构分析。
A: transmembrane helices (TMH) analysis; B: tertiary structure analysis.
2.4 ospin5c突变体表型分析
7 d幼苗的表型分析表明,突变体与野生型植株在苗高、根长和不定根数上没有显著差异(图7-A), 但随着培养时间的延长, 突变体的生长受到明显影响。培养至第14天时,突变体的苗高比野生型降低约14%, 根长降低约18%, 不定根数降低了11%~15% (图7-B), 表明参与调控水稻幼苗的生长发育。
2.5 ospin5c突变体基因表达分析
组织特异性表达分析表明,主要在水稻根部表达, 其次是茎和叶鞘, 而在叶片和根茎结合部的表达量很低(图8-A), 故提取14 d野生型和突变体根总RNA, 通过qRT-PCR检测基因和生长素合成关键基因基因家族的表达情况。结果表明,突变显著诱导了和及生长素合成关键基因、、和的表达(图8-B)。
2.6 ospin5c突变体根向重性分析
幼苗期表型(图7)和基因表达分析(图8)表明,突变体中生长素稳态很可能受到影响, 而生长素的稳态又与根的向重性密切相关[36]。进一步检测突变对根向重性的影响, 结果表明, 水平放置3 h时, 虽然突变体根的弯曲角度小于野生型植株, 但未达到显著性差异; 处理6 h和9 h时仅有突变体根向重性受到显著抑制, 处理12 h和24 h时2种突变体根的向重性弯曲角度与野生型的差异逐渐增大, 显著小于野生型植株(图9), 说明的突变部分影响了根的向重性。
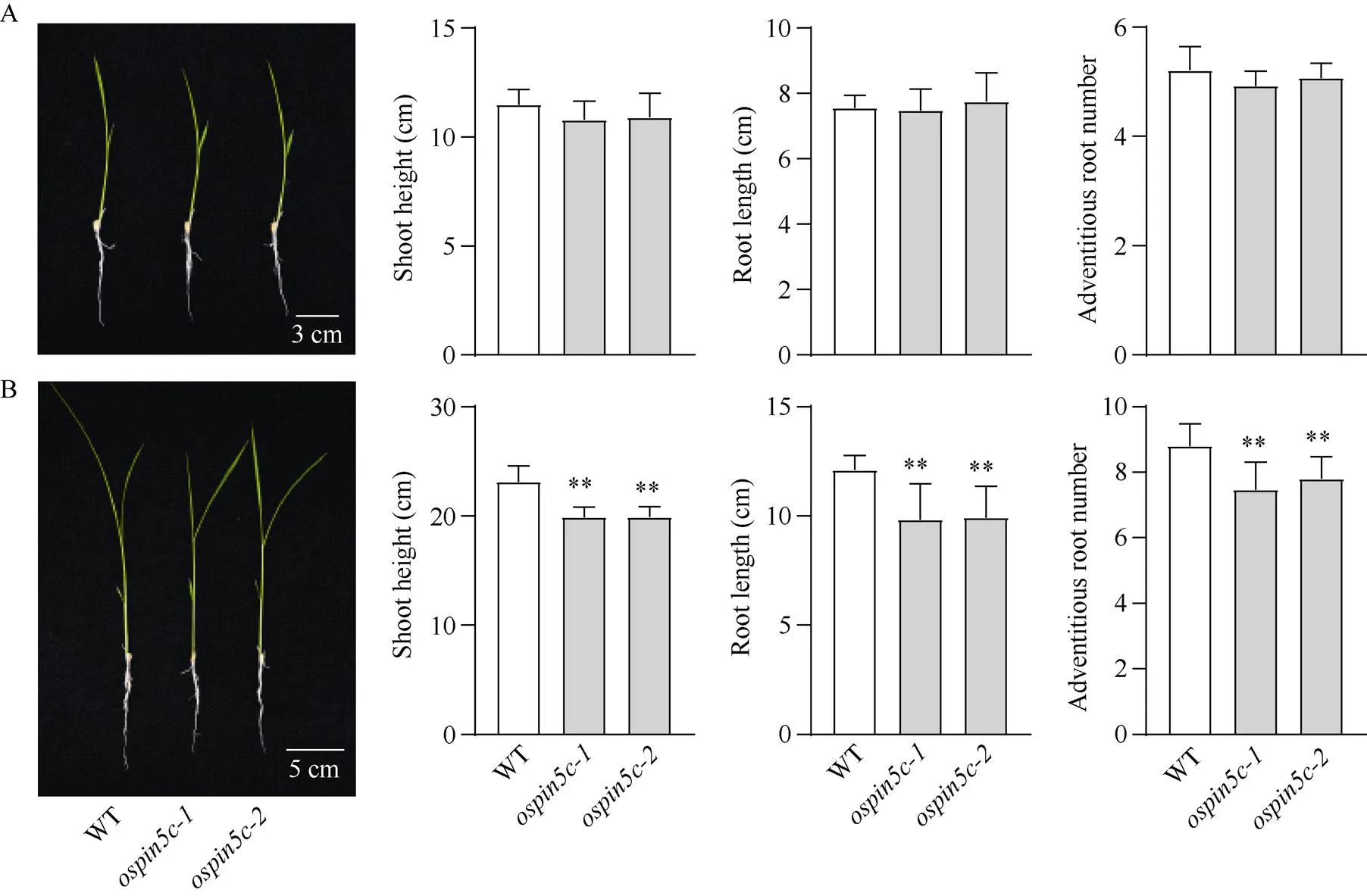
图7 幼苗期ospin5c突变体表型
A: 7 d突变体表型分析。B: 14 d突变体表型分析(≥ 15)。**表示< 0.01水平差异。
A: phenotypic analysis of 7-day-oldmutants. B: phenotypic analysis of 14-day-oldmutants (≥ 15). **:< 0.01.
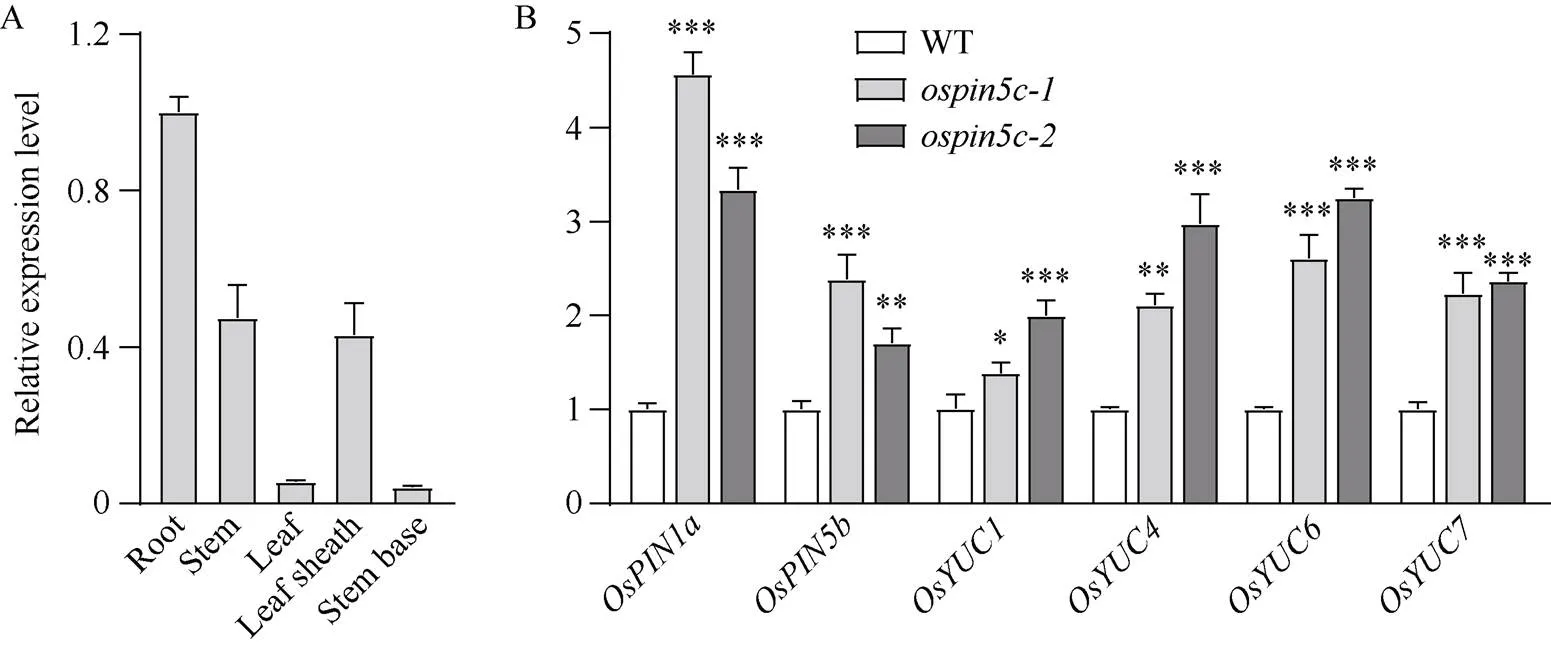
图8 基因表达分析
A:组织特异性表达分析。B:和表达分析; *表示< 0.05水平差异, **表示< 0.01水平差异, ***表示< 0.001水平差异。
A: tissue-specific analysis of. B: the relative expression levels ofandgenes. *:< 0.05; **:< 0.01; ***:< 0.001.

图9 野生型(WT)和ospin5c突变体根向重性分析
A: 重力刺激后野生型(WT)和突变体表型, 0~24 h表示重力刺激处理时间。B: 重力刺激后野生型(WT)和突变体根弯曲角度统计分析(≥ 12); *表示< 0.05水平差异, **表示< 0.01水平差异。
A: the phenotypes of WT andmutants after gravity stimulus; 0–24 h indicate time after gravity stimulus. B: statistical analysis of root reorientation angle in WT andmutants after gravity stimulus (≥ 12). *:< 0.05; **:< 0.01.
3 讨论
近年来, CRISPR/Cas9技术在植物基因功能研究以及遗传改良等方面得到广泛应用。如Zhang等[37]通过CRISPR/Cas9技术编辑小麦基因提高了小麦(L.)的氮素利用效率及田间产量, Zhang等[38]通过CRISPR/Cas9技术降低稻米直链淀粉含量, Wang等[39]通过CRISPR/Cas9技术创制香味玉米(L.), Zhang等[40]在异源六倍体高羊茅()中实现基因编辑等。此外, CRISPR/Cas9技术也在大豆(L.)、花生(L.)以及油菜(L.)等作物中得到广泛应用[41-43]。同时, 基因编辑技术也发展迅速, 如华南农业大学亚热带农业生物资源保护与利用国家重点实验室刘耀光院士团队开发的多基因编辑系统为研究者提供便利的基因编辑方法[44], 中国科学院上海植物逆境生物学研究中心朱健康院士团队开发的高效片段靶向敲入和替换技术,其靶向敲入效率可达50%, 极大地方便植物研究及育种工作[45]; 上海师范大学张辉课题组开发出高效的碱基编辑系统, 在水稻中的编辑效率几乎达到100%[46]; 随后在双子叶植物中也开发出一套高效的植物腺嘌呤单碱基编辑器, 其效率达到71.4%[47]。这些基因编辑系统的开发为进一步研究基因功能进行遗传改良奠定了基础。CRISPR/Cas9系统可造成不同形式的突变类型, 如碱基的插入、缺失或替换[48-50]。本实验中基因的突变类型主要是少数碱基的缺失, 在筛选获得的3种纯合突变中, 缺失碱基均导致移码突变及转录提前终止。氨基酸序列比对、蛋白跨膜结构域以及三级结构分析表明OsPIN5c蛋白均发生不同程度的变化。选择不同的位点进行编辑也可能会获得基因表达水平不同的株系, 为研究基因功能奠定基础。通常, 利用CRISPR/Cas9对基因进行编辑时, 其T0株系代大多数为双等位突变、纯合突变或者杂合突变[51]。本研究最终获得3种不同突变类型的纯合突变体。
对于基因功能的研究以前多采用反义RNA或RNA干涉的方法使基因沉默, 但由于反义RNA及RNA干涉一般使用较长的目的片段, 故有可能造成非特异沉默。因此对于核苷酸序列相似性高的基因家族来说, 对其功能的研究可能有限制[15]。CRISPR/Cas9基因编辑技术所使用的靶序列更短, 可以精确地对基因进行定点编辑, 这为多基因家族的功能研究提供了有效的技术手段。与拟南芥的氨基酸序列相似性较高[13-14], 暗示其功能可能与拟南芥类似。在拟南芥中蛋白定位于内质网, 很可能主要介导生长素从细胞质到内质网腔生长素的转运, 从而调控不同亚细胞结构间生长素的平衡;突变抑制侧根形成, 且突变体主根与下胚轴发育迟缓; 过表达则导致拟南芥胚胎发育异常, 叶片呈窄长型, 且拟南芥中游离态生长素含量略有降低[52]。OsPIN5b蛋白也定位于内质网, 在水稻生长发育中起着重要作用[21], 但目前基因功能未有报道。
本研究通过CRISPR/Cas9基因编辑技术定点突变, 获得3种不同类型的纯合突变体 (图3); 蛋白质序列比对结果显示野生型OsPIN5c蛋白长度大于OsPIN5c突变蛋白(图5); 突变蛋白的跨膜螺旋结构域消失, 三级结构也相对更简单(图6), 说明OsPIN5c蛋白功能可能受到影响。表型分析表明, 7 d突变体幼苗表型与野生型没有显著差异, 而培养至14 d时,突变体的苗高、根长和不定根数都要显著低于野生型植株(图7), 说明参与调控水稻幼苗株型。组织特异性表达分析表明,主要在水稻根、茎和叶鞘中表达(图8-A),功能缺失导致突变体苗高、根长、不定根数受到影响(图7)。基因表达分析表明,突变体中基因(和)和基因(、、和)表达提高, 说明突变体中生长素的运输及稳态很可能受到影响。在根、茎、叶等组织中均有表达, 而在根中主要在根尖部位表达[13], 很可能与植物根的发育有关。研究表明和确实共同参与调控水稻根系的发育[15]。是否与共同参与调控水稻根系发育仍有待研究。利用RNA干涉技术敲除基因导致根和分蘖等增多, 而过表达则同样导致根系发育受阻[21]。与过表达植株中生长素合成关键基因、和的表达量提高类似[21],突变体中这3个基因也受到诱导表达, 故突变体根系发育异常很可能也与表达量提高有关, 但其潜在联系还需进一步实验验证。
向重性反应一般由重力感应、信号传导以及不对称生长反应3个连续的步骤组成[53], 根的向重性与生长素浓度及分布密切相关。已有的研究表明,基因在植物根的向重性反应中起着重要作用, 如拟南芥中和参与调控根的向重性。主要在向重性反应发生弯曲的根尖伸长区表达[54], 参与根的向重性反应[10-11]。AtPIN3定位于根尖小柱细胞的下侧, 可能介导根尖生长素的分布从而引起不对称生长[2,55],的缺失也降低了根的向重性反应, 同时也抑制了下胚轴以及根的生长[12,56-57]。水稻也在根的向重性反应中起着重要作用[58],的表达受到重力刺激的强烈诱导, 很可能也参与调控根的向重性[59], 但是否参与调控根的向重性尚不明确。本研究中,突变引起根中基因和基因表达的变化(图8), 很可能扰乱了生长素的稳态,进而可能影响根的向重性。向重性试验表明,突变体根的向重性确实受到部分影响, 向重性处理12 h和24 h后, 突变体根的弯曲角度明显小于野生型植株(图9), 表明基因直接或间接参与调控根的向重性, 但其潜在的调控机制仍有待研究。
本研究利用CRISPR/Cas9基因编辑技术, 首次成功获得3种不同类型的纯合突变体, 并对基因功能进行了初步探索。的突变影响了水稻幼苗地上部和地下部的发育, 同时其他几个基因以及生长素合成关键基因的表达量提高, 这些可能共同组成一个调控网络影响植株的生长发育。同时,突变体根的向重性受到部分抑制。纯合突变体的获得, 不仅为深入研究基因功能提供了材料, 也为进一步通过杂交获得的双突及多突等突变体奠定基础。
4 结论
利用CRISPR/Cas9基因编辑技术, 首次对水稻生长素输出载体基因进行定点突变, 成功获得3种类型的无转基因成分的纯合突变体。突变影响了水稻幼苗的正常生长发育、和基因表达以及根的向重性反应。
[1] Benkova E, Michniewicz M, Sauer M, Teichmann T, Seifertova D, Jurgens G, Friml J. Local, efflux-dependent auxin gradients as a common module for plant organ formation., 2003, 115: 591–602.
[2] Friml J. Auxin transport—shaping the plant., 2003, 6: 7–12.
[3] Petrasek J, Mravec J, Bouchard R, Blakeslee J J, Abas M, Seifertova D, Wisniewska J, Tadile Z, Kubes M, Covanova M, Dhonukshe P, Skupa P, Benkova E, Perry L, Krecek P, Lee O R, Fink G R, Geisler M, Murphy A S, Luschnig C, Zazimalova E, Friml J. PIN proteins perform a rate-limiting function in cellular auxin efflux., 2006, 312: 914–918.
[4] Karki S, Rizal G, Quick W P. Improvement of photosynthesis in rice (L.) by inserting the C4pathway.(New York), 2013, 6: 1–8.
[5] Woodward A W. Auxin: regulation, action, and interaction., 2005, 95: 707–735.
[6] Zhao Y. Auxin biosynthesis and its role in plant development., 2010, 61: 49–64.
[7] Michiewicz M, Brewer P B, Friml J. Polar auxin transport and asymmetric auxin distribution., 2007, 5: e0108.
[8] Krecek P, Skupa P, Libus J, Naramoto S, Tejos R, Friml J, Zazimalova E. The PIN-FORMED (PIN) protein family of auxin transporters., 2009, 10: 249.
[9] Okada K, Ueda J, Komaki M K, Bell C J, Shimura Y. Requirement of the auxin polar transport system in early stages offloral bud formation., 1991, 3: 677–684.
[10] Luschnig C, Gaxiola R A, Grisafi P, Fink G R. EIR1, a root-specific protein involved in auxin transport, is required for gravitropism in., 1998, 12: 2175–2187.
[11] Muller A, Guan C, Galweiler L, Tanzler P, Huijser P, Marchant A, Parry G, Bennett M, Wisman E, Palme K.defines a locus offor root gravitropism control., 1998, 17: 6903–6911.
[12] Friml J, Wisniewska J, Benkova E, Mendgen K, Palme K. Lateral relocation of auxin efflux regulator PIN3 mediates tropism in., 2002, 415: 806–809.
[13] Wang J, Hu H, Wang G, Li J, Chen J, Wu P. Expression ofgenes in rice (L.): tissue specificity and regulation by hormones., 2009, 2: 823–831.
[14] Miyashita Y, Takasugi T, Ito Y. Identification and expression analysis ofgenes in rice., 2010, 178: 424–428.
[15] Li Y, Zhu J, Wu L, Shao Y, Wu Y, Mao C. Functional divergence ofparalogous genes in rice., 2019, 60: 2720–2732.
[16] Chen Y, Fan X, Song W, Zhang Y, Xu G. Over-expression ofleads to increased tiller numbers, angle and shorter plant height through suppression of., 2012, 10: 139–149.
[17] Inahashi H, Shelley I J, Yamauchi T, Nishiuchi S, Takahashi-nosaka M, Matsunami M, Ogawa A, Noda Y, Inukai Y., which encodes a member of the auxin efflux carrier proteins, is involved in root elongation growth and lateral root formation patterns via the regulation of auxin distribution in rice., 2018, 164: 216–225.
[18] Wu D, Shen H, Yokawa K, Baluska F. Overexpressingenhances aluminium internalization by elevating vesicular trafficking in rice root apex., 2015, 66: 6791–6801.
[19] Wu D, Shen H, Yokawa K, Baluska F. Alleviation of aluminium-induced cell rigidity by overexpression ofin rice roots., 2014, 65: 5305–5315.
[20] Zhang Q, Li J, Zhang W, Yan S, Wang R, Zhao J, Li Y, Qi Z, Sun Z, Zhu Z. The putative auxin efflux carrieris involved in the drought stress response and drought tolerance., 2012, 72: 805–816.
[21] Lu G, Coneva V, Casaretto J A, Ying S, Mahmood K, Liu F, Nambara E, Bi Y M, Rothstein S J.modulates rice () plant architecture and yield by changing auxin homeostasis, transport and distribution., 2015, 83: 913–925.
[22] Hou M, Luo F, Wu D, Zhang X, Lou M, Shen D, Yan M, Mao C, Fan X, Xu G, Zhang Y. OsPIN9, an auxin efflux carrier, is required for the regulation of rice tiller bud outgrowth by ammonium., 2021, 229: 935–949.
[23] 单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展. 遗传, 2015, 37: 953–973.
Dan Q W, Gao C X. Research progress of genome editing and derivative technologies in plants.(Beijing), 2015, 37: 953–973 (in Chinese with English abstract).
[24] Liu W, Xie X, Ma X, Li J, Chen J, Liu Y G. DSDecode: a web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations., 2015, 8: 1431–1433.
[25] 吴世洋, 杨晓祎, 张艳雯, 侯典云, 胥华伟. 利用CRISPR/Cas9基因编辑技术构建水稻突变体. 中国农业科学, 2021, 54: 3805–3817.
Wu S Y, Yang X Y, Zhang Y W, Hou D Y, Xu H W. Generation ofmutants in rice by CRISPR/Cas9 genome editing technology., 2021, 54: 3805–3817 (in Chinese with English abstract).
[26] Xin Z, Chen J. A high throughput DNA extraction method with high yield and quality., 2012, 8: 26.
[27] Ma X, Chen L, Zhu Q, Chen Y, Liu Y. Rapid decoding of sequence-specific nuclease-induced heterozygous and biallelic mutations by direct sequencing of PCR products., 2015, 8: 1285–1287.
[28] 刘巧泉, 陈秀花, 王兴稳, 彭凌涛, 顾铭洪. 一种快速检测转基因水稻中潮霉素抗性的简易方法. 农业生物技术学报, 2001, 9: 264.
Liu Q Q, Chen X H, Wang X W, Peng L T, Gu M H. A rapid simple method of assaying hygromycin resistance in transgenic rice plants., 2001, 9: 264 (in Chinese with English abstract).
[29] 张洪, 马骏, 胡国成, 斯华敏, 付亚萍, 戴良英, 孙宗修. 用叶片检测转基因水稻对潮霉素反应的可靠性研究. 浙江农业学报, 2005, 17: 341–345.
Zhang H, Ma J, Hu G C, Si H M, Fu Y P, Dai L Y, Sun Z X. Reliability study on the response ofgene in transgenic rice to hygromycin by leaf assay., 2005, 17: 341–345 (in Chinese with English abstract).
[30] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33: 1870–1874.
[31] Waterhouse A M, Procter J B, Martin D M A, Clamp M, Barton G J. Jalview Version 2: a multiple sequence alignment editor and analysis workbench., 2009, 25: 1189–1191.
[32] Krogh A, Larsson B, von Heijne G, Sonnhammer E L L. Predicting transmembrane protein topology with a hidden markov model: application to complete genomes., 2001, 305: 567–580.
[33] Madeira F, Park Y M, Lee J, Buso N, Gur T, Madhusoodanan N, Basutkar P, Tivey A R N, Potter S C, Finn R D, Lopez R. The EMBL-EBI search and sequence analysis tools APIs in 2019., 2019, 47: W636–W641.
[34] Iino M, Tarui Y, Uematsu C. Gravitropism of maize and rice coleoptiles: dependence on the stimulation angle., 1996, 19: 1160–1168.
[35] Bennett T, Brochington S F, Rothfels C, Graham S W, Stevenson D, Kutchan T, Rolf M, Thomas P, Wong G K, Leyser O, Glover B J, Harrison C J. Paralogous radiations of PIN proteins with multiple origins of noncanonical PIN structure., 2014, 31: 2042–2060.
[36] Rahman A, Takahashi M, Shibasakei K, Wu S, Inaba T, Tsurumi S, Baskin T I. Gravitropism ofroots requires the polarization of PIN2 toward the root tip in meristematic cortical cells., 2010, 22: 1762–1776.
[37] Zhang J, Zhang H, Li S, Li J, Yan L, Xia L. Increasing yield potential through manipulating of anortholog related to nitrogen use efficiency in wheat by CRISPR/Cas9., 2021, 63: 1649–1663.
[38] Zhang J, Zhang H, Botella J R, Zhu J K. Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of thegene in elite rice varieties., 2018, 60: 369–375.
[39] Wang Y, Liu X, Zheng X, Wang W, Yin X, Liu H, Ma C, Niu X, Zhu J K, Wang F. Creation of aromatic maize by CRISPR/Cas., 2021, 63: 1664–1670.
[40] Zhang L, Wang T, Wang G, Bi A, Wassie M, Xie Y, Cao L, Xu H, Fu J, Chen L, Zhao Y, Hu T. Simultaneous gene editing of three homoeoalleles in self-incompatible allohexaploid grasses., 2021, 63: 1410–1415.
[41] 侯智红, 吴艳, 程群, 董利东, 芦思佳, 南海洋, 甘卓然, 刘宝辉. 利用CRISPR/Cas9技术创制大豆高油酸突变系. 作物学报, 2019, 45: 839–847.
Hou Z H, Wu Y, Cheng Q, Dong L D, Lu S J, Nan H Y, Gan Z R, Liu B H. Creation of high oleic acid soybean mutation plants by CRISPR/Cas9., 2019, 45: 839–847 (in Chinese with English abstract).
[42] 张旺, 冼俊霖, 孙超, 王春明, 石丽, 于为常. CRISPR/Cas9编辑花生基因研究. 作物学报, 2021, 47: 1481–1490.
Zhang W, Xian J L, Sun C, Wang C M, Shi L, Yu W C. Preliminary study of genome editing of peanutgenes by CRISPR/Cas9., 2021, 47: 1481–1490 (in Chinese with English abstract).
[43] 石育钦, 孙梦丹, 陈帆, 成洪涛, 胡学志, 付丽, 胡琼, 梅德圣,李超. 通过CRISPR/Cas9技术突变基因提高甘蓝型油菜的抗病性. 作物学报, 2022, 48: 801–811.
Shi Y Q, Sun M D, Chen F, Cheng H T, Hu X Z, Fu L, Hu Q, Mei D S, Li C. Genome editing ofgene by CRISPR/Cas9 for the improvement of disease resistance inL., 2022, 48: 801–811 (in Chinese with English abstract).
[44] Ma X, Zhang Q, Zhu Q, Liu W, Chen Y, Qiu R, Wang B, Yang Z, Li H, Lin Y, Xie Y, Shen R, Chen S, Wang Z, Chen Y, Guo J, Chen L, Zhao X, Dong Z, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8: 1274–1284.
[45] Lu Y, Tian Y, Shen R, Yao Q, Wang M, Chen M, Dong J, Zhang T, Li F, Lei M, Zhu J. Targeted, efficient sequence insertion and replacement in rice., 2020, 38: 1402–1407.
[46] Wei C, Wang C, Jia M, Guo H X, Luo P Y, Wang M G, Zhu J K, Zhang H. Efficient generation of homozygous substitutions in rice in one generation utilizing an rABE8e base editor., 2021, 63: 1595–1599.
[47] Niu Q, Wu S, Xie H, Wu Q, Liu P, Xu Y, Lang Z. Efficient A.T to G.C base conversions in dicots using adenine base editors expressed under the tomato EF1alpha promoter., 2021, doi: 10.1111/pbi.13736.
[48] Shan Q, Wang Y, Li J, Zhang Y, Chen K, Liang Z, Zhang K, Liu J, Xi J J, Qiu J, Gao C. Targeted genome modification of crop plants using a CRISPR-Cas system., 2013, 31: 686–688.
[49] Li J, Norville J E, Aach J, McCormack M, Zhang D, Bush J, Church G M, Sheen J. Multiplex and homologous recombination-mediated genome editing via guide RNA/Cas9., 2013, 31: 688–691.
[50] Xie K, Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system., 2013, 6: 1975–1983.
[51] 马兴亮, 刘耀光. 植物CRISPR/Cas9基因组编辑系统与突变分析. 遗传, 2016, 38: 118–125.
Ma X L, Liu Y G. CRISPR/Cas9-based genome editing systems and the analysis of targeted genome mutations in plants.(Beijing), 2016, 38: 118–125 (in Chinese with English abstract).
[52] Mravec J, Skupa P, Baill Y A, Hoyerova K, Krecek P, Bielach A, Petrasek J, Zhang J, Gaykova V, Stierhof Y D, Dobrev P I, Schwarzerova K, Rolcik J, Seifertova D, Luschnig C, Benkova E, Zazimalova E, Geisler M, Friml J. Subcellular homeostasis of phytohormone auxin is mediated by the ER-localized PIN5 transporter., 2009, 459: 1136–1140.
[53] Fukaki H, Fujisawa H, Tasaka M. How do plant shoots bend up? The initial step to elucidate the molecular mechanisms of shoot gravitropism using., 1996, 109: 129–137.
[54] Chen R, Hilson P, Sedbrook J, Rosen E, Caspar T, Masson P H. Thegene encodes a component of the polar-auxin-transport efflux carrier., 1998, 95: 15112–15117.
[55] Grunewald W, Friml J. The march of the PINs: developmental plasticity by dynamic polar targeting in plant cells., 2010, 29: 2700–2714.
[56] Harrison B R, Masson P H. ARL2, ARG1 and PIN3 define a gravity signal transduction pathway in root statocytes., 2008, 53: 380–392.
[57] Keuskamp D H, Pollmann S, Voesenek L A, Peeters A J, Pierik R. Auxin transport through PIN-FORMED 3 (PIN3) controls shade avoidance and fitness during competition., 2010, 107: 22740–22744.
[58] Wang L, Guo M, Li Y, Ruan W, Mo X, Wu Z, Sturrock C J, Yu H, Lu C, Peng J, Mao C., encoding OsPIN2, is involved in root system architecture in rice., 2018, 69: 385–397.
[59] Farooq M, Jan R, Kim K. Gravistimulation effects onamino acid profile, growth pattern and expression ofgenes., 2020, 10: 17303.
Function analysis ofgene by CRISPR/Cas9
YANG Xiao-Yi1, WANG Hui-Hui1, ZHANG Yan-Wen1, HOU Dian-Yun1, ZHANG Hong-Xiao1, KANG Guo-Zhang2, and XU Hua-Wei1,*
1College of Agriculture, Henan University of Science and Technology, Luoyang 471000, Henan, China;2National Key Laboratory of Wheat and Maize Crop Science, Henan Agricultural University, Zhengzhou 450046, Henan, China
Polar auxin transport (PAT) plays a key role in plant growth and development, and auxin efflux carriers PIN-FORMED (PIN) are the crucial proteins controlling PAT. Although the functions of somegenes have been reported, the function ofgene is still unclear. In this study, the target site was designed at the first exon ofand the recombinant CRISPR/Cas9 vector ofwas constructed. Twenty-four independent transgenic rice lines were obtained by transformation using Nipponbare (L.ssp.) as the materials. PCR product sequencing indicated that 15 lines were identified as mutants and the corresponding mutation rate was 62.5%, the mutation type were biallelic heterozygous mutations. Three independenthomozygous mutants were further obtained in T1generation lines and named as,, and, respectively. Sequence alignment analysis showed that the three types of mutations resulted in frame-shift mutation and premature translation termination, which were shortened from 398 amino acid (aa) in wild-type (WT) plants to 109 aa, 106 aa and 250 aa, respectively. Transmembrane helices (TMH) indicated that the TMH of these three mutation proteins were disappeared totally. Protein structure demonstrated that the helix of three mutation proteins were obviously reduced than the native OsPIN5c protein. Phenotype structure indicated that, compared to the WT, the shoot height, root length and adventitious root number were significantly decreased inmutants at seedling stage. Tissue-specific analysis showed thatwas highly expressed in roots, andgenes (and),genes (,,, and) were up-regulated significantly inmutants. The gravitropism response ofmutants was partially inhibited. In conclusion, threehomozygous mutants were obtained via CRISPR/Cas9 technology and the function ofwas investigated in this study, providing the potential gene resources for crop genetic improvement by usinggene.
; CRISPR/cas9; gene function; rice
10.3724/SP.J.1006.2023.22002
本研究由河南省自然科学基金项目(182300410012, 202300410151, 202300410340)和小麦玉米作物学国家重点实验室开放课题项目(SKL2021KF03)资助。
This study was supported by the Natural Science Foundation of Henan Province (182300410012, 202300410151, 202300410340) and the Open Research Fund of National Key Laboratory of Wheat and Maize Crop Science (SKL2021KF03).
胥华伟, E-mail: xhwcyn@163.com
E-mail: 1961827259@qq.com
2022-01-13;
2022-06-07;
2022-07-08.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220707.1446.012.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
