春播早熟区高粱杂种优势群及杂种优势模式分析
2023-02-10陈冰嬬葛占宇李洪奎李海青石贵山高士杰周紫阳
陈冰嬬 于 淼 葛占宇 李洪奎 黄 炎 李海青 石贵山 谢 利 徐 宁 闫 峰 高士杰 周紫阳,* 王 鼐,*
春播早熟区高粱杂种优势群及杂种优势模式分析
陈冰嬬1于 淼1葛占宇2李洪奎3黄 炎3李海青1石贵山1谢 利1徐 宁1闫 峰4高士杰1周紫阳1,*王 鼐1,*
1吉林省农业科学院作物资源研究所, 吉林长春 130033;2赤峰市农牧科学研究所, 内蒙古赤峰 024031;3吉林省白城市农业科学院, 吉林白城 137000;4黑龙江省农业科学院齐齐哈尔分院, 黑龙江齐齐哈尔 161006
杂种优势群的划分对于拓宽亲本间遗传基础、提高育种效率, 培育突破性新品种具有重要的指导作用。本研究利用全基因组重测序技术对55份春播早熟区40余年生产中主推杂交种亲本系进行全基因组扫描, 分析其群体结构, 估算遗传距离, 划分杂种优势群, 分析主推杂交种的杂种优势模式。结果表明, 利用全基因组5×测序, 过滤到1,304,623个高质量SNP标记用于群体结构分析和杂种优势类群划分。55份高粱亲本系平均遗传距离为0.704, 变幅0.627~0.927。多态信息含量(polymorphism information content, PIC)平均为0.2935, 变幅为0.1~0.5。群体结构和主成分分析将55份亲本系划分为4个杂种优势群: 都拉群(Durra, D群)、卡佛尔/都拉群(Kafir/Durra, KD群)、俄罗斯/卡佛尔群(Russia/Kafir, RK群)、中国高粱群(Kaoliang, K群)。25个主推杂交种中76%的杂交种杂种优势模式为Kafir/Durra×Kaoliang模式, 主推高粱杂交种的不育系主要来源于引自国外的Kafir和Durra群, 恢复系多来源于我国自产的Kaoliang群。本研究的群体结构分析及其划分的杂种优势群阐明了春播早熟区高粱亲本系的遗传基础, 为亲本系改良和杂种优势模式创新研究提供科学依据。
高粱; 亲本系; 单核苷酸多态(SNP); 杂种优势; 杂种优势群; 杂种优势模式
杂种优势群和杂种优势利用模式能够为作物的杂种优势利用提供重要信息[1]。与玉米[2-3]、水稻[4]、小麦[5]、谷子[6]等作物的杂种优势群研究相比, 高粱杂种优势群的研究比较薄弱。在高粱杂种优势研究中, 我国学者以高粱种质的地理来源为依据将种质分为南非类型、印度类型、中国类型、亨加利类型[7]。自1956年TX3197AB引入国内[8], 利用TX3197A与在中国高粱中鉴选的恢复系组配杂交种, TX3197AB属于南非高粱, 形成了南非类型×中国类型杂优模式。随后采用中国类型保持材料与TX3197B、TX623B杂交选育中国类型不育系, 与中国类型恢复系选配杂交种, 形成了中国类型×中国类型杂优模式[9]。20世纪80年代从国际热带半干旱研究所引进421A等印度高粱材料后, 国内先后培育出7050A[10]、A2V4A[11]为代表性的倾南非类型、印度类型不育系, 形成了倾南非类型×中国类型、印度类型×中国类型杂优模式[12-13]。我国高粱育种家们采用不同类型间杂交, 在后代中选育趋向不同血缘类型的亲本系, 但是长期这种选育亲本的育种方法, 使亲本遗传差异与地理来源的关系变得不太明显, 组配的杂交组合杂种优势不强且综合抗性没有明显增强, 高粱育种处于艰难的爬坡阶段。
高粱杂种优势研究初期主要采用农艺性状进行分析[8,14]。随着分子标记技术的发展, 分子标记被逐渐应用于高粱种质资源分群和杂种优势研究。Wang等[15]利用41个SSR标记分析142份高粱种质资源, 群体结构分析显示形成了Kafir、Kaoliang、Feterita、Shallu、Hegari、Milo和Durra 7个亚群。王瑞等[16]利用SSR标记对20世纪70年代以来最具代表性的中晚熟区杂交种及亲本进行遗传距离分析, 从分子水平上分析了中国高粱杂交种优势类群, 将55份高粱材料聚成4类。高旭等[17]利用SSR标记根据地理来源将156份高粱材料划分为3个类群。肖松等[18]利用28对SSR标记将145个酱香白酒用糯高粱种质资源划分为2个类群。汪灿等[19]利用GBS测序技术将120份高粱材料划分为3个类群。尽管前人开展了一些高粱种质分类、杂种优势利用研究, 但是大部分并未从杂种优势群及杂种优势模式的角度进行分析。高粱杂种优势群划分和杂种优势模式研究鲜有报道。
本研究以高粱春播早熟区1973—2014年间生产中主推杂交种的亲本系为试验材料, 利用全基因组5×重测序技术, 获得覆盖全基因组的SNP标记, 分析55份高粱亲本系群体结构、计算遗传距离, 划分杂种优势群, 揭示主推杂交种杂种优势模式, 为春播早熟区高粱亲本系改良、组配强优势杂交种、建立新型杂优模式提供依据。
1 材料与方法
1.1 试验材料
选用春播早熟区55份高粱种质为试验材料, 其中保持系27份、恢复系25份、地方品种3份, 试验材料多数为1973—2014年间生产上大面积推广的杂交种的亲本系。供试材料见表1。
1.2 试验方法
1.2.1 试验材料种植 2019年将55份亲本系种植在吉林省农业科学院公主岭试验基地(43º31¢N, 124º49¢E), 采用随机区组设计, 每份材料种植3 m, 垄宽0.65 m, 株距15 cm, 3次重复。田间管理同一般试验田。抽穗期选择整齐一致的单株套袋, 去除杂株, 成熟期选择整齐一致、籽粒饱满的单穗收获, 晾干, 脱粒。
1.2.2 SNP分型及统计分析 在发芽盒中播种55份高粱亲本系种子, 每份材料播种10~20粒种子, 生长至三至四叶。对每份材料的幼嫩叶片进行混合取样。采用CTAB法提取样本基因组DNA[20], 在IlluminaHiSeqPE150测序平台上进行测序, 去除低质量的数据。采用Burrows-Wheeler Aligner (BWA)方法将高质量测序碱基数据与高粱BTA623参考基因组v3.1进行比对(the Phytozome database, http:// phytozome.jgi.doe.gov/), 采用Sentieon软件对样品SNP进行检测, 将检测出的SNP进行过滤, 过滤的标准为: (1) 选择测序深度大于4的标记; (2) 删除低频等位基因频率小于0.05、缺失率大于0.2的标记; (3) 去掉多于一种突变型的位点。最终获得了1,304,623个高质量的SNP标记用进一步分析。利用 Power Marker V3.25计算群体的低频等位频率(minor allele frequency, MAF)、杂合率以及多态信息含量(PIC)[21]。

表1 春播早熟区高粱亲本系名称和系谱

(续表1)
1~26号为保持系、27~55号为恢复系。1–26 were B-Lines, and 27–55 were R-Lines.
1.2.3 遗传距离分析、热图和邻近树的构建 采用Plink软件计算遗传距离[22]; 采用TASSEL5.0软件制作热图, 其数据采用遗传相似度(identity-by-state, IBS)数据[23]。IBS越小, 表明种质之间的亲缘关系越近。运用Treebest-1.9.2软件计算距离矩阵, 采用邻接法(Neighbor-Joining Method)构建系统进化树[24]。
1.2.4 群体结构分析和主成分分析(PCA) 使用fast Structure软件进行群体结构分析[25], GCTA软件进行主成分分析(PCA)分析[26]。
2 结果与分析
2.1 春播早熟区高粱亲本系杂合度和遗传距离分析
1,304,623个SNP平均最小等位基因频率0.205, 变幅为0.05~0.50。55份亲本系平均观测杂合度为0.241, 变异范围为0.090~0.319。杂合度比较低的是TX623B、辽宁黏B404B, 分别是0.090、0.091、0.135。杂合度比较高的是南108、吉2731B、299, 分别是0.308、0.308、0.319。67%亲本系的杂合度在0.203~0.300之间。平均多态信息含量(PIC)为0.2935, 变幅为0.1~0.5。55份亲本系的遗传距离变异范围为0.627~0.927, 平均遗传距离为0.704。55份材料两两之间的遗传距离见图1。
2.2 春播早熟区高粱亲本系群体结构与聚类分析
依据群体结构和聚类分析将55份亲本系分成4类(图2-A)。第1类包含SX44B、5222B、L407B、A2V4B、406B、521B共6份种质, 群体结构显示该类种质由Durra类群选育而来, 因此命名为Durra类群(简称D群); 第2类包含TAM428B、352B、7050B、辽宁黏B、TX623B、404B、QL33B、繁8B、5535B、吉2055B、314B、MHB、晋长早B、赤6B、2316B、哲15B、4190B共17份种质, 群体结构显示该类种质多由Kafir和Durra杂交选育而来, 命名为Kafir/Durra类群(简称KD群); 第3类仅包含忻4B、1230B、黑30B、哈恢144、赤恢428共5份种质, 群体结构显示该类种质多Russia和Kafir高粱杂交选育而来, 因此命名为Russia/Kafir类群(简称RK群); 第4类包含吉R159、LR9198、吉R117、吉2731B、299、哈恢75号、盘陀早、5933、7788、6115、7313、7384、2598、吉R9060、南108、忻粱52、吉R105、吉R107、吉R8036、吉R109、吉恢13、LR116、南133、T180、0-30、7616-533、三尺三共27份种质, 其群体结构显示该类种质多由kaoliang类群改良而来, 有少量的Kafir和Durra亲缘, 故命名为kaoliang类群(简称K群)。其中第1、第2、第3类群以保持系为主, 第4类群以恢复系为主。
2.3 春播早熟区高粱亲本系主成分分析
为更准确地划分55份亲本系的杂种优势群, 进行了主成分分析(PCA)。主成分分析也将55份亲本系分成了4类(图2-B)。主成分1 (PC1)、主成分2 (PC2)和主成分3 (PC3)解释的遗传方差分别为15.47%、8.29%和5.24%, 共解释供试材料中SNP总变异的29%。
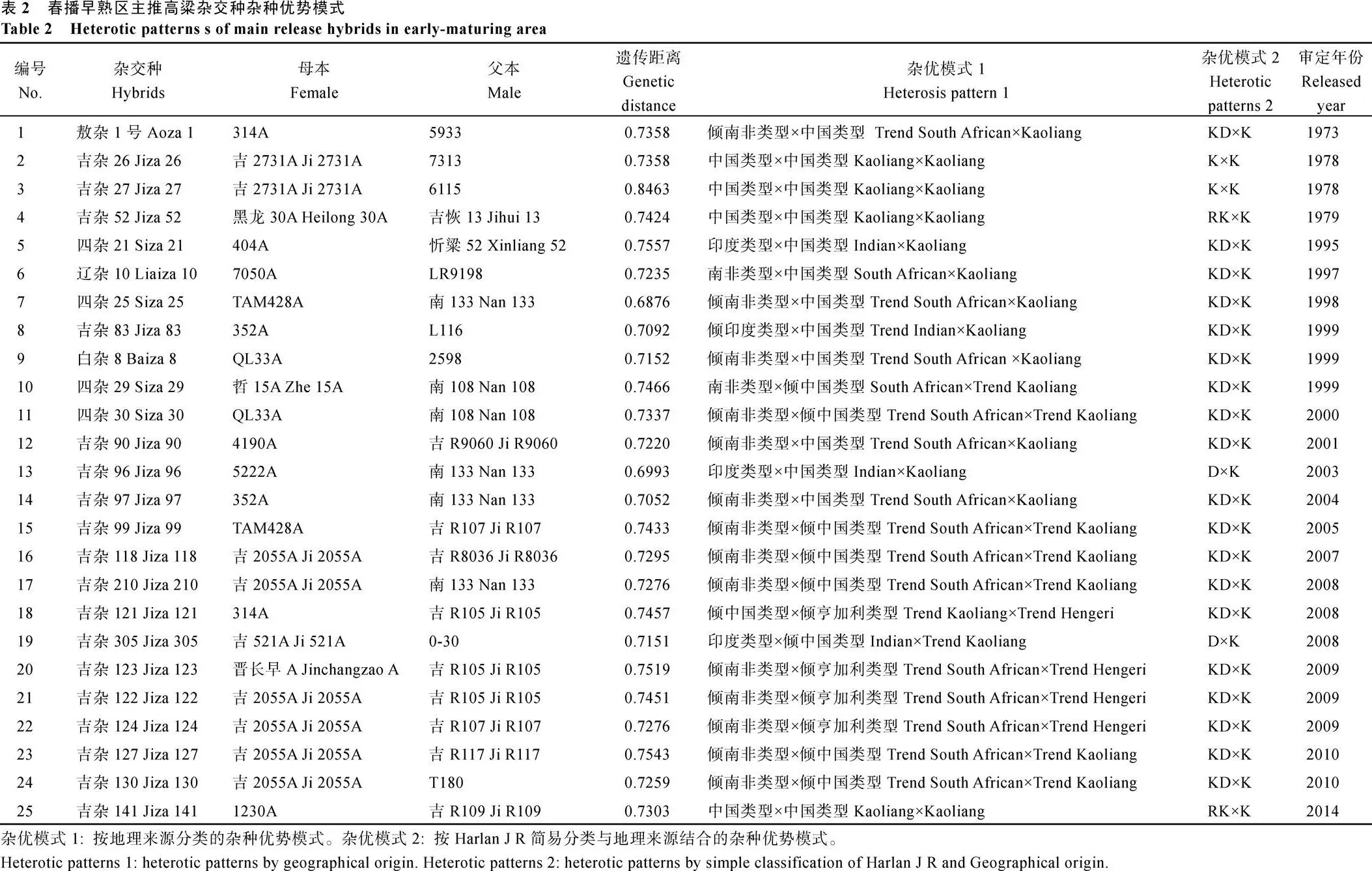
本试验55份亲本系中33份亲本系是1973—2014年间春播早熟区生产中主推的25个杂交种的亲本, 根据本试验杂种优势类群划分的结果, 这25个主推杂交种的杂种优势利用模式如表2所示, 其中有18个杂交种的杂种优势利用模式为KD×K, 2个杂交种的杂种优势利用模式为K×K, 2个杂交种的杂种优势利用模式为D×K, 2个杂交种的杂种优势利用模式为RK×K。同时有4个杂交种的杂优模式与先前认知的不同, 比如吉杂52、吉杂141被认为是中国类型×中国类型, 本试验结果为RK×K; 辽杂10被认为是南非类型×中国类型, 本试验结果为KD×K; 四杂21被认为是印度类型×中国类型, 本试验结果为KD×K。吉杂121、吉杂123、吉杂122和124四个杂交种的父本被认为是倾亨加利类型, 本试验结果均属于Kaoliang类群。

图1 春播早熟区高粱亲本系遗传距离
红色表示遗传距离较远, 蓝色表示遗传距离较近。
Red represents a high IBS value, showing a greater distance between two lines. Blue represents a low IBS value, showing a close distance between two lines.
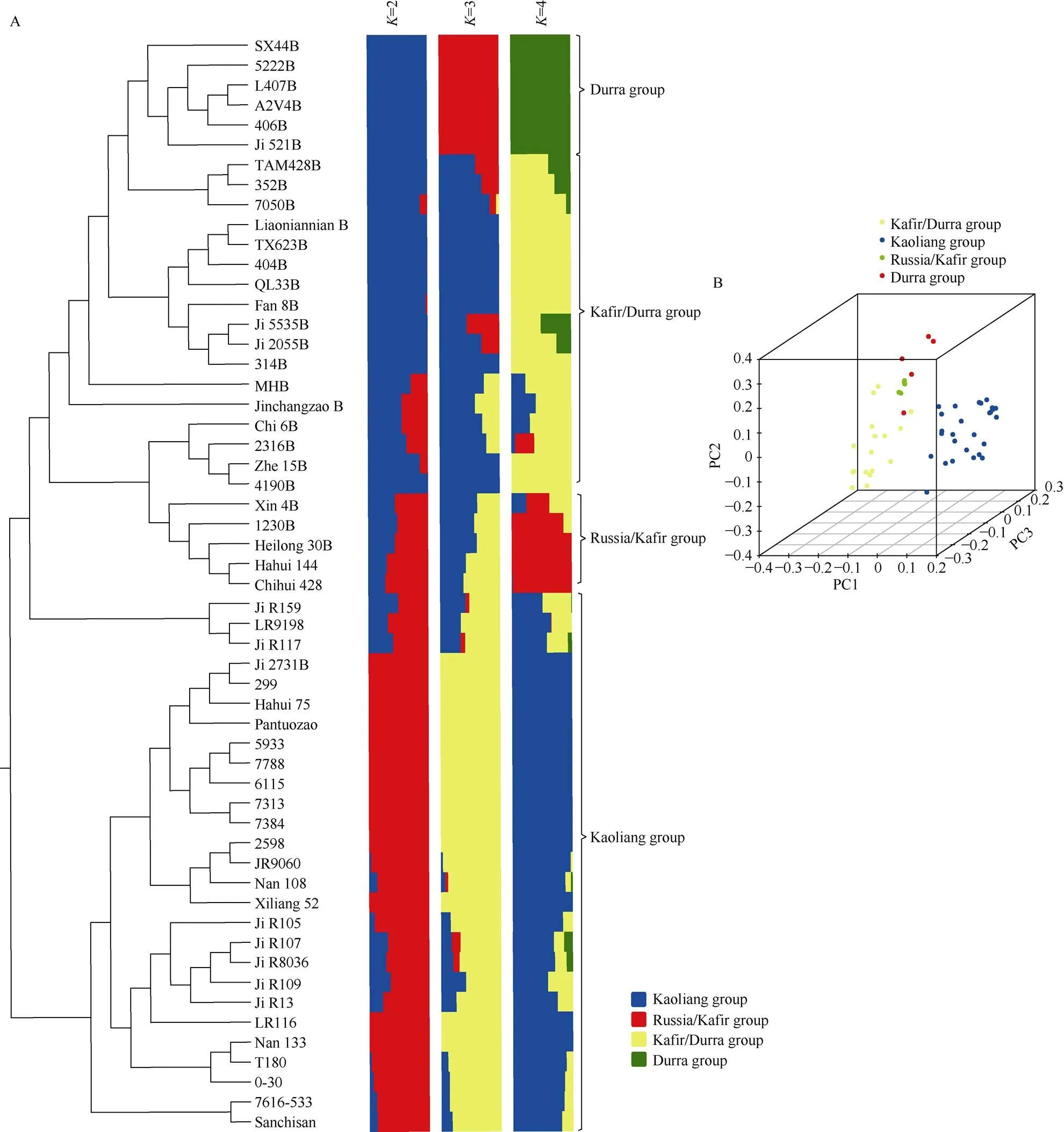
图2 春播早熟区高粱亲本系聚类分析、群体结构和主成分分析
A: 55个高粱亲本系聚类分析、群体结构结果图。55份高粱亲本系=2~4的群体结构。每条横线代表一个亲本系, 不同颜色代表基因型来源不同。当=4时, 群体可分为都拉群、卡佛尔/都拉群、俄罗斯/卡佛尔群、中国高粱群。B: 55个高粱亲本系3个主成分分析结果图。黑点表示卡佛尔/都拉群、蓝点表示中国高粱群、绿点表示都拉群和红点表示俄罗斯/卡佛尔群。
A: the clustering and population structure of 55 sorghum parental lines. The population structure analysis using 1,304,623 SNP. Each individual is represented by a horizontal bar, partitioned into colored segments with the length of each segment representing the proportion of the individual’s genome from=2–4 groups. When=4, the genetic groups named the Durra, Kafir/Durra, Russia/Kafir, Kaoliang. B: PCA plot of the three components (PC1, PC2, and PC3) of the 55 sorghum parental lines. Black plot represents Kafir/Durra group, blue plot represents Kaoliang group, green plot represents Durra group and red plot represents Russia/Kafir group.
2.4 春播早熟区主推杂交种杂种优势利用模式
2.4.1 春播早熟区主推杂交种杂种优势利用模式 本试验55份亲本系中33份亲本系是1973— 2014年间春播早熟区生产中主推的25个杂交种的亲本,根据本试验杂种优势类群划分的结果,这25个主推杂交种的杂种优势利用模式如表2所示,其中有18个杂交种的杂种优势利用模式为KD×K,2个杂交种的杂种优势利用模式为K×K,2个杂交种的杂种优势利用模式为D×K,2个杂交种的杂种优势利用模式为RK×K。同时有4个杂交种的杂优模式与先前认知的不同,比如吉杂52、吉杂141被认为是中国类型×中国类型,本试验结果为RK×K;辽杂10被认为是南非类型×中国类型,本试验结果为KD×K;四杂21被认为是印度类型×中国类型,本试验结果为KD×K。吉杂121、吉杂123、吉杂122和124四个杂交种的父本被认为是倾亨加利类型,本试验结果均属于Kaoliang类群。
2.4.2 春播早熟区主推杂交种父母本间遗传距离分析 25个杂交种的2个亲本之间的遗传距离变幅为0.6876~0.8463, 平均为0.7342。遗传距离最大的是1978年培育的吉杂27, 其父母本的遗传距离达到了0.8463, 其2个亲本均来自于Kaoliang类群。父母本遗传距离最小的杂交种是四杂25, 为0.6876。春播早熟区目前主推杂交种吉杂210、吉杂124、吉杂127亲本间的遗传距离为0.7276、0.7276、0.7543。2个D×K杂优模式的杂交种吉杂96、吉杂305的亲本遗传距离并不突出, 分别为0.6993和0.7151。
3 讨论
由于生产中越来越多的亲本具有2个或2个以上的亲缘, 依据种质地理来源分类的方法已不能将种质详细划分类群。从分子水平解析种质类群归属、合理准确地划分作物亲本杂种优势群, 建立相应的杂种优势模式[27], 对于培育新型亲本、选配突破性杂交组合, 提高高粱杂种优势育种效率具有重要意义[28]。
3.1 春播早熟区高粱亲本系杂种优势群与杂种优势利用模式分析
本研究利用SNP标记对55份亲本系划分类群, 并采用Harlan和de Wet的5个基本型和10个中间型[29]与地理来源结合的方法对4个杂种优势群命名, 即Durra、Kafir/Durra、Russia/Kafir和Kaoliang类群。划分的结果与系谱来源有较好的一致性, 比如SX44B、V4B、406B具有类似的亲缘, 被聚为同一类群。恢复系吉R117从LR9198的天然变异株中选育而来, 吉R159又具有吉R117的亲缘, 被聚为同一类群。与按地理来源分类的杂种优势群相比, 本研究中划分杂种优势群可清晰看出亲本的来源, 尤其是被认为是倾南非类型的亲本均明确为Kafir/Durra类群。表明采用SNP标记对高粱亲本系划分杂种优势群可以获的与其他作物一样比较理想的结果[30]。
25个主推杂交种中有19个杂交种的杂种优势模式为Kafir/Durra×Kaoliang, 占到76%。春播早熟区目前主推杂交种吉杂210、吉杂124、吉杂127均是Kafir/Durra×Kaoliang杂优模式。回顾春播早熟区高粱不育系、恢复系培育历程, 20世纪60至80年代 Tx3197AB、Tx6A系列不育系逐渐引入早熟区, 但是Tx3197A、Tx6A系列不育系在早熟区组配的杂交组合生育期比较长, 不能正常成熟。育种家们采用该生态区中国高粱保持类型材料(生育期短)与Tx3197B、Tx6B系列进行人工去雄杂交, 在后代中选育适合早熟区种植的保持系再回交转育不育系[31],比如早熟区主推杂交种敖杂1号的母本314A由Tx3197B×库班红杂交选育而来[32], 此时不育系以Kafir群为主。20世纪80年代之后, 由美国、国际半干旱研究所和澳大利亚逐渐引入TAM428AB、421AB、QL33AB等Kafir/Durra、Durra群引入之后, 逐渐培育出4190A、7050A、吉2055A等Kafir/Durra群不育系。而恢复系均是由我国Kaoliang类型改良而来。因此形成了Kafir/Durra×Kaoliang杂优模式。然而从1973年至2014年40余年间主推杂交种的杂优模式没有发生较大的变化, 尤其1995年至今的杂交种都是Kafir/Durra×Kaoliang模式, 且杂交种双亲之间的遗传距离也没有显著增加。
3.2 春播早熟区高粱不育系、恢复系遗传基础与改良
春播早熟区高粱不育系主要来源于Kafir和Durra类群, 少部分极早熟不育系属于Russia类群。春播早熟区著名不育系吉2055A是76%的Kafir亲缘、24%的Durra亲缘。新培育的糯性不育系吉5535A具有50% Kafir亲缘和50% Durra亲缘。4190A和314A则属于纯Kafir类群。通过本试验分析1230A、黑30A是带有少部分Kafir亲缘的Russia类群。
恢复系多是Kaoliang类群或在Kaoliang类群基础上引入少量Kafir、Durra亲缘。主推品种吉杂124父本吉R107具有69% Kaoliang、17% Kafir和14% Durra亲缘, 主推杂交种吉杂127的父本吉R117具有58% Kaoliang亲缘、35% Kafir亲缘和7% Durra亲缘。已知具有亨加利高粱血缘的恢复系南133[33]、吉R105[34]、吉R107[35]被划分在具有一定Kafir亲缘或Durra亲缘的Kaoliang类群中, 推断亨加利高粱不是一个独立的类型, 属于Kafir类群。
春播早熟区包括吉林、黑龙江、内蒙古等省区全部, 河北省承德地区、张家口坝下地区, 山西、陕西北部等地区。春播时杂交种和制种田容易受低温冷害, 造成出苗缺苗断条的情况, 主要内因还在于不育系亲缘主要来源于来自热带地区的Kafir和Durra类群, 不育系耐冷性不够突出, 若将不育系亲缘改良成我国自产的Kaoliang类群, 即可解决这个生产上的大问题。比如可多采用吉2731B, 吉2731B是从中国地方品种红棒子与黑壳打锣锤杂交选育而来, 具有耐冷、出苗快, 籽粒颜色黄红色、颜色好看等优点, 本试验分析吉2731B与南133、吉R105等多个亲本系的遗传距离均0.8以上。在选育恢复系时, 注意向Kafir或Durra亲缘选育, 增强杂交种的杂种优势, 将杂种优势模式变为Kaoliang×Kafir、Kaoliang×Durra或Kaoliang×Kafir/Durra。
4 结论
本研究利用1,304,623个高质量SNP标记对春播早熟区55份高粱亲本系估算遗传距离、划分杂种优势群, 研究主推杂交种的杂种优势模式。群体结构和PCA分析将55份亲本系划分为4个杂种优势群, 采用Harlan J R的简易分类与地理来源结合的方法对4个杂种优势群命名为Durra、Kafir/Durra、Russia/Kafir、Kaoliang。主推高粱杂交种的不育系主要来源于引自国外的Kafir和Durra群, 恢复系多来源于我国自产的Kaoliang群。Kafir/Durra× Kaoliang是春播早熟区高粱育种的主要杂种优势模式。本研究为春播早熟区高粱育种拓宽种质遗传基础、杂种优势深入研究以及开创新的杂种优势模式提供理论基础。
[1] 王胜军. 杂交籼稻亲本杂种优势群的划分及杂种优势模式的构建. 南京农业大学博士论文, 江苏南京, 2006.
Wang S J. Grouping of Parental Lines and Establishment of Heterotic Patterns inHybrid Rice (L.). PhD Dissertation of Nanjing Agricultural University, Nanjing, Jiangsu, China, 2006 (in Chinese with English abstract).
[2] 姜思奇, 郭瑞, 张敖, 赵艳贺, 时免免, 邓丽霞, 崔震海, 阮燕晔. 利用核心SNP标记划分辽宁省常用玉米自交系杂种优势群的研究. 玉米科学, 2018, 26(4): 17–23.
Jiang S Q, Guo R, Zhang A, Zhao Y H, Shi M M, Deng L X, Cui Z H, Ruan Y Y. Heterotic grouping by core SNP markers for maize inbred lines widely used in Liaoning province., 2018, 26(4): 17–23 (in Chinese with English abstract).
[3] 卢柏山, 史亚兴, 宋伟, 徐丽, 赵久然. 利用SNP标记划分甜玉米自交系的杂种优势类群. 玉米科学, 2015, 23(1): 58–62.
Lu B S, Shi Y X, Song W, Xu L, Zhao J R. Heterotic grouping of sweet corn inbred lines by SNP markers., 2015, 23(1): 58–62 (in Chinese with English abstract).
[4] 刘炜, 史延丽, 李白超, 马洪文, 王坚, 张洪亮. 粳型水稻杂种优势生态型与杂种优势模式的研究. 西北农林科技大学学报(自然科学版), 2005, 33(1): 108–114.
Liu W, Shi Y L, Li Z C, Ma H W, Wang J, Zhang H L. Studies on heterotic ecotypes and heterotic patters ofrice.(Nat Sci Edn) 2005, 33(1): 108–114 (in Chinese with English abstract).
[5] 逯腊虎, 李振兴, 倪中福, 彭惠茹, 聂秀玲, 孙其信. 小麦杂种优势群研究: VI. 普通小麦与穗分枝小麦、轮回选择后代材料、西藏半野生小麦和斯卑尔脱小麦早熟诱变系的SSR分子标记遗传差异研究. 麦类作物学报, 2007, 27(2): 201–206.
Lu L H, Li Z X, Ni Z F, Peng H R, Nie X L, Sun Q X. Study on wheat heterotic group: VI. Genetic diversity revealed by SSR marker between common wheat, ear branched wheat, wheat lines derived from recurrent selection, Tibetan wheat and early spelt wheat mutant lines., 2007, 27(2): 201–206 (in Chinese with English abstract).
[6] 刘正理. 谷子杂种优势群的构建方法及研究进展. 河北农业科学, 2010, 14(11): 102–104.
Liu Z L. Establish method of heterotic group in foxtail millet and its research progress., 2010, 14(11): 102–104 (in Chinese with English abstract).
[7] 陈冰嬬, 李继洪, 王阳, 李淑杰, 胡喜连, 李伟, 马英慧, 高鸣, 高士杰. 高粱((L.) Moench)种质资源研究进展. 西北农林科技大学学报(自然科学版), 2013, 41(1): 67–72.
Chen B R, Li J H, Wang Y, Li S J, Hu X L, Li W, Ma Y H, Gao M, Gao S J. Advances in germplasm resources of sorghum ((L.) Moench).(Nat Sci Edn), 2013, 41(1): 67–72 (in Chinese with English abstract).
[8] 高士杰, 韩少颖. 中国杂交高粱的研究与利用. 北京农业大学学报, 1993, 19(增刊1): 92–94.
Gao S J, Han S Y. A study and application of sorghum hybrids in China., 1993, 19(S1): 92–94 (in Chinese with English abstract).
[9] 马鸿图. 高粱杂种优势与类型血缘关系的探讨. 沈阳农业大学学报, 1974, (1): 60–68.
Ma H T. The relation of heterosis in sorghum and consanguinity of type., 1974, (1): 60–68 (in Chinese with English abstract).
[10] 邹剑秋, 朱凯, 王艳秋, 杨晓光. 高粱雄性不育系7050A的选育与应用. 作物杂志, 2010, (2): 101–104.
Zou J Q, Zhu K, Wang Y Q, Yang X G. Development and application of sorghum males sterile line 7050A., 2010, (2): 101–104 (in Chinese with English abstract).
[11] 李团银, 张福耀, 李三棉, 韦耀明, 柳青山. 高粱新型细胞质雄性不育系A2V4A选育及其应用研究, 中国农学通报, 1995, 11(5): 10–13.
Li T Y, Zhang F Y, Li S M, Wei Y M, Liu Q S. Development of a new type cytoplasmic male sterile line A2V4A and its application in sorghum., 1995, 11(5): 10–13 (in Chinese with English abstract).
[12] 卢庆善. 我国高粱杂种优势利用回顾与展望. 辽宁农业科学, 1992, (3): 40–44.
Lu Q S. Retrospect and prospect on heterosis utilization of sorghum in China., 1992, (3): 40–44 (in Chinese).
[13] 高士杰, 刘晓辉, 郭中校, 李继洪. 中国杂交高粱的种质基础及优势利用模式研究. 中国农学通报, 2005, 21(10): 106–108.
Gao S J, Liu S H, Guo Z X, Li J H. Chinese hybrid sorghum germplasm foundation and superiority use pattern., 2005, 21(10): 106–108 (in Chinese with English abstract).
[14] 王方, 张凤昌. 高粱不同类群杂种优势研究. 吉林农业科学, 1982, (3): 6–12.
Wang F, Zhang F C. The Heterosis analysis of different groups of sorghum., 1982, (3): 6–12.
[15] Wang L M, Jiao S J, Jiang Y X, Yan H D, Su D F, Sun G Q, Yan X F, Sun LF. Genetic structure analysis of sorghum parent lines based on SSR markers., 2013, 41: 359–365.
[16] 王瑞, 王金胜, 张福耀, 程庆军, 田承华, 凌亮. 1970s–2000s中国高粱杂交种亲本遗传距离演变的SSR分析. 中国农业科学, 2015, 48: 415–425.
Wang R, Wang J S, Zhang F Y, Cheng Q J, Tian C H, Ling L. Evolution of genetic distance between parental lines of Chinese sorghum hybrids from 1970s–2000s based on SSR analysis., 2015, 48: 415–425 (in Chinese with English abstract).
[17] 高旭, 周棱波, 张国兵, 邵明波, 张立异. 基于SSR 标记的粒用高粱资源遗传多样性及群体结构. 贵州农业科学, 2016, 44(9): 13–19.
Gao X, Zhou L B, Zhang G B, Shao M B, Zhang L Y. Genetic diversity and population structure of grain sorghum germplasm resources based on SSR marker., 2016, 44(9): 13–19 (in Chinese with English abstract).
[18] 肖松, 周棱波, 张国兵, 邵明波, 乙引, 张立异. 酱香型白酒用糯高粱种质遗传多样性分析. 江苏农业科学, 2016, 44(4): 45–49.
Xiao S, Zhou L B, Zhang G B, Shao M B, Yi Y, Zhang L Y. Genetic diversity analysis of Maotai-flavor liquor-making waxy sorghum., 2016, 44(4): 45–49 (in Chinese with English abstract).
[19] 汪灿, 周棱波, 高旭, 张国兵, 程斌, 曹宁, 丁延庆, 徐燕, 邵明波, 张立异. 基于分型测序技术的粒用高粱的遗传多样性和群体结构分析. 植物遗传资源学报, 2019, 20: 677–684.
Wand C, Zhou L B, Gao X, Zhang G B, Cheng B, Cao N, Ding Y Q, Xu Y, Shao M B, Zhang L Y. Genetic diversity and population structure analysis of grain-use sorghum based on genotyping by sequencing technology., 2019, 20: 677–684 (in Chinese with English abstract).
[20] Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA., 1980, 8: 4321–4325.
[21] Liu K, Muse S V. Power Marker: an integrated analysis environment for genetic marker analysis., 2005, 21: 2128–2129.
[22] Chang C C, Chow C C, Tellier L C, Vattikuti S, Purcell S M, Lee J J. Second-generation PLINK: rising to the challenge of larger and richer datasets., 2015, 4: 7.
[23] Bradbury P J, Zhang Z, Kroon D E, Casstevens T M, Ramdoss Y, Buckler E S. TASSEL: software for association mapping of complex traits in diverse samples., 2007, 23: 2633–2635.
[24] Heng L. http://treesoft.Sourceforge.net/treebest.shtml. Version: 1.9.2, 2014 Apr.
[25] Raj A, Stephens, Pritchard J K. Fast STRUCTURE: variational inference of population structure in large SNP data sets., 2014, 1975: 573–589.
[26] Yang J, Lee S H, Goddard M E. GCTA: a tool for genome-wide complex trait analysis., 2011, 88: 76–82.
[27] 任羽, 王得元, 张银东. 分子标记预测作物杂种优势的研究进展. 分子植物育种, 2004, 2: 401–406.
Ren Y, Wang D Y, Zhang Y D. Progresses in the heterosis molecular prediction in the crops., 2004, 2: 401–406 (in Chinese with English abstract).
[28] 李新海, 袁力行, 李晓辉, 张世煌, 李明顺, 李文华. 利用SSR标记划分70份我国玉米自交系的杂种优势群. 中国农业科学, 2003, 36: 622–627.
Li X H, Yuan L X, Li X H, Zhang S H, Li M S, Li W H. Heterotic grouping of 70 maize inbred lines by SSR markers., 2003, 36: 622–627 (in Chinese with English abstract).
[29] Harlan J R, Wet J M. A simplified classification of cultivated sorghum., 1972, 12: 172–176.
[30] 吴金凤, 宋伟, 王蕊, 田红丽, 李雪, 王凤格, 赵久然, 蔚荣海.利用SNP标记对51份玉米自交系进行类群划分. 玉米科学, 2014, 22(5): 29–34.
Wu J F, Song W, Wang R, Tian H L, Li X, Wang F G, Zhao J R, Wei R H. Heterotic grouping of 51 maize inbred lines by SNP markers., 2014, 22(5): 29–34 (in Chinese with English abstract).
[31] 王富德, 卢庆善. 我国主要高粱杂交种的系谱分析. 作物学报, 1985, 11: 9–14.
Wang F D, Lu Q S. An analysis of pedigrees of the main hybrid sorghums in China., 1985, 11: 9–14(in Chinese with English abstract).
[32] 张辉, 曲文祥, 李书田. 内蒙古特色作物. 北京: 中国农业科学技术出版社, 2010. pp 166–167.
Zhang H, Qu W X, Li S T. Special Crop of Inner Mongolia. Beijing: China Agricultural Science and Technology Press, 2010. pp 166–167 (in Chinese).
[33] 马忠良, 张淑君, 周紫阳, 王江红, 闫鸿雁, 李光华, 马英慧. 优良高粱恢复系南133的选育与利用. 杂粮作物, 2006, 26(3): 178–179.
Ma Z L, Zhang S J, Zhou Z Y, Wang J H, Yan H Y, Li G H, Ma Y H. Breeding and utilization of grain sorghum excellent restorer line Nan 133., 2006, 26(3): 178–179 (in Chinese with English abstract).
[34] 高士杰, 李继洪, 李伟. 高粱优良恢复系吉R105的选育与利用. 种子, 2009, 28(5): 107–108.
Gao S J, Li J H, Li W. Breeding and utilization of grain sorghum excellent restorer line Ji R105., 2009, 28(5): 107–108 (in Chinese with English abstract).
[35] 陈冰嬬, 李继洪, 高士杰, 王阳, 马英慧. 高粱长穗型恢复系吉R107的选育与应用. 现代农业科技, 2011, (24): 107–108.
Chen B R, Li J H, Gao S J, Wang Y, Ma Y H. Breeding and application of sorghum long panicle restorer line Ji R107., 2011, (24): 107–108 (in Chinese).
Analysis of heterotic groups and heterosis patterns of sorghum in early-maturing area
CHEN Bing-Ru1, YU Miao1, GE Zhan-Yu2, LI Hong-Kui3, HUANG Yan3, LI Hai-Qing1, SHI Gui-Shan1, XIE Li1, XU Ning1, YAN Feng4, GAO Shi-Jie1, ZHOU Zi-Yang1,*, and WANG Nai1,*
1Institute of Crop Germplasm Resources, Jilin Academy of Agricultural Sciences, Changchun 130033, Jilin, China;2Chifeng Academy of Agricultural and Animal Husbandry Sciences, Chifeng 024031, Inner Mongolia, China;3Jilin Province Baicheng Academy of Agricultural Sciences, Baicheng 137000, Jilin, China;4Qiqihar Sub-academy of Heilongjiang Academy of Agricultural Sciences, Qiqihar 161006, Heilongjiang, China
The division of heterotic groups can play an important guiding role in broadening the genetic basis of parents, improving breeding efficiency and developing breakthrough new hybrids. In this study, we used the whole genome resequencing technology to scan the whole genome of 55 parent lines of dominant hybrids lines in spring sowing early maturing region for more than 40 years, analyzed their population structure, estimated genetic distance, divided heterosis groups, and analyzed heterosis patterns of dominant hybrid lines. The results showed that the genetic distance of 55 germplasm were 0.704, with a range of 0.627–0.927, and the average polymorphism information content (PIC) was 0.2935, range from 0.1 to 0.5. Fifty-five parents’ lines were classified into 4 heterotic groups that Durra, Kafir/Durra, Russia/Kafir, Kaoliang by the population structure and principal components analysis. Meanwhile, heterosis models of 76% hybrids of 25 main release hybrids from 1973–2014 years were Kafir/Durra × Kaoliang. The sterile line of main release hybrids was derived from Kafir and durra groups from abroad, and the restorer lines were derived from the Kaoliang group produced in China. The heterotic groups divided in this study clarified the genetic basis of sorghum parents’ lines in early- maturing area in China, which provided a scientific basis for the improvement of parental lines and the innovation of heterotic patterns.
sorghum; parental lines; SNP; heterosis; heterotic groups; heterotic patterns
10.3724/SP.J.1006.2023.24035
本研究由高粱遗传与种质创新山西省重点实验室项目青年基金课题(2016K-01), 吉林省科技发展计划项目(20210202001NC), 吉林省农业科技创新工程项目(CXGC2021DX005)和财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-06)资助。
This study was supported by the Youth Fund of Key Laboratory of Sorghum Genetics and Germplasm Innovation in Shanxi Province (2016K-01), the Science and Technology Development Program of Jilin Province (20210202001NC), the Science and Technology Innovation Project of Jilin Province (CXGC2021DX005), and the China Agriculture Research System of MOF and MARA (CARS-06).
王鼐, E-mail: wang-nai@163.com; 周紫阳, E-mail: ziyang_z@163.com
E-mail: chenbingru1979@163.com
2022-02-07;
2022-05-05;
2022-05-31.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220530.2128.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)
