青狗尾草RNAi途径相关基因的全基因组鉴定和表达分析
2023-02-02罗皓天王龙王禹茜王月李佳祯杨梦珂张杰邓欣王红艳
罗皓天 王龙 王禹茜 王月 李佳祯 杨梦珂 张杰 邓欣 王红艳
(辽宁大学生命科学院,沈阳 110036)
RNA 介导的DNA 甲基化(RNA-directed DNA Methylation,RdDM),是生物体内DNA 甲基化从头构建的主要途径[1]。在植物中分为典型的RdDM 途径和非典型的RdDM 途径,其中非典型的RdDM 途径还包括转录后沉默(post-transcriptional gene silencing,PTGS),即RNA 干扰(RNA interference,RNAi)途径[2]。RNAi 途径首先利用 DNA 或 RNA 合成双链 RNA(double-stranded RNA,dsRNA),RNase 会将 dsRNA 切割成小 RNA(small RNA,sRNA),这些小 RNA 可对目标序列的 mRNA 进行降解,从而特异性地沉默或抑制该基因表达[3]。这一途径在对抗病毒RNA、转座子的沉默等过程中发挥重要作用[2]。在RNAi 途径中有3 种蛋白质发挥主要作用,分别是识别并结合sRNA 序列的Argonaute(AGO)、切割dsRNA 成为sRNA 的Dicer-like(DCL)和RNA依赖的RNA 聚合酶(RDR)。AGO 家族蛋白可以与21-24 nt 的小RNA(small RNA,sRNA)相结合,并通过与SPT5L、NRPE1 和IDN2-IDP 复合物的相互作用和帮助下,将DRM2 招募到DNA 中[3-5]。不同AGO 蛋白功能也有不同,例如AGO1 可结合21-22 nt 的sRNA,AGO4 和AGO6 可结合24 nt 的sRNA;AGO9 可在生殖组织中发挥作用;AGO18 为禾本科特有的一类AGO 蛋白,最早在水稻中被发现,主要与病毒外源RNA 入侵的防御有关[3,6]。DCL 家族是一类具有核酸内切酶活性的蛋白质,它们可以将双链小RNA(dsRNA)剪切成21-24 nt 的sRNA[5]。DCL1 可切割miRNA 前体或具有反向重复序列的mRNA,如转座元件,并产生21 nt 的sRNA[7-10]。DCL2 可切割mRNA 并产生22 nt 的sRNA[5]。DCL3会优先靶向Pol Ⅳ-RDR2 复合物产生的dsRNA,切割并产生24 nt 的sRNA,但也可以切割其他dsRNA底物[5,7-8]。DCL4 可产生21 nt 的sRNA,在禾本科中SHO 为DCL4 的同源蛋白[7-8]。DCL1/2/3/4 之间会互相竞争底物,当DCL2/4 参与的PTGS 通路饱和时,DCL3 可以介入并处理DCL2/4 的dsRNA 底物,触发PTGS 向RdDM 介导的TGS 转换[7-8,11]。RDR家族是一类RNA 依赖型RNA 聚合酶[5]。RDR2 可以与Pol Ⅳ结合,将Pol Ⅳ产生的单链RNA 产物加工成双链RNA[12]。RDR6 可将AGO1 产生的单链sRNA 加工成dsRNA,从而导致PTGS[5,13]。
目前已知的所有动植物中均存在AGO、DCL 和RDR 基因的多个拷贝,并且部分基因的功能已被阐明[14]。以禾本科为例,水稻(Oryza sativa)中含有19 种AGO 蛋白,8 个DCL 类蛋白和5 个RDR 蛋白[14]。在谷子(Setaria italica)中鉴定出AGO 蛋白13 个,DCL 蛋白7 个以及RDR 蛋白4 个[15]。基因拷贝数的多样性为基因功能的变化提供了空间,有利于物种的进化[16]。青狗尾草(Setaria viridis)是谷子的野生近缘种,为禾本科2 倍体(2n=2x=18)C4植物,广泛分布在世界各地。大约在9 000-6 000年前,青狗尾草被驯化成为谷子[17]。在作物驯化与改良过程中,落粒性和株高是两个重要的目标性状。对青狗尾草群体测序,人们发现控制落粒性状的基因SiLes1由于转座元件的插入使其发生了突变,从而使得青狗尾草丧失了落粒性,并重现了青狗尾草驯化的初始阶段[18]。可见物种驯化的过程是极其复杂的,但表观遗传修饰基因是否受到驯化影响还有待研究。
因此,本研究通过生物信息学和比较基因组学方法,对青狗尾草的RNAi 途径相关的3 种主要基因家族进行全基因组的挖掘和鉴定,并对其亚细胞位置、系统发育关系、保守结构域、染色体位置、Ka/Ks 比值以及基因表达水平进行分析。同时比较了各家族成员在青狗尾草和谷子间的异同。本研究为上述基因在调控青狗尾草的表观遗传修饰中的功能和作用提供初步的理论依据,为青狗尾草与谷子之间的驯化和基因进化的分子机制提供参考。
1 材料与方法
1.1 青狗尾草RNAi途径相关基因的鉴定
青狗尾草基因组下载自Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)。利用已知的谷子RNAi 途径相关基因的蛋白质序列作为参考序列[15],在青狗尾草的蛋白质序列中做blastp,得到的结果送至Pfam(https://pfam.xfam.org/)做进一步验证。
1.2 蛋白质理化性质及亚细胞定位
将得到的青狗尾草RNAi 途径相关基因的蛋白质序列上传至ExPASy(https://web.expasy.org/protparam/)和BUSCA(http://busca.biocomp.unibo.it/)得到其蛋白质的理化性质和预测的亚细胞定位。
1.3 系统发育分析、保守结构域和染色体定位
利用MEGA X(v5.1.1)对拟南芥、水稻、谷子和青狗尾草的RNAi 途径相关基因的蛋白质全长分别构建NJ 系统发育树,使用Bootstrap 测试和1 000个重复来评估每个节点的统计一致性。
将青狗尾草的RNAi 途径相关基因的蛋白质序列上传至Pfam 数据库进行分析,得到序列的保守结构域信息。
根据青狗尾草和谷子的基因组信息,利用在线软件MapGene2Chrom(http://mg2c.iask.in/mg2c_v2.0/)定位青狗尾草和谷子的RNAi 途径相关基因的染色体位置。
1.4 Ka/Ks和跨膜结构分析
利用TBtools(v1.082)分析青狗尾草和谷子的RNAi 途径相关基因的CDS 序列的Ka/Ks[19]。
1.5 转录组分析
青狗尾草和谷子的转录组数据下载自Phytozome(PRJNA633601,SRX116346-SRX116357)[18,20]。
2 结果
2.1 青狗尾草RNAi途径相关基因的全基因组鉴定、蛋白质理化性质和亚细胞定位
为了鉴定出青狗尾草RNAi 途径相关基因,利用已知的谷子AGO、DCL 和RDR 蛋白质序列在青狗尾草中进行同源探寻,并鉴定保守结构域。每个谷子AGO、DCL 和RDR 蛋白质均能在青狗尾草基因组中找到同源蛋白。结果共得到青狗尾草的13 个AGO、7 个DCL 和4 个RDR 蛋白质序列,它们与谷子同源蛋白的相似度在95%-100%之间(表1),蛋白质长度在313(SvDCL1b)-1 933(SvDCL1a)氨基酸之间,相对分子质量在35 044.76(SvDCL1b)-216 220.21(SvDCL1a)Mw/Da 之间,理论等电点5.01(SvDCL1b)-9.55(SvAGO1c) 之间。此外,SvDCL1b(38.74)、SvDCL1c(29.3)、SvAGO2(38.85)和SvRDR1(39.69)的不稳定指数小于40,说明其为稳定蛋白,其余为不稳定蛋白。亚细胞定位的预测结果显示SvDCL3b 和SvAGO1b 被定位在细胞间隙;SvMEL1 定位在叶绿体类囊体腔;SvRDR2 定位在叶绿体类囊体膜,其余均定位在细胞核中,说明同一家族的不同成员可能在不同细胞组分内发挥作用(表2)。
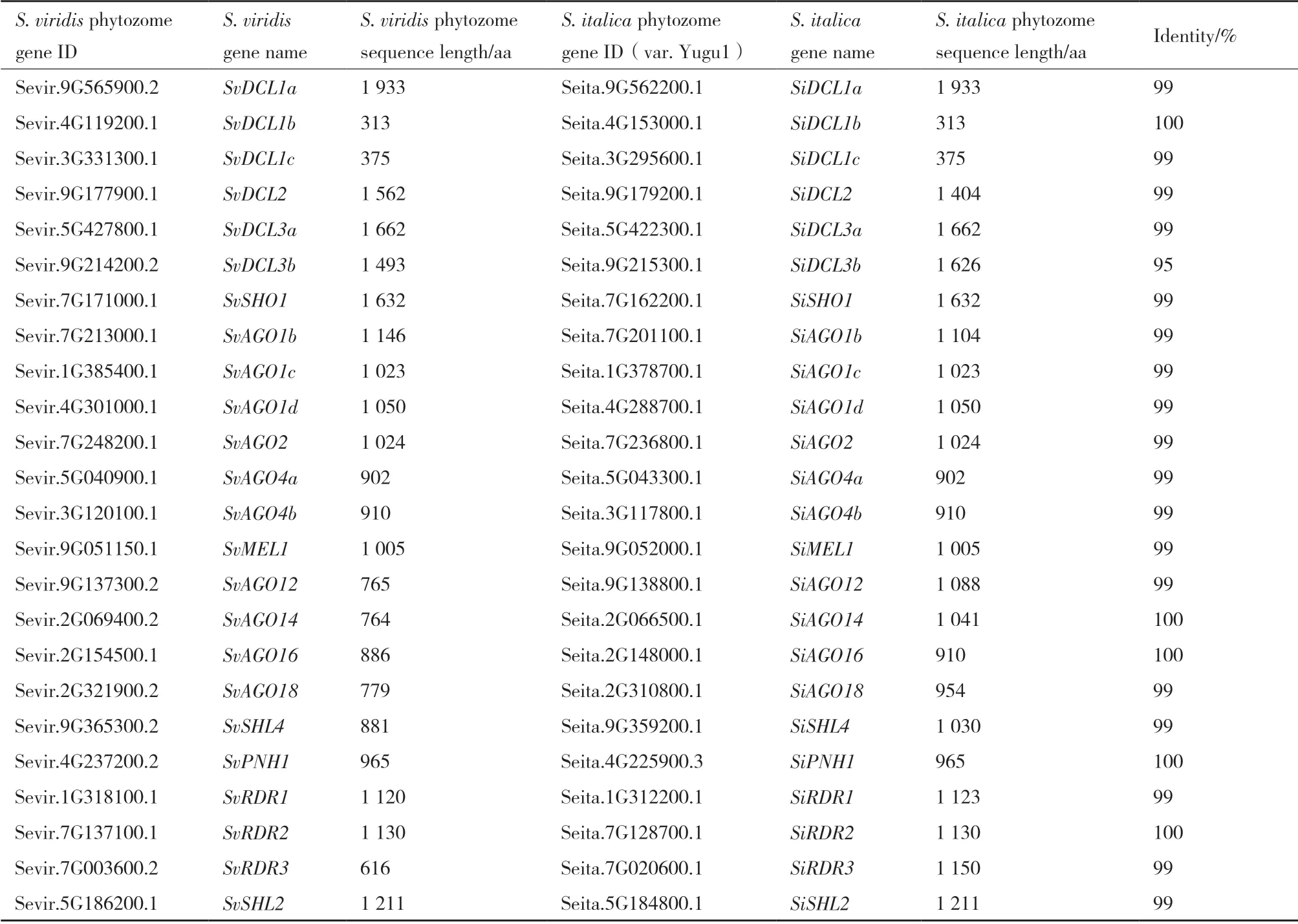
表1 青狗尾草与谷子的基因序列信息Table 1 Gene sequence information of S.viridis and S.italica
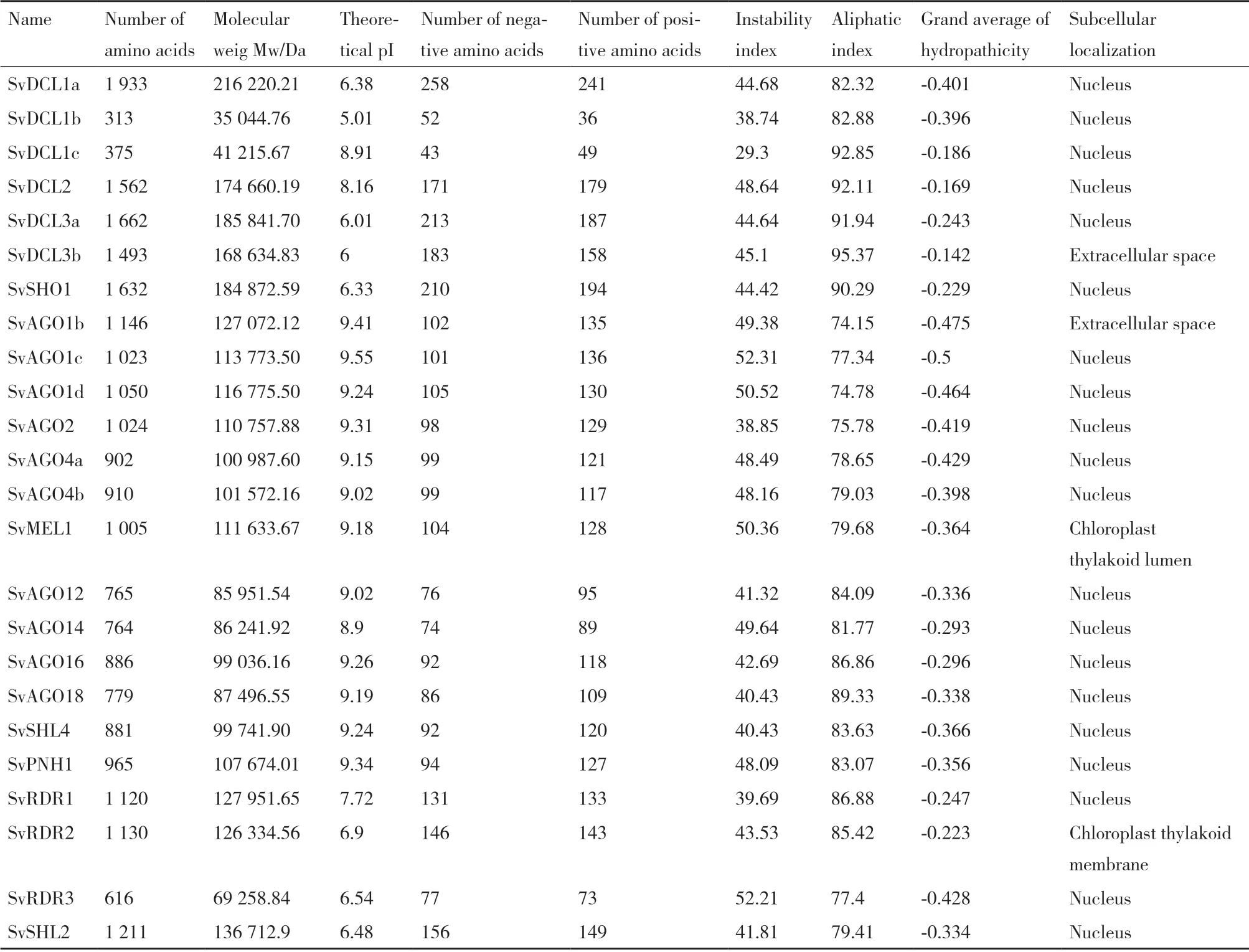
表2 青狗尾草的蛋白质序列信息Table 2 Protein sequence information of S.viridis
2.2 系统进化分析
为了了解青狗尾草RNAi 途径相关基因的蛋白质系统进化关系,利用3 个单子叶植物水稻、谷子和青狗尾草(O.sativa,S.italica,S.viridis)和一个双子叶植物拟南芥(A.thaliana)共94 个RNAi 途径相关基因的蛋白质序列绘制系统进化树(图1)。13 个SvAGO 家族蛋白被分成6 个亚组(AGO1,AGO2/7,AGO4/16,AGO10,AGO12/13/14,AGO18)。其中SvPNH1 在AGO10 亚组;SvMEL 在AGO12/13/14亚组;SvSHL4 在AGO2/7 亚组。AGO18 为禾本科植物特有的一类AGO 蛋白,被单独分为一组。7个SvDCL 家族蛋白被分成4 个亚组(DCL1,DCL2,DCL3,DCL4)。其中SvSHO1 分到DCL4 亚组中。4个SvRDR 家族蛋白被分成4 个亚组(RDR1,RDR2,RDR3,RDR6)。其中SvSHL2 与RDR6 分到一组。作为谷子的野生近缘种,青狗尾草的蛋白质序列与谷子的同源蛋白质亲缘关系最近,因而在同一亚组中它们总是聚在一起的,其次是水稻,外类群双子叶植物拟南芥的蛋白质序列总会单独聚在一起。
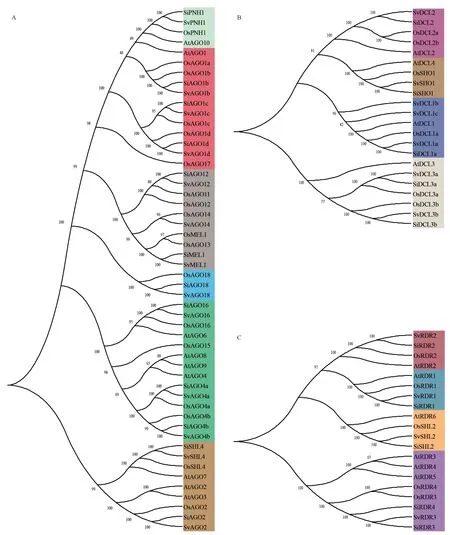
图1 拟南芥、水稻、谷子和青狗尾草的蛋白质系统进化树Fig.1 Protein phylogenetic tree of A.thaliana,O.sativa,S.italica and S.viridis
2.3 基因结构的比较分析
为了初步了解这些基因在青狗尾草和谷子之间的进化关系,对青狗尾草和谷子RNAi 途径相关同源基因的结构进行了比较分析(图2)。研究发现,虽然大部分基因在青狗尾草和谷子中的结构相似度很高,但有两个基因AGO1b和RDR3在青狗尾草和谷子间有明显差异。具体表现为,SiAGO1b比SvAGO1b的第一个外显子内插入了一个6 974 bp 的内含子序列;SiRDR3与SvRDR3相比,SiRDR3的基因结构更为复杂,其在5 176-31 746 bp 多了11 个内含子和外显子,导致编码的蛋白质更长。结构差异必然会导致功能差异,因此,在后续分析中我们对这两对基因进行了深入分析。
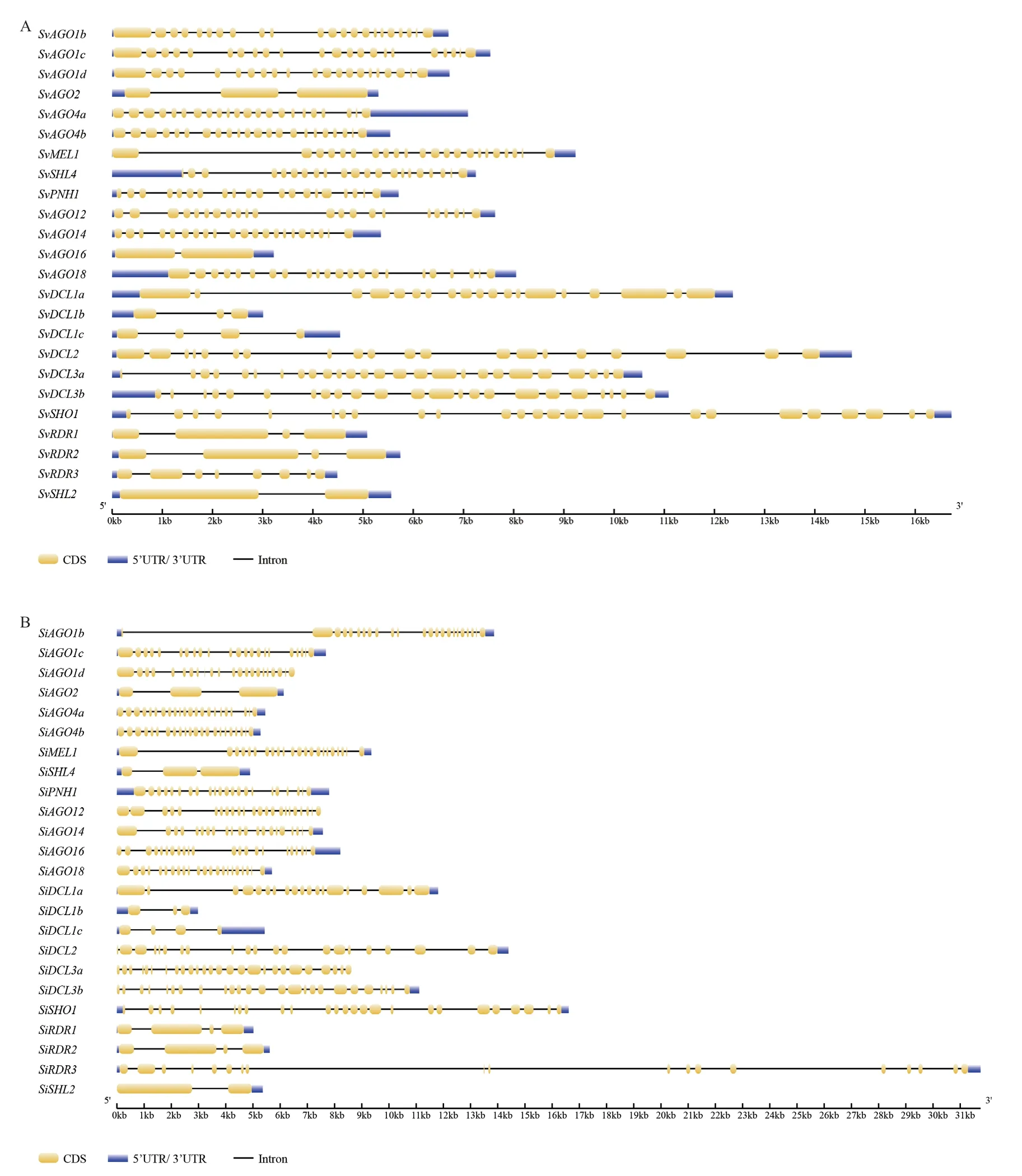
图2 青狗尾草(A)和谷子(B)的基因结构Fig.2 Gene structure of S.viridis(A)and S.italica(B)
2.4 保守结构域分析
本研究进一步分析了青狗尾草AGO、DCL、RDR 蛋白质的保守结构域,并将青狗尾草与课题组前期已报道的谷子同源蛋白的保守结构域进行比较(图3)[15]。研究表明,青狗尾草的AGO 家族中,全部AGO 家族成员都带有ArgoN(PF16486)结构域、ArgoL1(PF08699)结构域、PAZ(PF02170)结构域和Piwi(PF02171)结构域,此外SvAGO1b/SiAGO1b 和SvAGO1d/SiAGO1d 带有Gly-rich_Ago1(PF12764)结构域,个别成员还带有ArgoMid(PF16487)结构域。除了SvSHL4/SiSHL4,其他AGO 成员还带有ArgoL2(PF16488) 结构域。说明AGO 家族蛋白功能较为保守,且各成员之间功能可能相同或相似。与SiAGO12/14/18 相比,SvAGO12/14/18 的AgoN 结构域较短可能不完整,且功能可能受损。而SiAGO12/14/18 具有更加完整的AgoN 结构域,意味着它们具有完整的AGO 蛋白的功能。
在DCL 家族中,DCL1a、DCL3a、DCL3b 和SHO1 具有相似的结构域(图3),包括Helicase_C(PF00271)结构域、Dicer_dimer(PF03368)结构域、PAZ 结构域和Ribonuclease_3/3_3(PF00636)结构域。另外,SvDCL1a/SiDCL1a 和SvDCL3a/SiDCL3a和SiDCL3b 还带有Res Ⅲ(PF04851)结构域,而SvDCL3b 不带有Res Ⅲ结构域。青狗尾草和谷子的DCL1a 和SHO1 带有dsrm(PF00035) 结构域和DND1_DSRM(PF14709) 结构域。但是,青狗尾草和谷子的DCL1b 和DCL1c 蛋白质丧失了大部分DCL 蛋白家族的保守结构域,只包含一个Ribonuclease_3_3 结构域,以及一个dsrm 结构域或DND1_DSRM 结构域。
RDR 家族蛋白都包含一个RdRP(PF05183)结构域(图3)。但相较于其他青狗尾草RDR 蛋白,SvRDR3 的RdRP 结构域较短,表明SvRDR3 的功能可能受损,无法正常结合和复制单链RNA。不同于青狗尾草的是在谷子RDR 蛋白中,SiRDR3 的RdRP结构域更长,可能发挥正常的RDR 蛋白的功能。
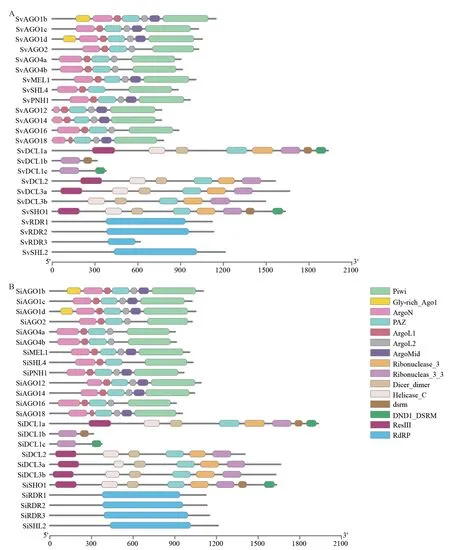
图3 青狗尾草和谷子的蛋白质保守结构域Fig.3 Conserved protein domains of S.viridis and S.italica
2.5 染色体定位
为了探明青狗尾草到谷子驯化过程中,RNAi 途径相关基因在染色体定位是否有变化,我们分别确定了青狗尾草和谷子RNAi 途径相关基因在各自染色体上的位置(图4)。青狗尾草各基因分布在7 条染色体上,与谷子的同源基因在染色体上的位置也基本一致。说明在物种分化后,青狗尾草/谷子的自然选择/驯化过程中,RNAi 途径相关基因的共线性并未发生明显改变。
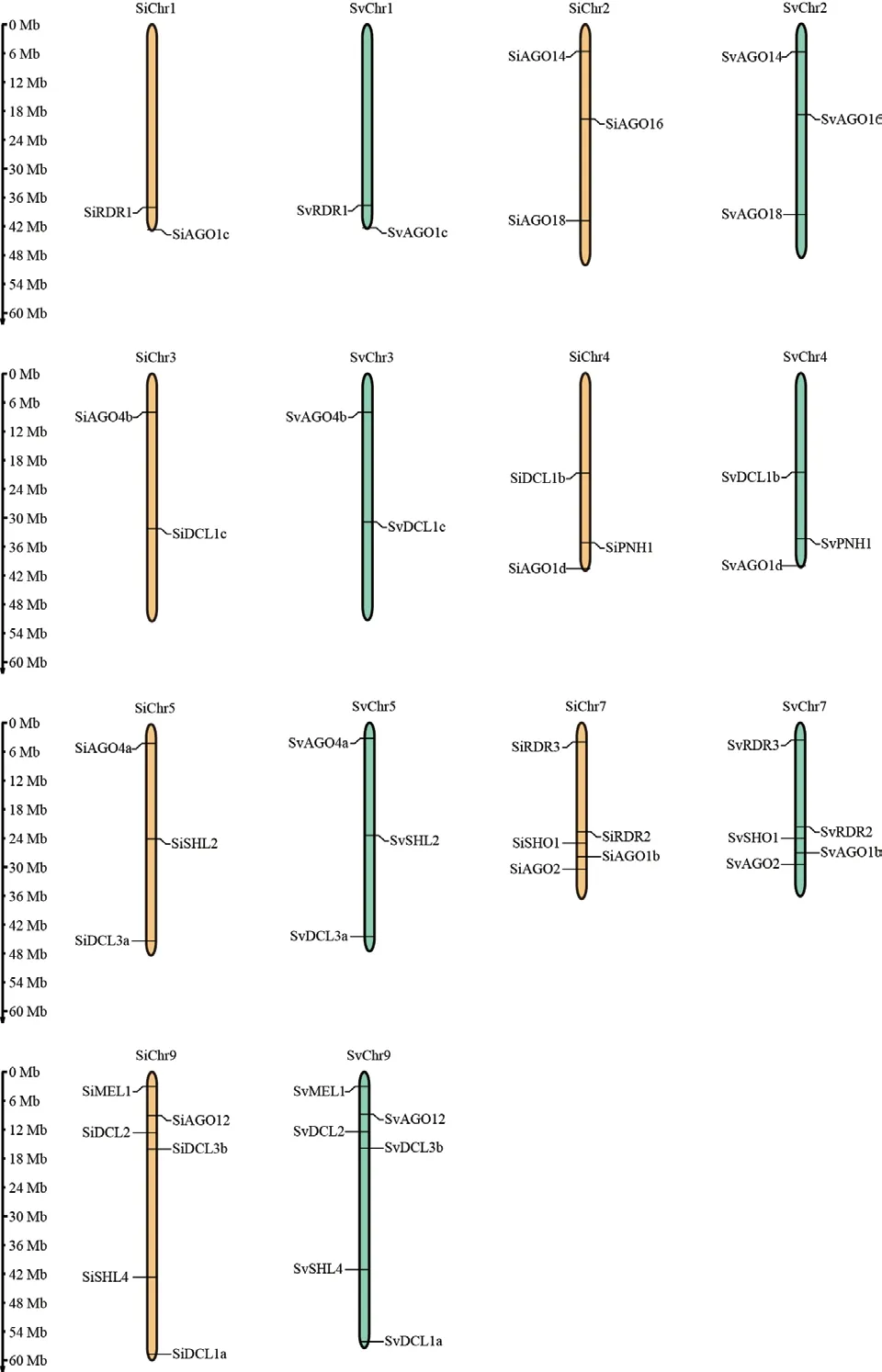
图4 谷子和青狗尾草基因的染色体位置Fig.4 Chromosomal locations of genes of S.italica and S.viridis
2.6 进化选择压力(Ka/Ks)分析
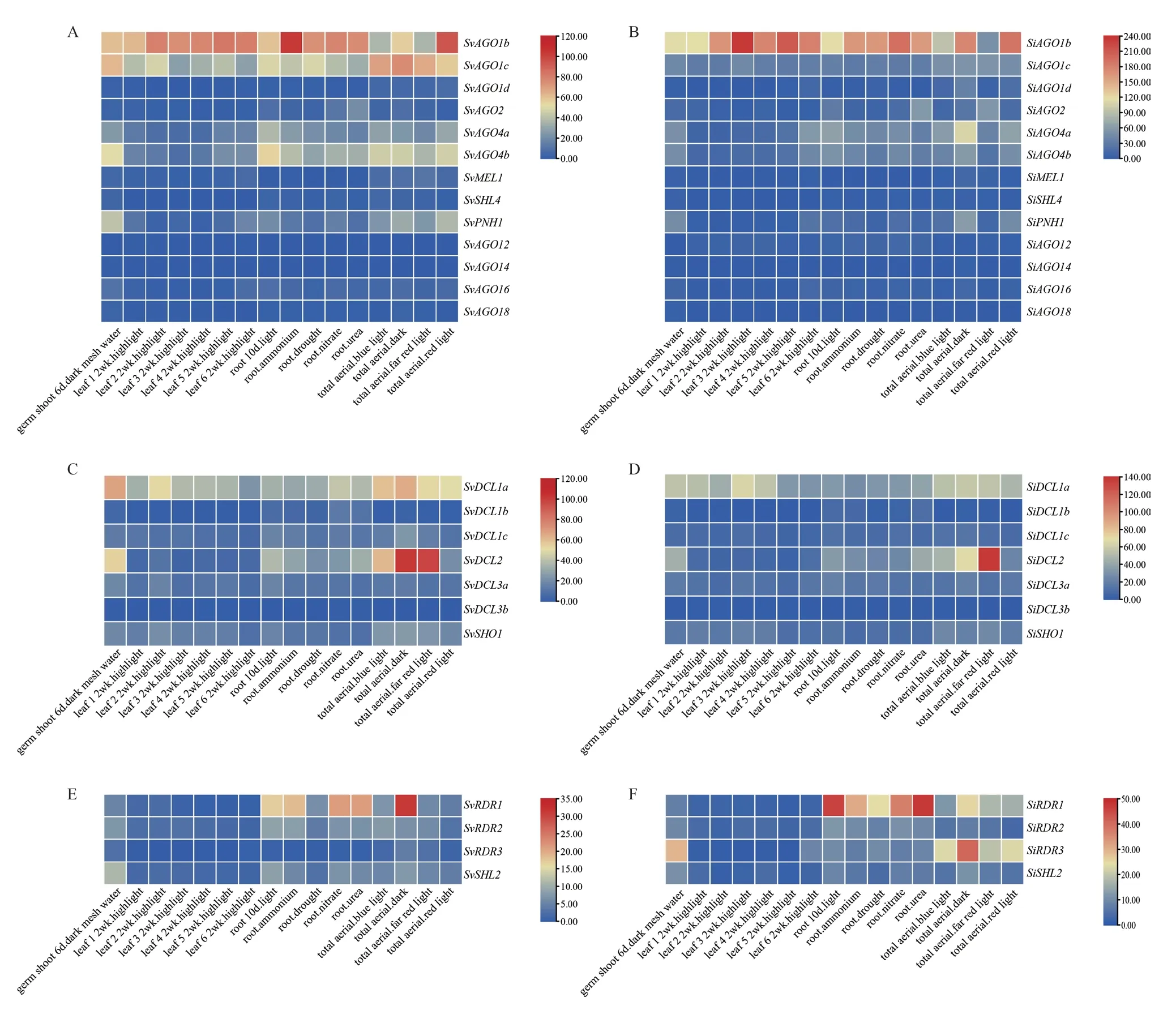
为了了解青狗尾草到谷子的驯化过程是否影响了同源基因的变化,我们计算了青狗尾草和谷子RNAi 途径基因之间的Ka/Ks(表3)。DCL1b、AGO16 和PNH1 的Ka/Ks=1,即Ka=Ks,说明它们在进化上受中性选择。AGO1b 的Ka>Ks,即Ka/Ks>1,说明SvAGO1b到SiAGO1b进化过程中受到正向选择,说明这个基因在青狗尾草与谷子分化过程中具有重要作用,可能对物种的形成有影响。其余基因均为Ka 表3 青狗尾草和谷子的Ka/KsTable 3 Ka/Ks of S.viridis and S.italica 进一步对SvAGO1b和SiAGO1b的序列进行分析,发现在SvAGO1b 蛋白质的N 端存在一个22 nt的信号肽结构,而在SiAGO1b 中则不存在这种结构。说明在青狗尾草/谷子的自然选择/驯化过程中,AGO1b 的N 端的结构可能受到了不同程度的选择压力。 基因结构和功能域的变化往往预示着表达水平的变化。为了验证这一观点,利用转录组数据,分析了青狗尾草和谷子RNAi 途径相关基因在16 种不同生长时期、不同生长条件下各基因的表达量(图5)。在青狗尾草的AGO 家族中,除了SvAGO1b/c、SvAGO4b和SvPNH1,其余多数AGO 成员表达量则较低。SvAGO1b的表达水平普遍高于SvAGO1c,而SvAGO1d则几乎不表达。具体表现为,SvAGO1b在6 天龄的芽、叶片、不同处理条件下的根和红光处理的地上部分中均有着较高的表达水平(TPM>40);SvAGO1c在6 天龄的芽、蓝光、黑暗和远红光处理的地上组织中表达水平较高(TPM>40)。表明SvAGO1b在多数生长发育阶段可能发挥着重要作用,尤其在叶片和不同处理下的根中;SvAGO1c响应光变化,在功能上可能与SvAGO1b有着一定差异;SvAGO1d则可能不发挥作用或在特定时期发挥功能。SvAGO4b则在6 天龄的芽、10 天龄的根、蓝光、黑暗和远红光处理的地上组织中表达量较高(TPM>40),且SvAGO4b的表达水平均略高于SvAGO4a,不同于前者的是SvAGO4a与SvAGO4b在不同处理条件下的表达趋势基本一致。 图5 青狗尾草和谷子RNAi 相关基因组织特异性表达分析Fig.5 Tissue-specific expression analysis of RNAi-related genes in S.viridis and S.italica 青狗尾草的DCL 家族中,SvDCL1a在光处理下有着较高的表达水平,并在暗处理下的地上组织中表达量最高(TPM=64.46)。表明在地上组织中,DCL 家族的SvDCL1a可能发挥主要作用,其他成员则可能存在着时空特异性表达。此外,相比于不同处理,SvDCL2仅在暗处理和远红光处理下的地上部分中有较高的表达水平(TPM=101.89),表明SvDCL2可能是潜在的光响应基因。其他DCL 成员则表达量较低或几乎不表达(TPM<20)。 青狗尾草的RDR 家族中,整体表达水平也较低,但相比于不同处理,SvRDR1在暗处理的地上部分中表达量相对较高(TPM=31.34)。此外,SvRDR2在不同组织、不同处理条件下的表达量则略高于SvRDR3。结果表明,青狗尾草RDR 家族成员的表达模式基本一致,但在叶中几乎不表达。不同于青狗尾草,SiRDR3在6 天龄的芽和不同光处理的地上组织中表达量较高。说明青狗尾草RDR 家族蛋白功能类似,SvRDR1可能在根中和暗处理的地上部分发挥着主要作用,且SvRDR3和SiRDR3在功能上可能有分化。 我们通过比对SvAGO1b和SiAGO1b的表达量,分析N 端的信号肽结构的差异是否对AGO1b 的转录水平产生了影响。发现在多数时期和处理下,SiAGO1b和SvAGO1b的表达模式相似。说明N 端的信号肽结构的有无几乎不影响AGO1b 的跨膜能力及其功能。 可以看出,除RDR3 外的青狗尾草和谷子的RNAi 途径相关基因在不同生长发育时期下各基因的表达模式基本一致,且基因结构和蛋白质保守结构域存在差异的同源基因间表达水平也没有发生明显的变化,说明在青狗尾草和谷子中,RNAi 途径相关基因行驶的功能相对重要和稳定,自然选择和驯化过程并没有使其结构和功能发生大的变化,因而其表达水平也相对稳定。 驯化是人类最伟大的发明之一,驯化可以使野生物种在强大的人工选择压力下获得对人类有利的优良性状变成栽培/家养品种[21]。人类对农作物的驯化历史悠久,例如9 000年前野生水稻被驯化为栽培稻[22],8 000年前野生棉被驯化成为栽培棉[23]。又有大刍草驯化成玉米[24],野生大豆驯化成栽培大豆[25],莴苣驯化成生菜等[26]。自青狗尾草和谷子基因组信息公布以来,越来越多的研究集中于青狗尾草到谷子的驯化过程的分子机制。因此,研究青狗尾草基因组中表观遗传修饰相关基因家族的序列和结构特征及其表达水平,并与谷子同源基因进行进化比较分析,将有助于加深理解表观遗传修饰在青狗尾草和谷子驯化过程中的功能和作用。 表观遗传学中的 RNAi 途径在DNA 甲基化[2,27]、转录后沉默[13,28]、抵抗病原微生物入侵[29-30]和调控植物生长发育[31-33]等方面发挥重要作用。本研究利用同源比对和保守结构域探寻方法在青狗尾草全基因组水平鉴定到13 个AGO、7 个DCL 和4 个RDR 蛋白。它们与谷子同源蛋白的相似度在95%-100%之间,说明在驯化或进化过程中青狗尾草和谷子的这些同源基因间是保守和稳定的。进一步对其基因结构和蛋白质结构域分析发现,在青狗尾草中SvAGO12/14/18、SvDCL3b 和SvRDR3 的蛋白质保守结构与谷子相比有差异。Res Ⅲ和RdRP 结构域分别是DCL 家族和RDR 家族中极其重要的结构域,这些结构域的变化可能会导致蛋白质功能的改变。此外,青狗尾草和谷子的DCL1b 和DCL1c 只包含极少的保守结构域,而在水稻中,与OsDCL1a相比,OsDCL1b和OsDCL1c丢失了大部分外显子及其对应的结构域,被认为是假基因不发挥功能[14],说明DCL1b 和DCL1c 的结构域或功能丧失可能是在禾本科物种中普遍存在的现象。转录组结果显示,SvAGO1b在叶片和不同处理下的根中发挥着重要作用,其中TPM 值最高可达103.75;SvAGO1c在不同光处理下的地上组织样品中表达量较高,SvDCL1a在叶片中和光处理下地上组织中表达量较高,且SvRDR1在多数处理下的根组织中和暗处理的地上组织中表达量相对较高,而各家族的其他成员表达量较低或几乎不表达,暗示各家族仅需要个别成员表达以发挥主要作用,其他成员则低表达作为补充,或与时空性表达有关。我们发现,SvAGO1b和SvRDR1在根中表达量相对较高,暗示其在青狗尾草应对土壤微生物是可能发挥重要作用。此外,我们的研究结果还表明,除RDR3 外各同源基因在青狗尾草和谷子中表达模式相似,说明谷子在驯化过程中,其各同源基因在功能上与青狗尾草相比未发生明显分歧。 本研究利用比较基因组学在青狗尾草中鉴定到13 个AGO 蛋白、7 个DCL 蛋白和4 个RDR 蛋白。SvDCL3b 和SvRDR3 的蛋白质结构域有缺失。转录组分析发现,SvAGO1b、SvDCL1a和SvRDR1在各家族中表达量较高。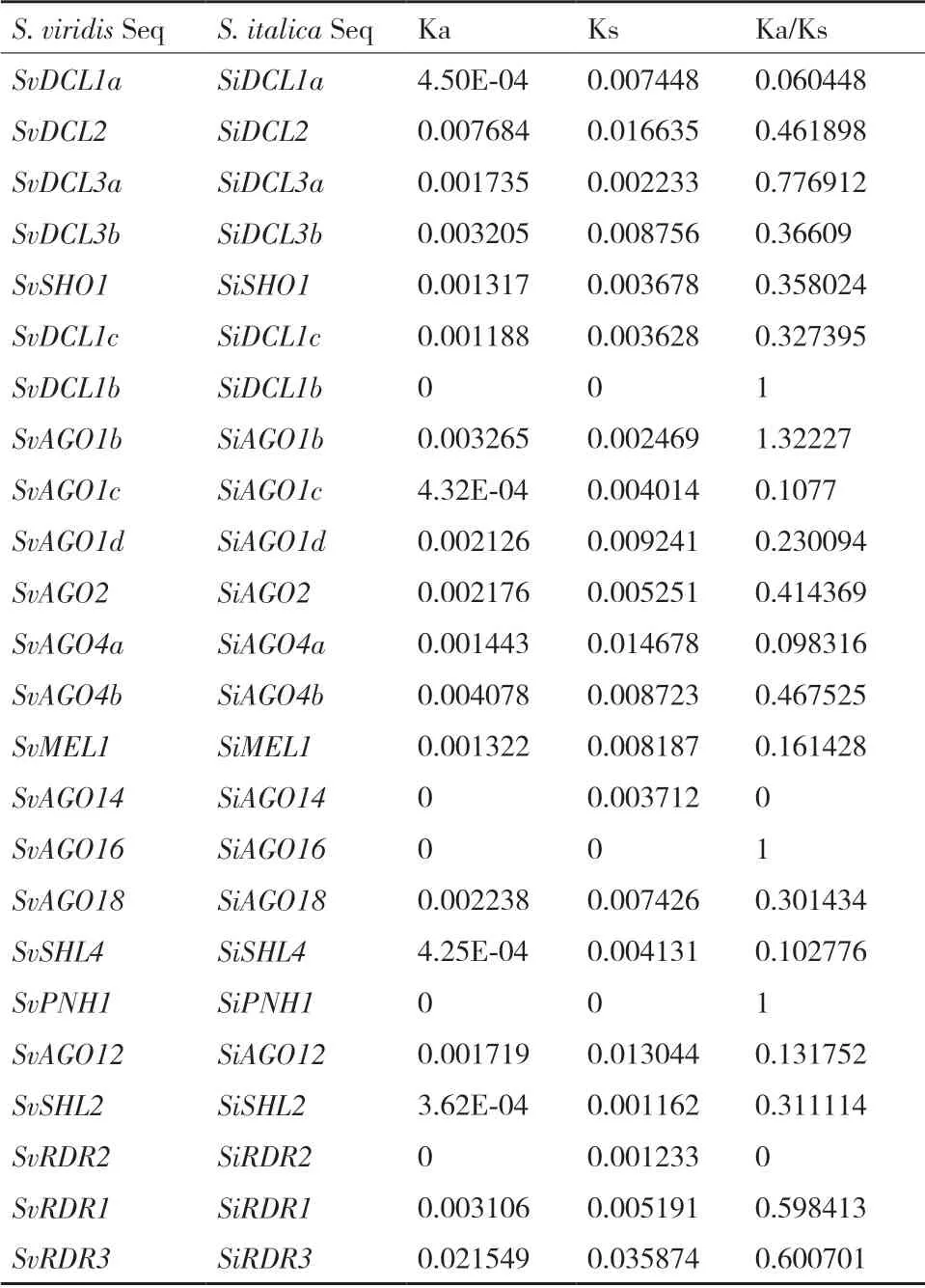
2.7 基因表达分析
3 讨论
4 结论
