基于多尺度特征与通道特征融合的脑肿瘤良恶性分类模型
2023-01-07姜林奇宁春玉余海涛
姜林奇,宁春玉,余海涛
(长春理工大学 生命科学技术学院, 吉林 长春 130022)
1 引 言
根据2018年全球癌症统计报告可知,大脑及神经系统癌变发生率约为2.9人/(年·10万人),约占全身肿瘤的1.6%,死亡率高达2.5%[1]。根据病因可以将大脑及神经系统癌变分为原发性和继发性。其中,胶质瘤(glioma)作为最常见的原发性脑肿瘤被广泛关注。根据磁共振肿瘤成像所呈现的特点,可将其恶化的程度分为低级别胶质瘤(Low Grade Gliomas, LGG)和高级别胶质瘤(High Grade Gliomas, HGG)[2]。如果医生不能准确地对肿瘤的恶化程度进行分类,随着时间的推移良性肿瘤很有可能会恶化成恶性肿瘤。因此,肿瘤的良恶性分类尤其重要[3]。
基于传统机器学习的图像分类方法通过提取图像的纹理信息,比如形状特征、颜色等,再利用支持向量机(Support Vector Machines, SVM)或其他分类器实现分类[4-6]。 Raju等人[7]采用贝叶斯模糊聚类方法进行脑肿瘤分割,并利用HCS(Harmony-Crow Search)优化算法来训练multi-SVNN分类器的权重,计算图像特征,得到的分类精度为93%。Narmatha等人[8]提出了将模糊算法与Brain-storm优化算法相结合的Fuzzy Brain-storm optimization 算法用于脑肿瘤图像的分割与分类。该方法有效地缩短了图像的分割时间,但最终的分类准确率仅为93.85%。
传统的图像分类方法在图像特征的选择上更依赖于操作者的先验知识,工作量大并且过程较复杂。近几年,深度学习(Deep Learning,DL)作为一种新兴技术被广泛应用于图像分割、图像分类、图像识别等任务中[9-12]。面对图像特征复杂的医学图像,学者们提出将DL引进到医学影像领域当中[13-14]。
目前,脑肿瘤分类系统可分为非端到端式和端到端式。非端到端的分类系统是指由多个独立部分组成的系统,卷积神经网络(Convolutional Neural Network,CNN)担任其中一个或多个部分。Sharif等人[15]提出一种基于深度学习的方法来实现肿瘤的分割与分类。该方法使用预训练后的Inception V3网络对分割结果图进行特征提取,并将分别由传统方法和深度学习提取的特征进行拼接,通过粒子群优化算法(Particle Swarm Optimization,PSO)进行特征向量的优化,最后,通过Softmax分类器进行分类,得到了96.90%的准确率。Khan等人[16]提出一种基于极限学习机(Extreme Learning Machine, ELM)的自动分类方法。该方法利用预训练过的VGG16提取图像特征,然后通过ELM对融合的鲁棒特征进行分类,有效减少了特征选取与融合的时间。Rehman等人[17]提出将3D CNN模型用于脑肿瘤的检测,利用预训练的VGG19网络用于特征提取,利用前反馈神经网络(Feed-forward Neural Network,FNN)选取最佳特征用于分类。该方法取得了较高的准确率,但肿瘤检测过程耗费时间较长。以上非端到端的分类系统对脑部肿瘤的分类准确率都有所提升,但此类系统大多基于图像分割,分类过程复杂,故会引入较多干扰因素,不可避免地会对分类结果造成影响。
端到端的分类系统是指仅通过卷积神经网络实现对肿瘤图像的分类。Seetha等人[18]针对脑肿瘤区域结构异化的问题,提出利用小卷积核提取肿瘤区域的细微特征,实现对肿瘤良恶性分类,最终得到了97.50%的准确率。赵尚义等人[19]提出了3D U-Net模型的分类算法。该方法在原始UNet 网络中引入了特征融合层,将浅层特征与深层特征相融合,实现了基于分割网络U-Net的分类系统。但该方法没有对浅层特征与深层特征的特征通道权重进行重新分配,过分表达冗余特征,导致最终的分类准确率仅达到91.67%。端到端式的分类系统基于设计好的网络结构,利用深度学习自动提取图像特征,降低了分类过程复杂性。但是上述用于脑肿瘤的分类方法没有充分利用图像的全局、局部显著特征以及特征图的通道特性,识别准确率还有待进一步提高。
针对上述问题,本文提出一种基于多尺度特征与通道特征融合的脑肿瘤良恶性自动分类方法(Improved MDCA-ResNeXt),利用ResNeXt网络结构的分组卷积策略,在增加网络宽度的同时提升了网络提取特征信息的能力,并通过多尺度特征提取模块融合全局特征与局部显著特征,通过通道注意力机制模块提升网络对病灶区域的关注度,减少冗余信息对分类结果造成的影响,最终达到提高肿瘤良恶性分类精度的目的。
2 基于Improved MDCA-ResNeXt的脑肿瘤良恶性分类模型
2.1 MDCA-ResNeXt网络
在对图像特征信息复杂、数据特征维度高的医学图像进行分类时,不仅要考虑网络提取特征信息的能力,还需要考虑训练过程的计算复杂度等问题,经过多番考虑,本文选用ResNeXt[20]网络作为脑肿瘤图像分类任务的主干网络,对其进行改进,以提高网络分类能力、降低参数冗余度、减少计算时间。首先,将基于空洞卷积的多尺度特征提取模块(Multi-scale Feature Extraction Module based on Dilated Convolution, MD)代替一般的卷积层,扩大第一层卷积层的感受野,同时保留HGG图像中增强区域的局部显著特征,将全局特征与局部显著特征融合;其次,添加通道注意力机制模块(Channel Attention Module,CA),引入特征通道信息,对特征通道重新分配,减少特征冗余引起的影响,提高肿瘤区域在整幅图像中的关注度;最后,简化ResNeXt网络,减少ResNeXt结构个数,避免因网络过深导致的过拟合现象。图1为本文提出的MDCA-ResNeXt网络结构。
2.1.1 ResNeXt结构
ResNeXt结构采用Inception结构[21]中的拆分-转换-合并的思想,沿用ResNet[22]的残差结构,构造重复的多分组卷积层。ResNeXt结构的本质是分组卷积,它通过变量基数C来控制组的数量,从而达到两种策略的平衡。图2为C=32的Res-NeXt结构。图中“弯曲的箭头”表示残差结构的恒等映射,⊕表示逐个像素点相加。
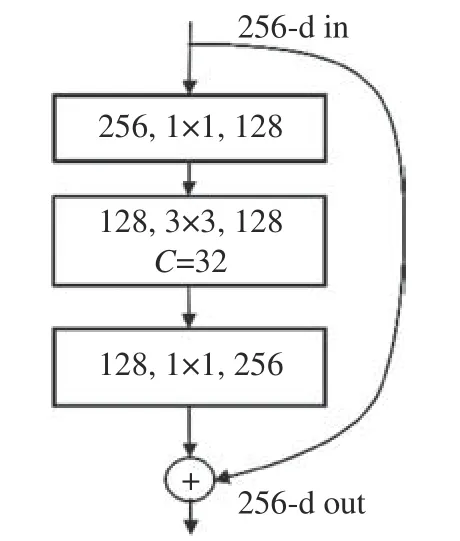
图2 C =32的ResNeXt结构[20]Fig. 2 ResNeXt structure with C =32[20]
ResNeXt结构的运算过程可用数学公式表达为:
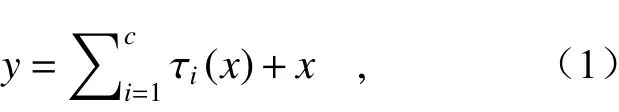
其中τ表 示 1×1→3×3→1×1一系列的卷积操作。
2.1.2 多尺度特征提取模块
空洞卷积(dilated convolution)[23]相对于正常卷积模板多一个膨胀率d,它表示在卷积核中插入权重为0的行和列的数量。图3为不同膨胀率的空洞卷积。当d=1时,卷积模板的感受野与3×3的 卷积模板相同;当d=2时,卷积模板的感受野与5 ×5的 卷积模板相同;当d=3时,卷积模板的感受野与7 ×7的卷积模板相同。
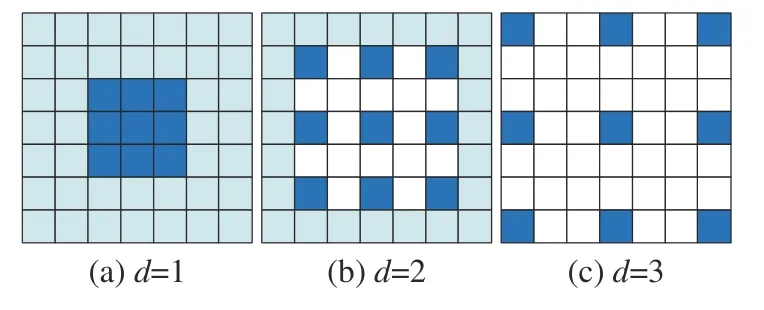
图3 不同膨胀率的空洞卷积Fig. 3 Dilated convolution results with different dilation rates
图4为本文提出的MD模块,该模块的第一层卷积层包括了1 ×1的普通卷积模板以及卷积尺寸为 3×3、 膨胀率d分别为1 ,2,3的空洞卷积模板。第二层的卷积层是由1 ×1卷积模板组成,其作用是将上一层的输出特征图拼接在一起,得到同时包含全局与局部显著特征信息的特征图。

图4 MD模块Fig. 4 MD module
2.1.3 通道注意力机制模块
图5为注意力机制[24]中的通道注意力机制模块(Channel Attention Module,CA),该模块通过建立特征图之间的通道关系,生成通道注意力图,对特征通道重新分配权重,提高网络对输入图像中重要信息的关注度,降低网络对冗余信息的关注度。其计算过程如下:

图5 CA模块Fig. 5 CA module
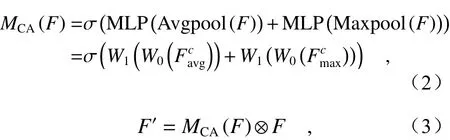
其中, Avgpool 和 Maxpool 分 别表示对输入特征F进行全局平均池化操作和全局最大池化操作,MLP表示多层感知器(Multi-layer Perceptron, MLP),W0和W1为MLP层之间的共享权重,其维数分别为:W0∈RC/r×C,W1∈RC×C/r,r表示降维系数,σ表示sigmoid函数,⊗表示像素点相乘。
2.2 基于MDCA-ResNeXt网络的优化策略
在设计分类模型过程中,网络结构对图像特征的提取起着关键作用,但是在训练过程中需要设置的超参数也会影响网络的学习能力。此外,提高网络的学习能力是一个比较漫长、复杂的过程。在脑肿瘤的前期分类任务中,本文作者提出了学习率的线性衰减策略、图像的标签平滑策略以及基于医学图像的迁移学习策略3种优化策略的组合[25]。为了提高网络的学习能力,减少训练的迭代次数,本文采用相同的优化策略对MDCAResNeXt网络进行优化,经过优化的网络简记为Improved MDCA-ResNeXt网络。预训练过程中网络的训练迭代次数为50,批次大小为8,优化算法为带有动量随机梯度下降优化算法(Stochastic Gradient Descent with Momentum, SGDM),初始学习率为0.01,学习率衰减迭代次数分别为30和40,标签平滑系数为0.01,采用Cheng等人[26]提供的CE-MRI数据库作为预训练数据集。该数据库包含了T1C模态下233名病人的3 064张脑肿瘤MRI切片图像,其中包含了1 426张胶质瘤图像、930张脑膜瘤图像和708张垂体瘤图像。
3 实验设计
3.1 实验数据
3.1.1 BraTS数据库
本文使用的BraTS2017和BraTS2019数据库分别为2017年和2019年举办的多模态脑肿瘤分割挑战赛提供的开源数据库。BraTS2017数据库中包含210例HGG和75例LGG,BraTS2019数据库包含259例HGG和76例LGG。数据库中每个病例都包括FLAIR、T2、T1和T1C 4种模态的MRI序列,每个序列包含155张切片图像,图像分辨率为240 pixel×240 pixel。图6、图7为4种模态下的HGG和LGG肿瘤图像。通过对比不同模态MRI图像可知,添加了造影剂的T1C模态能够更加凸显病灶区域的特征,因此本文选用该模态下的图像作为原始输入。
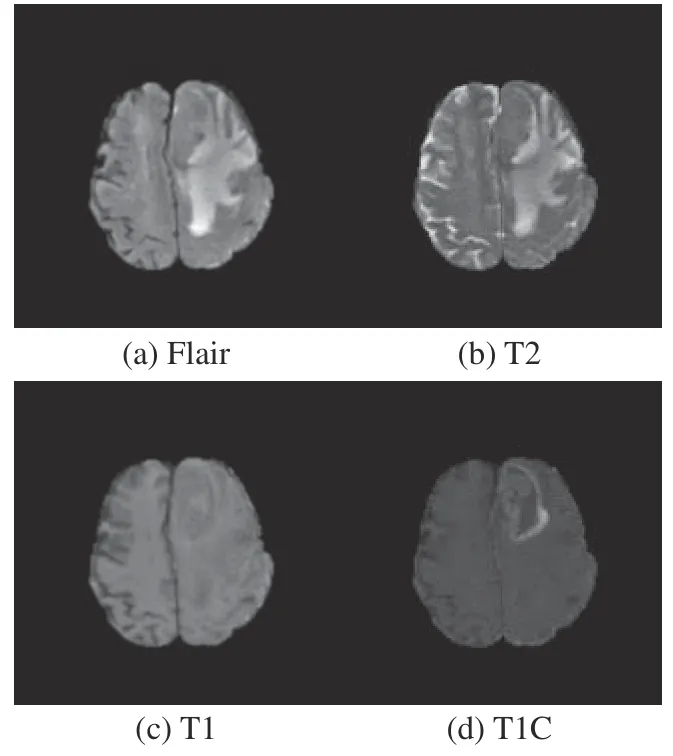
图6 4种模态下的HGG图像Fig. 6 HGG images in four modalities
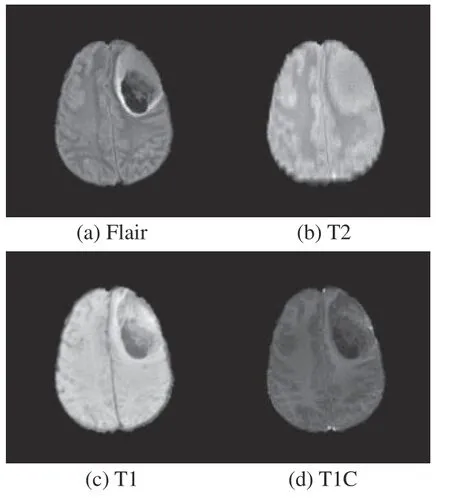
图7 4种模态下的LGG图像Fig. 7 LGG images in four modalities
3.1.2 数据预处理
图8为去噪前后对比图。图8(a)中矩形虚线框内图像中含有一些不均匀的信号,这是由于设备老化等问题造成的灰度变化不均。这种现象称为偏置场效应。如果在图像预处理阶段不对其进行处理,很容易造成网络对图像的错误判断,所以本文使用优化后的N3(Nonparametric nonuniform intensity normalization)[27]算法——N4ITK算法[28]对原始图像数据进行偏置场校正。图8(b)是N4ITK算法校正后的MRI图像,可见虚线框内的亮度变得比较均匀。

图8 去除噪声前后对比图Fig. 8 Comparison before and after preprocessing
在分类任务中,不同类别下的图像数量分布不均可能会造成过拟合,因此需要对LGG样本进行数据扩充。本文利用随机翻转、旋转等操作对LGG样本进行扩充。并通过对训练集中的图像进行随机旋转、随机擦除[29]等数据增强操作来增加数据集中来自同一病例的切片图像的差异性,从而提高网络分辨能力的鲁棒性。
3.1.3 实验数据集
为了验证所提出的分类模型对胶质瘤图像的分类能力,本文将数据增强后的BraTS2017和BraTS2019数据集分别划分成5个图像数量相等的子集,进行五折交叉验证实验。表1所示为实验数据集的分布情况。
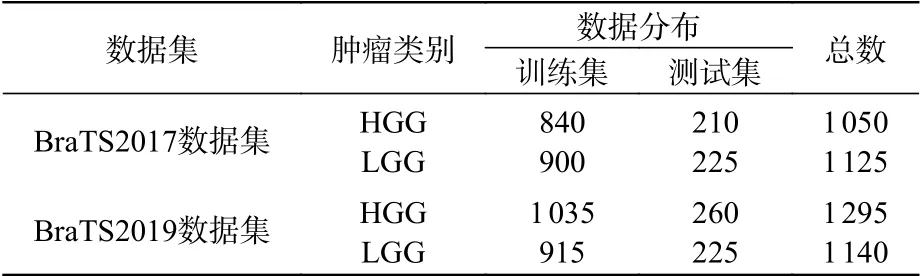
表1 实验数据集分布Tab. 1 Distribution of experimental datasets
3.2 实验环境及参数设置
本文基于Pytorch深度学习模型框架,采用Python作为编程语言,在操作系统为Windows 10、GPU为NVIDIA Tesla K40m的实验平台搭建了基于Improved MDCA-ResNeXt网络模型的脑肿瘤图像良恶性分类框架。网络训练过程中的训练迭代次数为30,批次大小为8,优化算法为SGDM,初始学习率为0.01,学习率衰减迭代次数为20,标签平滑系数为0.01,网络初始化参数为迁移学习得到的。
3.3 评价指标
本文采用准确率(Accuracy)、灵敏度(Sensitivity)、特异度(Specificity)、阳性预测值(Positive Predictive Value)以及阴性预测值(Negative Predictive Value)对分类结果进行性能评价。上述指标分别记为ACC、SEN、SPE、PPV及NPV,计算公式如式(4)~式(8)所示。

式中α 和β分别表示真实标签与预测标签相同的HGG和LGG样本个数,δ和 γ分别表示真实标签与预测标签不相同的HGG和LGG样本个数。
3.4 实验结果与分析
3.4.1 网络结构消融实验
本小节将仅含有MD模块的MD-ResNeXt网络和仅含有CA模块的CA-ResNeXt网络与MDCA-ResNeXt网络进行对比,以验证MDCAResNeXt的有效性。为了保证实验结果的科学性,所有网络的初始化参数均由相同的随机数种子生成,实验数据集为BraTS2017。
图9(彩图见期刊电子版)为MD-ResNeXt、CA-ResNeXt和MDCA-ResNeXt 3个网络在5个交叉验证集中获得的分类结果评价图,图中数字为评价标准的平均值,线段为标准差。从图9可以看出,MDCA-ResNeXt对HGG样本和LGG样本的分辨能力,相较于MD-ResNeXt和CA-Res-NeXt都有提升,并且标准差相对较小,说明MDCA-ResNeXt在不同验证集的表现都相对稳定,鲁棒性强。
为探究MDCA-ResNeXt工作机制的有效性,对特征图进行了可视化处理,图10和图11分别为HGG样本、LGG样本的原始图像和特征可视化图。在图10(a)中,肿瘤区域位于右侧大脑颞部,并且核心肿瘤的外围有增强区域包裹,在图像中表现为高频信号。图10(b)~10(e)分别表示4种网络结构在同一层输出的特征图。对比图10(b)和图10(c)可以看出,MD-ResNeXt相较于Res-NeXt更好地保留了脑部组织结构,对肿瘤的增强区域有积极的“响应”;对比图10(b)和图10(d)可以看出,CA-ResNeXt相较于ResNeXt更好地突出了肿瘤的增强区域,减少了冗余的特征信息;对比图10(c)和图10(e)可以看出,MDCA-Res-NeXt只对肿瘤的增强区域有积极的“响应”,相较于MD-ResNeXt减少了对冗余信息的“响应”,充分说明了在网络结构中添加CA模块可以有效地避免冗余特征信息对分类结果造成的影响;对比图10(d)和图10(e)可以看出,相较CA-ResNeXt,添加了MD模块的MDCA-ResNeXt能够较好地体现肿瘤区域的图像特征。

图10 HGG的原始图像和特征可视化图Fig. 10 Original image and feature visualizations of HGG

图11 LGG的原始图像和特征可视化图Fig. 11 Original image and feature visualization of LGG
在图11(a)中,肿瘤区域位于左侧大脑颞前部,而良性的胶质瘤在图像中呈现低频信号,并且肿瘤外围没有出现增强区域。对比图11(b)、图11(c)、图11(d)和图11(e)可以看出,MDCAResNeXt网络能够更有效地保存脑肿瘤图像的特征信息,能够更好地区分HGG样本和LGG样本。
3.4.2 经典网络对比实验
为验证本文提出的MDCA-ResNeXt网络以及优化后的Improved MDCA-ResNeXt网络在脑肿瘤良恶性分类任务中的优势,本文设置了两组对比实验,一组实验是将ResNet网络、Res-NeXt网络和SENet网络[30]应用在BraTS2017和BraTS2019数据集中,把3种经典网络的分类结果与MDCA-ResNeXt网络进行比较;另一组是将优化后的3种经典网络与Improved MDCA-Res-NeXt网络的分类结果进行对比。
根据表2和表3可知,MDCA-ResNeXt的分类结果与其他3个经典网络相比都是最高的。对比不同网络的SPE值和NPV值可以看出,MDCA-ResNeXt提高了对LGG样本的分类能力。从表4和表5可以看出,本文提出的Improved MDCA-ResNeXt的各项分类评价标准同样都是最高的。

表2 优化前BraTS2017数据集的分类结果评价表Tab. 2 Evaluation of classification results on BraTS2017 before optimization

表3 优化前BraTS2019数据集的分类结果评价表Tab. 3 Evaluation of classification results on BraTS2019 before optimization

表4 优化后BraTS2017数据集的分类结果评价表Tab. 4 Evaluation of classification results on BraTS2017 after optimization

表5 优化后BraTS2019数据集的分类结果评价表Tab. 5 Evaluation of classification results on BraTS2019 after optimization
根据以上4个分类结果评价表可以得出以下结论:首先,本文提出的MDCA-ResNeXt网络是以ResNeXt为主干网络进行的改进,改进后的网络能够有效区分良恶性脑肿瘤,并且提高了网络在不同分类任务的鲁棒性;其次,本文采取3种优化策略的组合可以提高网络的分类准确率和泛化能力。
3.4.3 其他先进方法的对比结果
为充分说明本文提出的基于Improved MDCA-ResNeXt网络的脑肿瘤良恶性分类模型的性能,将本文方法与其他先进方法进行对比,对比结果如表6所示。
表6中,文献[7]和文献[8]为传统机器学习的分类方式,文献[19]、[15]、[16]和[17]为深度学习的分类方式。上述对比方法中大多是基于分割结果图进行肿瘤分类的,而图像分割是基于图像像素点进行的0-1分类过程,对分割算法和分割模型的性能要求更高,并且分割结果会直接影响分类结果。除此之外,先分割后分类的方法,增加了分类过程中的不可控因素。基于此,本文提出的Improved MDCA-ResNeXt分类算法在肿瘤良恶性分类任务中更具有优势,分类准确率更高。

表6 先进方法分类结果对比表Tab. 6 Comparison of classification results of advanced methods
4 结 论
本文提出一种基于Improved MDCA-ResNeXt网络的脑肿瘤良恶性分类方法,不仅能将原始图像中细微的局部特征和全局特征相融合,保留HGG的肿瘤增强区的特征信息,还能将特征图的特征通道重新排列,提高网络对肿瘤区域的关注度,降低网络对冗余特征的关注度;采用学习率的线性衰减策略、图像标签平滑策略以及基于医学图像的迁移学习策略的组合优化策略可提高网络的学习能力和泛化性能。在BraTS2017和BraTS2019数据集上的实验结果表明,本文方法相较经典网络和其他先进方法的分类准确率、鲁棒性有所增强,其中准确率分别达到 98.11%和98.72%。在未来的研究工作中需要进一步优化网络结构,可以考虑引入新的技术,减少具有相似性的冗余特征对分类结果的影响和计算消耗,进一步提升分类准确率、降低运算时间。