基于nrDNA和cpDNA序列的甘肃省4种肉苁蓉种质资源分子鉴定
2022-12-28康舒淇徐丽张明惠陈博晋玲朱田田
康舒淇,徐丽,张明惠,陈博,晋玲,2,3,朱田田,2,3
1.甘肃中医药大学,甘肃 兰州 730000;2.西北中藏药省部共建协同创新中心,甘肃 兰州 730000;3.甘肃省珍稀中药资源评价与保护利用工程研究中心,甘肃 兰州 730000
肉苁蓉为列当科肉苁蓉属植物肉苁蓉Cistanche deserticolaY.C.Ma和管花肉苁蓉Cistanche.tubulosa(Schenk)Wight的干燥带鳞叶肉质茎[1]。全球约有22种肉苁蓉属植物,多分布于北半球欧亚温暖的沙漠、荒漠等干燥地带。据《中国植物志》记载,中国的肉苁蓉属植物主要有5种,包括肉苁蓉C.deserticolaY.C.Ma、管花肉苁蓉C.tubulosa(Schenk)Wight、盐生肉苁蓉Cistanche salsa(C.A.Mey.)G.Beck、沙苁蓉Cistanche sinensisG.Beck及兰州肉苁蓉Cistanche.lanzhouensisZ.Y.Zhang,主要分布在新疆、内蒙古、甘肃等西北部地区[2]。肉苁蓉不仅具有较高的药用价值,因其有利于沙漠治理和土壤改善,也具有较高的生态效益[3]。甘肃省作为2020年版《中华人民共和国药典》规定正品肉苁蓉的主要分布省份之一,野生蕴藏量和人工种植面积的合理规划和采收尤为重要[4]。随着经济发展及国民健康需求的不断增加,肉苁蓉需求量日益增长,通过对甘肃肉苁蓉资源现状进行实地调查[5],发现在皋兰、靖远等多个地区仍然存在对野生肉苁蓉的大规模、灭绝式采挖现象,致使野生资源受到严重破坏。非药典种在民间大量使用的现象时有发生,大量伪品肉苁蓉进入药市[6],种质混乱势必造成商品肉苁蓉质量参差不齐,严重影响其经济价值,因此对不同种质的肉苁蓉进行准确鉴定尤为重要。
分子鉴定技术因样品用量少、速度快、准确性高,已被广泛用于动植物的物种鉴定[7],而当前对甘肃肉苁蓉的种质资源分子鉴定研究还未见报道。本研究以核基因片段nrDNA的ITS和CD序列、叶绿体基因片段cpDNA的matK和rbcL序列为研究对象,对甘肃省不同种质肉苁蓉进行分子鉴定,以期寻找适合肉苁蓉分类的基因序列,并探究种质内与种质间的差异,实现肉苁蓉种质的快速准确鉴别,以此探讨肉苁蓉种质间的分类地位,为甘肃肉苁蓉资源的评价、保护和利用提供参考依据。
1 仪器与试药
OHAUS Corporation DV SERIES型电子天平,奥豪斯仪器有限责任公司;DK-98型数显恒温水浴锅,天津市泰斯特仪器有限公司;TGL-16M型高速离心机,湖南湘仪实验仪器开发有限公司;G1000改进型基因聚合酶链式反应(PCR)仪,杭州博日科技有限公司;4100型凝胶成像处理系统,上海天能科技有限公司;Vortex-Genie 2T型涡旋混匀器,德国IKA公司;BCD-451WDEMU1型冰箱,青岛海尔股份有限公司。
NEP003-2型植物基因组提取试剂盒(批号B00B00103),北京鼎国昌盛科生物技术有限责任公司;β-巯基乙醇(批号20191103)、Agarose琼脂糖(批号0000590598)、BSA(批号20191203)、引物、Taq DNA Polymerase(批号W9910e)、dNTPs(批号R6409)、GoldView核酸染料(批号321523BB)等PCR试剂,上海生工生物工程股份有限公司;DNA Marker(批号20190601),大连宝生物股份有限公司;乙醇、氯仿、异戊醇、异丙醇为分析纯,天津市大茂化学试剂厂;水为屈臣氏纯净水。
肉苁蓉样品共27份,多数为自采,部分为市场购买,其中肉苁蓉(C.deserticola)16份、沙苁蓉(C.sinensis)6份、盐生肉苁蓉(C.salsa)4份、兰州肉苁蓉(C.lanzhouensis)1份。经甘肃中医药大学晋玲教授鉴定为以上基原肉苁蓉的肉质茎,所有样品采集后于干燥硅胶中保存,样品来源信息见表1。
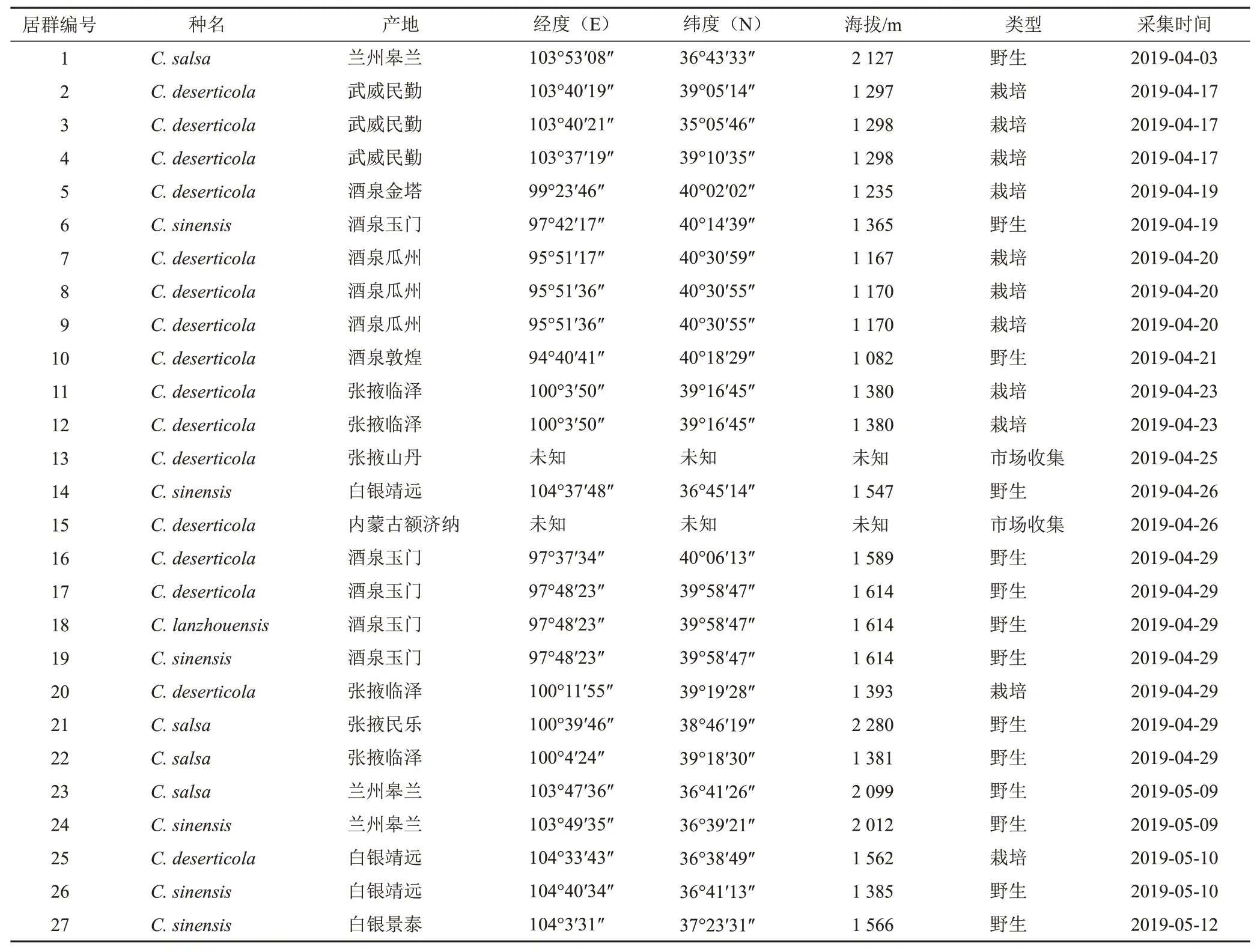
表1 肉苁蓉样品来源信息
2 方法与结果
2.1 肉苁蓉基因组DNA提取与质量检测
用75%乙醇擦拭样品表面后置于2 mL离心管中,粉碎成细粉。采用植物基因组DNA快速抽提试剂盒提取样品的基因组DNA,提取操作方法参照试剂盒说明书。由于肉苁蓉药材多糖含量较高,对DNA提取有一定干扰,故在此方法基础上用75%乙醇纯化DNA 3~4次。提取的基因组DNA于-20℃冰箱中保存。采用电泳1%琼脂糖凝胶,1×TAE缓冲液,于电泳仪(220 V,50 mA,30 min)进行基因组DNA完整性检测。电泳完成后,置于凝胶成像仪下观察,选取条带清晰明亮,无明显拖尾杂带的DNA用于后续分析。
2.2 PCR扩增及测序
2.2.1 引物信息
在查阅文献[8-10]的基础上进行筛选,选用扩增成功率较高的4对引物:ITS2-ITS3、CD1F-CD1R、matK3F-matK1R和rbcL1F-rbcL724R。引 物 信 息见表2。
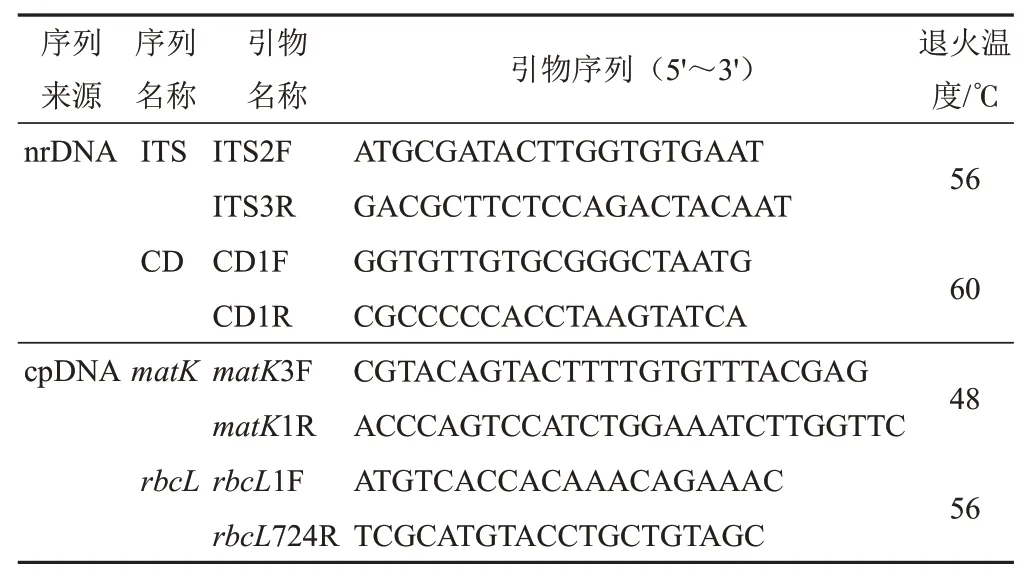
表2 肉苁蓉基因组序列PCR引物信息
2.2.2 反应体系与检测
PCR扩增总体系为25μL:10×PCR Buffer 2.5μL,dNTPs(10 mmol/L)2.0μL,BSA 0.5μL,TaqDNA聚合酶0.5μL,正向及反向引物各0.5μL,DNA模板2μL,ddH2O 15.5μL。PCR扩增程序为:95℃预变性5 min,94℃变性30 s,根据不同引物的退火温度复性30 s,72℃延伸45 s,35个循环,72℃延伸7 min,4℃保存。27份肉苁蓉样品经4对引物扩增后电泳检测,其扩增条带清晰明亮,扩增成功率均为100%(见图1)。将符合测序条件的PCR产物送上海生工生物工程股份有限公司进行双向测序。
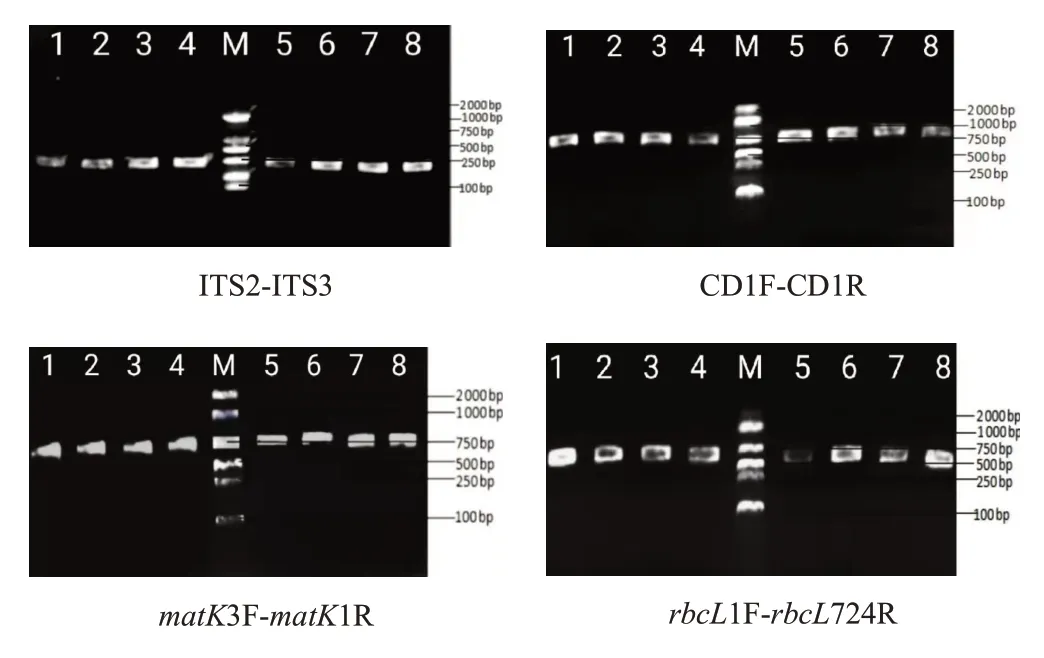
图1 肉苁蓉基因组序列部分PCR产物电泳
2.3 结果分析
2.3.1 样品测序结果
将测序所得序列文件用Chromas和ContigExpress软件去除两端不稳定信号的低质量碱基,用以校对峰图,并将每个样品测序后的正反向序列拼接[11],对拼接后的每条序列峰图进行检查及人工校对,将处理得到的样品序列及采集信息上传至GenBank数据库。ITS2F-ITS3R序列登录号为OL827059-OL827085,CD1F-CD1R序列登录号为OL827086-OL827112,matK3F-matK1R序列登录号为OL827113-OL827139,rbcL1F-rbcL724R序列登录号为OL827140-OL827166(见表3)。

表3 肉苁蓉样品序列GenBank登录号
2.3.2 肉苁蓉序列特征
27份肉苁蓉样品均被成功扩增和测序,使用Mega X软件[12]中ClustalW程序将4对引物扩增出的序列进行比对,再分别将nrDNA和cpDNA序列联合得到2组合并序列的序列特征(见表4)。4对序列的GC含量分别为56.8%、56.9%、30.3%和41.3%,而cpDNA合并序列GC含量比nrDNA合并序列低,为34.6%。
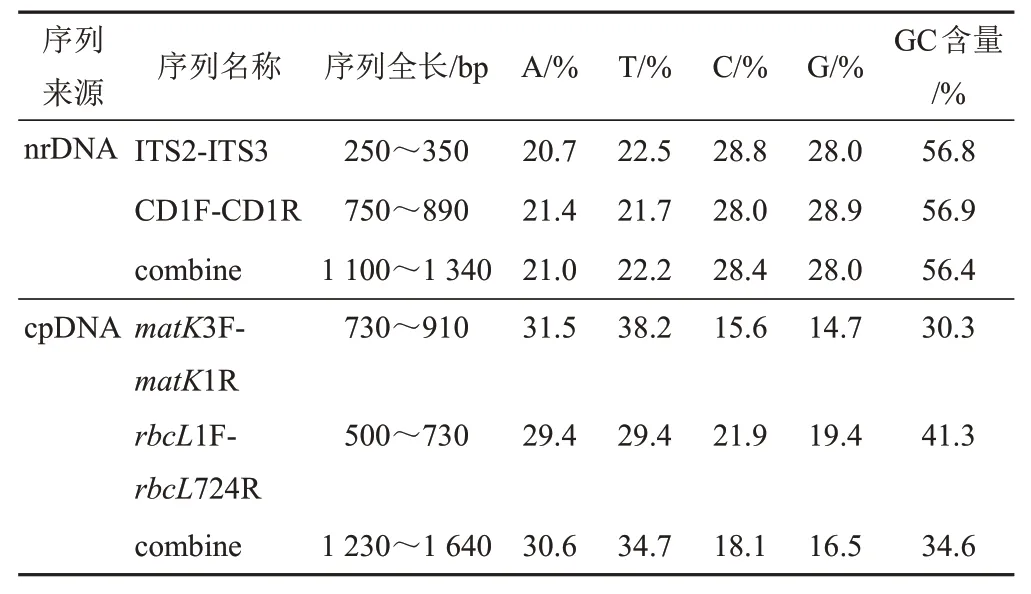
表4 肉苁蓉样品序列特征
2.3.3 核苷酸多态性
运用DanSP5.10.01软件[13]进行核苷酸多态性分析,肉苁蓉样品在ITS、CD、matK和rbcL序列的碱基位点中存在着不同程度的变异,其中,matK序列的变异位点、单一突变位点、简约信息性位点、插入-缺失位点及平均核苷酸差异数均明显高于其余3对序列,共检测到481个变异位点、369个单一突变位点、112个简约信息位点和88个插入-缺失位点,平均核苷酸差异数为62.761。cpDNA合并序列的变异位点、单一突变位点、简约信息性位点、插入-缺失位点及平均核苷酸差异数均明显高于nrDNA合并序列,共检测到534个变异位点、388个单一突变位点、146个简约信息位点和120个插入-缺失位点,平均核苷酸差异数为77.276,但其核苷酸多样性低于nrDNA合并序列(见表5)。
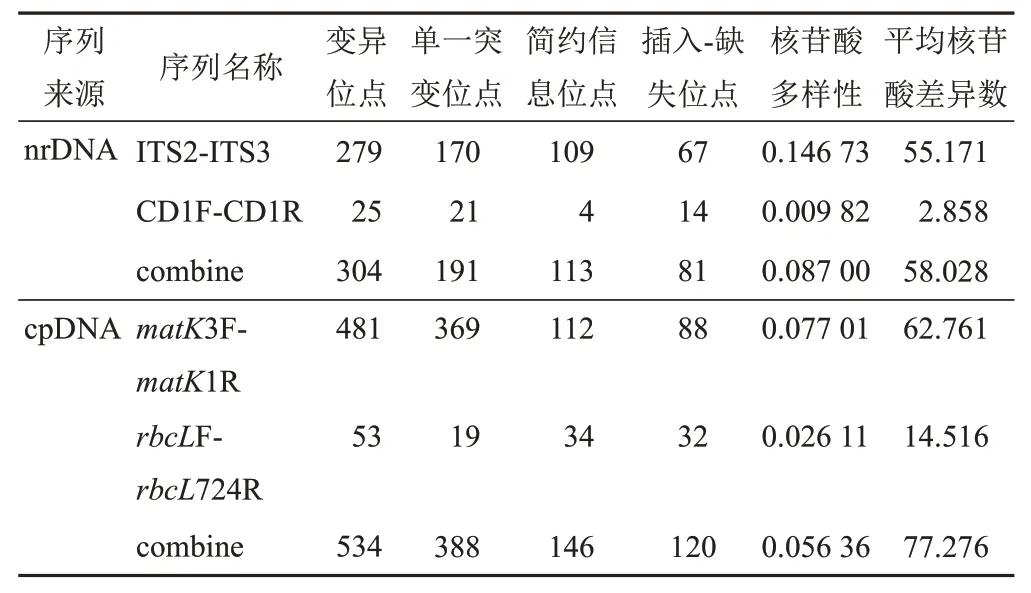
表5 肉苁蓉样品核苷酸多态性信息
2.3.4 单倍型多样性
根据DanSP5.10.01软件分析结果,发现肉苁蓉样品的4对序列和2组合并序列的单倍型多样性均大于0.5,值越大,则可推断多样性程度越高。在4对序列中,matK序列中单倍型数量最多,共形成21个单倍型;2组合并序列中,cpDNA序列的单倍型多样性更高,其单倍型数量达到24个(见表6)。
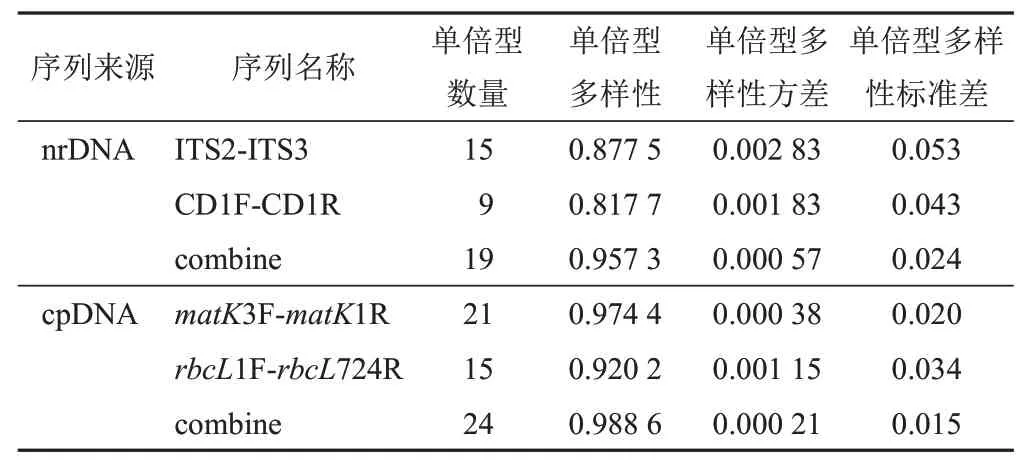
表6 肉苁蓉样品单倍型多样性
2.3.5 种质遗传距离分析
基于kimura2-parameter(K2P)模型计算肉苁蓉ITS、CD、matK和rbcL4对序列的种质遗传距离(见表7)可知,4对序列的平均遗传距离为0.209 3、0.010 8、0.109 4、0.027 1,经比较其遗传距离ITS>matK>rbcL>CD。4对序列得到的所有样品遗传距离范围分别为0~1.405 6、0~0.064 1、0~0.992 5、0~0.077 3。对比可得,ITS和matK序列的变异性较大,CD和rbcL序列较保守。此外,计算遗传距离均显示兰州肉苁蓉与肉苁蓉的遗传距离最近,使用matK序列通过GraphPad Prism 8.0.1软件绘制遗传距离矩阵热图,遗传距离为0.001 2(见图2)。
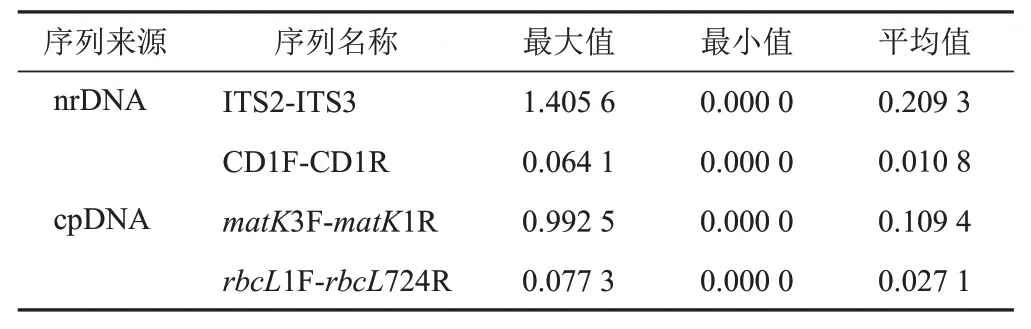
表7 肉苁蓉不同序列种质遗传距离
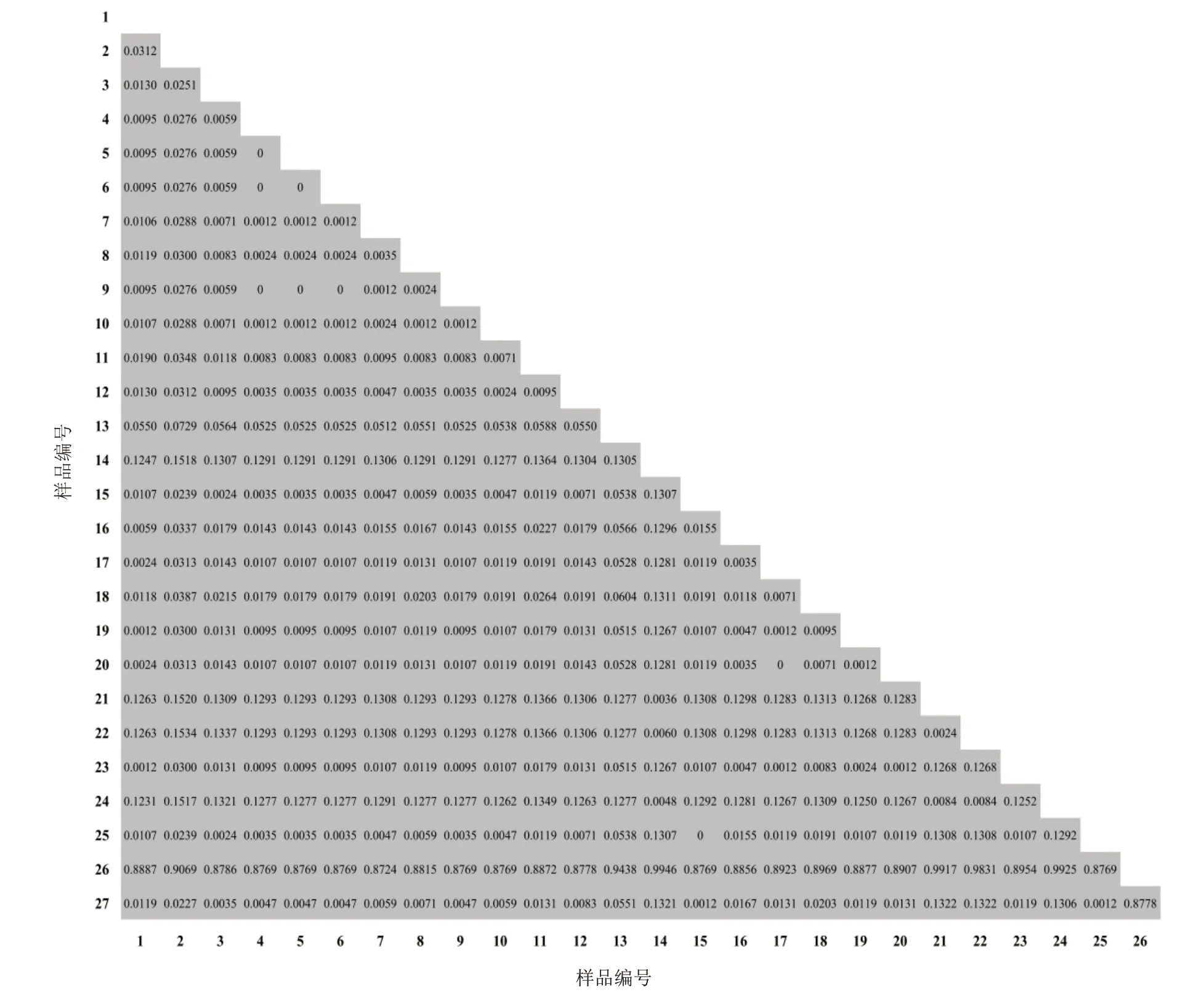
图2 肉苁蓉matK序列遗传距离矩阵
2.3.6 聚类分析
通过GenBank数据库下载草苁蓉属Boschniakia植物丁座草Boschniakia himalaicaHook.f.et Thoms.序列信息(ITS和CD为AY911212.1,matK为MH659839.1,rbcL为MG546905.1),以其为外类群与nrDNA和cpDNA序列及2组合并序列构建邻接法(NJ)聚类树[14],结果表明,外类群丁座草可单独聚为一支,4对序列在属水平上的鉴定成功率均为100%。ITS和CD序列聚类图显示,27份肉苁蓉样品可聚为3类,其中样品27单独为一个进化支,样品6、11、12、13、14、19、21、22、26、24为一个进化支,其余为一个进化支(见图3A、图3B)。matK序列聚类图显示,27份肉苁蓉样品可聚为4类,其中样品13单独为一个进化支,样品22、21、24、14为一个进化支,样品19、1、23、16、20、18、17为一个进化支,其余为一个进化支(见图3C)。rbcL序列聚类图显示,27份肉苁蓉样品可聚为4类,其中样品26、6、2分别为一个进化支,其余为一个进化支(见图3D)。2组合并序列聚类图表明,nrDNA合并序列可将大多数沙苁蓉、盐生肉苁蓉与正品肉苁蓉区分开,只有样品11、12、13未能区分(见图3E);cpDNA合并序列则可将样品13单独聚为一个进化支,其余肉苁蓉聚为另一个进化支,从而进行有效鉴别区分(见图3F)。所有构建的NJ聚类树均显示样品1、23、21、22(盐生肉苁蓉)分别为不同进化支,样品1和23为一个进化支,样品21和22则为另一个进化支,且兰州肉苁蓉始终与肉苁蓉聚为一支。
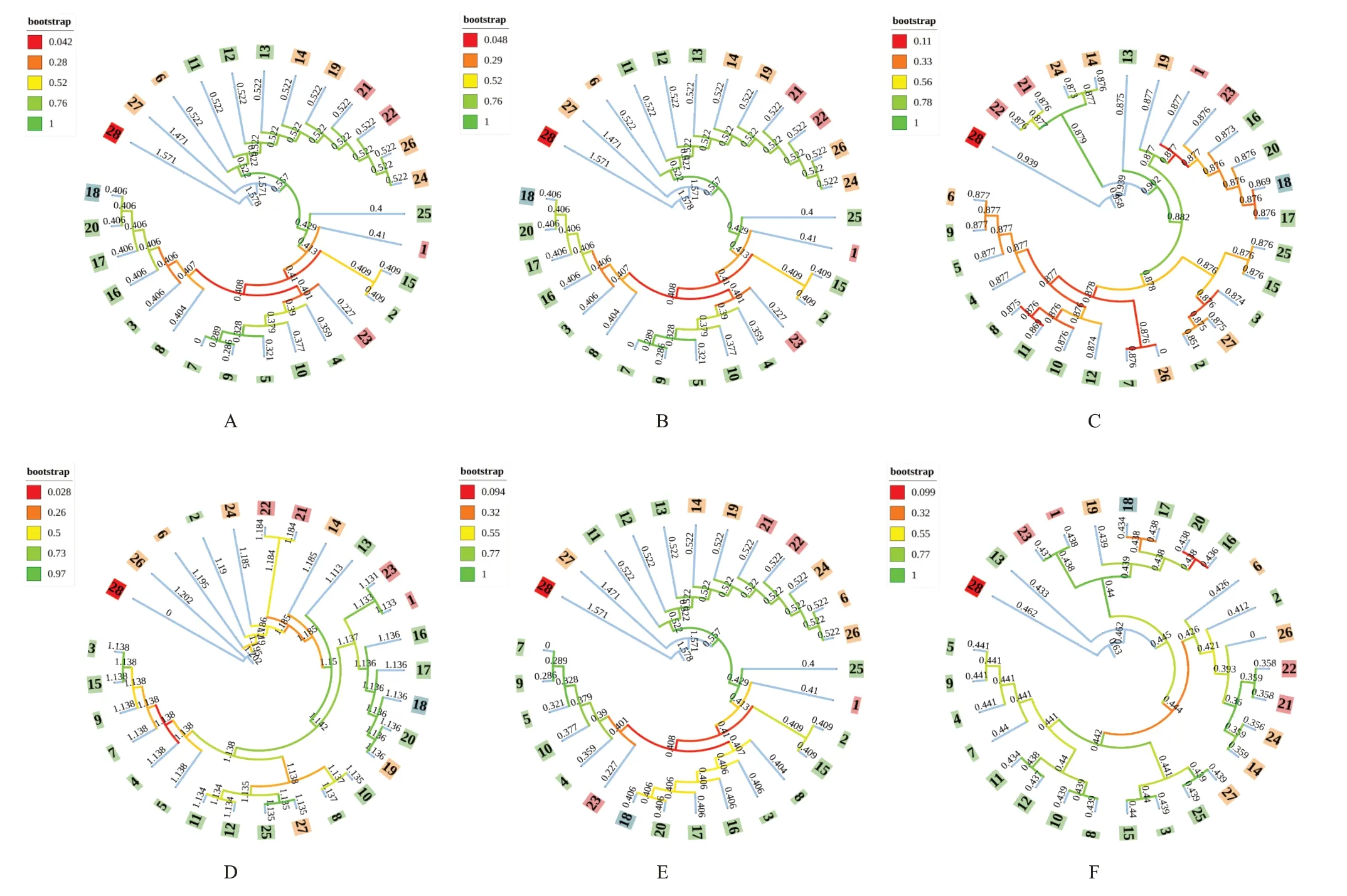
图3 肉苁蓉样品基因组序列NJ系统聚类树
2.3.7 方差分析
将采样地不同但属于同种的肉苁蓉样品归为一组,共分为3组,即肉苁蓉组、盐生肉苁蓉组和沙苁蓉组,采用Arlequin3.5软件[15]对nrDNA和cpDNA合并序列进行方差分析(见表8),结果表明,肉苁蓉遗传变异主要来自组内变异,即同一物种不同种质间分化明显。ITS和CD的合并序列遗传分化指数(Fst)为0.206 8,基因流(Nm)为1.01;matK和rbcL的合并序列Fst 为0.230 7,Nm为0.92,说明肉苁蓉不同种质间存在一定基因流动,但基因交流不频繁。
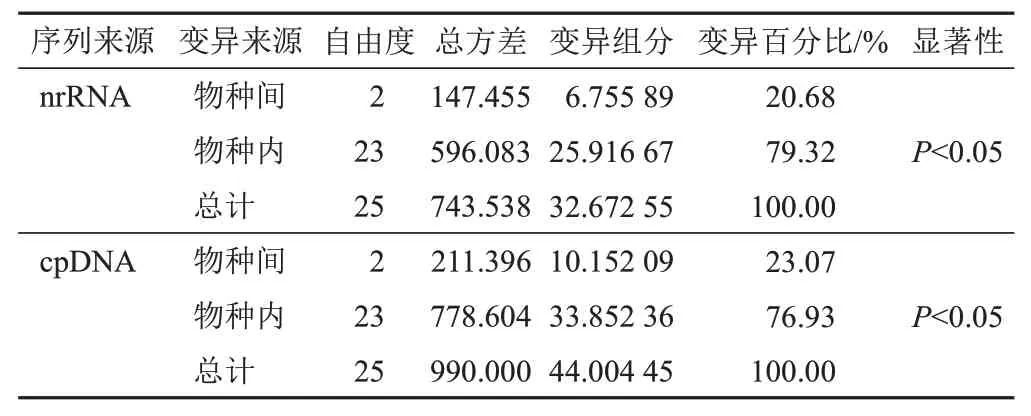
表8 肉苁蓉样品基因组序列方差分析
3 讨论
以往研究表明,nrDNA ITS序列进化速率快,变异信息位点较丰富,被广泛应用于植物的遗传多样性分析[16]。而cpDNA序列则可提供丰富的遗传信息,为评估物种遗传多样性和亲缘关系提供科学参考[17]。本研 究 利 用4对 引 物(ITS2-ITS3、CD1F-CD1R、matK3F-matK1R和rbcL1F-rbcL724R)进行序列比对、遗传距离分析及NJ聚类树的构建,结果表明,肉苁蓉种质资源多样性程度较高,且在种质间水平上均有丰富的变异性。毕毓芳等[18]研究表明,matK序列是进化速率最快的叶绿体基因组序列,常在科级、属级、种间和种内水平上被用于系统进化研究。马丽等[19]在对石榴种质资源进行基因组序列分析时发现石榴物种的matK基因片段扩增及测序成功率高,聚类准确度较强。董斌等[20]研究发现,在matK序列变异位点较多的情况下,遗传差异也较大,有利于鉴别同一物种的不同种质。本研究中,matK序列是4对序列中变异位点最多的序列,具有481个变异位点,其中单一突变位点为369个,所占比例最大,是变异的主要来源。按Grant等[21]提出的标准,单倍型多样性以0.5为临界值,核苷酸多样性以0.005为临界值,二者值越大,群体的多样性程度越高。4对序列中matK序列单倍型多样性最高,而cpDNA合并序列的单倍型多样性明显高于nrDNA合并序列,因此,matK序列更适合作为肉苁蓉遗传多样性分析的序列,也证明了叶绿体基因片段在群体遗传学研究中的优越性。此外,对盐生肉苁蓉,由于采集样品地理位置的差异,发现不同地区的同种肉苁蓉也存在地理遗传差异性,与杨俊誉等[22]研究结果一致。
分子鉴定技术可有效对中药材进行准确鉴别,是对传统经典鉴定方法的有效补充,弥补了经典鉴定方法的不足[23]。近年来,许多学者为实现对物种进行快速鉴定,对适合植物物种鉴定的基因序列进行了深入研究,其中主要包括ITS、ITS2、psbA-trnH、matK和rbcL序列[24]。徐素素等[25]认为,单独类型的序列鉴别存在一定局限性,分子鉴定体系的完善需要多条序列联合应用。李波等[26]在研究中使用核基因片段与叶绿体片段相结合的方法,从而达到提高物种鉴别效率。本研究以nrDNA和cpDNA序列构建NJ聚类树,发现单独某对序列并不能在种水平上有较好的鉴别效率。ITS和CD序列聚类图表明,多数肉苁蓉与沙苁蓉、盐生肉苁蓉亲缘关系较远,会分别聚于不同进化支,但也存在小部分肉苁蓉与其他物种聚为一支的情况;matK和rbcL序列聚类图表明,沙苁蓉与盐生肉苁蓉亲缘关系较近,可聚为同一大支,而两物种又会分别聚为不同小支,但部分肉苁蓉和盐生肉苁蓉存在聚类混乱的情况,不能准确鉴别。故将nrDNA和cpDNA序列联合使用,可进一步实现对肉苁蓉种质的快速、准确鉴别。
兰州肉苁蓉在《中国植物志》中作为单独的物种具有一定植物学分类地位,但近年来也有学者建议将兰州肉苁蓉及沙苁蓉归为同个物种[27]。而陈博等[6]在对甘肃肉苁蓉进行指标性成分含量测定研究发现,兰州肉苁蓉与沙苁蓉成分含量存在一定差异,与肉苁蓉成分含量较为相似。本研究中,通过计算遗传距离及构建NJ聚类树,结果兰州肉苁蓉与肉苁蓉的遗传距离较近,均可聚为同一个进化支。由此,建议将兰州肉苁蓉与其他肉苁蓉进行分类区别,考虑作为正品肉苁蓉的药源补充,提高其资源开发利用度,后续可就此问题进行深入研究。
近年来,随着对肉苁蓉药材需求量的提升,为保证临床安全用药,对肉苁蓉种质资源的鉴别、评价及保护至关重要。本研究基于nrDNA和cpDNA序列,对甘肃省4种肉苁蓉进行分子鉴定,结果表明肉苁蓉种质资源多样性程度较高,可为进一步细化肉苁蓉不同种质资源的分类提供参考。
