绿豆遗传连锁图谱加密及生育期相关性状的QTL定位
2022-12-10蔡德宝丁冬会杨树琼孙新颖陈吉宝
蔡德宝, 丁冬会, 马 乐, 杨树琼, 孙新颖, 陈吉宝
(南阳师范学院/南水北调中线水源区水安全河南省协同创新中心,河南南阳 473061)
绿豆(Vignaradiata)是豆科(Leguminosae)菜豆族(Phaseoleae)豇豆属(Vigna)植物中的一个栽培种,因其相对耐旱性强、具有固氮作用以及较短的生育期等特性而广受欢迎,是豆类植物中重要的作物[1-2]。生育期是作物高产育种中极为重要的指标,它不仅决定了品种的种植地区与季节适应性,而且与抗逆性、品质以及产量密切相关[3-7]。绿豆属于短日照作物,其生育期相关性状主要包括始花期、开花期、始熟期、成熟期等,这些性状属于数量性状,受多基因控制,遗传力高且遗传稳定性强,在分离群体中呈连续分布,可以通过数量性状位点(quantitative trait locus,简称QTL)定位和图位克隆的方法获得这些基因[8-10]。
获得一张高质量的遗传图谱是基因定位、图位克隆乃至基因组结构与功能研究的基础[11],在绿豆中,叶卫军等利用RFLP、RAPD、AFLP、SSR、SNP 等分子标记和F2群体、回交群体、重组自交系群体(recombinant inbreed line,简称RIL)等作图群体构建了至少20张遗传图谱[12]。在绿豆转绿组和基因组数据公布之前,可用的分子标记十分有限,前人所绘制的绿豆遗传连锁图谱主要基于栽培绿豆和野生绿豆杂交形成的群体,其主要原因是利用野生绿豆和栽培绿豆的种间杂交更容易筛选到多态性分子标记[13]。Kang等公布的首张绿豆基因组序列草图和大量SSR分子标记[1],为利用2个栽培绿豆品种杂交构建群体绘制高质量的遗传连锁图谱提供前提[13-14]。近年来,绿豆生育期相关性状的QTL定位也取得一定进展。梅丽等用RIL群体在广西壮族自治区和北京2个生境下进行绿豆生育期相关性状的QTL定位[7],共鉴定出了4个与开花期相关的基因位点,贡献率在4.95%~34.53%之间;共鉴定了4个成熟期QTL,分布LG2、LG9上。Isemura等发现了4个与初花期紧密相关的QTL,其中位于LG2上的QTL贡献率最大[15]。Kajonphol等鉴定了6个始熟期QTL位点,位于第2、4、6、7、9、11连锁群上[16]。Somta等在雨季和旱季2种环境下对绿豆开花期进行QTL定位,共检测到7个QTL,其中位于LG2A连锁群上的2个标记区间在2种环境下均检测到开花期QTL[17]。
尽管对绿豆的生育期相关性状已有大量研究,鉴定出了一些生育期相关QTL位点,但是还存在以下问题需要解决:(1)这些研究限于绿豆开花期、成熟期等生殖生长阶段,缺乏对绿豆营养生长阶段的研究,更没有对各生育期相关性状进行系统分析;(2)还有新的生育期相关性状位点没有完全分离出来,需要继续发掘;(3)尽管目前研究者绘制了多张绿豆遗传连锁图谱,但由于标记数量少,分辨率低,不能满足基因图位克隆的条件。本研究以RIL群体为材料,在自然条件下通过3年的田间表型评价,研究绿豆生育期相关性状的遗传特征,通过对Liu等构建的图谱[14]加密,整合1张高密度绿豆遗传连锁图谱,应用新图谱定位绿豆生育期相关性状QTL,为绿豆生育期研究提供标记信息,推动绿豆熟性分子育种的发展。
1 材料与方法
1.1 试验材料
试验所用绿豆RIL群体由中国农业科学院作物科学研究所食用豆课题组提供,该群体以晚熟材料鹦哥绿和早熟材料VC2917为父母本构建而成,包含261个株系[14,18],目前已经繁殖至F9代。
1.2 田间试验设计与数据分析
大田试验于2018—2020年春季在河南省南阳师范学院农田进行,于每年4月下旬播种,完熟后收获。绿豆生长期间按照正常田间管理措施进行,于分枝形成前中耕1次,并起垄培土,整个生长期间不人为浇水。试验采用完全随机区组设计,重复3次。
出苗期(GD)、三叶期(TLD)、分枝期(BS)、始花期(FFD)、始花期-分枝期(FFD-BS)、开花期(FD)、开花期-分枝期(FD-BS)、成熟期(MD)严格按照《绿豆种质资源描述规范和数据标准》进行调查统计[8]。
参照陈吉宝的方法[19]计算2018、2019、2020年3个年份8个性状的表型数据的BLUE值。用SPSS 24.0进行描述性统计分析、方差分析(ANOVA)和相关性分析,并计算广义遗传率。
1.3 SSR分子标记的筛选和基因分型
本研究总计筛选SSR引物275对,其中250对为Kang等开发[1],下载自NCBI网站绿豆基因组数据库(GenBank/EMBL/DDBJ数据代码JJMO00000000),前缀为“Vr”;25对为Liu等从绿豆近源种小豆中筛选[14],前缀为“CEDG”。
取绿豆RIL群体苗期鲜嫩叶片,用研钵在液氮环境中研磨成粉状,绿豆RIL群体单株及亲本基因组DNA采用试剂盒(EasyPure® Plant Genomic DNA Kit)提取;PCR扩增采用10 μL反应体系:酶(2×EasyTaq® PCR SuperMix for PAGE+dye)5 μL,引物0.5 μL (10 μmol/L),模板1 μL(50 ng/μL),ddH2O 3.5 μL。PCR反应程序及银染检测参考王建花等的方法[11]。
1.4 遗传连锁图谱的加密
选择2个亲本间具有明显多态性的SSR标记进行RIL群体的基因分型。读板时母本带型记作“A”,父本带型记作“B”,杂合带型记作“H”,空缺或其他带型记作“X”。利用卡平方测验分析每个标记在0.05、0.01和0.001水平上是否符合1 ∶1的分离规律,用于推断标记偏分离情况。
利用QTL IciMaping V4.0软件[20]加密绿豆遗传连锁图谱,加密前遗传连锁图谱由Liu等构建[14]。加密参照软件提供的Sampledata进行数据格式化,用Grouping命令对所有标记进行分组,比较不同LOD值下的分组情况,选择最优分组;运算法则选用“nnTwoOpt”,其他参数采用软件默认值。
1.5 生育期性状的QTL分析
本研究基于Liu等构建的图谱[14],利用加密后的图谱进行QTL定位,采用QTL IciMapping V4.0软件改进复合区间作图法定位[20],Permutation次数设置为1 000次,LOD值以大于等于2.5为标准。
2 结果与分析
2.1 绿豆生育期性状遗传分析
从表1可以看出,RIL群体内分枝期、始花-分枝期、开花期、成熟期的最大值和最小值均分别高于亲本最大值和小于亲本最小值,群体内除开花期、开花-分枝、成熟期外,其余各性状均值均小于双亲均值。各性状变异系数为5.01%~28.81%,其中变异系数最小的是分枝期,始花期-分枝期变异系数最大,以上结果说明各性状在群体个体之间均得到较好的分离。8个性状的遗传力在42.24%~82.38%之间,其中三叶期、始花期、始花-分枝期和成熟期的遗传力较高,均大于70%;分枝期、开花-分枝期的遗传力均小于50%,表明二者的遗传力较低。除出苗期、三叶期外,其余性状偏度绝对值均小于或近似等于1,说明这些性状近似符合正态分布;除分枝期和开花-分枝期外,其余性状偏度均大于0,分枝期和开花-分枝期表型数据分布向右偏,其他性状表型数据分布向左偏,其中出苗期的偏度为1.6,呈明显向左偏。除始花期、始花-分枝外,其他性状峰度的绝对值均大于0.5;始花期、始花-分枝的峰度小于0,表明这2个性状表型分布不太集中。从图1可以看出,群体各性状值均呈连续分布,说明这些性状适合QTL分析。

表1 RIL群体各生育期性状的分布特征
2.2 绿豆生育期性状相关性分析
依据8个性状的BLUE数据进行各生育期性状之间的相关分析,结果(表2)表明,出苗期与分枝期、始花期、开花-分枝间隔期,三叶期与成熟期以及分枝期与成熟期均呈显著正相关;三叶期与分枝期,分枝期与开花-分枝间隔期,始花期与始花-分枝间隔期、开花期、开花-分枝间隔期、成熟期,始花-分枝间隔期与开花期、开花-分枝间隔期、成熟期,开花期与开花-分枝间隔期、成熟期,开花-分枝间隔期与成熟期均呈极显著正相关;其中始花期与始花-分枝间隔期相关性最强(0.861),表明始花期在决定始花-分枝间隔期起关键性作用,其次为开花期与成熟期(0.737);分枝期与始花-分枝间隔期呈极显著负相关(-0.302),表明分枝期对始花-分枝间隔期起负作用。
2.3 遗传连锁图谱的构建
共得到亲本间具有多态性且在RIL群体中带型清晰的引物20对,多态性比率7.2%(图 2)。这些分子标记连同Liu等所使用的313个标记[14],通过QTL IciMaping V4.0作图[20],得到1张新的绿豆遗传图谱,加密后图谱的各项特征较原图谱均有所改变(图3)。

表2 绿豆生育期相关性状间的相关性分析
Liu 等的原图谱由11个连锁群组成,包含313个分子标记,总长为1 010.18 cM[14]。新图谱包含11个连锁群,333个标记位点,图谱总长为 1 417.29 cM。原图谱各连锁群标记数为19~74个,平均28.45个,其中标记最少的是LG2、LG7,各连锁群平均标记数为19个,LG4标记数最多,包含74个标记。各标记间距离在 2.14~4.29 cM之间,平均3.37 cM,加密后各连锁群密度为 2.14~6.24 cM,平均密度为4.26 cM。新图谱标记间平均距离有所增大,是由于新图谱LG1、LG2连锁群中存在2个大于40 cM的空隙,整合后图谱有所延长,对绿豆基因组的覆盖率也极大增加。除LG4、LG9未新加标记外,其余连锁群的长度都发生了较大改变,如LG1连锁群新增标记4个,长度由59.74 cM 延长为 128.07 cM;LG10连锁群新增标记3个,长度由82.42 cM 延长到116.63 cM;LG5连锁群新增2个标记,长度由65.46 cM 延长到91.96 cM。
标记位置随着新标记的加入而有所变动。如第1连锁群上MUS275~HAAS_VR_1620这9个标记、第6连锁群HAAS_VR_2489~CEDG-144这9个标记在新标记加入后,标记间的方向、距离均有所改变,同时相邻标记间的位置也有所改变,但未发生连锁水平上的改变。新建图谱333个标记中,偏分离标记数72个(21.62%,P<0.05),各连锁群偏分离标记数在1(LG6、LG10)~17(LG1)之间(表3)。新加标记中严重偏分离(P<0.001)的标记主要位于LG1上,且偏分离的标记主要偏向于父本,与Liu等的图谱[14]类似。
2.4 生育期相关性状的 QTL分析
本研究对绿豆8个生育期相关性状的BLUE数据进行QTL分析,共检测到15个QTL位点,成熟期检测到4个QTL位点,出苗期检测到3个QTL位点,三叶期、开花期各检测到2个QTL位点,分枝期、始花期、始花-分枝间隔期以及开花-分枝间隔期均检测到1个QTL位点。
共检测到3个出苗期(GD)QTL位点。其中第1连锁群(LG1)的MUS275~CEDG-150、第2连锁群(LG2)的Vr11-473~Mchr11-45以及第6连锁群(LG6)的MUS118~MUS35标记之间检测各到1个QTL位点(GD1a、GD2a、GD6a),贡献率分别为59.24%、63.85%、6.97%,其中GD1a、GD2a位点对出苗期有较大贡献,是决定出苗期的大效应位点。3个QTL位点加性效应均为负值,平均加性效应为-0.51,平均可使出苗期减少0.51 d。
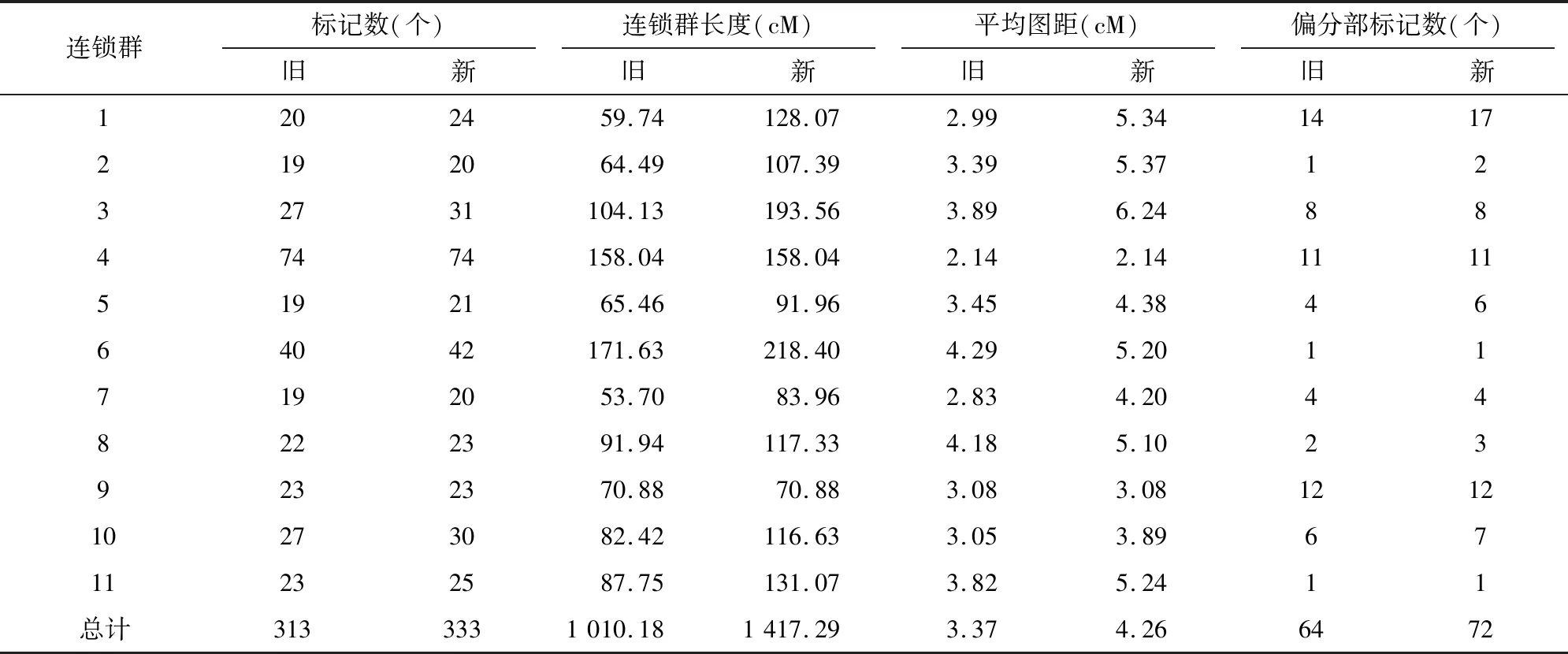
表3 图谱加密前后定位的QTL比较
共检测到2个三叶期(TLD)QTL位点。其中LG1的MUS275~CEDG-150、LG6的MUS168~Mchr7-23之间各检测到1个QTL(TLD1a、TLD6a),贡献率分别为26.76%、10.29%,表明前者是决定三叶期的大效应位点。TLD1a加性效应为负,可使三叶期减少1.86 d,TLD6a加性效应为正,可使三叶期增加0.64 d。
检测到1个分枝期(BS)QTL位点(BS9a),位于第9连锁群的Mchr10-9~Mchr5-37标记之间,该位点贡献率为5.95%,可能属于微效基因。该位点加性效应为负,说明该位点可缩短分枝期。
检测到1个始花期(FFD)QTL位点(FFD2a),位于第 2 连锁群的MUS171~MUS617标记之间,该位点贡献率较大(11.61%),说明该位点对FFD的贡献较大,可能属于大效应位点。该位点加性效应为-0.96,说明该位点可使FFD缩短0.96 d。
检测到1个始花-分枝间隔期(FFD-BS)QTL位点(FFD-BS2a),位于第2连锁群的MUS171~MUS617标记之间,该位点贡献率为10.44%,说明该位点对FFD-BS的贡献较大。位点加性效应为负,说明该位点可缩短FFD-BS。
共检测到2个开花期(FD)QTL位点。其中LG1的MUS275~CEDG-150、LG2的Vr11-473~Mchr11-45标记之间各检测到1个QTL位点(FD1a、FD2a),单个位点贡献率分别为36.95%、51.30%,说明二者对FD的贡献均较大,均属于大效应位点。2个位点加性效应均为负,平均为 -3.56,平均可使FD减少3.56 d。
检测到1个开花-分枝间隔期(FD-BS)QTL位点(FD-BS11a),位于第11连锁群的Mchr10-49~HAAS_VR_1519标记之间,该位点贡献率为11.09%,说明该位点对FD-BS的贡献较大。该位点加性效应为0.62,说明该位点可使FD-BS增加0.62 d。
共检测到4个成熟期(MD)QTLs 位点。分别为位于第1连锁群MUS275~CEDG-150与第2连锁群Vr11-473~Mchr11-45标记之间的MD1a、MD2a,以及LG11 Mchr10-1~Mchr10-5与Mchr10-27~Mchr10-30标记之间的MD11a、MD11b。MD1a、MD2a均属于大效应位点(贡献率分别为37.77%、44.76%),且加性效应均为负,累计可使MD减少8.95 d。MD11a、MD11b可能属于微效基因(贡献率分别为6.19%、8.36%),前者加性效应为负,后者加性效应为正(图4、表4)。
3 讨论与结论
3.1 绿豆生育期性状统计学性分析
生育期是豆类作物产量形成的重要因素[21],研究绿豆生育期相关性状的遗传规律是了解其产量形成机制的重要途径之一。绿豆生育期相关性状主要包括出苗期、三叶期、分枝期、始花期、开花期、始熟期、成熟期[8]。本研究中,出苗期、三叶期、始花-分枝间隔期的变异系数均大于10%,最高达28.81%,开花期、成熟期变异系数均小于10%,说明营养生长前期的遗传受环境影响大于生殖生长期,与马乐等的研究结果[22,6]类似。Sompong等研究发现,绿豆成熟期与始花期呈极显著正相关[23,16]。梅丽研究表明, 与生育期的关联度由高到低排序为开花期、始花期、出苗期[6]。绿豆开花期和成熟期遗传力高且遗传稳定性强,Yimram等对340个绿豆种质材料的分析表明,开花期、成熟期广义遗传力分别为0.75、0.71[24]。Kajonphol等研究发现,绿豆始花期、始熟期遗传力分别为0.89、0.91[16]。马乐等研究表明,绿豆开花期和成熟期的广义遗传力在0.95以上[18]。本研究中绿豆始花期、开花期、成熟期遗传力均较高,分别为0.77、0.59、0.74。以上结果说明,始花期、开花期在影响绿豆生育期中扮演重要角色,育种实践中关注始花期、开花期对选育不同生育期长短的绿豆新品种更有效,开花期、成熟期遗传力高,有利于相关基因的定位和图位克隆[25,26]。
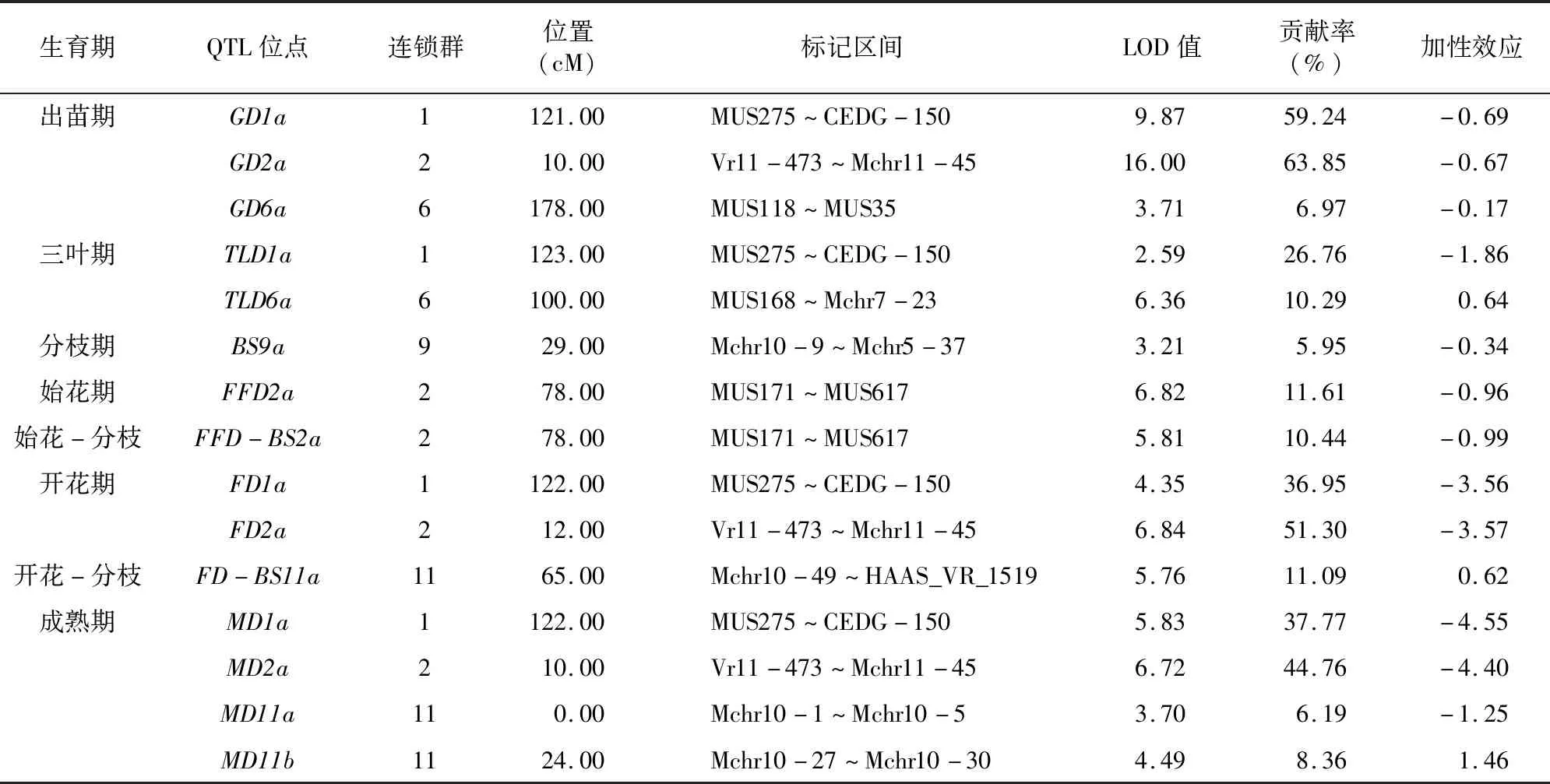
表4 绿豆生育期相关性状的QTL
3.2 遗传连锁图谱的构建
构建高质量的遗传连锁图谱是基因定位前提条件之一[27]。前人所绘制的绿豆遗传连锁图谱主要基于栽培绿豆和野生绿豆杂交形成的群体,主要原因是栽培绿豆与栽培绿豆的种间杂交很难筛选出多态性标记[13]。Chankaew 等利用绿豆的433个SSR标记对2种栽培绿豆KPS1和V4718之间进行多态性进行评估,结果发现只有27个(8.85%) SSR标记在亲本之间存在多态性[2];Liu等使用Isemura等的JP211874×JP229096(野生绿豆×栽培绿豆)绿豆群体中430个表现出多态性SSR标记,只有24个显示出多态性[14-15]。本研究应用275对SSR引物对亲本VC2917和鹦哥绿进行筛选,亲本间共得到多态性引物20对,亲本间多态性为7.2%,使得本研究设计的多态性SSR标记具有价值。
整合后遗传连锁图与Liu等的原图谱[14]基本一致,但也有一些标记位置随着连锁图谱整合发生变动,主要变动为第1、第6连锁群上的位置发生颠倒。赵丹等整合绿豆遗传连锁图谱时也发生类似状况,认为这可能是因标记偏分离现象的影响[28]。本研究整合的绿豆遗传连锁图谱中有72个偏分离标记,偏分离比率为21.75%,低于赵丹等构建的绿豆遗传连锁图谱[28-30],高于Isemura等构建的绿豆遗传连锁图谱[15]。这可能是因为本研究所用群体为栽培绿豆×栽培绿豆构建的RIL群体,避免了野生亲本携带的不利位点,但与回交群体相比,RIL群体偏分离比率一般更高,主要原因是RIL群体构建时人为抽样的偏差以及多代的自然选择[14,28]。
3.3 生育期相关性状的 QTL分析
本研究通过1点3年大田试验,对8个生育期相关性状的BLUE数据进行QTL定位,各性状均检测到QTL位点。数量性状的表达极易受环境的影响,在生长环境不同时定位的结果可能不同[7],本研究与马乐使用相同群体在同一试验地点不同年份进行QTL定位,将三叶期QTL定位在LG6(MUS168~Mchr7-23)[22]。本研究在该区间外检测1个QTL,位于LG2的MUS275~CEDG-150标记之间,同时也说明该性状可能为由位于不同染色体的多个QTL调控。大效应QTL一般是在不同生境下均可以检测到的稳定QTL,存在相关基因的概率更大,是实现图位克隆的关键区域[31-32]与梅丽研究结果[6]类似,本研究将出苗期定位在第1、第2连锁群;本研究将开花期QTL定位在LG1、LG2,与Isemura等(LG2、LG4、LG6 和 LG11)[15]、梅丽等(LG2、LG9、和 LG12)[7]及Somta等(LG2、LG4、LG5和LG6)[17]的研究结果类似,且大效应位点位于LG2,说明第2连锁群上存在决定开花期性状的关键基因;与Sompong等的研究结果[23]相比,本研究在LG2检测到1个成熟期大效应QTL,与Isemura等类似[15-16]。上述大效应 QTL 可以为今后该性状进一步研究提供借鉴,可为今后分子标记辅助选育新型早熟绿豆品种提供新的基因位点。
本试验发现2个QTL富集区,第1连锁群的MUS275~CEDG-150标记区间富集了开花期、成熟期2个性状的QTL位点,第2连锁群的MUS171~MUS617标记区间检测到控制始花期、始花-分枝间隔期的QTL,这种QTL的富集现象在梅丽等的研究中也有报道[6,14,19]。Guan等认为,QTL簇(QTL cluster)或QTL富集区是一因多效或者多个基因紧密连锁的结果[33]。本试验还发现,在QTL成束的区域,第2连锁群Vr11-473~Mchr11-45区间的不同位点检测到控制出苗期、开花期、成熟期关键性状QTL位点,Tuberosa 等认为,控制同一性状的QTL可能分布于染色体上的一个集中区域,而不是随机分布[34]。然而目前所用图谱依然无法满足图位克隆的需要,今后有必要借助全基因组关联分析(genome-wide association study)技术,有针对性地进行精细定位。在此基础上,开发与这些生育期性状密切相关的分子标记,通过回交育种与分子标记辅助选择相结合的方法,选育出新的绿豆早熟品种,为绿豆育种创造优异新材料。
