基于3D U-Net 模型实现T2WI 图像中前列腺的区域分割及临床验证
2022-12-10刘想高歌韩超朱丽娜张耀峰王祥鹏张晓东王霄英
刘想,高歌,韩超,朱丽娜,张耀峰,王祥鹏,张晓东,王霄英
(1.北京大学第一医院医学影像科,北京 100034;2.郑州大学第一附属医院影像科,河南 郑州 450052;3.北京赛迈特锐医学科技有限公司,北京 100011)
多参数磁共振成像(Multiparametric MRI,mpMRI)是发现前列腺可疑病灶并进行定位、检测的重要成像方式[1-2]。前列腺影像报告和数据系统(Prostate imaging and report data system,PI -RADS)v2.1 建议根据分带解剖来进行病灶的定位。T2WI 上前列腺分带解剖显示清楚,因此临床上常基于T2WI 来进行解剖定位[3]。
自2011 年Makni 等提出应用C-means 聚类方法进行前列腺的自动分割后[4],关于前列腺自动分割的方法和研究已有很多报道。尤其是近些年,随着深度学习技术的发展,以U-Net 为代表的卷积神经网络在前列腺分割中表现出色[5-7]。但目前这些研究仅实现了前列腺的腺体分割或简单的区域分割(中央腺体+外周带),并未满足前列腺系统穿刺的6 分区定位标准及影像医生对前列腺癌定位的分割精度要求。我科已实现在前列腺T2WI 上使用U-Net 模型进行前列腺的全腺体分割[8]并已证实基于该分割模型的腺体径线测量植入结构化报告的可行性[9]。本研究在此基础上进一步利用3D U-Net 模型进行精细的前列腺区域分割并利用临床实际前列腺癌患者病例,验证该模型是否可满足临床上影像医生对癌灶的定位需求,以期为后续的前列腺癌自动检出、定位奠定基础。
1 资料与方法
本研究获得了伦理审查委员会批准(批件号:[2021-060]),按照本单位人工智能(Artificial intelligence,AI)模型训练规范执行研究方案。
1.1 用例定义
根据本单位AI 训练管理方法,首先定义研发前列腺区域分割的用例。包括:前列腺区域分割AI 模型的ID、临床问题、场景描述、模型在实际工作中的调用流程、模型输入输出数据结构等。AI 模型返回结果定义为:前列腺外周带、移行带、中央带、前纤维基质带及尿道的坐标,以及上述分割区域的体积等。AI 模型结果返回至前列腺mpMRI 结构化报告中。
1.2 一般资料
本研究收集数据包括用于模型训练的数据集(数据集A)和用于临床验证的数据集(数据集B)(图1)。从本院PACS 系统回顾性收集2019 年6月—2020 年1 月的302 例患者的前列腺mpMRI 图像,纳入标准为:①因前列腺特异性抗原(Prostate specific antigen,PSA)增高和(或)直肠指诊怀疑为前列腺癌而行mpMRI 扫描;②扫描规范的T2WI 序列。按照排除标准:①前列腺癌根治术后或经尿道前列腺切除术后(n=4);②前列腺癌辅助治疗后(n=5);③有明显的运动伪影等(n=5),最终有288 例患者纳入研究,用于模型训练(数据集A),其中包括PI-RADS 评分≤3 分患者198例,≥4 分患者90 例。
回顾性收集2020 年2—5 月的经病理证实的35 例前列腺癌患者,排除5 例治疗后患者,最终有30 例患者(共33 个前列腺癌灶)用于前列腺分割模型的临床验证(数据集B)。其中包括Gleason 评分3+3 分4例,3+4 分9例,4+3 分9例,4+4 分6例,5+5 分2 例。
1.3 扫描参数
根据前列腺mpMRI 扫描方案,所有患者的扫描序列均包括T1WI、T2WI、扩散加权成像及动态增强成像。扫描设备为3.0T Ingenia(Philips Healthcare,Best,the Netherlands)。其中,前列腺行轴位、矢状位及冠状位扫描的T2WI 参数为:重复时间(time of repetition,TR)=2 905 ms;回波时间(echo time)=90 ms;显示野(FOV)=240×240 mm,矩阵=324×280,层厚=3 mm;无间距扫描;未压脂。
1.4 分割模型训练
将所有DICOM 格式的T2WI 图像导入数据管理平台并转换为Nifty 格式。由一位低年资影像医生(阅片经验3年)使用ITK-SNAP软件(Version 3.6.0,http://www.itksnap.org/)逐层标注前列腺T2WI上的外周带、中央带、移行带、前纤维基质带及尿道结构。由另一名具有15 年阅片经验的前列腺影像专家进行审核。以手工标注的标签作为评价模型分割性能的评价标准(图2)。
使用本科室已建立的前列腺分割模型[8]分割整个前列腺,以减少周围组织结构对前列腺区域分割的干扰。将288 例数据按照8∶1∶1 的比例随机分为训练集(n=231)、调优集(n=29)和测试集(n=28)。训练集用于模型的训练,通过对样本数据的学习确定模型参数从而建立一种分类的方式;调优集用于对已训练的模型进行参数调整即做模型的最终优化及确定;测试集则是对已训练好的最终模型进行性能测试。利用3D U-Net 深度学习网络[10]进行前列腺区域分割,训练过程均在GPU NVIDIA Tesla P100 16G 上进行,软件包括Python 3.6、Pytorch 0.4.Opencv、Numpy 和Simple ITK。使用Adam 作为训练优化器。模型训练时每批次输入数据量(batch size)设定为10,学习率(learning rate)为0.0001,模型迭代次数为300 个周期(epoch)。模型训练流程如图3 所示。
1.5 分割模型评估
利用测试集数据对3D U-Net 模型的分割性能进行定量评估,评估指标包括基于重合度(overlapbased)的指标:Dice 相似系数(Dice similarity coefficient,DSC)、精确度(precision)、灵敏度(recall)[11];基于体积(volume-based)的指标:体积相似度(volumetric similarity,VS);以及基于空间距离(Spatial distance-based)的指标:Hausdorff 表面距离(Hausdorff surface distance,HSD)[12]。三者联合使用可以从整体重叠程度、体积以及边缘匹配程度多方面评估分割性能。应用最小体积包围盒(minimum volume bounding box)算法[13]自动计算各结构的手工标注体积和模型预测体积,将两者进行比较,评估模型测量前列腺各个分带区域体积的效能。
1.6 模型临床验证
由一名低年资影像医生对临床验证数据集中的30 例前列腺癌患者进行两次阅片并记录癌灶的位置。第一次阅片是在未进行前列腺分割的基础上,在T2WI 图像上记录癌灶的位置。经过一个星期的洗脱期后,进行第二次阅片。第二次阅片是在模型对患者前列腺T2WI 进行分割的基础上记录癌灶的位置。以影像专家浏览mpMRI 各个序列的图像后确定的癌灶位置为参考标准,评估低年资影像医生两次阅片的准确率。
1.7 统计分析
应用SPSS 23.0 软件进行统计学分析。应用方差分析比较数据集A 中训练集、调优集和测试集之间的年龄差异及PSA 水平,t 检验用于比较训练集及测试集中各区域的DSC 及HSD 值。t 检验也用于比较数据集A 和数据集B 之间的年龄及PSA 水平。Bland-Altman 分析评价模型与参考标准对测量前列腺分带区域体积的一致性。P<0.05 认为差异有统计学意义。
2 结果
2.1 数据分布
不同数据集的患者特征及分布如表1 所示,训练集、调优集及测试集之间的患者年龄和PSA 水平均无显著差异(P=0.170 和0.140)。数据集A 和数据集B 之间的患者年龄无显著差异(P=0.542),但数据集B 的PSA 水平显著高于数据集A(P=0.002)。

表1 患者临床特征及分布
在数据集B 中的30 名前列腺癌患者中,共有33 个癌灶。其中17 个位于外周带,8 个位于移行带,4 个位于外周带+移行带中,4 个位于前纤维基质+移行带中。
2.2 模型分割性能
以手工标注的前列腺各区域和尿道区域标签为参考标准,评估测试集中模型的分割性能如表2 所示。在测试集中,模型对外周带、移行带和尿道区域的分割性能较好,其DSC 均大于0.70,精确度、灵敏度及VS 值也均在0.85 以上。中央带和前纤维基质带的DSC 值较低,分别为0.52±0.22 和0.63±0.22,且前纤维基质带的HSD 也为各区域中最大((17.86±14.05)mm)。
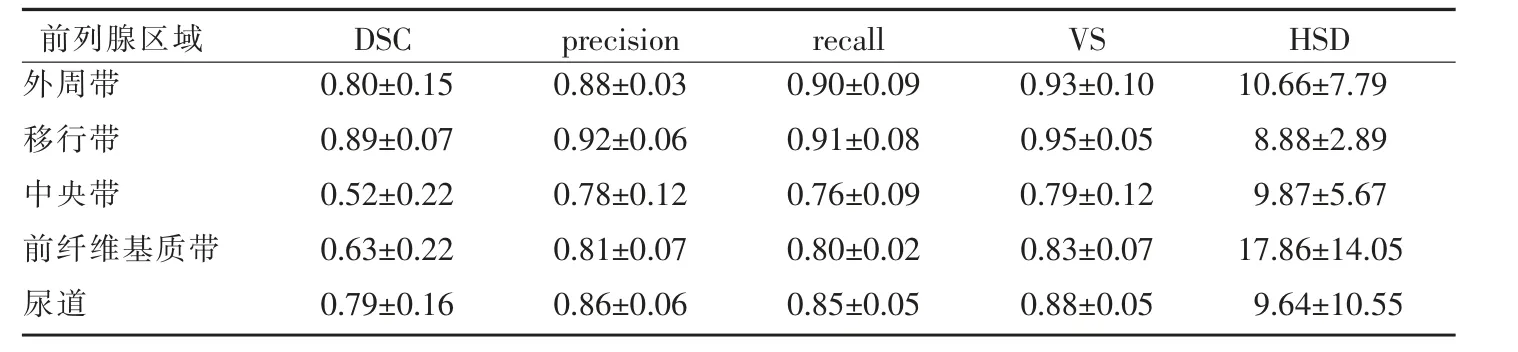
表2 模型对前列腺区域的分割性能
2.3 体积比较
测试集中手工标注及模型预测的前列腺各区域及尿道区域的体积如表3 所示。图4 所示为测试集中两者之间的Bland-Altman 一致性分析,模型预测与手工标注的各区域体积一致性较高,差值基本都位于95%一致范围(limits of agreement,LoA)之内。

表3 前列腺区域分割体积
2.4 临床验证结果
以病灶为单位分析低年资影像医生癌灶的检出及定位准确性如表4 所示。在两次阅片中,33 个病灶均被正确检出,第一次阅片的定位准确率为(93.93%),其中1 处位于外周带+移行带的癌灶被定位为外周带;1 处前纤维基质带+移行带的癌灶被定位为移行带。第二次阅片的准确率为100%(33/33),所有被检出的癌灶均被正确定位。
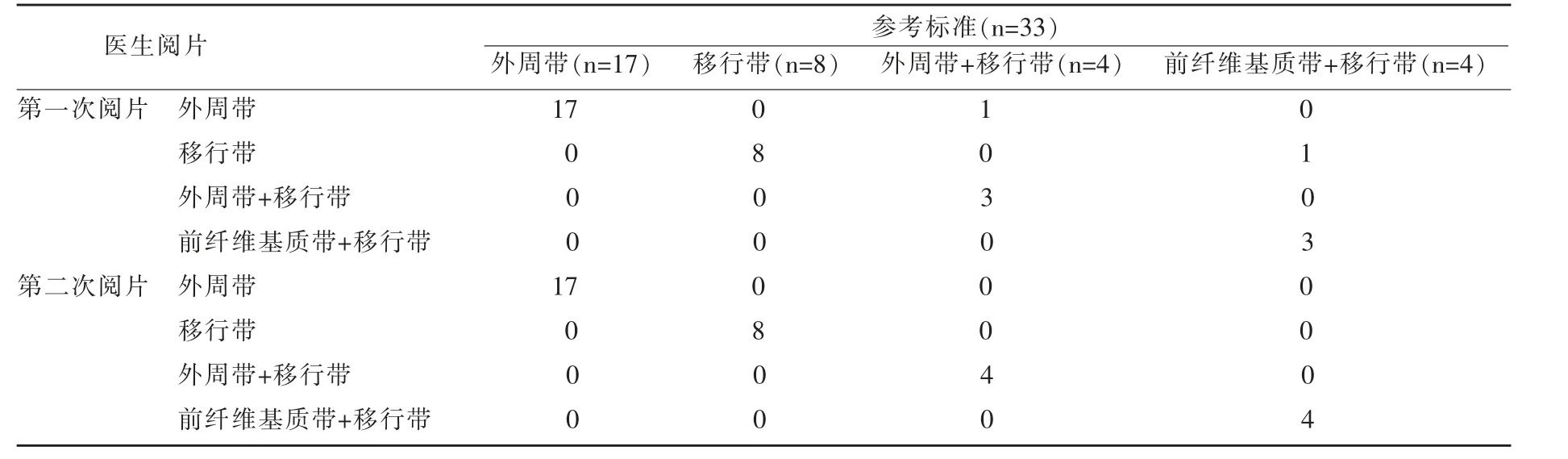
表4 影像医生的两次阅片结果
3 讨论
前列腺mpMRI 检查的主要任务是发现可疑癌灶并精确定位,以指导穿刺活检。根据PI-RADS 2.1指标,前列腺病灶定位时先将腺体分为不同分带,再按照上下关系、左右关系和前后关系将其细分为39个分区[3,14]。因此,mpMRI 诊断时识别腺体的分带解剖是必需的。但是对于经验较少的影像医生,快速准确地对识别腺体分带解剖存在一定的困难。在本研究中,我们应用3D U-Net 模型实现了前列腺分带解剖的精细分割,并在临床验证数据集中证实了该模型辅助医生对于前列腺癌灶定位的可行性。
T2WI 是前列腺癌mpMRI 检查中用于癌灶定位的常规序列,目前基于U-Net 模型在T2WI 进行前列腺区域的自动分割已有较多报道。Nader等[7]通过建立Dense-2 U-net 模型将前列腺自动分割为中央腺体和外周带,其DSC 值分别可达到0.89±0.02 和0.78±0.03。Carina等[15]应用2D U-Net 模型进行前列腺区域分割的DSC 值分别为0.79(中央腺体)和0.69(外周带)。Renato等[16]比较了不同分割模型(UNet vs.ENet vs.ERFNet)对前列腺区域分割的效果,结果显示ENet 模型对前列腺的分割效果最佳,其外周带的DSC 可达到0.71±0.08,移行带可达到0.87±0.05;U-Net 次之,其外周带和移行带的DSC分别为0.70±0.08,0.86±0.07。
与上述既往研究相比,本研究采用3D U-Net模型分割前列腺区域,结果显示模型对T2WI 图像中的前列腺区域分割性能较好,尤其是外周带和移行带,其在测试集中的DSC 可达0.80±0.15 和0.89±0.07。分析其原因,一方面可能是因为我们收集了更多的数据用于模型训练(288 vs.Nader:141 vs.Carina:40 vs.Renato:204)。另一方面,我们对前列腺的区域分割是在前列腺腺体分割的基础上进行的,这样可减少背景的干扰,从而更高效地利用图像信息。另外,本研究所用的3D U-Net 模型不仅可以提取到图像层面内的空间信息,同时还能提取到层间特征信息。相比于2D U-Net 网络能够多增加一个维度的特征[17]。
在本研究中,为了更全面的对模型的分割性能进行评估,我们应用了包括基于重合度、基于体积和基于空间距离的三类分割评估指标。研究结果显示,外周带、移行带及尿道区域的三类评估指标值均高于中央带和前纤维基质带,即模型对外周带、移行带及尿道区域具有较好的分割效果。分析其原因,我们认为可能是因为这三个区域相较于前纤维基质带和中央带而言体积更大且形态更为规则。
与既往的二分区(中央腺体+外周带)分割研究不同[18-19],本研究对前列腺进行了更为精细的区域分割。按照前列腺的解剖分带,我们将其分为四个腺体区域(外周带、移行带、中央带、前纤维基质带)和一个尿道区域。更为精细的分割可提供更为确切的定位信息。模型对于不同区域的分割性能稍有不同。模型对外周带、移行带及尿道的分割具有较好的表现,其DSC 值均在0.70 以上,中央带及前纤维基质带的效果则较差,分别为0.52 和0.63。其原因可能是,与外周带及移行带相比,中央带及前纤维基质带的体积较小,其在模型训练过程中可用于学习的体素和图像特征较少。且中央带、前纤维基质带和移行带在T2WI 图像上的信号特征相似,这三个区域的分割本身就存在一定的难度。虽然DSC 值低于0.70,但模型预测出的体积与手工标注一致性较高,考虑到分带模型的目的是用于定位,仍可认为大致符合临床要求。
为了进一步验证分带解剖分割模型是否可用于辅助医生定位,我们在训练模型之后,应用临床实际前列腺癌患者数据对该分割模型进行了验证。结果显示以癌灶为单位分析,影像医生在模型辅助时对癌灶定位准确率可达到100%,高于其医生直接进行癌灶定位的准确率(93.93%)。因此,我们认为其可满足临床对前列腺癌的定位要求,并有望为后续进行前列腺癌的全自动检出及定位奠定基础。
本研究存在一定的局限性,首先,我们当前只是在单独的非抑脂T2WI 序列上进行前列腺的区域分割,并未对其他如3D T2WI 图像或多模态图像(如增强期图像和DWI 图像)的前列腺分割性能进行研究。因此,在后续研究中,我们可考虑在现有模型中加入其他序列作为输入,来训练一个适用于多序列的前列腺分割模型。其次,本研究入组时排除了腺体结构被肿瘤明显破坏的患者和因术后、治疗后导致腺体结构不清晰的患者。该类患者的前列腺分带解剖会有明显变化,模型对上述情况是不适用的。因此本模型在实际临床应用过程中有必要先进行图像甄别,将明显结构异常的数据自动过滤,才能保证分带解剖分割模型的准确性。第三,当前用于模型训练的数据为单一中心来源,在未来应扩大样本量,加入多中心、多设备及多种临床场景下的患者数据对模型进行迭代和优化,并统一标准建立训练模型,将AI与人的主观能动性有机结合,从而提高前列腺早期癌定位准确率。
总之,本研究基于3D U-Net 训练的前列腺区域分割模型可满足临床对前列腺癌定位的需求,有利于引导穿刺活检,同时也可作为后续前列腺癌全自动检出的定位基础。
