青海省小麦品种基于55K SNP 芯片的遗传多样性分析
2022-12-09谢静敏侯万伟张小娟
谢静敏,侯万伟 ,张小娟
(1. 青海大学生态环境工程学院,青海西宁 810016; 2. 青海省农林科学院, 青海西宁 810016)
小麦是青海省主要的粮食作物之一,但前期为解决当地小麦产量和品质下降的问题,在进行品种培育过程中多次应用同一亲本,导致育成品种之间的遗传关系较近,不利于后续的小麦分子育种发展[1]。
作物遗传多样性在很大程度上影响作物的适应能力、产量等。作物遗传变异对品种改良至关重要,为后续培育出抗逆和优质新品种提供了选择。相较于通过传统表型性状进行遗传多样性评价分析,通过高通量分子标记检测技术能够快速得到高质量的数据,并为遗传多样性分析及分子育种提供大量的资源[2]。目前已经有多种分子标记应用于小麦的遗传育种中,包括RAPD[3]、ISSR[4]、SSR[4]、AFLP[5]、SNP[6]等。SNP在作物基因组中含量丰富,具有操作简单、识别准确、高通量等优势,是遗传作图和分子育种的理想标记[7],已广泛应用于作物的性状鉴定、遗传多样性分析以及遗传图谱的构建中[8-13]。相较于其他作物,小麦SNP芯片发展较晚,目前已开发出9K[14]、15K[15-16]、35K[17]、55K[18]、90K[19]、660K[20]等芯片,并应用于小麦诸多研究领域[21-23]。李珊珊等[24]利用90K SNP芯片对河北省143份小麦品种进行全基因组扫描,根据分型数据发现河北省小麦材料趋同,种质资源狭窄,拓展引进新种质,丰富河北省小麦育种资源已迫在眉睫。马艳明等[1]利用55K SNP芯片分析新疆冬小麦地方品种和育成品种的遗传多样性,发现地方品种与育成品种之间的遗传差异较大,可以利用地方品种来丰富育成品种的遗传基础。
本研究利用55K SNP 小麦芯片对93个青海省主栽小麦品种进行全基因扫描,通过分析多态性信息含量(PIC)和遗传距离来评估青海省小麦品种的遗传多样性,同时对93个小麦品种进行聚类分析,以期为后续青海省小麦的遗传育种提供数据支撑。
1 材料与方法
1.1 试验材料
试验材料包括93个青海省主栽小麦品种(表1),是目前青海省主要生产种植的育成及引进品种。所有供试小麦品种在青海大学试验农场种植,并收集保存。

表1 用于SNP分析的供试小麦品种Table 1 Wheat varieties tested for SNP genotyping
1.2 基因型分析
利用Affymetrix○RAxiom平台的小麦 55K SNP 芯片对93个品种进行全基因组扫描。筛选标准:数据质量控制(DQC)≥0.82,分型成功率(call rate,CR)≥0.95,筛选后得到53 063个原始SNP位点;利用Affymetrix 的Axiom Analysis Suite 软件对原始SNP位点进行深入分型,得到高质量的过滤数据,分型原则选择CR≥0.90、缺失率<10%、最小等位基因频率(MAF)>0.05 的SNP位点,筛选过滤后得到50 243个具有多态性的SNP 位点。
1.3 数据分析
利用PowerMarker V3.25[25]软件计算多态信息含量(polymorphic information content,PIC)和品种之间的Nei遗传距离。根据Nei遗传距离,利用MEGA 6[26]软件构建系统发育树,利用FigTree(http://tree.bio.ed.ac.uk/software/figtree/)和Evolview(http://www.evolgenius.info/evolview)在线作图软件[27]对聚类图进行修饰美化。
2 结果与分析
2.1 多态性SNP位点的分布
在小麦21条染色体上共检测到53 063个SNP位点,选择缺失率<10%、MAF>0.05的SNP位点,获得具有多态性的SNP位点50 243个,多态性比率为94.68%。每条染色体上分布855~2 575个多态性位点,平均每条染色体分布2 216个,6A染色体分布最多(2 575个),占比 5.12%;4D染色体分布最少(855个),占比 1.70%。SNP标记在A、B、D基因组中分布不均,B 基因组上的SNP标记数最多(17 556),占比 34.94%;D 基因组上的SNP标记数最少 (11 904个),占比23.69%(图1A)。
质控后的多态性 SNP 位点在小麦7个部分同源群中的平均分布数量为6 650个, 其中第 2 部分同源群的多态性位点数分布最多(7 152个),第4部分同源群的多态性位点数分布最少 (5 656个)(图1B)。
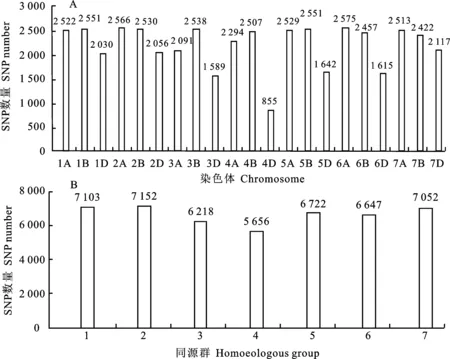
图1 多态性SNP位点在染色体(A)和同源群(B)中的分布
2.2 遗传多样性
PIC是DNA变异程度的指标,是评估遗传多样性的重要标准之一。根据Botstein等[28]提出的PIC衡量标准,PIC值大于0.50的标记属于高度多态性标记,PIC值介于0.25~ 0.50之间的标记属于中度多态性标记,PIC小于 0.25的标记则属于低多态性标记。基于93个材料的SNP标记数据,发现供试品种的PIC值变化范围为 0.00~ 0.59,平均值为0.33,主要分布在0.20~0.45之间,说明所用的大部分SNP标记属于中度多态性标记(图2)。
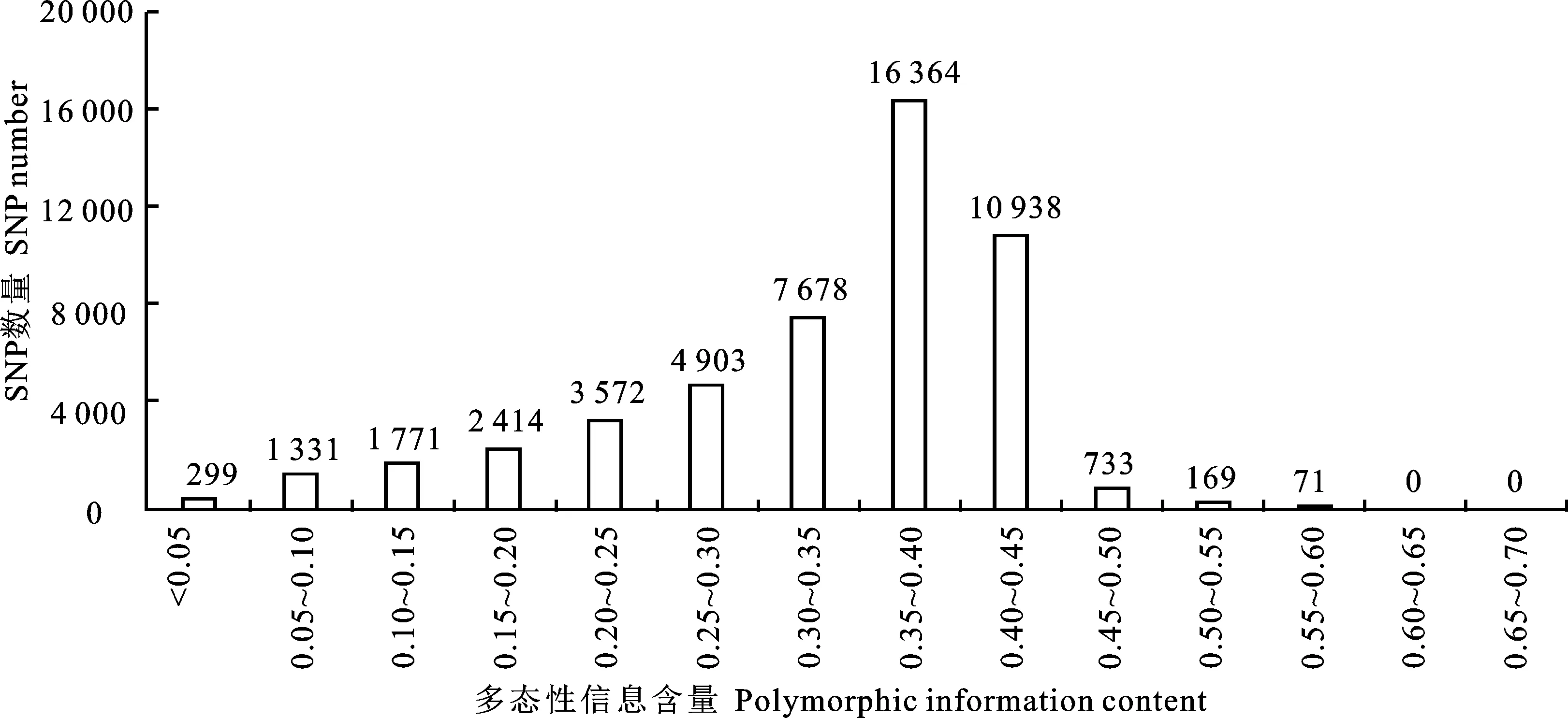
图2 SNP标记在93个小麦中的多态性信息含量分布
遗传距离是评估遗传多样性的重要标准。利用SNP数据计算93个小麦品种之间的遗传距离,共获得4 278个遗传距离值,数据范围在 0.00~0.67之间,平均值为0.41。93个小麦品种之间的遗传距离值分布比较集中, 有3 950个遗传距离值分布在0.30~0.55之间,占比 92.33%(图3)。13799和高原437两个品种之间的遗传距离最大,87号材料和青春10号两个品种之间的遗传距离最小。
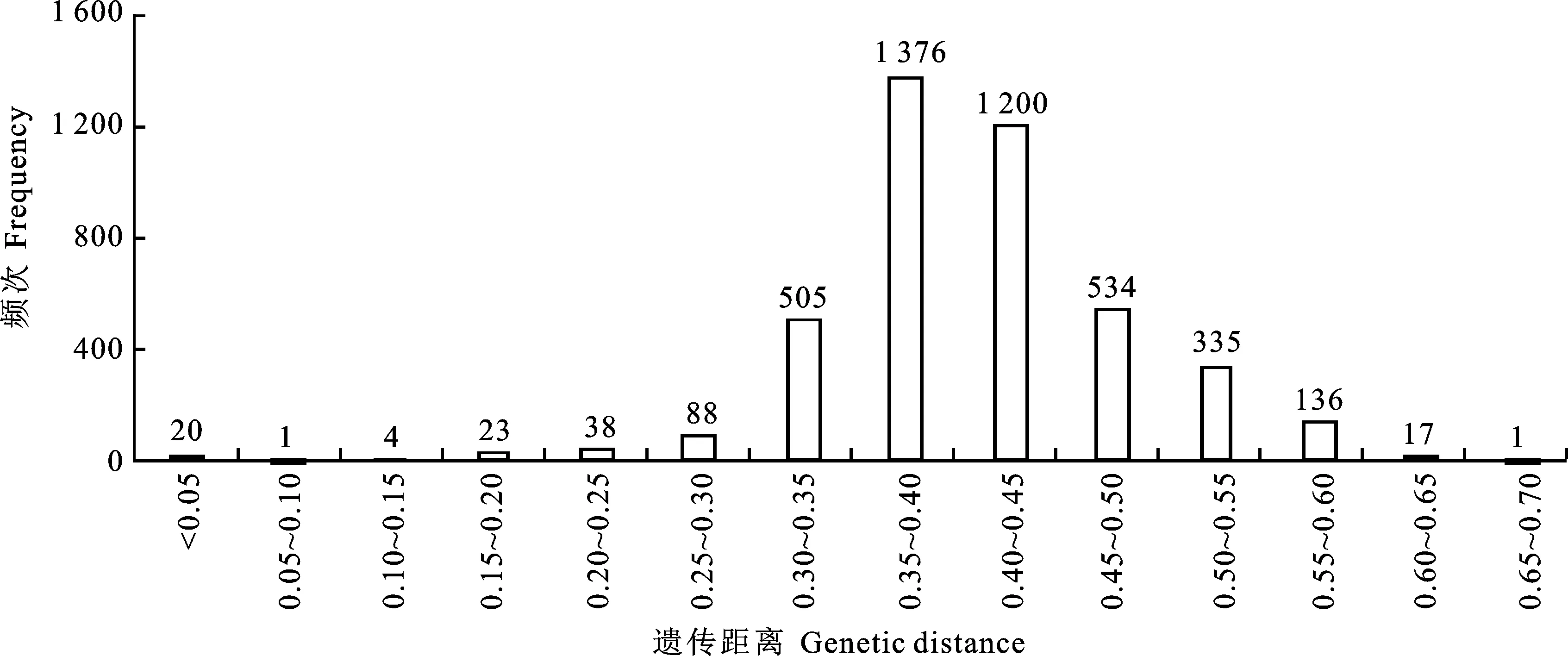
图3 品种间遗传距离的次数分布
2.3 聚类分析
根据所有小麦品种的SNP标记数据,利用Powermarker V3.25得到93个小麦品种两两之间的遗传距离矩阵,并构建系统进化树(图4)。通过聚类可将小麦品种分为8个类群。
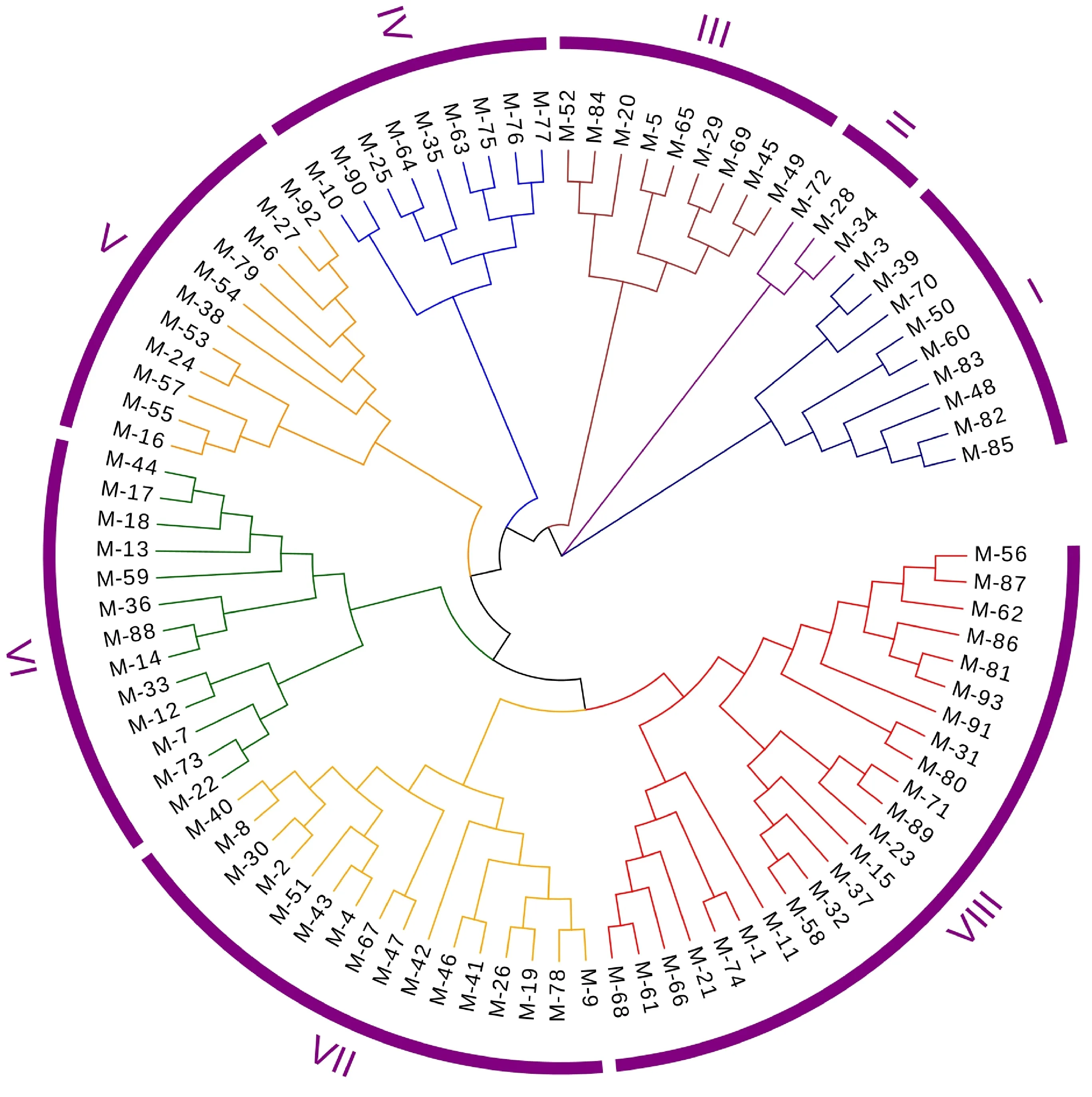
图4 93个小麦品种的 SNP 数据聚类图
第Ⅰ类群包含9个品种,遗传距离在0.13~0.63之间,柴春044与瀚海304之间的遗传距离最小,普冰133与87号材料之间的遗传距离最大。第Ⅱ类群包含3个品种,遗传距离在0.28~0.33之间,宁春26与墨波1号之间的遗传距离最小,宁春26与新麦繁4之间的遗传距离最大。第Ⅲ类群包含9个品种,遗传距离在0.01~0.48之间,ME-47与ME-45之间的遗传距离最小,高原V028与86号材料之间的遗传距离最大。第Ⅳ类群包含9个品种,遗传距离在0.00~0.53之间,青农469与吉农469之间的遗传距离最小,高原584与互助红之间的遗传距离最大。第Ⅴ类群包含11个品种,遗传距离在0.01~0.43之间,青春39与青春41之间的遗传距离最小,13繁530与9101之间的遗传距离最大。第Ⅵ类群包含13个品种,遗传距离在0.15~0.44之间,民和588与民和853之间的遗传距离最小,民和665与兰考906之间的遗传距离最大。第Ⅶ类群包含16个品种,遗传距离在0.05~0.43之间,高原466与高原465之间的遗传距离最小,高原932与杨麦16号之间的遗传距离最大。第Ⅷ类群包含23个品种,遗传距离在0.00~0.52之间,13799与高原437之间的遗传距离最小,阿勃与青春40号之间的遗传距离最大。
根据小麦品种的引进来源进行分析,93个材料主要包括青海省当地所育成的系列品种、外来引进品种(分别从山东、江苏、甘肃、河南、宁夏、意大利、墨西哥引进的品种)以及部分未有具体信息的小麦品种。从聚类结果看,第Ⅱ类群中,于1998年从宁夏引进的宁春26与该类群其他品种间的遗传关系较远;第Ⅵ类群中,于1999年从河南引入的兰考906与该类群其他品种间的遗传关系较远;第Ⅷ类群主要包括青春系列品种,而阿勃是青海省为提高当地小麦抗病能力,于20世纪60年代引进的意大利品种,与类群中其他品种的遗传关系较远,说明青海省当地的育成品种与外来引进品种间的遗传距离较大。青海省当地的育成品种和外来引进品种在8个类群中均有分布,表明研究材料间的遗传关系与引进来源无必然关系。高原系列品种中,选用同一亲本高原602进行培育的3个品种(高原448、高原776、高原437)聚在同一类群,表明品种间的遗传关系与育种所选的亲本相关。
3 讨 论
地方小麦品种具有克服当地特殊环境条件的遗传特性,是小麦分子育种的首选资源。为提高青海省小麦的产量,青海省当地小麦育种以提高作物产量为固定发展方向,以产量较高的小麦品种作为亲本进行连续培育,导致当地小麦的遗传多样性较低,品种的遗传资源基础也越来越窄,无法抵挡青海省当地的特殊气候变化和病害入侵[29-30]。
SNP是在基因组中广泛存在的一类DNA序列变异[31],本研究利用55K SNP对93个小麦材料进行全基因组扫描,发现SNP的多态性比率为 94.68%,在基因组之间SNP位点的分布具有差异。与张德强等[32]和Tehseen等[33]的研究结果一致,多态性SNP位点数在基因组上的分布均呈现为B>A>D。研究表明,小麦B基因组的遗传多样性较高,而来源于粗山羊草的D基因组遗传多样性较低,这可能和六倍体小麦的起源相关,A、B基因组之间能够形成较多的四倍体小麦品种,而D基因组与A、B基因组之间都无法形成四倍体,在一定程度上减少了D基因组的遗传多样性[19]。SNP多态性位点在第4部分同源群中分布最少,特别是4D染色体,这与白彦明等[34]的研究结果一致。
刘易科等[35]利用18 741个标记对240个小麦品种进行全基因组扫描,发现PIC值的变化范围为0.01~ 0.38,平均值为0.26;Kumar等[36]利用35K SNP对483个小麦品种进行基因分型,筛选到 14 650个SNP位点,进一步分析得到PIC值的变化范围为0.09~0.38,平均值为 0.29;左煜昕等[17]利用15 947个SNP标记分析124个抗旱冬小麦品种,发现平均每个位点的PIC值为0.31。本研究利用Powermarker计算93份小麦材料的PIC值,发现PIC值变化范围为0.00~0.59,平均值为0.33,主要分布在0.20~0.45之间,说明所用的SNP标记属于中度多态性标记,与前人研究结论一致。本研究利用SNP数据计算93个小麦品种之间的遗传距离,共获得4 278个遗传距离值,数据范围在0.00~0.67之间,平均值为0.41,主要分布在0.30~0.55之间,占比92.33%,说明93个品种之间的遗传距离分布较为集中,与李珊珊等[24](河北省小麦品种的遗传距离主要分布在 0.52~0.73之间)、曹延杰等[37](河南省小麦品种的遗传距离主要分布在0.63~0.83之间)和马艳明等[1](新疆小麦小麦品种的遗传距离主要分布在0.06~0.12和 0.68~0.72之间)的研究结果相比,青海省小麦品种的遗传距离较小,遗传多样性要小于河北省和河南省小麦品种,但高于新疆部分小麦。
本研究通过聚类分析,将93份材料分为8个类群,当地育种品种与引进品种的遗传关系较远,同时结合品种的来源进行分析,发现遗传关系与品种的来源无必然关系,而与育种所选取的亲本相关,与陈丽华等[38]的研究结论一致。以上研究结果表明,青海省当地的小麦品种遗传多样性较低,在后续的育种工作中可加强外来品种的引用,并结合青海省当地品种,培育出能够适应青海省当地气候且高质量的小麦品种。
4 结 论
利用小麦55K SNP 标记芯片,对93个小麦品种进行全基因组扫描。通过基因分型筛选得到50 243个SNP多态性位点,SNP多态性比率为 94.68%,多态性位点在部分同源群、基因组以及21条染色体上的分布不均,其中第4同源群和D基因组上的多态性位点分布最少,特别是4D染色体。利用Powermarker软件计算小麦品种的基因多样性、PIC值和遗传距离,发现青海省当地主栽品种间的遗传多样性较低。根据两两品种间的遗传距离将93个品种分为8个类群,当地育成品种与引进品种的遗传关系较远。后续可通过利用外来品种和当地优良农家品种进行杂交,培育出可抵御当地特殊气候环境且质量较高的优良品种,丰富青海省当地小麦的遗传多样性。
