影像与基因特征分析方法在阿尔茨海默病中的研究进展
2022-11-24韩立婷姚旭峰赵从义
韩立婷 姚旭峰 金 宇 赵从义 黄 钢
1(上海理工大学医疗器械与食品学院,上海 200082)
2(上海健康医学院医学影像学院,上海 201308)
3(上海市分子影像学重点实验室,上海健康医学院,上海 201308)
引言
阿尔茨海默病(Alzheimer's disease,AD)是最常见的神经退行性疾病[1]。AD 在神经病理学上表现为大脑严重萎缩,细胞外淀粉样斑块和细胞内神经原纤维缠结积聚,这种病变在脑内分布不均且不可逆。AD 已带来巨大的社会和个人压力和沉重的经济负担[2-3],目前尚缺乏对其发病机制的认识以及有效的诊断和治疗方法,也缺乏有效的预防手段,突破这一困境迫在眉睫。
现有研究表明磁共振成像(magnetic resonance imaging,MRI) 和正电子发射断层成像(positron emission tomography,PET)在AD 的诊断和预后评价中发挥重要作用[4-5]。此外,研究也证实AD 患者70%的风险是由复杂的遗传风险因素引起的,其中APOEε4 是导致晚发性AD 发生的主要遗传危险因素,但其单一效应仅占该疾病遗传度的27.3%[6]。
迄今为止,AD 发病的遗传基础尚难以揭示,因为单个基因变异对该疾病遗传风险的贡献微弱,即其很可能是受数百个风险基因和多个环境风险因素的共同作用[7-8]。为此,许多研究者将影像特征与基因特征联合分析,对AD 患者进行分类和预测。
文中首先概述与AD 相关的影像特征和基因特征,随后阐述统计学及机器学习方法在影像与基因特征联合分析中的应用,并讨论其优缺点,最后对影像与基因特征分析方法在AD 应用中的前景进行展望。
1 AD 相关的影像与基因特征
随着研究的不断深入,影像与基因特征在揭示复杂疾病特异信息方面显示了独特优势,特别是在AD 中的应用较为广泛[9]。目前,影像技术中PET和MRI 成像应用最为广泛,其为揭示AD 患者疾病进展提供了可靠的生物标记物,见表1。PET 依标记物可分为氟脱氧葡萄糖PET(fluorodeoxyglucose positron emission tomography,FDG-PET) 和淀粉样PET,FDG-PET 应用较广泛,其主要能显示AD 患者在后扣带回、楔前叶和颞叶皮质等区域的大脑葡萄糖代谢率异常。如Christopher[10]等进行了关于后扣带回葡萄糖代谢率下降的全基因组关联研究,发现后扣带回中葡萄糖代谢的减少与PPP4R3A 基因中的rs2273674 位点存在显著关联,从而揭示了脑内糖代谢的病理机制。与PET 相比,MRI 在AD 诊断中得到了更为广泛的应用,MRI 主要包括sMRI和fMRI。MRI 的相关研究主要是发现了AD 患者的海马、内嗅皮质以及全脑的萎缩速度都在加快。如Kim[3]等利用全脑的平均皮质厚度进行全基因组关联分析(genome-wide association studies,GWAS),发现了4 个基因与皮质厚度有相关性,并鉴定了基因生物功能信息,为AD 的治疗提供了一定的帮助。

表1 主要成像方法的优缺点Tab.1 Advantages and disadvantages of the main imaging method
虽然影像提供了许多重要的AD 疾病表型信息,但由于基因的异质性,要对大量的遗传信息进行联合分析仍需要深入探索。单核苷酸多态性(single nucleotide polymorphism,SNP)是目前研究最为广泛的基因组变体,其可影响蛋白质的表达。起初的研究只能单独分析每个SNP 位点,加上大多数基因关联研究提供的样本量可能太小,难以统计,因此随后逐渐发展为更复杂的GWAS,并迅速成为相关基因研究的主要方法。
Coon[11]等首次完成了针对AD 的GWAS 分析,但仅发现APOE 为AD 的易感基因座,其发现的19号染色体上的SNP(rs4420638)位于APOEε4 变异远端的14 个碱基对,与其他检测的SNP 相比,该SNP 与AD 的患病风险的关联更强。Lambert[12]等使用GWAS 分析,在欧洲血统的人群中新发现了11个AD 的易感基因座,发现的新位点中最强关联的是ZCWPW1 基因,第二强关联的是SORL1 基因,相关基因与常染色体显性遗传和AD 的患病风险增加相关,该研究是截至2018年针对AD 最大的GWAS分析。在2018 和2019年,Marioni[13]等和Jansen[14]等用了更大样本量进行GWAS 分析,该研究发现了更多与AD 相关的易感基因,使其增加至40 个,见表2。除此之外,该研究的基因分析还提供了多种方法,如主坐标分析(principal co-ordinates analysis,PCOA) 和非度量多维尺度分析( nonmetric multidimensional scaling,NMDS)等[15]。随着GWAS应用的不断发展,促进了影像与基因特征联合进行相关性分析[16],逐步发展成脑研究新兴领域,联合分析技术旨在揭示遗传对脑表型的影响,可用于检测AD 的潜在生物标志物,帮助开发新的治疗方法。

表2 基因特征相关的主要研究Tab.2 Major studies related to genetic characteristics
早期的影像与基因特征联合分析研究包括大脑形态和功能与SNP 之间的关联分析[17-18],后来的相关研究更多是利用影像、基因特征等信息对AD进行分类预测,为AD 的诊断提供相应的支持。神经成像能够捕捉一个人从正常衰老到神经退行性疾病的发展过程,而基因变异能够提供最终发展为复杂表型特征倾向的准确信息,因此通过两者联合分析来研究AD,有助于实现更准确的诊断和预测[19]。
2 统计学分析在阿尔茨海默病中的应用
统计学分析方法分为两类,即单变量统计分析方法和多变量统计分析方法。
2.1 单变量分析方法
早期的影像与基因特征联合分析在AD 中的应用研究大多集中于特定的候选基因,对于特定的候选基因变异或感兴趣区,单变量分析是最常用的方法。如表3 所示,Habes 等[20]构建了一个线性回归模型, 通过MRI 与APOEε4 联合分析来确定APOEε4 对患者整个成年期脑萎缩的影响。

表3 单变量分析方法的优缺点Tab.3 Advantages and disadvantages of univariate analysis method
由于单变量分析在特征空间范围内具有极高维度,因此被扩展到基于大量成对单变量分析来处理全脑范围的全基因组扫描。Stein 等[21]提出了基于体素的全基因组关联研究(voxelwise genome-wide association study,VGWAS),探讨了740 名受试者全脑31 622 个体素中的448 293 个单核苷酸多态性之间的关系,共使用300 个计算集群节点并行执行所需的计算,为发现遗传对大脑结构的影响提供了一种新的方法。为解决该方法在速度方面的局限性,Huang 等[22]在此基础上提出了一种更有效的方法,即快速体素式全基因组关联分析(fast genome-wide association study, FVGWAS), 以此加速传统的VGWAS 的计算,FVGWAS 比传统的VGWAS 快几十倍,通过该方法最终成功地确定了3 个基因,即ANK3、MEIS2 和TLR4 与智力低下、学习障碍和年龄有显著的关联。
遗传数据的大规模单变量分析方法目前仍占主导地位,这是因其分析方法简单,且数据所需的模型相对易于拟合,但因局限性,如需进行多项测试,连锁不平衡(linkage disequilibrium,LD)带来的冗余,以及缺乏上位性效应的分析,而对这些局限效应必须进行明确建模,因此单变量分析方法的应用受到很大限制。
2.2 多变量分析方法
单变量分析的局限在于:首先通常需要非常大的样本量才有可能发现有意义的关联,其次其没有考虑到潜在的基因交互作用,很可能导致重大关联的发现力度不足。因此,影像基因特征联合分析的最新方法学引入了多变量方法来捕捉有意义的信息。如偏最小二乘法(partial least-square,PLS)、独立成分分析(independent component analysis,ICA)、典型相关分析(canonical correlation analysis,CCA)等。常用的多变量方法是整合两种或两种以上数据类型,其基本思想是最大化来自不同数据类型变量的线性组合之间的相关性,以此找到相互的关联性。在这些算法的基础之上,又有了改进版本,如:稀疏典型相关分析、稀疏偏最小二乘法和稀疏降秩回归等,并被证明在检测多变量基因组学和脑成像关联方面有效[23-26]。
如表4 所示,Du 等[27]在稀疏典型相关分析基础之上,提出了一种新颖的结构化稀疏规范相关分析模型和优化算法,通过施加两个新的惩罚项,使新方法识别了更高的典型相关系数,其在揭示具有生物学意义的影像遗传关联方面有良好的能力。

表4 多变量分析方法的优缺点Tab.4 Advantages and disadvantages of the multivariate analysis method
Zhou 等[28]提出了一种新的联合投影和稀疏回归模型,以此来揭示表型和基因型之间的关联。为了解决数据异质性、复杂的表型-基因型关联、高维数据(例如,数千个SNPS)和表型异常值等问题,上述方法在基因组到表型预测的平均均方根误差方面优于几种以往的最先进方法。此外,该研究也证实了以往的AD 相关研究中发现的相关SNP 和脑区,从而验证了该方法在AD 发病机制研究中的有效性和潜力。
Lu 等[29]基于贝叶斯广义低秩回归模型(Bayesian generalized low-rank regression,GLRR)扩展形成贝叶斯纵向低秩回归模型(Bayesian longitudinal low-rank regression,L2R2),用来检测40个AD 候选基因的1071 个SNP 对93 个感兴趣区的纵向成像测量的影响,通过对纵向神经影像变量的相关性建模提高了检测能力。Zhu 等[30]提出了一种用于全脑和全基因组关联研究的稀疏回归方法,通过对联合改进框架中的低秩回归和变量的选择来优化目标函数,提高了在SNP 选择方面的性能。Soheili-Nezhad 等[31]使用基于独立成分分析的预处理的脑sMRI 数据,在一组老年受试者的纵向队列中,得出了数据驱动的AD 和轻度认知障碍脑影像特征,即内侧颞路(medial temporal circuit,MTC),并对此进行了全基因组搜索,寻找与这种大脑特征相关的遗传变异。
每种多变量分析方法都有其特殊的优点和局限性,这些方法主要用于单个数据集或者数据量相对较小的研究中,可以在全基因组水平上鉴定出重要的关联。由于大量的输入变量和参数,导致可能存在过拟合和模型无法推广的情况,因此需要在成像或遗传域降维,随着数据集的扩充,可以逐步克服对降维方法的需求。
3 传统机器学习在AD 中的应用
目前对于影像与基因联合分析在AD 中的应用,尚存在诊断环境相对不准确、脑脊液(cerebrospinal fluid,CSF)等诊断的侵入性以及缺乏具有足够AD 诊断专业知识的临床医生等问题,而机器学习可以提供从MRI 等影像数据中获得高精度预测的方法,已在疾病预测与分类方面得到了应用,已有研究引入了机器学习方法,表明其亦可用于AD诊断。
机器学习的能力来自其从大量数据中获得预测模型的能力,这些模型很少或在某些情况下完全不需要数据的先验知识或有关数据的任何假设。当前对AD 诊断方面的应用,大多数机器学习方法都是基于探索MCI 到AD 转换之间的单变量关联,由于不断努力,已逐步构建了基于临床、MRI、实验室和遗传数据融合的多变量预测模型。常用的传统机器学习算法包括线性回归(linear regression,LR)、 逻辑回归、 支持向量机( support vector machine,SVM)和朴素贝叶斯分类器[32-34]。
基于传统机器学习方法的研究如表5 所示。Dukart 等[35]通过构建朴素贝叶斯分类器,研究了多模式成像(MRI、FDG-PET 和淀粉样PET)、神经心理学和遗传数据,将其作为潜在生物标记物,识别其在未来MCI 患者转为AD 的过程中所起的作用,并使用上述数据模态的不同组合来区分AD 和NC。之后,他们将学习到的分类器应用于MCI 队列,以预测AD 转换状态,发现使用FDG-PET 数据达到76%的准确率,使用多模式成像和遗传数据达到87%的准确率。

表5 机器学习方法的优缺点Tab.5 Advantages and disadvantages of machine learning methods
Zhang 等[36]研究了几种机器学习方法,通过结合多模式成像(MRI 和FDG-PET)、CSF 和SNP 数据,比较了3 种最先进的特征选择方法,即:1)多核学习方法(multiple kernel learning,MKL);2)基于高阶图匹配的特征选择(high-order graph matching based feature selection,HGM-FS);3)稀疏多模型学习(sparse multimodal learning,SMML)。经过实验研究发现:1)FDG-PET 是预测精度最高的模式;2)将SNP 数据加入其他模式可以提高预测精度;3)HGM-FS 在3 种特征选择方法中效果最好。
除了这些传统的机器学习方法之外,最近有研究在此基础上进行了一些创新,如Peng 等[37]提出了一种使用多模态成像(MRI 和FDG-PET)和SNP数据进行AD 预测的结构化稀疏核学习(structured sparse kernel learning,SSKL)模型。该研究用内核描述每个特征,并使用模态信息对内核进行分组,以便于在特征和组级别进行变量选择,并进一步引入了一种创新的结构化稀疏正则化项,以实现每个模态内的特征稀疏性,实验结果表明不同的模态可以提供互补的信息,并取得了不错的效果。
机器学习方法对于促进医学和基因组学的进步非常重要。然而,传统的机器学习技术通常需要人工进行特征工程设计,需要对数据降维以精准选择最佳的特征,从而限制了它们在需要实时决策的情况下的实用性。应该看到机器学习的优势在于:1)在小样本上能够实现更好的性能;2)计算成本不高,可以在短时间内尝试不同的技术;3)算法易于理解,在需要调参和更改模型设计时也更简单。总之,每种模型都有其优缺点,在选择时应考虑自身特征和样本等因素,以高性能为目标设计优良的模型。未来构建适宜的机器学习模型任重道远,整合各种模态的信息用于AD 的早期诊断意义重大。
4 深度学习在AD 中的应用
深度学习与传统机器学习的不同之处在于,如何从原始数据中自动发现表示形式。与作为浅层特征学习技术的ANN 相比,深度学习算法使用感知器的多个深层来捕获数据的低级和高级表示,从而使他们能够学习更丰富的输入抽象。这消除了对特征的手动工程的需要,并允许深度学习模型自然地发现以前未知的模式,更好地刻画数据的丰富内在信息[38-39]。深度学习算法可运用单模态到多模态的影像特征,但大多数模型都是在单一数据集上进行训练和测试,多用于疾病的预测和分类。目前,利用深度学习对AD 进行分类预测大多都基于影像特征,关于影像基因联合并用深度学习来预测分类的研究较少。
如表6 所示,Ning 等[40]构建神经网络(neural network,NN)框架,并用sMRI 和SNP 数据对NN 进行训练,发现以大脑和SNP 特征为预测指标的模型AUC 值达0.992。此外,还发现了右侧海马旁回与右侧枕叶回、右侧颞上沟和左侧后扣带回以及rs10838725 与左侧枕叶回之间的关系,表明该模型不仅能够对AD 的进展进行分类和预测,还能够识别重要的AD 风险因素以及之间的相互作用。Pelka[41]等融合社会人口统计数据、遗传数据(APOEε4)和MRI 等信息作为基于长短时记忆网络(long short-term memory,LSTM)的循环神经网络的输入,最终分类精度达到77%的准确度。随着深度学习的不断发展,有研究者使用深度学习模型集成了多个数据域,以发现无法由单个数据域解释的集成特征,即使用基于深度学习的框架将多模态神经影像数据集合并,以区分NC 与AD,从而显著改善了性能。Zhou[42]等提出了一种三阶段深度特征学习和融合框架,将多模态神经影像数据(MRI 和PET)和遗传数据(SNP)融合在一起用于AD 的预测诊断。在第一阶段,独立学习每种模态潜在特征;在第二阶段,组合来自不同模态的潜在特征,并学习组合的联合潜在特征;在第三阶段,使用所有具有完整MRI,PET 和SNP 数据的样本对模型进行训练,该方法主要克服了数据异质性、高维问题和不完整的多模态数据问题,可以提高AD 诊断的准确性,是第一个将多模态神经成像和遗传数据融合在一起用于AD 诊断的深度学习框架。
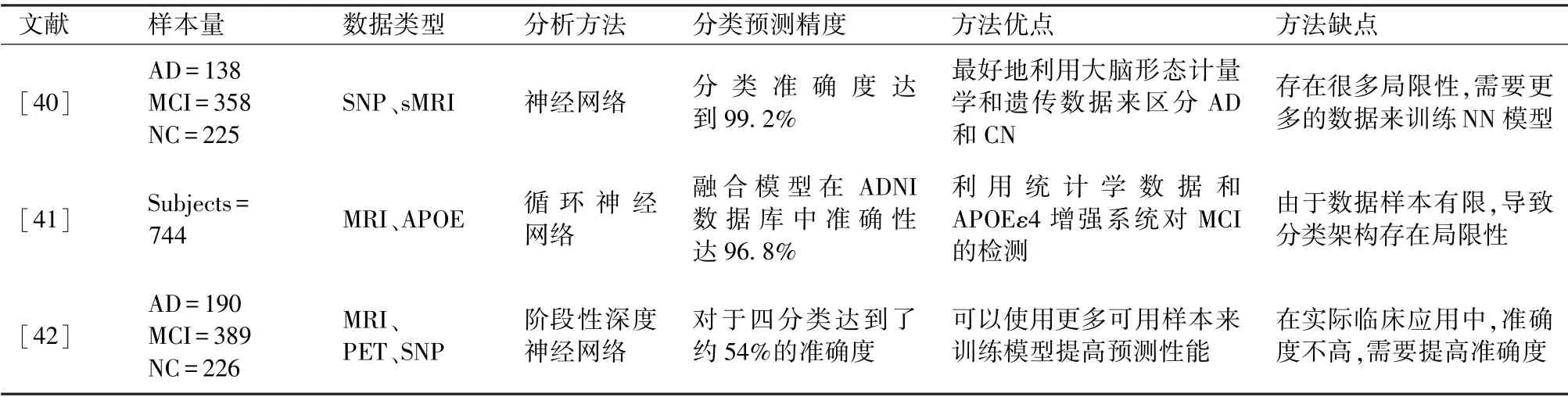
表6 深度学习方法的优缺点Tab.6 Advantages and disadvantages of deep learning method
目前,深度学习在影像基因特征用于结果预测方面尚未广泛应用,已有的研究大多是单独利用影像特征来进行结果的预测,部分原因是受影像和基因组学数据集样本大小的限制。目前深度学习已在AD 早期预测上有了一些尝试,但仍存在诸多缺点,如需要大量的样本训练模型;鲁棒性不强;深度学习是一个“黑盒子”,研究者并不能完全理解网络的内部等缺点,因此相关应用仍旧需要不断完善。未来,深度学习方法是发展影像基因特征联合分析应用中一个有前途的方向。
5 纵向研究
最近纵向研究已逐渐成为影像与基因特征联合分析可依赖的新方向,其目标是为了研究相对于时间AD 的主要变化。Wachinger 等[43]使用纵向成像数据和基因数据来探讨AD 神经解剖的不对称性,提示这些基因可能在AD 中发挥影响的机制或途径。Tabarestani 等[44]在一项研究中通过循环神经网络(recurrent neural networks,RNNs)的两种不同变体,即LSTM 和门控递归单元(gated recurrent unit,GRU)对1458 例受试者的纵向研究中进行了AD 进展的预测。通过利用前三个时间点的患者历史记录,该模型可以在其他三个后时间点跟踪疾病进展,其准确性优于仅依赖基线记录的方法。Du等[45]提出了一种新颖的时间多任务稀疏规范相关分析(multi-task sparse canonical correlation analysis,T-MTSCCA)框架,可以使用纵向神经影像数据来揭示SNP 在一段时间内如何影响脑部定量特征(quantitative traits,QT),结合纵向成像数据和SNP内的关系,T-MTSCCA 可以确定一段时间内渐进式成像遗传模式的轨迹。最后作者验证了ADNI 数据库中408 名受试者的T-MTSCCA,并获得了纵向磁共振成像数据和遗传数据。实验结果表明,TMTSCCA 的性能优于或等同于最新技术。
当前纵向研究的主要挑战是患者在随访中失访或者退出研究造成的数据样本缺失,但发展纵向研究方法仍具有重要意义,它可以进一步帮助揭示多种遗传因素在疾病进展过程中影响大脑变化的作用。
6 总结与展望
影像与基因特征联合分析是一个新的研究热点,是随着获取高通量组学数据和多模式成像数据的最新发展而兴起的一个新兴研究领域。它主要是对基因组的数据、结构、功能和分子成像数据进行综合分析,有可能为正常或紊乱的生物结构、功能的表型特征和遗传机制提供重要的见解。在影像与基因这两个领域中,统计研究对其产生了重大影响,由此也为其统计分析提供了许多大数据挑战。本文重点回顾了分析这类数据的各种方法,其中重点是单变量方法、多变量方法,以及利用传统机器学习方法和深度学习方法对AD 进行分类预测。目前,影像与基因特征联合分析除了应用于AD、PD 等神经退行性疾病,在其他疾病中也有相应的应用。
尽管近年来在技术和方法上有了很大的进步,但有关AD 的影像与基因研究仍然困难重重,且设计、实施和分析的成本高昂。当前,在实践中存在的挑战概况有:1)样本的数量和类型有限;2)这些数据具有高维和复杂的结构,正面临着重大的计算和生物信息学挑战;3)遗传力的缺失通常可以部分归因于遗传变异内或遗传与环境因素之间的相互作用效应(或上位效应),目前所发掘的基因只能解释所研究性状的部分遗传力等问题。随着临床医学中人工智能的深度应用,尤其是深度学习的兴起,相信结合多个数据源的更复杂的模型可以克服诸多挑战,在神经退行性疾病的临床应用方面带来更多突破。
