CUT&Tag技术在代谢组织细胞的实验操作
2022-11-21张子寅周燕萍孟卓贤
张子寅,周燕萍,孟卓贤
实验操作指南
CUT&Tag技术在代谢组织细胞的实验操作
张子寅1,2,周燕萍1,2,孟卓贤1,2
1. 浙江大学医学院病理学与病理生理学系,杭州 310058 2. 浙江大学医学院浙江省疾病蛋白质组学重点实验室,杭州 310058
染色体靶向切割和标签化(clevage under target and tagment, CUT&Tag)技术利用Tn5转座酶与Protein A/G的融合蛋白,引导Tn5酶至与靶蛋白结合的抗体附近,对靶蛋白结合的附近染色质区域进行切割,随后通过标签化处理对片段化染色质进行文库制备,并利用高通量测序技术获取特定位点或蛋白质结合位置的染色质信息。CUT&Tag技术在蛋白质和DNA相互作用的研究领域起到了重大作用,不仅可以了解组蛋白修饰发生的位置,而且可以明确转录因子结合的区域。相较于传统的染色质免疫沉淀高通量测序(chromatin immunoprecipitation- sequencing, ChIP-seq)技术,CUT&Tag技术具有信噪比高、可重复性好、实验周期短、细胞投入量低等优点,在早期胚胎发育、干细胞、肿瘤以及表观遗传学等研究领域体现出巨大优势。本文将针对CUT&Tag在代谢组织细胞(以小鼠原代胰岛细胞为例)的具体操作步骤进行描述,以提供一种研究代谢细胞的表观遗传学方法。
CUT&Tag;组蛋白修饰;操作步骤;代谢组织细胞
蛋白质与DNA的相互作用是基因转录调控的关键,也是启动基因转录的前提。能够与DNA互作的蛋白质主要包括组蛋白、转录因子、DNA甲基化酶和染色质重塑复合物等。为了研究蛋白质-DNA互作,科学家发明了很多方法:凝胶阻滞、DNaseⅠ足迹实验、甲基化干扰、体内足迹、酵母杂交、ChIP- seq等。其中ChIP-seq可以真实、完整地反映结合在DNA序列上的靶蛋白,是一种研究蛋白质-DNA互作的传统且经典的方法[1]。但该技术需要投入大量细胞、步骤繁琐、耗时较长、得到的数据背景较高且需要的测序量大、实验重复性差,这些缺点限制了其在低起始细胞量实验上的应用。因此,研究者在不断寻找新的替代方法。CUT&Tag是近年兴起的一种新型蛋白质–DNA相互作用研究方法[2],该方法利用Protein A/G能和抗体相互作用的原理,通过融合的Protein A/G-Tn5转座子,将转座体富集在靶抗体周围的区域,从而实现目标特异性的片段化反应。相较于传统的ChIP-seq,CUT&Tag操作相对简单、无需甲醛交联、信噪比较高、所需测序量少,且适用于极低起始量和单细胞的应用场景[3]。CUT&Tag的主要流程包括:(1)ConA beads和细胞结合;(2)表面活性剂digitonin透化细胞,并进行一抗和二抗孵育;(3)洗去多余的抗体,通过ProteinA/ G-Tn5转座体使其结合在靶位点附近;(4)洗去多余的转座体,通过镁离子的活化转座体作用,在37℃的条件下进行片段化反应;(5)加入SDS终止反应,乙醇沉淀DNA片段;(6)进行PCR建库以及二代测序获取文库信息[4]。本文将以小鼠原代胰岛细胞为例,针对CUT&Tag在代谢组织细胞的具体操作步骤进行描述。
1 仪器与材料
实验所需的仪器、试剂和耗材详见表1。
2 试剂配制
2.1 结合试剂(Binding buffer)
取200 μL 1 mol/L pH 7.9 HEPES、100 μL 1 mol/L KCl、10 μL 1 mol/L CaCl2、10 μL 1 mol/L MnCl2,使用双蒸水定容至10 mL配制成Binding buffer。Binding buffer可在4℃保存6个月。
2.2 洗涤试剂(Wash buffer)
取1mL 1 mol/L pH 7.5 HEPES、1.5 mL 5 mol/L NaCl、12.5 μL 2 mol/L Spermidine、500 μL Protease inhibitor(100×),使用双蒸水定容至50 mL配制成Wash buffer。Wash buffer试剂可在4℃保存1周。
2.3 毛地黄皂苷洗涤试剂(Dig-wash buffer)
取40 mL Wash buffer,加入400 μL 5% Digitonin,配制成Dig-wash buffer。Dig-wash buffer可在4℃保存2天。5% Digitonin需使用DMSO配制。
2.4 抗体结合试剂(Antibody buffer)
取2 mL Dig-wash buffer,加入8 μL 0.5 mol/L EDTA和20 μL 10% BSA,配制成Antibody buffer。Antibody buffer配制完后需置于冰上预冷,并保证现配现用。
2.5 毛地黄皂苷-300试剂(Dig-300 buffer)
取1 mL 1 mol/L pH7.5 HEPES、3 mL 5 mol/L NaCl、12.5 μL 2 mol/L Spermidine、100 μL 5% Digitonin、500 μL Protease inhibitor(100×),使用双蒸水定容至50 mL配制成Dig-300 buffer。未添加5% Digitonin的Dig-300 buffer可于4℃放置1周,Digitonin应在使用当天加入,以减少沉淀的生成。
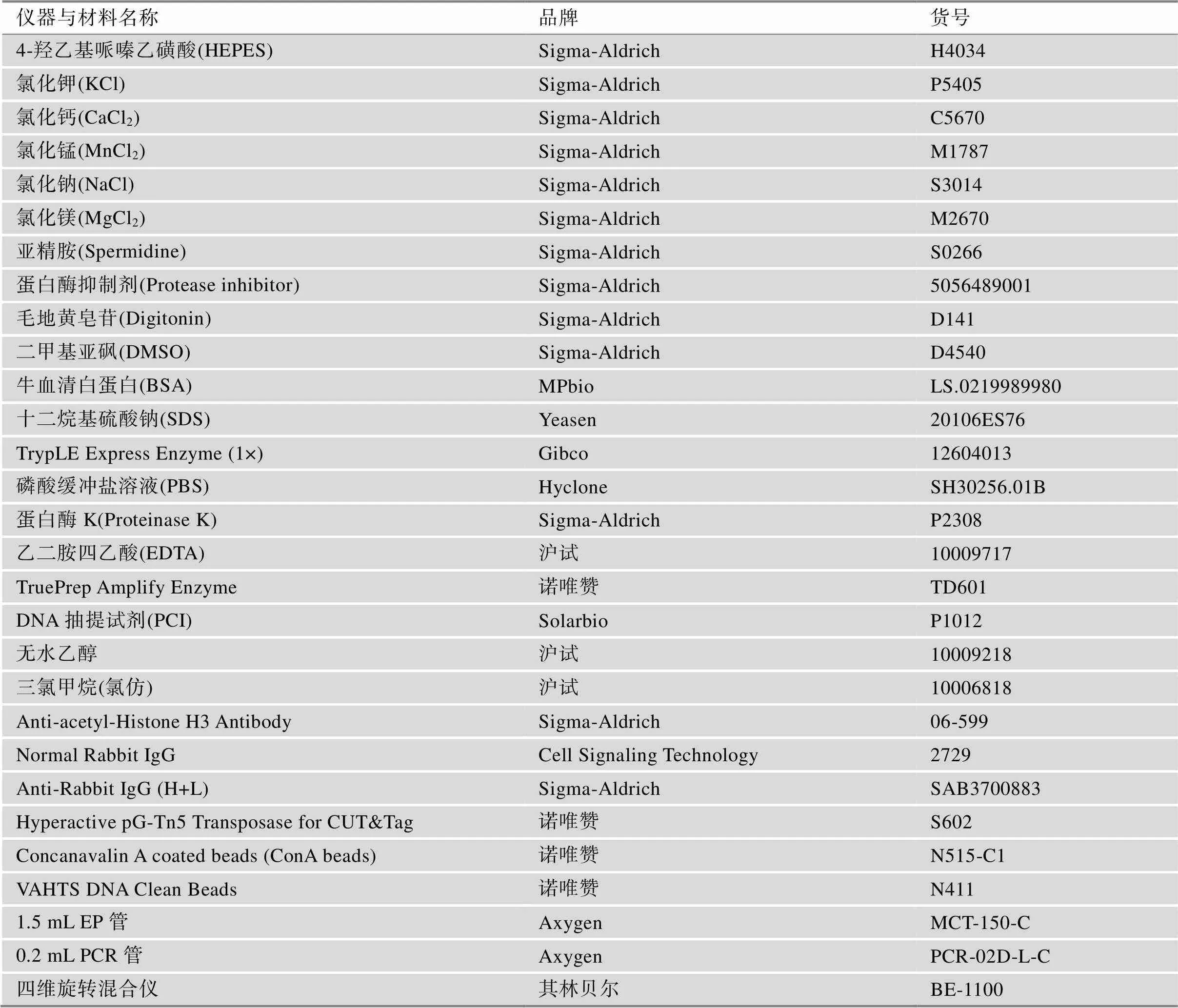
表1 CUT&Tag实验所需的仪器、试剂及耗材信息
2.6 片段化试剂(Tagmentation buffer)
取5 mL Dig-300 buffer,加入50 μL 1 mol/L MgCl2配制成Tagmentation buffer。Tagmentation buffer需要现配现用。
3 细胞获取及ConA beads处理
3.1 ConA beads预处理(以一个样品为例,如有多个样品可按倍数增加试剂用量)
3.1.1 取出10 μL混匀的ConA beads至1.5 mL EP管中。
3.1.2 加入100 μL Binding Buffer(按照体积比Binding Buffer:ConA beads=10∶1),轻轻吹打混匀ConA beads(请勿振荡混匀)。
3.1.3 将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
3.1.4 重复步骤3.1.2、3.1.3一次。
3.1.5 加入10 μL Binding Buffer重悬ConA beads。
注意:
实验操作过程中避免剧烈的震荡,破坏磁珠完整性。
3.2 小鼠原代胰岛细胞的准备
3.2.1 收取大约200个正常大小的小鼠胰岛至新的1.5 mL EP管中,短暂离心(<100 ×)5 s,弃上清。
3.2.2 取2 mL TrypLE Express Enzyme至15 mL管中,转移所有胰岛至该15 mL管中。
3.2.3 于37℃水浴锅中消化10 min,每隔1 min轻弹几下15 mL管,使胰岛完全重悬,每隔5 min,用1 mL移液枪缓慢吹打胰岛8~10下,第一次吹打需使胰岛完全解体至肉眼不可见。
3.2..4 加入4 mL 含2 mmol/L EDTA,0.5% BSA的PBS缓冲液以终止消化,将细胞于600 ×,室温离心5 min,弃掉上清。
3.2.5 室温条件下加入1 mL Wash Buffer重悬细胞,将细胞于600 ×室温离心5 min,弃尽上清。
3.2.6 重复步骤3.2.5一次。
3.2.7 视所得细胞量加入0.5~1 mL Wash Buffer重悬细胞,进行细胞活力染色,计数并计算细胞活力,保证细胞活力在80%以上。
3.2.8 取适量细胞(本实验适用的细胞投入量为60~ 100000个/样品)于新的1.5 mL EP管中,使用Wash Buffer定容至100 μL。
注意:
1) 细胞活力对于实验成功与否至关重要,一定要保证细胞的活力在80%以上,最好在90%以上。
2) 在进行细胞染色、计数等涉及到吸取细胞的过程前,需要混匀细胞,以确保细胞吸取数量的准确性。
3) 在细胞通透之前的所有步骤都在室温下进行,使细胞受到的刺激最小化。
4 CUT&Tag文库构建操作
4.1 ConA beads与细胞结合
4.1.1 低速振荡含有细胞的EP管,同时加入10 μL混匀的ConA beads悬液,室温下置于四维旋转混合仪上旋转孵育10 min。
4.1.2 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
注意:
1) 使用ConA beads前应进行预实验计算beads富集效率。
2) ConA beads和细胞结合后的所有步骤应避免开盖时间过长,否则会出现暴露于空气导致beads干燥从而影响实验结果。
4.2 孵育一抗
4.2.1 按照一定比例(1:50)配制含有目的一抗的Antibody buffer,置于冰上。
4.2.2 在ConA beads与细胞结合复合物中加入50 μL预冷的 Antibody Buffer,轻轻吹打混匀,置于冰上。
4.2.3 置于四维旋转混合仪上旋转室温孵育2 h或4℃孵育过夜。
注意:
1) 四维旋转混合仪转动时,请确保液体一直保持在管的底部和侧面,中途可轻弹管底混匀,缓解磁珠干结。
2) 每一次实验需要设置阳性对照抗体(如H3Ace、H3K4me3)和阴性对照抗体(如Normal Rabbit IgG)
4.3 孵育二抗
4.3.1 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
4.3.2 按照一定比例(1∶100)配制含有目的二抗的Dig-wash Buffer。
4.3.3 在ConA beads与细胞结合复合物中加入100 μL Dig-wash Buffer,轻轻吹打混匀。
4.3.4 置于四维旋转混合仪上旋转室温孵育1 h。
4.3.5 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
4.3.6 加入1 mL Dig-wash Buffer,轻轻吹打混匀。
4.3.7 重复步骤4.3.5、4.3.6两次。
注意:
四维旋转混合仪转动时,请确保液体一直保持在管的底部和侧面,中途可轻弹管底混匀,缓解磁珠干结。
4.4 pA/pG-Tn5 Transposase孵育
4.4.1 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
4.4.2 按照1∶100的比例配制含有Hyperactive pG/pA-Tn5 Transposase的Dig-300 Buffer(终浓度为0.04 μmol/L)。
4.4.3 在ConA beads与细胞结合复合物中加入100 μL pG/pA-Tn5-Dig-300 Buffer混合液,轻轻吹打混匀。
4.4.4 置于四维旋转混合仪上旋转室温孵育1 h。
4.4.5 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
4.4.6 加入1 mL Dig-300 Buffer,轻轻吹打混匀。
4.4.7 重复步骤4.4.5、4.4.6两次。
注意:
1) 四维旋转混合仪转动时,请确保液体一直保持在管的底部和侧面,中途可轻弹管底混匀,缓解磁珠干结。
2) NaCl浓度的增加会减缓Hyperactive pG /pA-Tn5 Transposase与染色质可开放区域的结合。
3) Dig-300 Buffer加入后有可能会出现白色沉淀,不影响孵育和清洗。
4.5 激活转座子,进行DNA片段化
4.5.1 短暂离心(<100 ×)收集反应液于管底,将EP管置于磁力架上,静置2 min左右,待溶液澄清后弃尽上清。
4.5.2 加入 300 μL Tagmentation Buffer,轻轻吹打混匀。
4.5.3 37℃孵育1 h,期间可轻弹混匀2~3次。
4.6 DNA提取
4.6.1 室温下,每个反应中加入10 μL 0.5 mol/L EDTA,3 μL 10% SDS和2.5 μL 20 mg/mL蛋白酶K,剧烈震荡混匀,终止片段化反应。
4.6.2 短暂离心收集液体于管底,50℃孵育1 h(或者37℃孵育过夜)。
4.6.3 加入300 μL DNA抽提试剂(PCI,苯酚:氯仿:异戊醇=25∶24∶1,注意不要取到上层水相),上下剧烈摇晃10次,然后高速震荡2 s。
4.6.4 16000 ×,室温离心5 min。
4.6.5 小心吸取上层水相(约250 μL)至新的1.5 mL EP管中,加入300 μL氯仿,上下颠倒10次(请勿振荡),16000 ×室温离心5 min。
4.6.6 小心吸取上层水相(约180 μL)至新的1.5 mL EP管中,加入750 μL无水乙醇,上下颠倒10次,置于冰上。
4.6.7 冰上冷却后,标记好离心方向,4℃,16000 ×离心15 min。
4.6.8 小心弃尽液体,并倒扣在纸巾上将液体沥干。
4.6.9 加入1 mL预冷的无水乙醇漂洗,4℃,16000 ×离心5 min。
4.6.10 小心地倒出液体并在纸巾上沥干后,在空气中倒扣晾干。
4.6.11 待管干燥后,加入30 μL 1 × TE(10 mmol/L Tris-HCl[pH 8.0], 1 mmol/L EDTA, RNsseA)。
4.6.12 37℃孵育10 min。
4.6.13 直接进行后续步骤或置于-80 °C储存。
注意:
无水乙醇沉淀DNA时往往沉淀物不可见,此时应小心弃尽上清,避免将沉淀物倒出。
4.7 文库扩增
4.7.1 在PCR管中按如下体系配制50 μL PCR反应体系:30 μL片段化的DNA、7μL双蒸水、10 μL 5×TAB、1 μL 20 μmol/L N5 index引物、1 μL 20 μmol/L N7 index引物、1 μL TAE。
注意:
CUT&Tag建库过程使用双端index引物,因此需在PCR体系中同时添加N5和N7 index引物,后续测序策略选择双端测序。
4.7.2 轻轻吹打混匀PCR体系,在PCR仪中进行如下反应:72℃ 3 min→98℃ 30 s→(98℃ 15 s、60℃30 s、72℃ 30 s)12~15个循环→72℃ 5 min→4℃保存。
注意:
1) 可根据实际情况或参考文献选择合适的扩增循环数。
2) 文库产量与细胞类型、细胞投入量、靶蛋白的丰度及PCR的循环扩增产物存在一定关系,实验时不宜追求过高的文库产量而设置过高的PCR循环数,防止因高PCR循环数引起过度扩增,使文库DNA片段呈现大片段分布。
4.8 文库纯化
4.8.1 提前20 min将VAHTS DNA Clean Beads置于室温平衡,随后震荡使其充分混匀,加60 μL VAHTSDNA Clean Beads 到上述PCR反应产物中,吹打混匀10次以上保证整个体系均匀。
4.8.2 室温孵育5 min。
4.8.3 将反应管短暂离心并置于磁力架上分离磁珠和液体。
4.8.4 保持PCR管在磁力架上,待溶液澄清(约5 min),弃去上清,注意不要扰动到磁珠。
4.8.5 保持PCR管在磁力架上,加入200 μL新鲜配制的80%乙醇,加入乙醇时不要扰动磁珠,孵育30 s后小心移除上清。
4.8.6 重复步骤4.8.5一次。
4.8.7 开盖干燥3~5 min。
4.8.8 磁珠晾干后,将PCR管从磁力架上取出,加入22 μL 双蒸水覆盖磁珠,吹打混匀磁珠。
4.8.9 室温孵育5 min(如果磁珠干燥开裂,适当延长孵育时间)。
4.8.10 将PCR管短暂离心收集置于磁力架中分离磁珠和液体,直到溶液澄清(约5 min)。
4.8.11 小心吸取20 μL上清转移到新的EP管中。
注意:
不同地区环境干湿程度有差别,磁珠晾干时间不一,磁珠刚好晾干,表面无光泽即可。过度干燥会导致洗脱困难,未完全晾干会有酒精残留影响后续实验反应。该纯化步骤也可以使用其他文献报道的纯化方式[5]。
5 文库质量检测
5.1 2%琼脂糖凝胶电泳初步判断文库质量
一般而言,H3K4Me3的CUT&Tag文库在凝胶电泳图中呈现150~1000 bp的弥散样条带,而IgG的文库则无明显条带(图1)。
5.2 Qubit检测文库浓度
使用Qubit定量技术,对文库进行浓度检测。一般而言,组蛋白修饰的文库所得DNA浓度偏高,而转录因子或其他靶蛋白所得文库浓度会较低。检测得到的文库浓度只能反映是否含有DNA及其浓度,无法作为文库构建成功的最终指标,需要经过下一步的文库片段分布检测才可判断文库是否构建成功,也可以不进行浓度检测,直接进行文库片段分布检测。
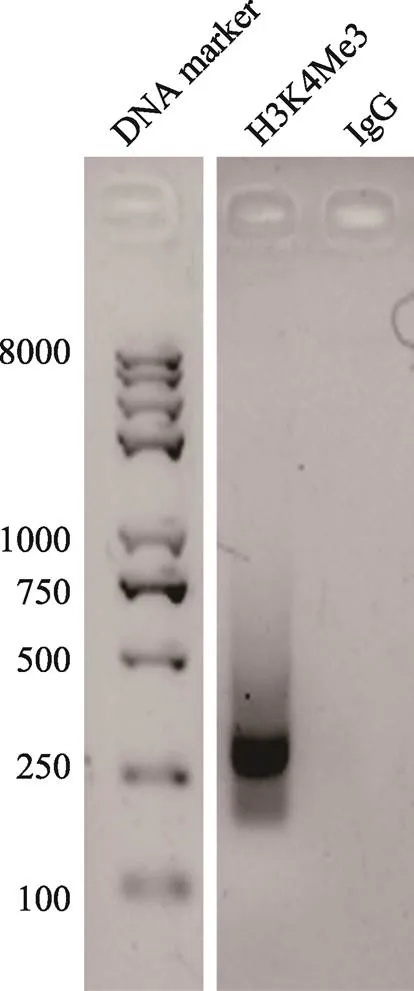
图1 使用琼脂糖凝胶电泳初步检测小鼠原代胰岛细胞的CUT&Tag文库质量
5.3 文库片段分布检测
使用Agilent 2100 Bioanalyzer或等效产品,进行文库长度分布检测。一般而言,H3K4Me3的CUT&Tag文库峰型图呈现核小体倍数大小的阶梯状,而IgG的文库峰应普遍很低(图2)。
注意:
实验中研究的靶蛋白不同,文库峰型存在一定差异。研究组蛋白修饰的文库峰型图呈现核小体倍数大小的阶梯状;转录因子存在靶蛋白特异性,无固定峰型,在180~1000 bp之间有片段分布便可进行测序。
6 小鼠胰岛细胞CUT&Tag测序、分析流程及典型结果
6.1 上机策略
样品送至安诺优达(北京)公司,使用Illumina测序平台进行高通量测序,测序读长为PE150。
6.2 分析流程
上游分析步骤参考弗雷德-哈钦森癌症研究中心疫苗和传染病部Ye Zheng提供的分析流程(https://yezhengstat.github.io/CUTTag_tutorial/),做完peaks在基因转录起始位点(transcription start site, TSS)和转录终止位点(transcription end site, TES)附近的可视化富集分析后,可以得到如下代表热图结果(图3)。同时使用IGV将校准后的bigwig文件可视化,可以得到H3K4Me3和IgG的peaks在小鼠基因附近分布的结果(图4)。
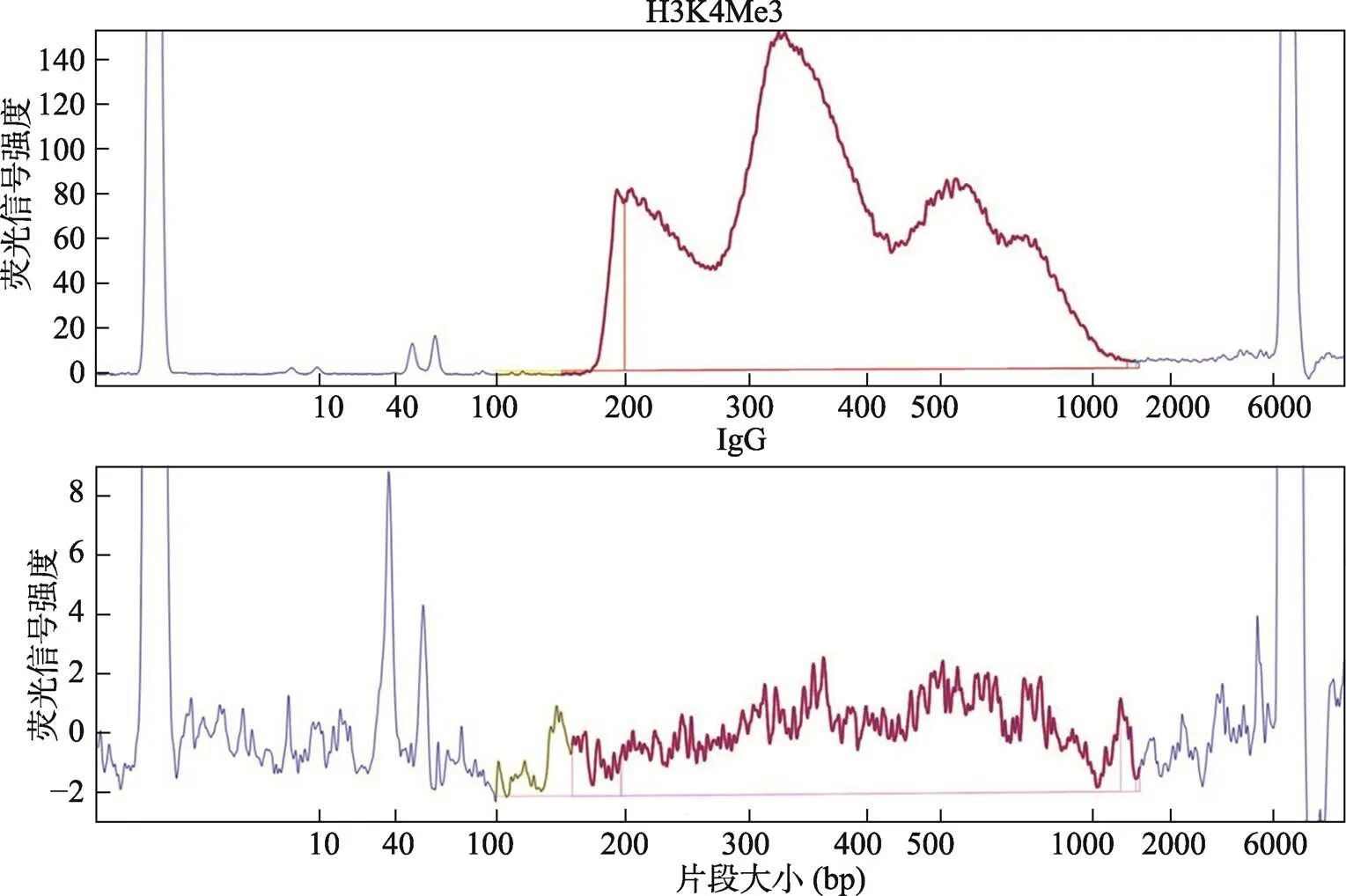
图2 使用LabChip Touch检测小鼠原代胰岛细胞的CUT&Tag文库质量
7 结语
由于传统的ChIP-seq实验需要的高细胞投入量,导致在胰岛领域研究蛋白质(转录因子)-DNA互作是一项极其困难的工作:为了拿到一个好的ChIP- seq实验结果往往需要投入上百只实验小鼠的胰岛细胞[6]。而CUT&Tag这种新兴的蛋白质-DNA互作研究方法的问世,则可以解决传统ChIP-seq方法所需要的高细胞投入量、产生结果的低重复性、低信噪比等问题。自问世以来,CUT&Tag已经被应用在多种细胞的研究中,但在小鼠原代胰岛细胞中仍未被证实适用。本文描述了针对小鼠原代胰岛细胞CUT&Tag的具体操作步骤,并利用本实验室已成功测序的案例,提供了一种研究胰岛细胞的表观遗传学方法,证明了CUT&Tag在胰岛细胞中的可行性。与此同时,本实验室以往的研究也表明CUT&Tag不仅可以检测胰岛细胞中组蛋白-DNA的互作,也可以用于研究胰岛转录因子-DNA之间的互作[7,8]。
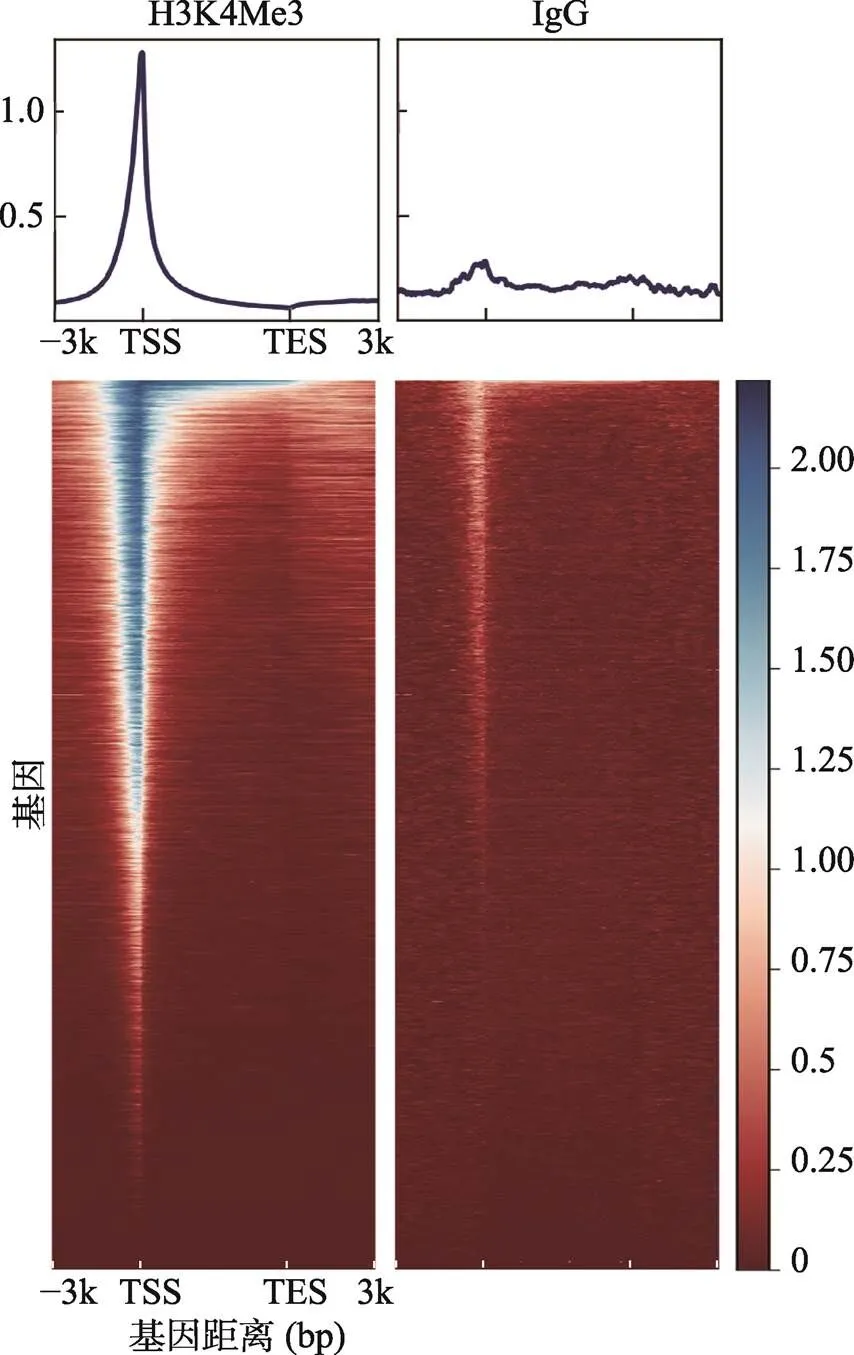
图3 小鼠胰岛细胞H3K4Me3和IgG的CUT&Tag peaks在基因转录起始位点(TSS)和转录终止位点(TES)周围富集的热图

图4 小鼠胰岛细胞H3K4Me3和IgG的CUT&Tag peaks在Slc2a2基因周围的分布
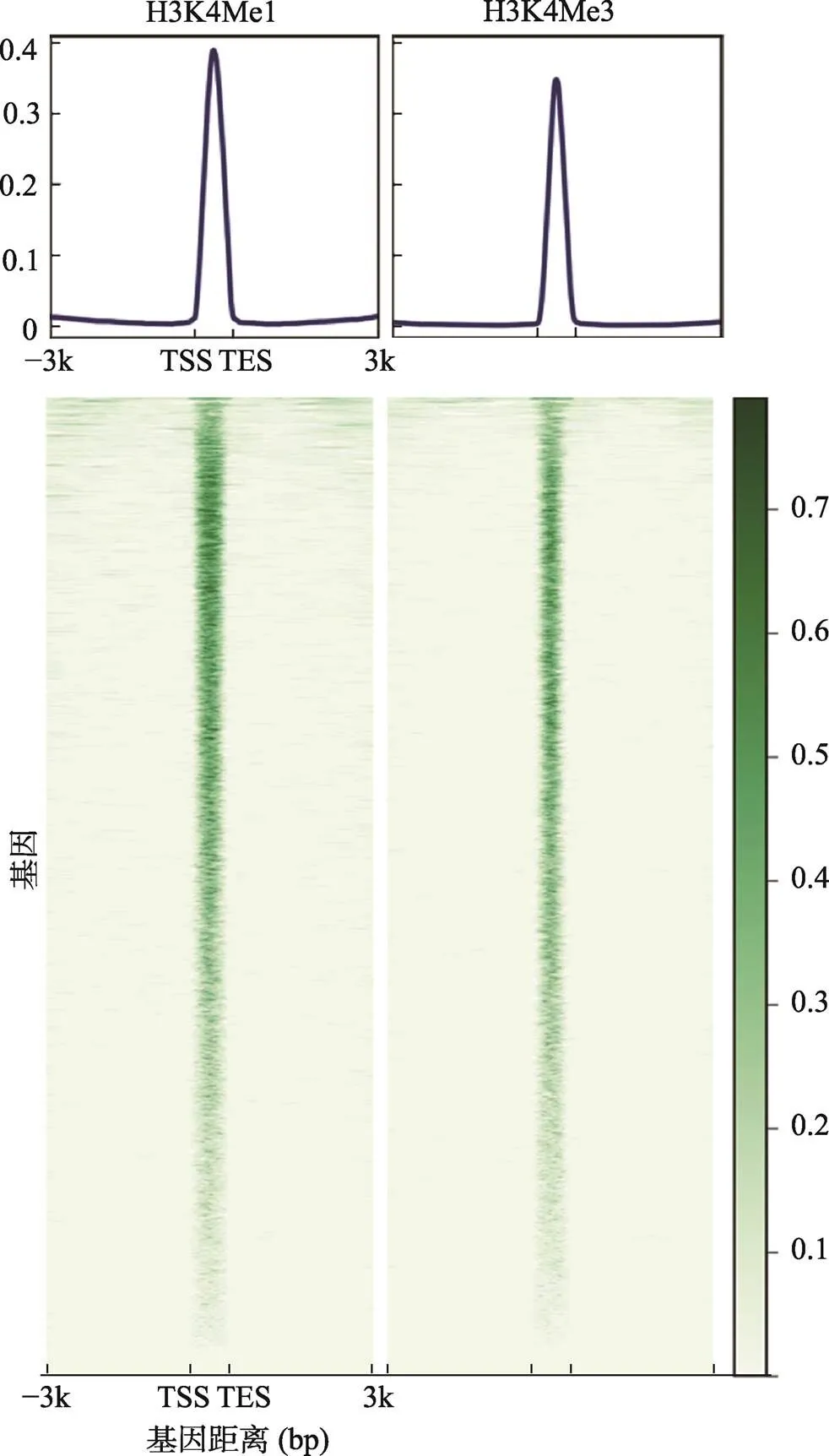
图5 小鼠胰岛细胞的CTCF CUT&Tag peaks在H3K4Me1、H3K4Me3 ChIP-seq数据中距离TSS-3到+3 kb区域的富集情况
Kaya-Okur等[2]在2019年最早介绍CUT&Tag技术的时,便比较了ChIP-seq和CUT&Tag在测序结果上的差异:当二者采用相同测序深度(8×106reads)时,CUT&Tag测到的组蛋白修饰基因位点约为ChIP-seq的20倍,同时CUT&Tag拥有比ChIP-seq更高的信噪比、更好的重复性。并且,Henikoff团队通过CTCF 的CUT&Tag表明了其对于转录因子结合位点的检测也更加灵敏[2]。在本文中,小鼠胰岛细胞H3K4Me3的CUT&Tag所读取到的peaks有大约1/2分布在启动子区域,这和组蛋白H3K4Me3会和启动子区域结合的特性一致,说明了该方法在小鼠胰岛CUT&Tag测序上的准确性。而且,本实验室之前的研究中也发现小鼠胰岛CTCF的CUT&Tag结合位点在启动子和增强子均有很强的结合(H3K4Me1和H3K4Me3 ChIP-seq数据来源于SRA数据库:SRP000660)(图5)。
综上所述,本文为实现小鼠原代胰岛细胞CUT&Tag提供了详细的操作步骤和结果,为研究胰岛细胞的蛋白质-DNA互作以及表观遗传学提供了一种可推广的方法。
[1] Kidder BL, Hu GQ, Zhao KJ. ChIP-Seq: technical considerations for obtaining high-quality data., 2011, 12(10): 918–922.
[2] Kaya-Okur HS, Wu SJ, Codomo CA, Pledger ES, Bryson TD, Henikoff JG, Ahmad K, Henikoff S. CUT&Tag for efficient epigenomic profiling of small samples and single cells., 2019, 10(1): 1930.
[3] Henikoff S, Henikoff JG, Kaya-Okur HS, Ahmad K. Efficient chromatin accessibility mapping in situ by nucleosome-tethered tagmentation., 2020, 9: e63274.
[4] Kaya-Okur HS, Janssens DH, Henikoff JG, Ahmad K, Henikoff S. Efficient low-cost chromatin profiling with CUT&Tag., 2020, 15(10): 3264–3283.
[5] Wei Y, Li K, Lu DR, Zhu HX. Optimization of CUT&Tag product recovery and library construction method., 2021, 43(4): 362–374.
韦晔, 李科, 卢大儒, 朱化星. CUT&Tag产物回收和建库方法的优化. 遗传, 2021, 43(4): 362–374.
[6] Taylor BL, Liu FF, Sander M. Nkx6.1 is essential for maintaining the functional state of pancreatic beta cells., 2013, 4(6): 1262–1275.
[7] Qiao JT, Zhang ZY, Ji SH, Liu TL, Zhang XN, Huang YM, Feng WL, Wang KL, Wang JY, Wang SS, Meng ZX, Liu M. A distinct role of STING in regulating glucose homeostasis through insulin sensitivity and insulin secretion., 2022, 119(7): e2101848119.
[8] Wang RR, Qiu XY, Pan R, Fu HX, Zhang ZY, Wang QT, Chen HD, Wu QQ, Pan XW, Zhou YP, Shan PF, Wang SS, Guo GJ, Zheng M, Zhu LY, Meng ZX. Dietary intervention preserves β cell function in mice through CTCF-mediated transcriptional reprogramming., 2022, 219(7): e20211779.
The protocol of CUT&Tag for metabolic tissue cells
Ziyin Zhang1,2, Yanping Zhou1,2, Zhuo-Xian Meng1,2
Cleavage under target and tagment (CUT&Tag) is a technology that utilizes the fusion protein of Tn5 transposase and protein A/G which can guide Tn5 enzyme to the antibody bound to target protein and cleave the chromatin regions adjacent to target protein. Chromatin libraries are then tagged and sequenced by the high-throughput sequencing to obtain chromatin information at specific sites or protein binding locations. CUT&Tag technology plays an important role in the research of DNA and protein interactions. It can be used to understand the modifications of histone and the bindings of transcription factors. Compared with the traditional chromatin immunoprecipitation-sequencing (ChIP-seq) technology, the CUT&Tag has the strengths of high signal-to-noise ratio, good repeatability, short experimental period, and low cell input. It shows great advantages in early embryonic development, stem cells, cancer, epigenetics and other research fields. In this article, we described the protocol of CUT&Tag for metabolic tissue cells (mouse primary islet cells), to provide an epigenetic method for studying metabolic cells.
CUT&Tag; histone modification; protocol; metabolic cell
2022-08-01;
2022-09-20;
2022-10-03
国家自然科学基金重大研究计划(编号:91857110),国家重点研究发展计划(编号:2018YFA0800403、2016YFC1305303),国家自然科学基金(编号:81670740),国家优秀青年科学基金(编号:81722012),浙江省自然科学基金(编号:LZ21H070001),浙江大学基础医学创新研究院、中央高校基本科研专项基金、杭州市医学重点学科建设基金(编号:OO20200055)、王宽诚教育基金资助 [Supported by the Training Program of the Major Research Plan of the National Natural Science Foundation of China (No. 91857110), the National Key Research and Development Program of China (Nos. 2018YFA0800403, 2016YFC1305303), the National Natural Science Foundation of China (No. 81670740), the National Natural Science Fund for Excellent Young Scholars of China (No. 81722012), the Zhejiang Provincial Natural Science Foundation of China (No. LZ21H070001), the Innovative Institute of Basic Medical Sciences of Zhejiang University, and the Fundamental Research Funds for the Central Universities, the Construction Fund of Medical Key Disciplines of Hangzhou (No. OO20200055), and K.C. Wong Education Foundation]
张子寅,硕士研究生,专业方向:病理与病理生理学。E-mail: ziyinzhang@zju.edu.cn
孟卓贤,博士,教授,研究方向:病理与病理生理学。E-mail: zxmeng@zju.edu.cn
10.16288/j.yczz.22-253
(责任编委: 周红文)
