水稻幼苗响应氰化物胁迫的基因表达谱差异分析
2022-11-16张伏中蔡青宋江燕胡龙兴吴根义
张伏中,蔡青,宋江燕,胡龙兴,吴根义
水稻幼苗响应氰化物胁迫的基因表达谱差异分析
张伏中1,蔡青1,宋江燕1,胡龙兴2,吴根义3*
(1.湖南省环境保护科学研究院,湖南 长沙 410004;2.湖南农业大学农学院,湖南 长沙 410128;3.生态环境部华南环境科学研究所,广东 广州 510655)
为筛选水稻参与氰化物胁迫响应的关键基因,采用Agilent4×44 K水稻全基因组芯片,分析了氰化钾和铁氰化钾胁迫下水稻幼苗叶片和根系的基因表达特征。结果表明:氰化钾和铁氰化钾处理下,从水稻根系中分别筛选到1841和3037个差异表达基因,从水稻叶片中分别筛选到734和1805个差异表达基因;氰化钾处理下,细胞壁代谢和抗逆相关的基因在根中显著上调表达;铁氰化钾处理下,水稻叶中解毒相关基因显著上调表达,说明氰化物能诱导水稻基因差异表达;与细胞解毒作用和抗逆相关的基因、与细胞壁和次生代谢途径相关的基因以及转录因子类基因的表达均显著上调,表明2种氰化物处理下水稻根系和叶片均可能通过调节其体内的一些代谢途径和一些酶的催化活性来应对氰化物的胁迫,积极诱导与防御相关的基因,并通过主动吸收和转运等代谢过程将氰化物代谢解毒,以降低对植物的伤害。
水稻;氰化物;胁迫;基因表达特征;差异分析
氰化物(cyanide)对生物有机体和环境都有剧毒。自然环境中存在多种含氰化合物,如:游离氰化物、易分解的弱酸性氰化物、铁氰络合物和硫氰酸盐等[1]。在环境中检测到的含氰根化合物中,铁氰化物是最常见、最稳定的化合物[2],它与游离氰化物(CN–与HCN)联系密切[3]。研究[1,4–8]表明,植物在一定的浓度范围内可以吸收、运输和同化氰化物,但植物对非生物胁迫的响应非常复杂,包括一系列的信号转导途径和转录调控等过程,传统的分子生物学手段已经不能满足科研的需要。
基因芯片技术的发展开启了从全基因组水平检测基因表达的时代[9–10]。常见的基因芯片包括表达谱芯片、miRNA芯片、SNP芯片、甲基化芯片等,可以用来检测基因表达谱、miRNA表达谱、基因突变或者进行多态性分析等,为“后基因组计划”时期基因功能研究提供了强有力的工具。高通量基因芯片技术的基因表达谱分析是当前功能性基因研究的重要手段,已被广泛应用于植物对逆境胁迫的研究[11–12]。许多物种的非生物胁迫研究中都采用了生物芯片技术,如KREPS等[13]使用生物芯片技术对拟南芥盐胁迫、渗透胁迫和冷胁迫3种逆境中的根和叶片中转录组的变化进行研究。在水稻研究领域,也有不少利用基因表达谱芯片进行研究的报道,如郭小勤等[14]、邢俊杰等[15]研究了培矮64S不同处理下减数分裂期不育幼穗和可育幼穗基因表达谱的差异;FUJII等[16]利用Aglilent芯片研究了CW型细胞质不育系与其保持系成熟花粉基因型的差异。
本研究中,采用Agilent 4×44 K水稻全基因组芯片分析了氰化物胁迫下水稻幼苗叶片和根系的基因表达特征,以期筛选到水稻参与应答氰化物胁迫的关键基因或水稻植株内代谢氰化物的关键基因,为从分子水平揭示植物吸收、累积、转运氰化物以及植物对氰化物胁迫的响应机制提供依据。
1 材料与方法
1.1 植物材料与培养
水稻种子(L. cv. XZX 45)来源于湖南省农业科学院。将水稻种子用5%次氯酸钠消毒8 min后用灭菌水清洗3次,播种于垫有双层灭菌滤纸的培养皿中,25 ℃恒温条件下发芽,期间每天适量补充蒸馏水。水稻种子发芽 (播种15 d) 后,将长势均匀一致的水稻幼苗转移至含有1 mmol/L CaCl2和2 mmol/L 2–(N–吗啉)乙磺酸–三羟甲基氨基甲烷缓冲液溶液的锥形瓶中预处理4 h,以去除细胞壁中的离子,然后将长势基本一致的10株幼苗转移至装有50 mL Hoagland营养液的锥形瓶中。将锥形瓶用锡纸包裹,以阻止水分的损失和抑制藻类的生长。将锥形瓶放置于(25 ± 0.5) ℃、相对湿度(60 ± 2)%、光照强度300 μmol/(m2·s)和光周期14/10 h(昼/夜)的植物生长室进行培养。
1.2 试验设计
设置3种不同的处理:①CK,营养液不添加氰化物;②在营养液中添加氰化钾,其浓度为2.0 mg/L;③在营养液中添加铁氰化钾,其浓度为10.0 mg/L;每个处理设6次重复(6个锥形瓶),处理48 h后取水稻叶片和根系于液氮中速冻后保存在–80 ℃超低温冰箱中,用于RNA的提取。
1.3 基因芯片分析
1.3.1RNA的提取与纯化
采用TRIZOL Reagent (Cat#15596–018,Life technologies,Carlsbad,CA,US)进行样品总RNA的提取,抽提所得总RNA,经Agilent Bioanalyzer 2100(Agilent technologies,Santa Clara,CA,US)电泳质检合格后,使用RNeasy micro kit (Cat#74004,QIAGEN,GmBH,Germany)和 RNase–Free DNase Set(Cat#79254,QIAGEN,GmBH,Germany)纯化总RNA。纯化后的RNA采用Agilent表达谱芯片配套试剂盒和Low Input Quick Amp Labeling Kit,One– Color (Cat.# 5190–2305,Agilent technologies,Santa Clara,CA,US)对样品总RNA中的mRNA进行放大和标记,并用RNeasy mini kit(Cat.# 74106,QIAGEN,Ver:201403 GmBH,Germany)纯化标记的cDNA。
1.3.2芯片杂交
试验基因芯片为Agilent水稻4×44 K全基因组芯片,单张玻片4个微阵列,寡核苷酸探针密度为42 477,代表约41 005个水稻基因。芯片试验在上海伯豪生物技术有限公司完成。按照Agilent基因芯片提供的杂交标准流程和配套试剂盒(Cat.# 5188–5242,Agilent technologies,Santa Clara,CA,US) 在滚动杂交炉中(65 ℃、10 r/min)滚动杂交17 h。杂交cDNA上样量为1.65 μg,并在洗缸中洗片,洗片所用的试剂为Gene Expression Wash Buffer Kit (Cat.# 5188–5327,Agilent technologies,Santa Clara,CA,US)。
1.3.3芯片扫描
完成杂交的芯片采用Agilent Microarray Scanner (Cat.# G2565CA,Agilent technologies,Santa Clara,CA,US) 进行扫描,软件设置Dye channel: Green,Scan resolution=5 μm,PMT 100%,16 bit。用Feature Extraction software 10.7(Agilent technologies,Santa Clara,CA,US)读取数据,采用GeneSpring Software 11.0 (Agilent technologies,Santa Clara,CA,US) 进行归一化处理,所用的算法为Quantile。
1.3.4差异基因的筛选
对照与处理间的荧光杂交信号归一化后的信号比值均取以2为底的对数,以信号比值≤0.5或者≥2倍的为差异表达基因的筛选标准。将筛选到的不同组间的差异基因通过散点图进行展示。
1.3.5差异基因Gene Ontology (GO)富集分析
借助在线工具agriGO (http://bioinfo.cau.edu.cn/ agriGO/)进行GO富集分析。
2 结果与分析
2.1 基因芯片的检测结果
基因芯片的原始数据经过标准化处理,转化为以2为底的对数后,在二维坐标系中绘制散点图来评估2组数据总体分布情况。在基因表达谱的散点图上,每个点表示1个基因探针,X轴和Y轴分别表示对照条件下水稻叶片或根系的信号强度与氰化物处理条件下水稻叶片或根系的信号强度(图1)。基因芯片数据分析常用2倍表示基因差异表达,图中红点表示上调表达的基因数量,绿点表示下调表达的基因数量,而在散点图中±2平行线以内的基因表达无显著差异。从图1可以看出,在2和–2线外有较多的散点,且分布较为分散,表明氰化物处理导致水稻叶片和根系许多表达基因发生了改变。
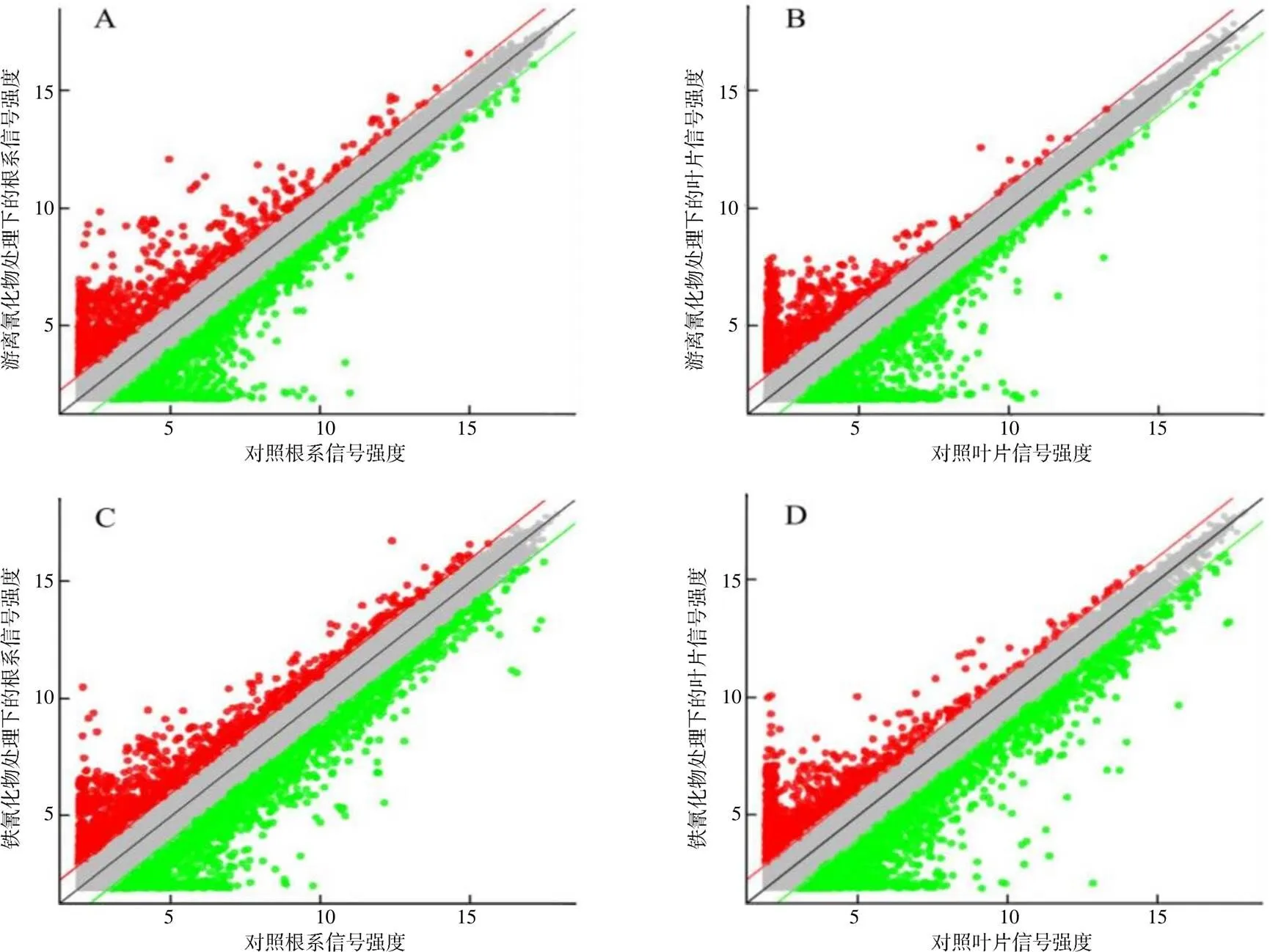
图1 氰化物胁迫下水稻叶片和根系基因表达谱芯片杂交信号散点图
2.2 氰化物胁迫下水稻叶片和根系差异基因表达分析
以未添加氰化物的营养液为对照,以添加氰化物为处理,按照表达强度变化在2倍或2倍以上(<0.05)的基因被定义为差异基因的标准。从表1可以看出,氰化钾处理下,在水稻根系中共筛选到了1841个差异表达基因,其中940个上调基因表达,901个下调表达基因;而在叶中差异表达的基因数显著低于根系中差异表达的基因数。在铁氰化钾处理下,水稻幼苗中差异表达的基因数远多于氰化钾处理下差异表达的基因数,根系中差异表达的基因数达3037个,其中上调表达1476个,下调表达1561个;而在叶中差异表达的基因数为1805个,其中747个上调表达基因,1058个下调表达基因。

表1 氰化物胁迫下水稻叶片和根系差异表达的基因数
2.3 不同氰化物处理下水稻幼苗的差异表达基因分析
氰化物对植物的胁迫反映在一系列的生物学过程中,包括植物对氰化物胁迫信号的感知、氰化物诱导基因的表达与调控、氰化物诱导代谢的改变及植物对氰化物的适应等。从表2至表5可以看出,不同氰化物诱导水稻幼苗基因上调表达的差异倍数较大,而且同一氰化物对水稻不同组织间基因上调表达的差异也比较明显。在氰化钾和铁氰化钾处理下,水稻根中与解毒相关的基因,如细胞色素P450家族蛋白基因(LOC_Os03g55250.1)、NADPH毒素还原酶基因(LOC_Os07g40974.2)、谷胱甘肽s–转移酶GST19基因(LOC_Os07g28480.1)和细胞色素P450单加氧酶CYP72A5基因(LOC_Os01g43740.1)等显著上调表达;在氰化钾和铁氰化钾处理下,水稻叶片中与光合作用相关的基因,如光系统II蛋白基因(LOC_Os08g10020.2)、叶绿素a/b结合蛋白III型基因(LOC_Os02g10390.2)、光系统I反应中心亚基基因(LOC_Os12g08770.1)和聚光复合体I基因(LOC_Os06g21590.1)等显著上调表达。水稻根系中上调表达的基因,如与细胞壁代谢相关的基因(LOC_Os04g53950.3/木葡聚糖内糖基转移酶基因)和抗逆相关的基因(LOC_Os12g06820.1/热激蛋白20基因)分别上调表达18.29倍和39.88倍(表2)。

表2 氰化钾处理下水稻根中与解毒和抗逆相关的上调表达基因
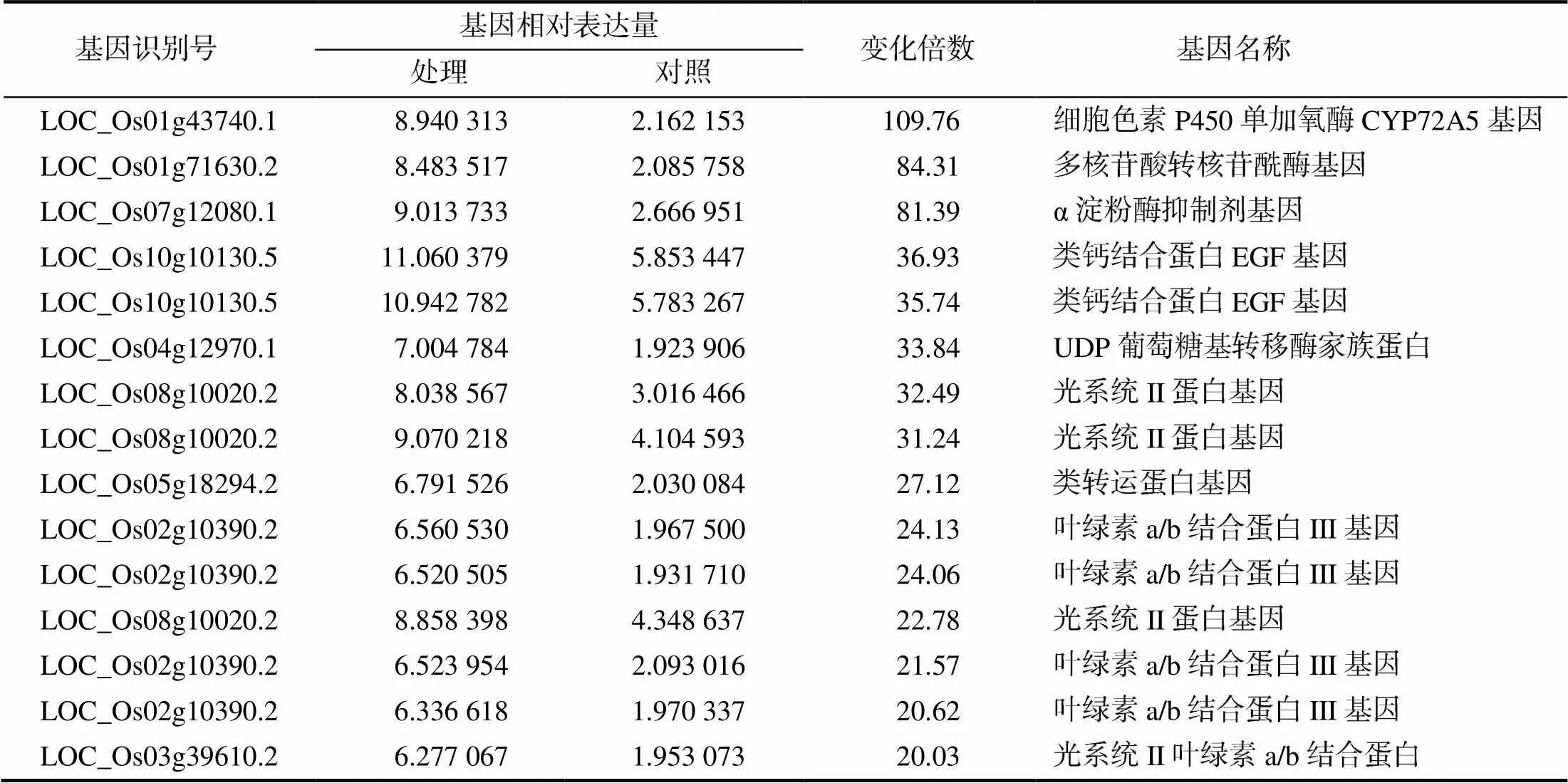
表3 氰化钾处理下水稻叶中与光合相关的上调表达基因

表4 铁氰化钾处理下水稻根中与解毒和抗逆相关的上调表达基因

表5 铁氰化钾处理下水稻叶中与光合相关的上调表达基因

表5(续)
2.4 差异表达基因的GO功能富集分析
基因本体论(Gene Ontology,GO)包括3个部分,即分子功能(molecular function)、生物学过程(biological process)和细胞组分(cellular component)。通过ID对应或者序列注释的方法,每个蛋白质或者基因均可以找到与之对应的GO号,进而对应到分类中,即功能类别或者细胞定位。如图2和图3所示,2种氰化物处理后,在细胞组分层面,GO类别中与“细胞组分”“细胞内”和“细胞间”相关的基因在水稻根和叶中均显著富集;在分子功能层面上,GO类别中与“蛋白结合”“核酸结合”“水解酶活性”和“转移酶活性”相关的基因在水稻根系和叶片中显著富集;在生物学过程层面上,水稻根系和叶片中显著富集的GO类别主要为“初级代谢过程”和“细胞代谢过程”。这些结果表明,2种氰化物处理下水稻根系和叶片均可能通过调节其体内的一些代谢途径和一些酶的催化活性来应对氰化物的胁迫,积极诱导与防御相关的基因,并通过主动吸收和转运等代谢过程将氰化物代谢解毒以降低对植物的伤害。

图2 铁氰化钾处理下水稻根系(A)和叶(B)中差异表达基因的GO功能分类

图3 氰化钾处理下水稻根系(A)和叶(B)中差异表达基因的GO功能分类
3 结论与讨论
本研究发现不同氰化物对水稻幼苗基因的表达都具有诱导作用,但其诱导差异表达基因的数量和变化的倍数差异较大,具有一定的组织器官特异性。不论是氰化钾还是铁氰化钾,其诱导差异表达的基因数量和变化倍数均在根中表现较高,在叶中相对较低,表明根系作为直接与氰化物接触的组织器官,其对氰化物处理的响应更为敏感,这与YU等[17–18]的研究结果一致。
在对差异表达的基因进行GO分类分析时发现,在细胞组分、分子功能和生物学过程类别中与“细胞组分”“细胞内”“细胞间”“水解酶活性”“转移酶活性”“初级代谢过程”和“细胞代谢过程”相关的基因在2种不同氰化物处理下均显著富集。不同氰化物处理下与细胞壁代谢相关的基因(LOC_ Os04g53950.3/木葡聚糖内糖基转移酶基因)和抗逆相关的基因(LOC_Os12g06820.1/热激蛋白20基因)、解毒相关基因(LOC_Os07g28480.1/谷胱甘肽s–转移酶 GST19,LOC_Os07g40974.2/NADPH毒素还原酶基因)均显著上调,说明在2种氰化物处理下,水稻根系和叶片均可能通过调节其体内的一些代谢途径和一些酶的催化活性来应对氰化物的胁迫,积极诱导与防御相关的基因,并通过主动吸收和转运等代谢过程将氰化物代谢解毒,以降低对植物的伤害。细胞色素P450家族蛋白基因、NADPH毒素还原酶基因和谷胱甘肽s–转移酶GST19基因可催化许多外源有毒物在细胞内的氧化和脱氢反应,从而降低有毒物的毒性。本研究中,细胞色素P450家族蛋白基因(LOC_Os03g55250.1)、细胞色素P450单加氧酶 CYP72A5(LOC_Os01g43740.1)、 NADPH毒素还原酶基因 (LOC_Os07g40974.2)和谷胱甘肽s–转移酶GST19基因(LOC_Os07g28480.1)在氰化物处理的水稻中显著上调表达,表明这些基因可通过上调表达来降低氰化物对水稻的毒性,从而提高水稻的适应性[19–21]。
植物抵抗氰化物胁迫或代谢氰化物的机制很复杂,涉及植物体内的多个代谢途径和解毒代谢过程,包括氧化还原酶系统、交替氧化酶、氨基酸和糖类物质的合成与降解等,目前其分子机理仍未得到解释。在今后的研究中需着重探索其分子机理,将分子机理和生理特性联系起来,以期破解植物抵抗氰化物胁迫和代谢氰代物的机理。
[1] EBBS S D,PICCININ R C,GOODGER J Q D,et al. Transport of ferrocyanide by two eucalypt species and sorghum[J].International Journal of Phytoremediation,2008,10(4):343–357.
[2] MANSFELDT T,LEYER H,BARMETTLER K,et al. Cyanide leaching from soil developed from coking plant purifier waste as influenced by citrate[J].Vadose Zone Journal,2004,3(2):471–479.
[3] EBBS S D,KOSMA D K,NIELSON E H,et al.Nitrogen supply and cyanide concentration influence the enrichment of nitrogen from cyanide in wheat(L.) and sorghum(L.)[J].Plant,Cell & Environment,2010,33(7):1152–1160.
[4] EBBS S D,BUSHEY J,POSTON S,et al.Transport and metabolism of free cyanide and iron cyanide complexes by willow[J].Plant,Cell & Environment,2003,26(9):1467–1478.
[5] SAMIOTAKIS M,EBBS S D.Possible evidence for transport of an iron cyanide complex by plants[J]. Environmental Pollution,2004,127(2):169–173.
[6] LARSEN M,TRAPP S.Uptake of iron cyanide complexes into willow trees[J].Environmental Science & Technology,2006,40(6):1956–1961.
[7] YU X Z,LI F,LI K.A possible new mechanism involved in ferro-cyanide metabolism by plants[J].Environmental Science and Pollution Research International,2011,18(8):1343–1350.
[8] YU X Z,LU P C,YU Z.On the role of β-cyanoalanine synthase(CAS) in metabolism of free cyanide and ferri-cyanide by rice seedlings[J].Ecotoxicology,2012,21(2):548–556.
[9] BARTELS D,SUNKAR R.Drought and salt tolerance in plants[J].Critical Reviews in Plant Sciences,2005,24(1):23–58.
[10] RIZHSKY L,LIANG H J,SHUMAN J,et al.When defense pathways collide.The response ofto a combination of drought and heat stress[J].Plant Physiology,2004,134(4):1683–1696.
[11] MANTRI N L,FORD R,CORAM T E,et al. Transcriptional profiling of chickpea genes differentially regulated in response to high-salinity,cold and drought[J]. BMC Genomics,2007,8:303.
[12] UEDA A,KATHIRESAN A,BENNETT J,et al. Comparative transcriptome analyses of barley and rice under salt stress[J].Theoretical and Applied Genetics,2006,112(7):1286–1294.
[13] KREPS J A,WU Y J,CHANG H S,et al.Transcriptome changes forin response to salt,osmotic,and cold stress[J].Plant Physiology,2002,130(4):2129–2141.
[14] 郭小勤,董海涛,郑康乐,等.水稻光温敏核不育系培矮64S在不同光周期/温度下表达谱的变化[J].科学通报,2005,50(22):2509–2513.
[15] 邢俊杰,李莉,李磊,等.水稻光温敏核不育基因表达产物初步研究[J].生命科学研究,2008,12(2):126–130.
[16] FUJII S,KOMATSU S,TORIYAMA K.Retrograde regulation of nuclear gene expression in CW-CMS of rice[J].Plant Molecular Biology,2007,63(3):405–417.
[17] YU X Z,GU J D,LI T P.Availability of ferrocyanide and ferricyanide complexes as a nitrogen source to cyanogenic plants[J].Archives of Environmental Contamination and Toxicology,2008,55(2):229–237.
[18] YU X Z,GU J D.Uptake,accumulation and metabolic response of ferricyanide in weeping willows[J].Journal of Environmental Monitoring,2009,11(1):145–152.
[19] YU X Z,GU J D.Differences in uptake and translocation of selenate and selenite by the weeping willow and hybrid willow[J].Environmental Science and Pollution Research International,2008,15(6):499–508.
[20] YU X Z,GU J D,LI L.Assimilation and physiological effects of ferrocyanide on weeping willows[J]. Ecotoxicology and Environmental Safety,2008,71(3):609–615.
[21] YU X Z,ZHOU P H,YANG Y M.The potential for phytoremediation of iron cyanide complex by willows[J]. Ecotoxicology(London,England),2006,15(5):461–467.
Characterization of gene expression profiles of rice seedlings in response to cyanide stress
ZHANG Fuzhong1,CAI Qing1,SONG Jiangyan1,HU Longxing2,WU Genyi3*
(1.Hunan Research Academy of Environmental Sciences, Changsha, Hunan 410004, China; 2.College of Agronomy, Hunan Agricultural University, Changsha, Hunan 410128, China; 3.South China Institute of Environmental Sciences, Ministry of Ecology and Environment of the PRC, Guangzhou, Guangdong 510655, China)
In order to screen out the key genes involved in the response of rice to cyanide stress and cyanide metabolism in rice plants, the gene expression characteristics of leaves and roots of rice seedlings under the stress of potassium cyanide and potassium ferricyanide were analyzed by Agilent 4×44 K rice genome chip. The results showed that 1841 and 3037 differentially expressed genes in rice roots and 734 and 1805 differentially expressed genes in rice leaves were found under potassium cyanide and potassium ferricyanide treatments, respectively. Under potassium cyanide treatment, the up-regulated expression multiples of genes related to cell wall metabolism and stress resistance in roots. Under potassium ferricyanide treatment, the up-regulated expression were the detoxification-related genes in rice leaves. The results showed that cyanide could induce the differential expression of genes in rice. There were great differences in the expression of genes related to detoxification and stress resistance, cell wall and secondary metabolic pathways and transcription factors. These results revealed detoxify cyanide metabolism through metabolic processes such as active absorption and transport to reduce plant damage. The roots and leaves of rice may serve the main organism for responsing to cyanide stress by self-regulation.
rice; cyanide; stress; genes expression characterization; variance analysis
张伏中,蔡青,宋江燕,胡龙兴,吴根义.水稻幼苗响应氰化物胁迫的基因表达谱差异分析[J].湖南农业大学学报(自然科学版),2022,48(5):528–535.
ZHANG F Z,CAI Q,SONG J Y,HU L X,WU G Y.Characterization of gene expression profiles of rice seedlings in response to cyanide stress[J].Journal of Hunan Agricultural University(Natural Sciences),2022,48(5):528–535.
http://xb.hunau.edu.cn

S511.01
A
1007-1032(2022)05-0528-08
10.13331/j.cnki.jhau.2022.05.004
2021–11–15
2022–09–12
国家自然科学基金项目(U20A2086);郴州国家可持续发展议程创新示范区建设专项(2021SFQ26)
张伏中(1983—),男,湖南岳阳人,博士,高级工程师,主要从事环境生态与修复研究,78697415@qq.com;*通信作者,吴根义,博士,教授,主要从事农业环境保护研究,wugenyi99@163.com
责任编辑:毛友纯
英文编辑:柳正
