基于全基因组测序的耐碳青霉烯类抗生素鲍曼不动杆菌耐药基因、毒力因子及同源性分析
2022-11-11胡小骞王琴
胡小骞 王琴
(安徽医科大学第二附属医院,合肥 230601)
鲍曼不动杆菌(Acinetobacter baumannii,AB)是一种易引起医院感染的非发酵革兰阴性杆菌,近年来在重症监护病房的检出率居高,主要感染免疫力低下的重症患者、侵入性治疗的患者及抗菌药物广泛使用的患者,易引起脑膜炎、肺炎、腹膜炎、菌血症等疾 病[1-2],感染患者的死亡率高达35%[3]。AB在环境中可长期存在,当感控措施落实不到位时,会增加患者间交叉传播以及耐药菌间传播的机会[4]。随着抗菌药物的广泛使用,AB的耐药性日渐严重,其对碳青酶烯类的耐药率逐年增高[5-6],耐碳青霉烯类抗生素鲍曼不动杆菌(carbapenem-resistantAcinetobacter baumannii,CRAB)的检出率逐渐增高,其在社区中、医院内传播的机会不断增加,对医疗机构构成了全球性威胁[7]。本研究收集该院重症监护病房、神经外科住院患者分离的CRAB,应用全基因组测序技术对其携带的耐药基因、毒力因子及同源性进行分析,为临床CRAB感染防控措施及抗菌药物的合理使用提供参考依据。
1 材料与方法
1.1 材料
1.1.1 菌株来源
收集该院2020年1月—3月ICU、神经外科分离的所有医院感染CRAB菌株,剔除同一患者的重复菌株及入院48 h内检出的社区感染菌株,共11株。菌株纳入依据为1月至3月期间该院ICU发现7例CRAB医院感染的患者,进一步调查发现神经外科有4例CRAB医院感染的患者与7例ICU患者存在时间及空间的交集,将此11株CRAB纳入研究。标本来源分别为痰(10株)、血(1株)。11例患者中男性11例(100%),见表1。以大肠埃希菌ATCC25922,肺炎克雷伯菌ATCC700603,铜绿假单胞菌ATCC27853,阴沟肠杆菌ATCC700323为质控菌株。
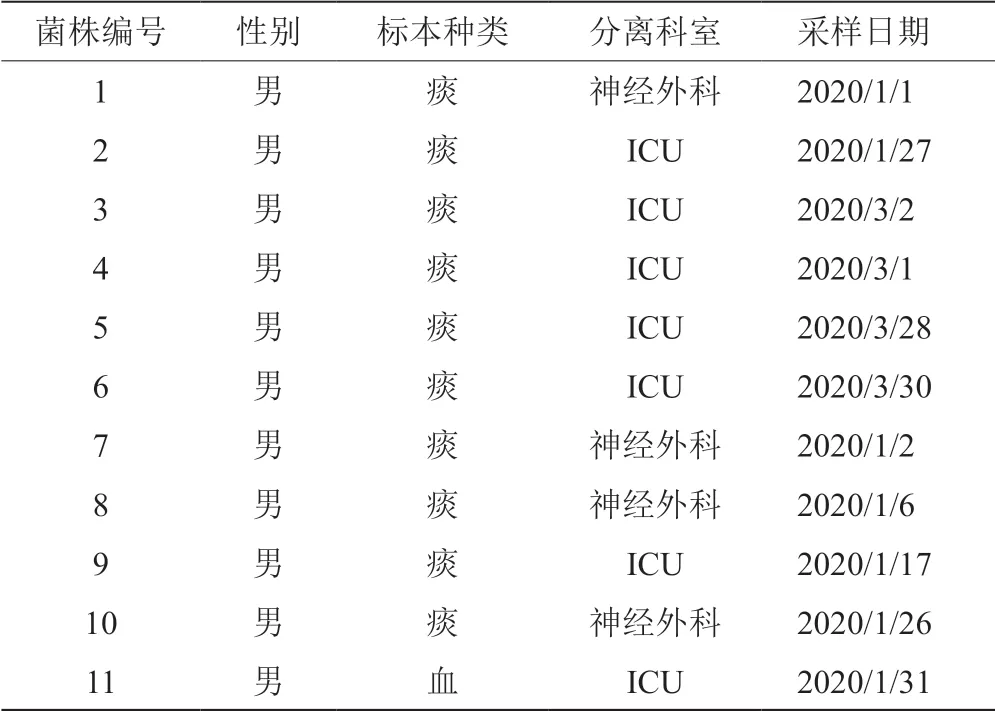
表1 11株CRAB菌株的分布情况Tab.1 The distribution of 11 strains of CRAB
1.1.2 仪器与试剂
法国生物梅里埃VITEK-2 Compat全自动细菌分析系统,英国Biochrom超微量分光光度计,上海一恒科学仪器有限公司恒温培养摇床,美国Bio-Rad电泳仪,美国Bio-Rad凝胶成像系统。药敏纸片、M-H培养基购自Oxoid公司,DNA Marker D、4S Red plus核酸染色剂、上样缓冲液购自上海生工生物工程股份有限公司,细菌DNA提取试剂盒购自天根生化科技有限公司,质控菌株购自国家卫健委临床检验中心。
1.2 方法
1.2.1 细菌鉴定及药敏实验
应用法国VITEK-2 Compat全自动细菌分析系统和配套试剂GN/GN13,进行革兰阴性菌的鉴定及药敏试验。参照临床实验室标准化研究所(Clinical and laboratory standard institute,CLSI)2019年标准对药敏结果进行判定,使用微量肉汤稀释法(MIC法)对亚胺培南、庆大霉素、头孢菌素、妥布霉素、复方磺胺甲恶唑、左氧氟沙星和环丙沙星进行药敏试验,使用K-B纸片扩散法对美罗培南和阿米卡星进行药敏试验,CRAB药敏结果判定标准为对任一种碳青霉烯类抗生素耐药的鲍曼不动杆菌。
1.2.2 细菌基因组DNA提取
复苏CRAB菌株于LB固体培养基上,37℃培养24 h后,挑取单克隆CRAB菌株于LB液体培养基中,37℃、220 r/min摇菌过夜。取细菌培养液1 mL,10000 r/min离心1 min,吸净上清液,保留菌体沉淀,使用DNA提取试剂盒提取菌株DNA。
1.2.3 基因组DNA提取物质量控制标准
将DNA提取产物进行琼脂糖凝胶电泳,并用超微量分光光度计检测纯度和浓度。纯度要求菌株DNA产物的A260:A280于1.8~2.0范围内,避免RNA污染。DNA浓度≥20 ng/μL,条带清晰无杂带,总量在1.0~2.0 μg范围内。
1.2.4 细菌全基因组测序
通过Illumina Hiseq2500第二代高通量测序平台进行2×100 bp Paired-End测序,基于Illumina平台的测序,是对核酸提取样本进行建库、扩增测序、质控的3个主要步骤。
1.2.5 测序结果组装
在Linux或Centos系统中运行SPAdes-2.0软件将测序原始FASTQ结果拼接为FASTA文件,运行命令参考SPAdes-2.0软件网址https://github.com/ablab/spades。
1.2.6 耐药基因筛选
应用丹麦技术大学(DTU)基因组流行病学中心(Center for genomic epidemiology,CGE)(https://cge.cbs.dtu.dk/services/ResFinder/)的ResFinder 4.0工具,对11株CRAB的contigs进行耐药基因筛选,获得其携带的耐药基因。应用MORPHEUS(https://software.broadinstitute.org/morpheus/)在线制作11株CRAB的耐药基因热图,实现耐药基因分布可视化。
1.2.7 毒力因子筛选
应用Linux系统运行Kaptive软件检测CRAB菌株的荚膜型,应用中国卫生健康委员会创建的病原生物学系统生物学重点实验室、CAMS&PUMC病原生物学研究所毒力因子数据库(Virulence factors of pathogenic bacteria,VFDB)(http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgifunc=VFanalyzer)的VFanalyzer在线工具,对11株CRAB进行毒力因子筛选。
1.2.8 同源性分析
应用Linux系统运行MLST软件,MLST软件将菌株的FASTA文件应用BLAST算法与PubMLST分子分型和微生物基因组多样性的公共数据库进行(https://pubmlst.org/)比对,运行命令参考MLST软件(https://github.com/tseemann/mlst),对细菌基因组的gapA、infB、mdh、pgi、phoE、rpoB和tonB7个管家基因进行对齐分类,每个基因组的每种管家基因被命名为一个随机整数,各种管家基因命名整数的唯一组合(等位基因谱)即为一种序列类型(ST型),根据细菌ST型初步分析其同源性。
同时,基于全基因组序列的单核苷酸多态性(single-nucleotide polymorphism,SNP)分析,以1号菌株序列作为参考,使用CGE的CSI Phylogeny 1.4工具(https://cge.cbs.dtu.dk/services/CSIPhylogeny/)对所有菌株序列进行SNP分析,应用FigTree v1.4.4软件针对分析结果构建11株CRAB的最大似然树,按同源性大小分组,分析其同源性。
1.2.9 统计学分析
应用WHONET5.6软件对数据进行分析处理,计数资料以例数进行描述。
2 结果
2.1 细菌药敏结果
11株CRAB均为多重耐药菌,其中11株对亚胺培南、美罗培南、头孢菌素和环丙沙星均表现耐药,而对阿米卡星和左氧氟沙星耐药的菌株株数较少,分别为4株和2株,见表2。
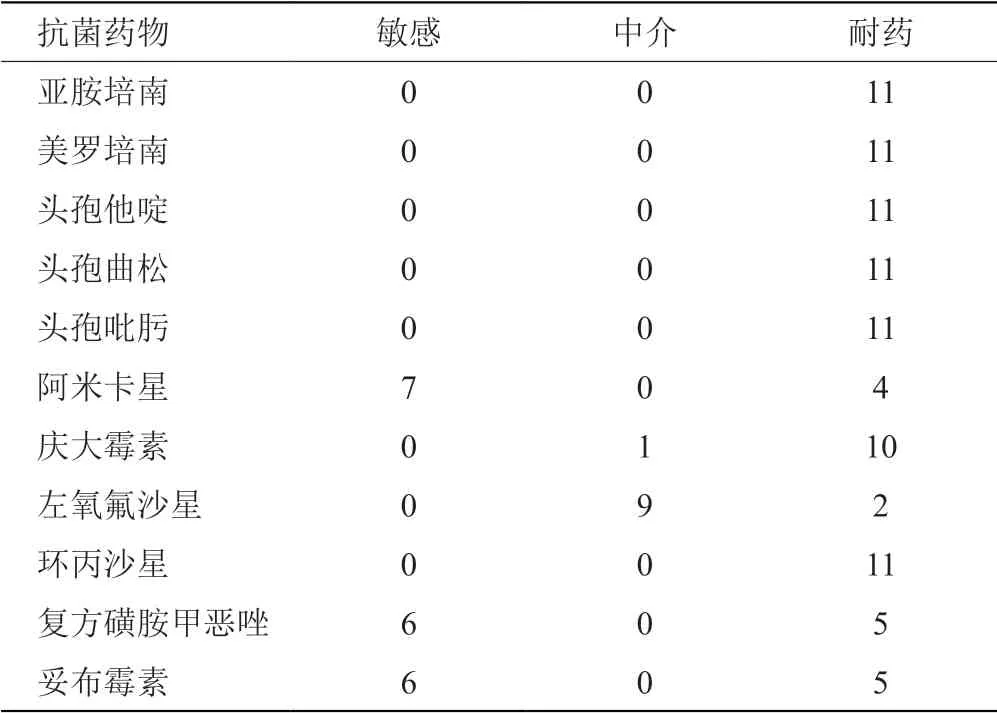
表2 CRAB对常用抗菌药物的耐药情况(株数)Tab.2 Resistance of CRAB to commonly used antibacterial drugs(number of plants)
2.2 耐药基因分析结果
通过对11株CRAB进行WGS分析,共发现18种耐药基因,包括碳青霉烯酶耐药基因、β-内酰胺酶耐药基因、多种外排泵相关耐药基因及抗菌药物修饰酶耐药基因,见图1和表3。碳青霉烯酶耐药基因以blaOXA-23和blaOXA-66为主,11株(100.0%)均携带此型。11株(100.0%)均携带β-内酰胺酶耐药基因,包括blaADC-25和blaTEM-1D。检出外排泵相关基因如tet(B),以及抗菌药物修饰酶耐药基因如aadA1、aph(3″)-Ib、aph(3')-Ia、aph(6)-Id、aac(3)-Ia和aac(6')-Ib3,7种氨基糖苷类耐药基因中,11株(100.0%)携带aadA1、aph(3″)-Ib、aph(3')-Ia和aph(6)-Id,9株(81.8%)携带aac(3)-Ia,5株(45.5%)携带aac(6')-Ib3和armA。检出大环内酯类耐药基因mph(E)(10株)、msr(E)(10株),磺胺类耐药基因sul1(11株),喹诺酮类耐药基因aac(6')-Ib-cr(5株),四环素耐药基因tet(B)(11株),氯霉素耐药基因catB8(5株),消毒剂耐药基因qacE(11株)。

表3 CRAB携带的耐药基因型分布情况Tab.3 The distribution of drug-resistant genotypes carried by CRAB
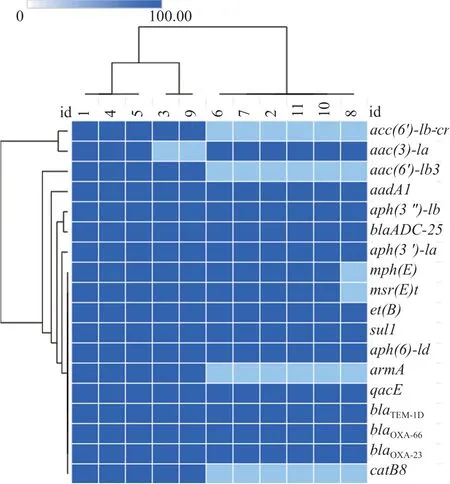
图1 11株CRAB耐药基因热图Fig.1 Heat map of 11 strains of CRAB resistant genes
2.3 毒力因子分析结果
通过WGS分析,发现11株CRAB均为K22荚膜型,其均携带多种毒力因子,包括外膜孔蛋白(OmpA)、脂多糖(LpsB)、生物膜(CsuABCDE、Bap和PgaABCD)、外排泵(AdeFGH)、磷脂酶(Plc、PlcD)、不动杆菌素生物合成蛋白(BauABCDEF)、外膜醋酸杆菌素蛋白(BauABCDEF)、肠杆菌素合成酶(EntE)、血红素加氧酶(HemO)、群体感应(QS系统)蛋白(AbaIR)、菌毛调节因子(BfmRS)、青霉素结合蛋白G(PbpG)和过氧化氢酶(KatA)。
2.4 同源性分析结果
11株CRAB均为ST 2-K22型,其最大似然树构建结果如图2所示,依据树分支节点的bootstrap值,将bootstrap值≥0.5的分支菌株分为一组[8-10],11株CRAB按同源性差异分成5组克隆株,10号为A组,7号为B组,1、3、4、5、6、8和9号为C组,11号为D组,2号为E组。C组组内同源性关系较近,组内的分支节点上的bootstrap值均≥0.7,C组感染患者的流行病学调查结果显示,7位感染患者均为入住ICU后分离出CRAB,其ICU住院时间存在先后或交集关系,其中6号与8号床位相同,其他感染患者的床号相近。

图2 11株CRAB最大似然树Fig.2 Phylogenetic tree diagram of 11 strains of CRAB
3 讨论
本研究对11株CRAB的药敏结果显示其耐药形势较严峻,其对临床常用抗菌药物呈现耐药,除了6株对妥布霉素、复方磺胺甲恶唑敏感,7株对阿米卡星敏感外,对其他抗菌药物敏感的菌株株数均较低。说明临床应加强抗菌药物合理规范使用的管理,减少抗菌药物对耐药菌的筛选机会,否则将会面临无药可用的局面。
CRAB的耐药机制较为复杂,主要包括外排泵的过度表达、膜孔通道蛋白表达下调、药物作用靶位变化、产碳青霉烯酶等方式实现耐药[11-15]。其中,产碳青霉烯酶是CRAB重要的耐药机制之一,本研究中碳青霉烯酶耐药基因blaOXA-23和blaOXA-66检出率均为100%,未检出其他类型碳青霉烯酶耐药基因。blaOXA-23和blaOXA-66属于Ambler D类酶,blaOXA-23通常由质粒或染色体介导传播[16-17],位于blaOXA-23上游的ISAba1基因对其具有协调作用[18-19],ISAba1作为插入序列可使carO基因失活,致使膜孔蛋白carO缺失[20],阻止药物进入菌株内部而产生耐药。说明blaOXA-23和blaOXA-66为该院CRAB对碳青霉烯类抗菌药物耐药的主要因素之一,这与国内外相关报道一致[12,17,21-23]。本研究结果中的aac(3)-Ia和aac(6')-Ib3属于氨基糖苷乙酰转移酶(AACs)基因,其检出率分别为81.8%和45.5%,检出率高于国内相关研究[24],氨基糖苷核苷转移酶(ANTs/aad)基因aadA1、以及氨基糖苷磷酸转移酶(APHs)基因aph(3″)-Ib、aph(3')-Ia和aph(6)-Id的检出率均为100.0%,说明这些基因为该院流行的主要耐药基因,其可通过多种途径在菌株间传播,如转座子、整合子及质粒等[22]。armA属于氨基糖苷类耐药基因中的16S rRNA甲基化酶基因,是AB耐氨基糖苷类药物的主要因素之一[25],本研究中的检出率为45.5%,临床科室应加强氨基糖苷类抗菌药物的合理使用,减少此类药物对AB的选择作用。喹诺酮类耐药基因aac(6')-Ib-cr由氨基糖苷乙酰转移酶基因aac(6')-Ib突变而来,可由质粒介导乙酰化含游离氨基的药物,可同时对氨基糖苷类和喹诺酮类药物进行酶解,其检出率为45.5%。四环素耐药基因tet(B)检出率为100.0%,Vilacoba等[26]发现tet(B)可与插入序列ISCR2协同实现耐药基因的传播。以上研究显示该院CRAB耐药基因类别较多、机制复杂,说明临床及相关主管部门应加强抗菌药物的管理,合理使用抗菌药物,避免药物滥用情况的发生。
AB的毒力因子主要包括磷脂酶、膜孔蛋白、脂多糖、铁载体及生物被膜相关蛋白等。本研究中11株CRAB均携带多种毒力因子。其中,生物膜因子包括CsuABCDE、Bap和PgaABCD,可保护细菌以适应乏养环境,CsuE和PgaB是其常见流行因子[27],其他因素也能促使生物膜的形成,如上调Csu和菌毛调节因子(BfmR)[28]、外排泵AdeFGH基因的过度表达等[29]。膜孔蛋白因子(OmpA)能够辅助菌株形成生物膜,并参与菌株对宿主的黏附作用,甚至能够诱导凋亡级联反应的发生[30]。脂多糖因子(LpsB)可提高菌株对宿主血清补体的抵抗力,以实现对宿主细胞的免疫逃逸,还可以预防宿主抗菌肽LL-37对菌株的杀伤作用[2,31]。不动杆菌素生物合成蛋白包括BasABCDFGHIJ,其中BasD能够促进菌株对于宿主细胞的铁摄取,保障菌株能量供应及持续生存。外膜醋酸杆菌素蛋白BauA与BasD作用相似[32]。另外,群体感应(QS系统)蛋白(AbaIR)可辅助菌株表面运动以增强毒力,相关研 究[33]表明菌株主要依赖AbaI介导的群体感应以实现运动能力。青霉素结合蛋白G(PbpG)基因可参与合成Pbp7/8[11],有研究[34]表明Pbp7/8的缺失可能破坏肽聚糖结构,从而影响菌株对宿主的防御作用。血红素加氧酶HemO会促进CRAB对宿主血红素及铁的有效利用[35],实现菌株的生存维持。本研究结果显示该院CRAB毒力机制复杂多样,各类毒力因子赋予菌株强大的生存力及侵袭力,说明实施有效防控措施以控制CRAB的传播至关重要。
本研究结果显示11株CRAB均为ST 2-K22荚膜型,与Rezaei等[36]的研究结果相似。最大似然树结果显示C组的1、3、4、5、6、8和9号菌株同源性较高,提示为该院CRAB的优势克隆株。有研究发现AB在医疗环境中可长期存活,在干燥环境中可存活60 d,当消毒隔离措施落实不到位,医务人员手卫生不达标等情况发生时,此类环境可能成为CRAB定植和交叉感染的温床[37]。同源性分析结果显示,C组的1、8号感染患者先于2019年12月在ICU入住治疗,转回神经外科后分离出CRAB,3、4、5、6、9号感染患者随后入住ICU治疗,并在ICU住院期间分离出CRAB,7位感染患者的ICU入住床号较为相近,其中,6号与8号床位相同。说明不能排除环境及医务人员等因素导致CRAB在ICU内的传播,此次传播原因可能与ICU的床单元消毒不到位、医务人员手卫生不规范以及医务人员无菌操作不达标有关。医院相关管理部门及临床科室应加强耐药菌防控的管理,包括严格落实病区的消毒隔离措施,严格执行无菌操作,规范手卫生等,医务人员应加强对国内、外细菌耐药趋势的了解,合理使用抗菌药物[38]。
多重耐药菌的耐药基因、毒力因子及亲缘性关系对于细菌耐药性流行病学的研究至关重要,传统的技术方法如PCR及qPCR基因芯片、脉冲场凝胶电泳等的分析结果不全面、灵活性低、耗时长、操作复杂,具有一定的局限性。WGS结合生物信息分析的模式能够快速全面地识别细菌的耐药性及毒力机理,精准地呈现菌株间的亲缘性关系,具有灵敏度、特异性、准确性高的特点,对于研究基因组的复杂性、多样性以及提高各类细菌发展进程的认知具有重要意义。WGS分析结果与流行病学调查的结合能够快速、准确地确认院感暴发、追踪感染源、明确传播路径,为耐药菌的防控提供更有效的目标干预信息,对于医院感染暴发的调查具有重要意义,提升了医院感染防控的技术水平。
本研究的菌株样本量偏少,在开展耐药性和毒力的统计学研究方面存在不足。同时,本研究选用的ResFinder 4.0数据库对于部分基因及突变基因的比对不全面,如喹诺酮类耐药基因gyrA、gyrB和parC等,使本研究在预测菌株的耐药性方面具有一定的局限性。后续将收集更多科室的不同种类标本,应用多样化的技术方法对菌株进行耐药性、毒力及同源性的分析。
综上所述,本研究中的CRAB对大多数抗菌药物均耐药,同时携带多种耐药基因,耐药形势较为严峻。其携带的毒力因子较多,毒力机制较为复杂,给临床治疗带来了挑战。CRAB菌株间存在一定的同源性,应加强对CRAB的耐药性监测及主动筛查,落实有效的感染防控措施,遏制耐药菌在医院内的传播。
