扁核木属植物叶绿体基因组特征分析
2022-10-17赵文植董章宏马路遥李卫英夏茂甜王正德辛培尧
王 飞,赵文植,董章宏,马路遥,李卫英,夏茂甜,王正德,辛培尧*
扁核木属植物叶绿体基因组特征分析
王 飞1,2,赵文植1,2,董章宏2,马路遥1,2,李卫英1,2,夏茂甜1,2,王正德1,2,辛培尧2*
1. 西南林业大学国家林业和草原局西南风景园林工程技术研究中心,云南昆明 650224;2. 西南林业大学西南地区生物多样性保育国家林业和草原局重点实验室,云南昆明 650224
扁核木属()植物在中国分布有4个种,均具有极高的食用和药用价值。为解析扁核木属植物的叶绿体基因组特征,基于现已发表的扁核木属植物叶绿体基因组,利用相关生物信息学手段进行其叶绿体基因结构、SSR、密码子偏好性和序列变异情况分析,并构建系统发育树。结果表明,2种扁核木属植物叶绿体基因组大小介于159 179~ 168 206 bp之间,平均GC含量为37.3%,从编码基因数目来看,仅相差2个tRNA,而蛋白编码基因和rRNA数目均一致,种间具有较高的保守性;在扁核木和蕤核叶绿体全基因组序列中各检测出分散重复49个,其中以正向重复和回文重复为主,占比达73%~82%,而串联重复数目分别为49个和58个,经简单重复序列SSR分析,分别在2种植物叶绿体基因组序列中分别筛选出100个和84个SSR位点,且多是以A/T碱基为主的单核苷酸重复类型;扁核木属植物叶绿体基因组密码子偏好性分析发现GC3的碱基含量显著低于GC1和GC2,说明密码子偏好以A、U结尾,ENC取值均大于48%,表明其密码子偏性较弱,中性绘图和PR2-plot分析发现自然选择是影响扁核木属植物密码子使用偏好性的主要原因,通过建立高、低基因表达库,以RSCU值为参考,确定了6个扁核木属最优密码子。经叶绿体基因组序列变异分析,根据核苷酸多态性指数>0.015筛选出-GUG-,UCCCAU和等3个高变区,以蕤核叶绿体基因组为参考,在扁核木叶绿体基因组编码区发现存在大量的插入、缺失和SNP突变位点,并在蕤核叶绿体基因组中发现了多个该物种的特有基因。基于叶绿体基因组的间隔区构建的系统发育树显示,4种扁核木属植物可分为南系(扁核木、台湾扁核木)和北系(蕤核、东北扁核木)两类。研究结果可为扁核木属植物的系统发育、分类鉴定及其资源的开发利用等相关研究提供理论基础。
扁核木属;叶绿体基因组;特征分析;系统发育
扁核木属(Royle)隶属蔷薇科(Rosaceae)李亚科(Prunoideae),具有极高的药用价值和保健作用。属内共有5个种,中国分布有4个种,分别是扁核木()、蕤核()、东北扁核木()和台湾扁核木()[12]。已有相关扁核木属植物的研究主要集中于其提取物药理作用、生理特性、地理谱系和形态学分类、资源分布以及繁殖等方面[13-18]。但有关扁核木属植物叶绿体基因组方面的研究报道相对较少,WANG等[19]基于扁核木质体基因组测序,解析了其质体基因组特征,通过与同科其他4个物种的叶绿体基因组比对,构建了系统发育树,为研究蔷薇科植物叶绿体基因组进化和突变模式提供了理论依据;而刘家奇等[15]则利用3个叶绿体DNA片段从谱系地理学的角度,分析了蕤核的遗传结构及种群历史动态。同年,研究人员报道了蕤核全叶绿体基因组序列,并简单阐述了其基本结构[20]。
本研究基于NCBI库中现已发表的2种扁核木属植物完整叶绿体基因组序列,对比分析了叶绿体基因组的基本特征,检测重复序列和SSR位点,解析密码子使用模式和影响因素;利用比较基因组学分析核酸变异程度,并统计基因编码区的突变数量及形式;通过系统发育树来确定属间各物种之间的亲缘关系。以期为扁核木属植物的分子鉴定、叶绿体基因组结构和功能的研究提供参考。
1 材料与方法
1.1 材料
从NCBI数据库中下载南北方扁核木属的代表植物扁核木和蕤核的完整叶绿体基因组序列,Genbank登录号分别为MW332618、MZ270554。
1.2 方法
1.2.1 可视化处理 利用Geneious 8.1.3软件对序列进行可视化处理,统计叶绿体基因组大小、GC含量、基因数目,区划叶绿体基因组,并计算各区长度和GC含量等信息,运用OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)在线软件绘制扁核木属植物叶绿体基因组物理图谱。
1.2.2 重复序列与SSR分析 重复序列包括散在重复和串联重复,其中散在重复又包含了正向重复(forward repeats, F)、反向重复(reverse repeats, R)、回文重复(palindromic repeats, P)和互补重复(complement repeats, C)。散在重复序列利用REPuter软件[21]进行预测,参数设置为:最小长度(minimum length)为30 bp,海明距离(hamming distance)为3;串联重复序列通过在线Tandem Repeats Finder工具[22]进行分析。使用MISA软件[23]检测扁核木属植物叶绿体基因组中的简单重复序列(simple sequence repeats, SSR),SSR搜索参数设置为:单核苷酸重复≥10,二核苷酸重复≥5,三核苷酸重复≥4,四核苷酸及以上核苷酸重复≥3。
1.2.3 叶绿体基因组学序列分析 以蕤核叶绿体基因组作为参考,利用在线工具MAFFT v.7 (https://mafft.cbrc.jp/alignment/server/)及Geneious 8.1.3软件检测分析2种扁核木属植物叶绿体基因组编码基因变异位点上的差异。
1.2.4 密码子偏好性分析 利用Geneious软件各获取了84条编码蛋白质的基因序列(CDS)。为减少实验误差,根据以下条件对CDS进行筛选:(1)组成CDS的碱基必为A、T、C、G;(2)CDS的起始和终止密码子正确,即ATG起始,TAA、TAG或TGA终止;(3)剔除长度小于300 bp以及重复的CDS序列。最终分别获得了52条符合条件的CDS用于后续分析。
(2)真实反映学习过程,暴露学习问题.动态生成的课堂给了师生更多的探索空间,教师和学生更容易发现探索过程中遇到的问题,进而针对性地解决这些问题,减少将来的麻烦.
①密码子组成分析。密码子第1、2、3位上碱基的种类和含量是评判密码子使用模式有效指标[24]。利用CodonW分析筛选出来的52条CDS的ENC值(有效密码子数)和各氨基酸所对应密码子的RSCU值(相对同义密码子使用度),并应用在线程序CUSP(http://imed.med.ucm.es/EMBOSS/)计算各基因总的GC含量及密码子第1~3位上的GC含量,分别以GCall、GC1、GC2和GC3表示。
②中性绘图。中性图是以GC3值作为横坐标,每个基因的GC1和GC2的平均值GC12作为纵坐标进行散点图的绘制,进而分析二者相关性,以此来初步判断影响密码子使用偏好性的因素,图中的每个点都作为独立的基因存在[25]。
③PR2-plot分析。统计各密码子第3位上各碱基含量,以A3/(A3+T3)值为纵坐标、G3/(G3+C3)值为横坐标绘制偏倚图,分析密码子第3位上嘌呤(A、G)和嘧啶(C、T)的使用情况及关系,当互补碱基含量相等时,即G=C,A=T,表明密码子无偏向性[26]。
④最优密码子分析。根据ENC值大小分别对2种扁核木属植物的叶绿体基因进行排序,从两端各选取基因总数的10%(5条)来建立高、低基因表达库。利用CodonW软件统计高、低表达基因的RSCU值,并计算ΔRSCU值(ΔRSCU= RSCU高表达-RSCU低表达),以RSCU>1且ΔRSCU≥0.08两个条件为标准来筛选扁核木属植物的最优密码子。
1.2.5 核苷酸变异分析 利用DnaSP 6软件[27]分析扁核木和蕤核叶绿体基因组的核苷酸多样性(),滑动窗口长度设为600 bp,步长设为200 bp。
1.2.6 系统发育分析 研究表明-间隔区能提供丰富的信息位点且引物通用,在种间及近亲属间的系统发育研究中应用广泛[28]。因此,为了确定扁核木属植物的系统发育位置及种间亲缘关系,基于现已发表的4种扁核木属植物叶绿体基因组的基因片段,利用RAxML 8.2.12软件下的模型构建系统发育树,进而分析扁核木属植物系统发育位置。
2 结果与分析
2.1 叶绿体基因组物理图谱与基本特征
扁核木和蕤核的叶绿体基因组基本信息如表1所示。2种植物叶绿体基因组总长度分别为168 206、159 179 bp,相差9027 bp。从编码基因数来看,扁核木比蕤核少2条tRNA,即-UAG和-UGC,而蛋白编码基因和rRNA数目没有发生变化,分别是84个和8个。叶绿体基因组的GC含量相差也不大,分别为38.0%和36.6%。经基因类别统计,在2种扁核木属植物叶绿体基因组中各发现了18个含有内含子的基因,其中含有1个内含子的基因有15个,含有2个内含子的基因有3个。值得注意的是,基因仅在蕤核叶绿体基因组中含有2个内含子,而在扁核木叶绿体基因组中只含有1个内含子。根据扁核木和蕤核的叶绿体全基因组序列,运用在线软件OGDRAW绘制出扁核木属植物叶绿体基因组物理图谱(图1)。
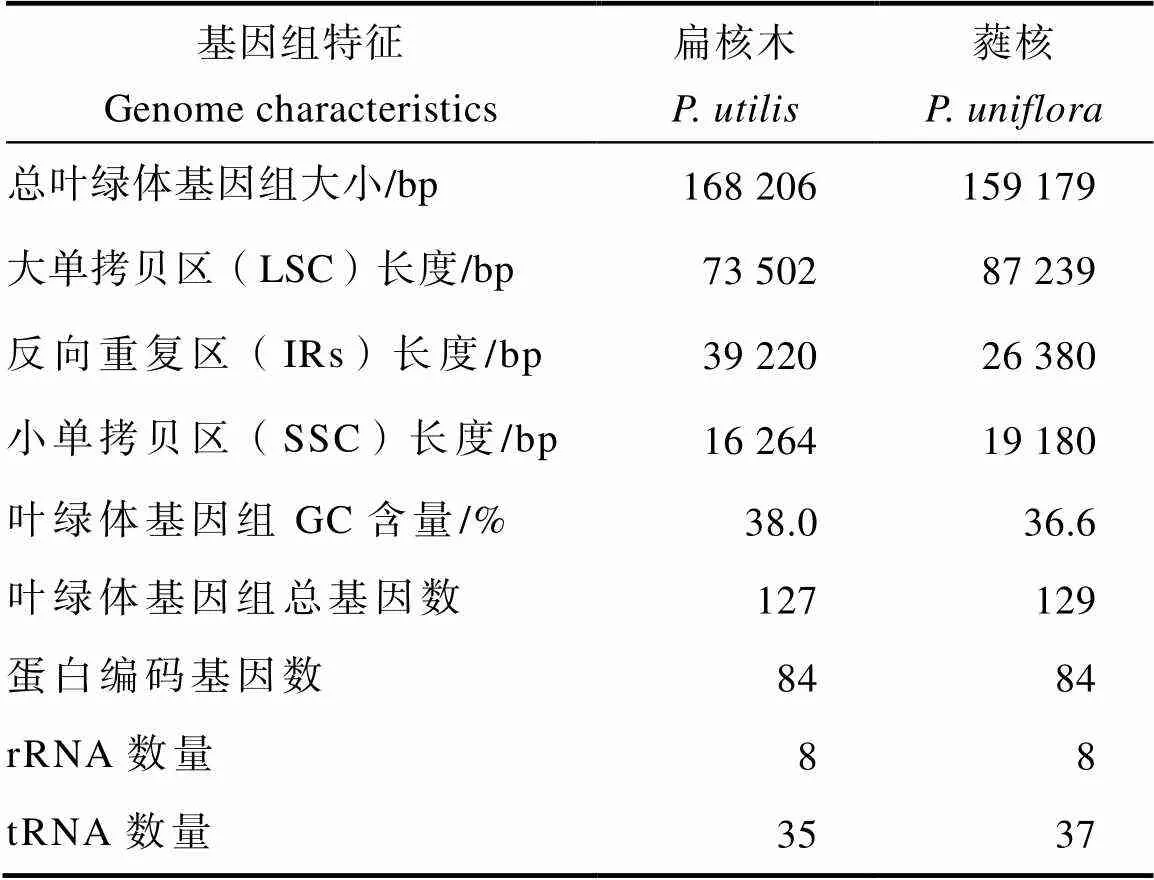
表1 扁核木和蕤核叶绿体全基因组特征
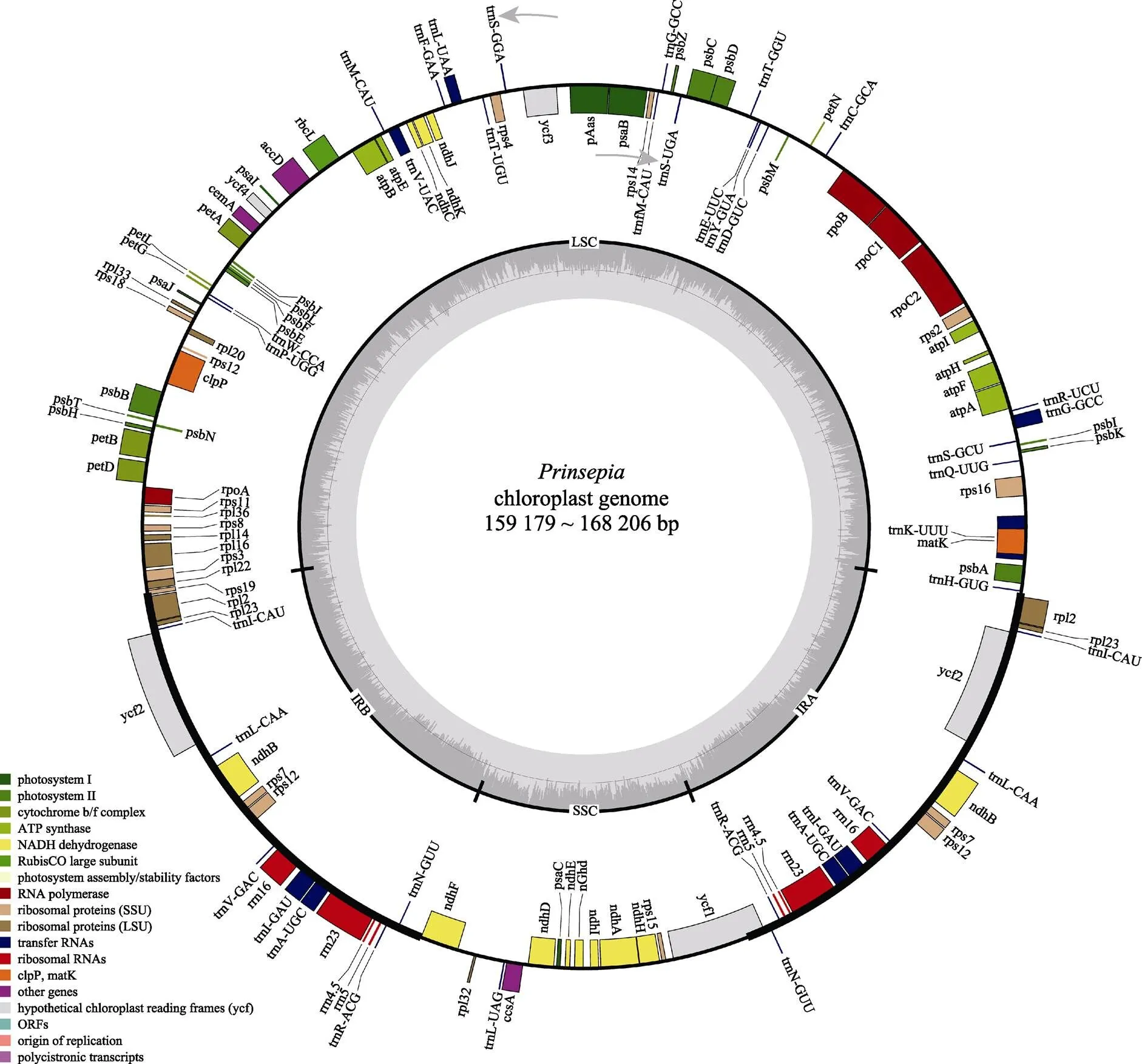
图1 扁核木属植物叶绿体全基因组物理图谱
2.2 重复序列与SSR分析
扁核木和蕤核叶绿体基因组中的分散重复和串联重复序列分别利用REPuter软件和Tandem Repeats Finder软件进行分析,在2种植物叶绿体基因组中各鉴定出49个分散重复序列,其中扁核木中有30个正向重复、16个回文重复、2个反向重复和1个互补重复;而蕤核中有18个正向重复、22个回文重复、6个反向重复和3个互补重复。串联重复序列分别有49个和58个(图2A)。经MISA软件分析,在扁核木叶绿体基因组中共筛选出100个SSRs位点,其中单核苷酸重复有77个(均是A/T碱基重复,没有C/G碱基重复)、双核苷酸重复有3个、三核苷酸重复有3个、四核苷酸重复有7个、六核苷酸重复有8个,没有五核苷酸重复类型;而在蕤核叶绿体基因组中仅筛选出84个SSRs位点,其中单核苷酸重复有59个(A/T碱基重复58个,C/G碱基重复1个),双核苷酸重复(AT)11个、三核苷酸重复2个、四核苷酸重复10个、五核苷酸重复3个,无六核苷酸重复类型(图2B)。

图2 重复结构与SSR类型
2.3 叶绿体基因组学序列分析
以蕤核叶绿体基因组作参考,检测扁核木叶绿体基因组中蛋白质编码基因变异位点上的差异。其中,基因上的插入和缺失位点数均大于10,而基因上的插入和缺失总位点数更是达55个。其次是和基因,基因上的插入和缺失位点均有3个,插入位点最大有276 bp(图3)。扁核木叶绿体基因组特有基因为,蕤核特有基因为UAGUGC和。
SNP(single nucleotide polymorphism,单核苷酸多态性)标记作为最丰富的突变类型,在扁核木叶绿体基因组编码区共检测到6971个SNP,包括4288个Ts(Transition,转换)和2683个Tv(Transversion,颠换),Ts与Tv的比率是1.60∶1。其中,C与T之间的Ts有2247个,A与G之间的Ts有2041个;而A与T之间的Tv有728个,C与G之间的Tv有409个。在众多替换位点中,基因上的替换位点占总替换位点数的20.63%,占有较高水平,说明基因具有较高的进化率(表2)。
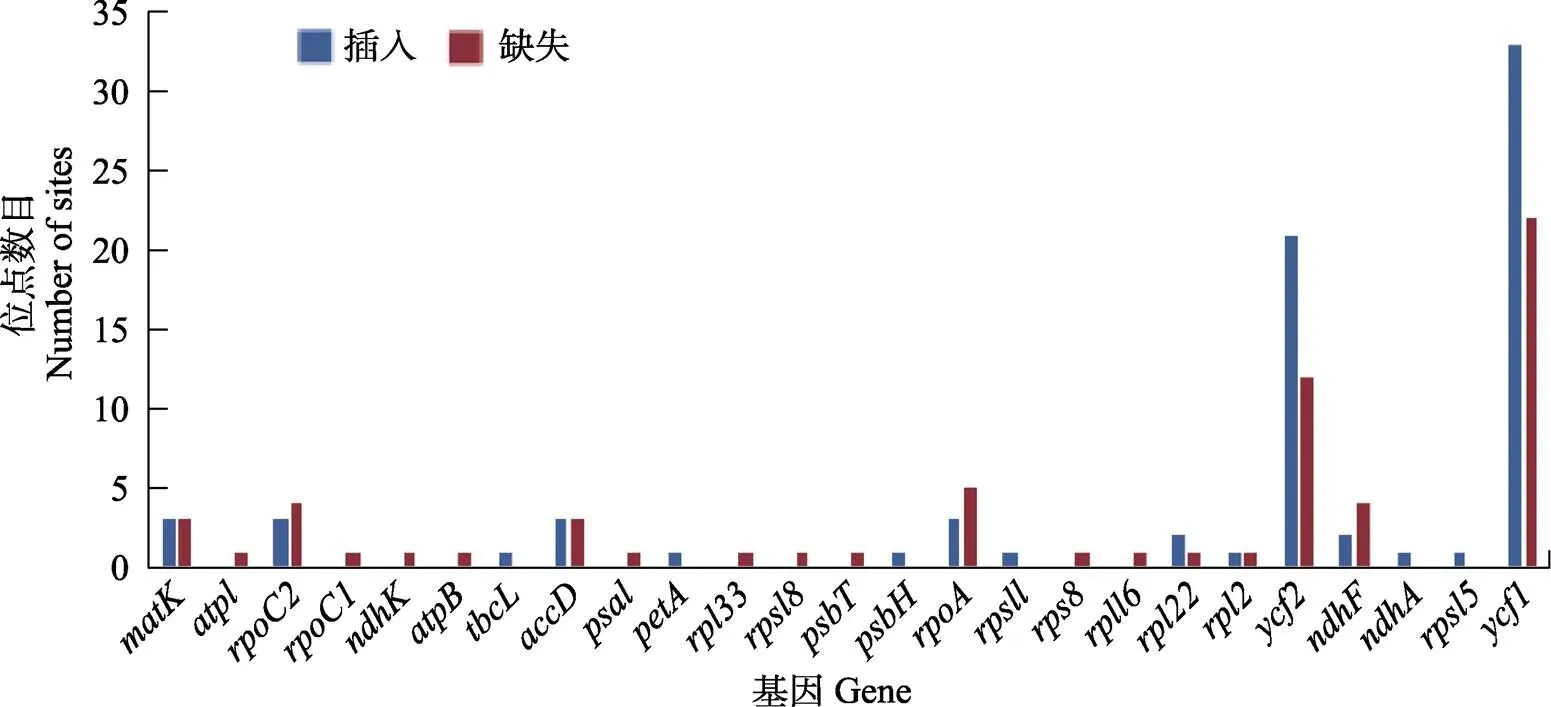
图3 扁核木叶绿体基因组的插入与缺失
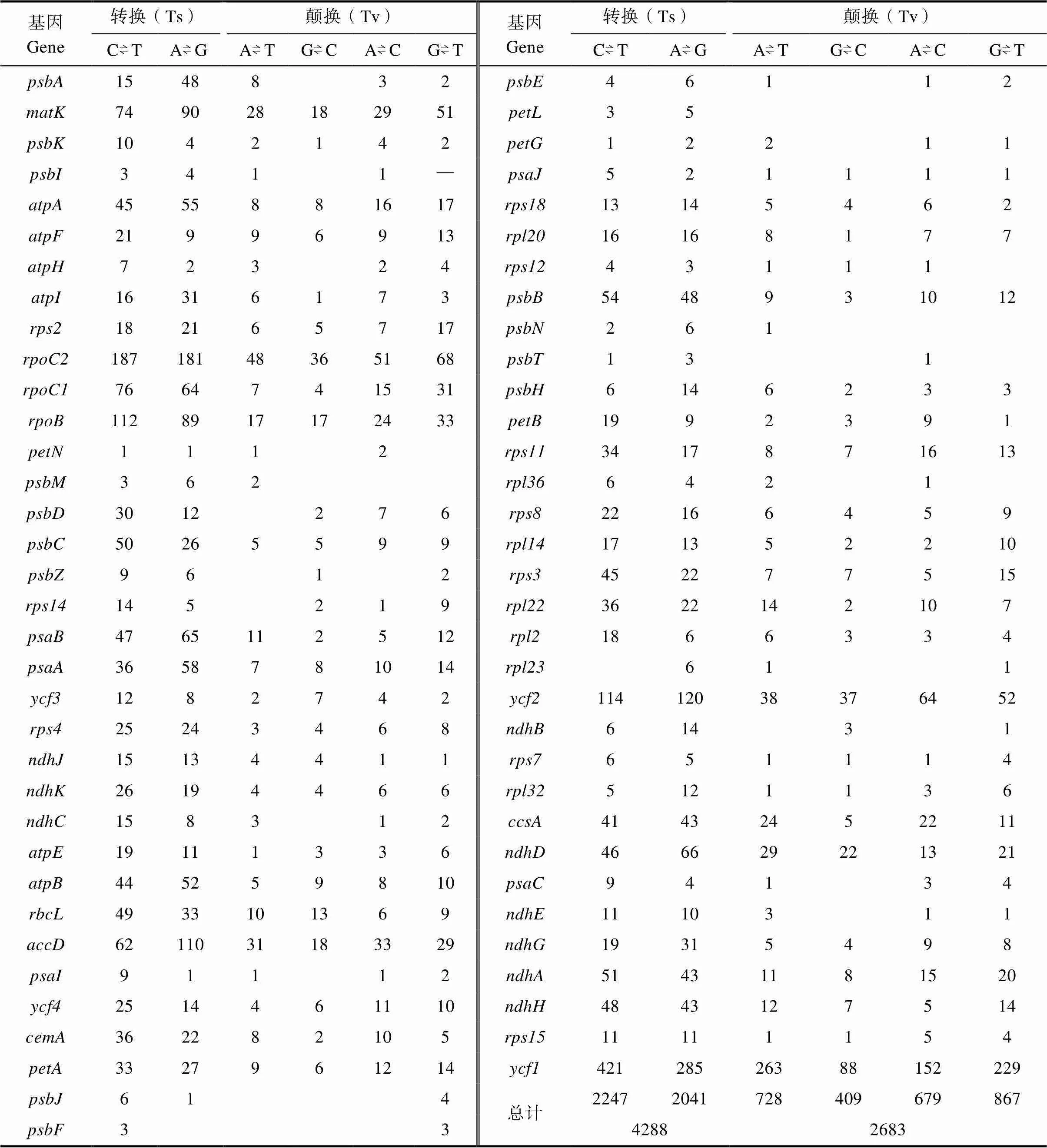
表2 扁核木、蕤核叶绿体基因组编码区单核苷酸突变情况
2.4 密码子偏好性分析
2.4.1 密码子组成分析 经CodonW 1.4.2和在线CUSP分析,如表3所示,扁核木和蕤核叶绿体基因组密码子第3位碱基中T3s分别是46.04%、46.46%,A3s分别是42.63%、43.09%,说明扁核木属植物的叶绿体基因组密码子偏好A/U结尾。20~61是ENC的理论取值范围,ENC值越接近20,其同义密码子偏性越强,反之越弱。扁核木、蕤核的ENC值分别为48.74和48.43,说明扁核木属植物的密码子偏好性较弱。
利用热图统计扁核木和蕤核编码基因密码子的相关参数值,行列名分别对应每个基因的名称和相关参数。如图4所示,在2种扁核木属植物中,基因不同,其GC含量也存在差异,多数基因的GC1和GC2含量高于GC3;扁核木和蕤核的ENC实际取值范围分别为38.44~61.00和35.94~60.48,所有基因的ENC值均大于35,进一步表明扁核木属植物叶绿体基因密码子偏性较弱。另外,和基因的ENC值在扁核木与蕤核间的极差均大于5,说明同属植物间部分基因的密码子偏好性存在较大差异。
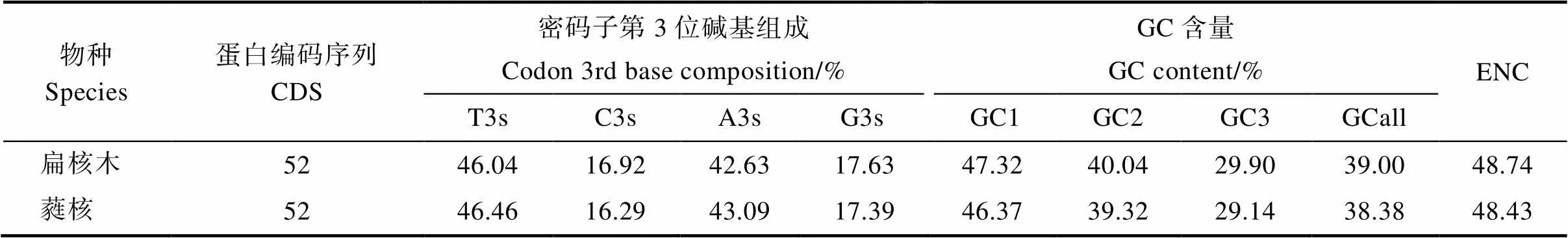
表3 扁核木和蕤核叶绿体基因组密码子相关参数
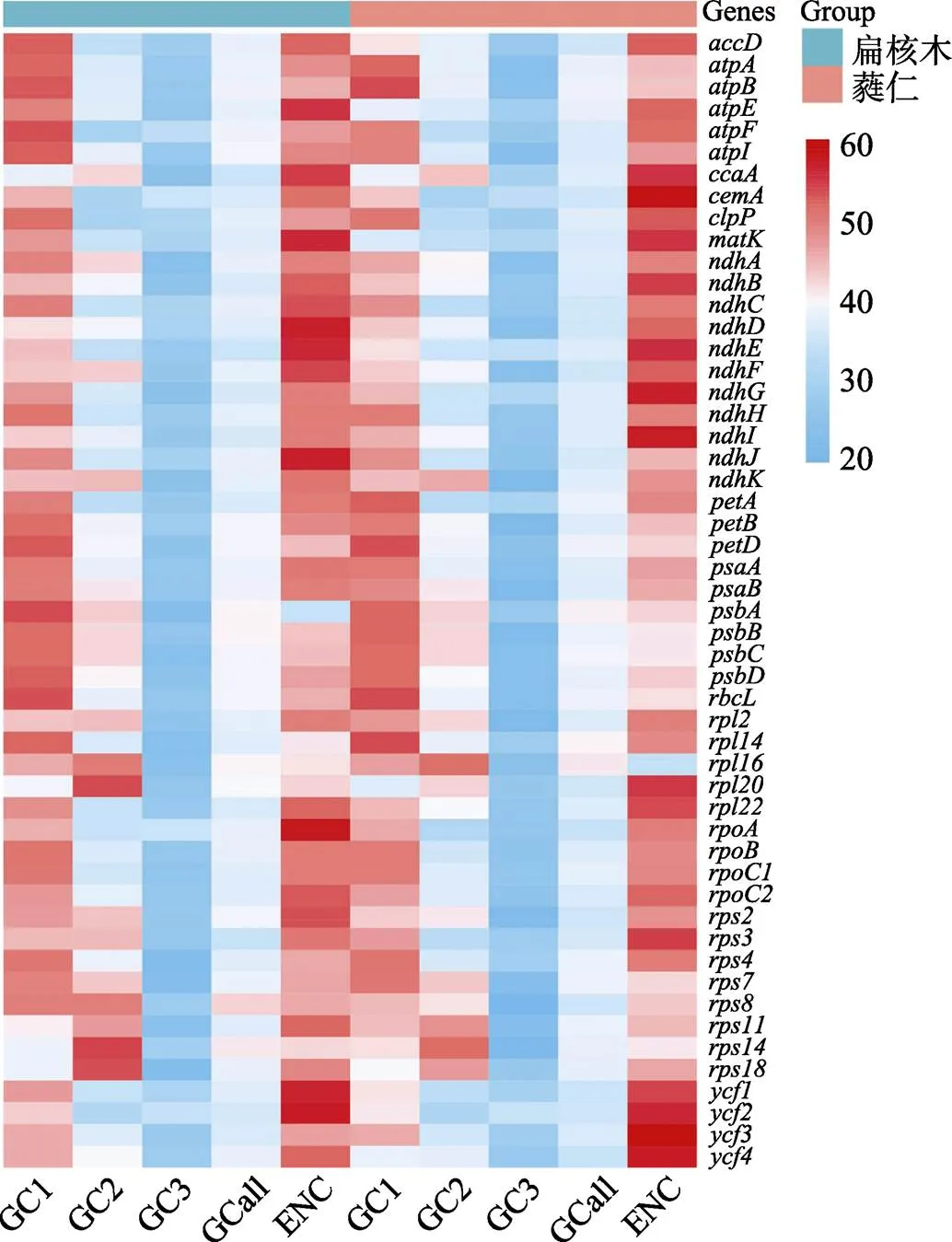
图4 扁核木和蕤核叶绿体编码基因密码子GC含量
2.4.2 中性绘图分析 从图5可以看出,扁核木GC12的取值介于0.34~0.52之间,GC3取值介于0.22~0.38之间,GC12与GC3相关系数=0.1975;蕤核GC12取值在0.31~0.54之间,GC3取值在0.22~0.37之间,GC12与GC3相关系数=0.2555,这说明扁核木属植物GC12与GC3的相关性弱,密码子第1、2位与第3位之间碱基的相关性不显著,进化方式存在一定差异,进而可判断选择压力是影响密码子偏好性的主要原因。

图5 中性绘图
2.4.3 PR2-plot分析 PR2-plot绘图可直观地看出密码子第3位嘌呤(A、G)和嘧啶(C、T)的使用频率,若各编码基因在4个区域内均匀分布,则4个碱基的使用频率相等,密码子偏好性完全受突变影响。从图6中可以看出2种扁核木属植物叶绿体基因分布并不均匀,多数基因汇集在右下方区域,从碱基使用频率来看,T>A、G>C,密码子第3位的嘧啶使用频率高于嘌呤,说明2种植物叶绿体基因组密码子偏好性除了受突变的影响,还与其他因素有关。

图6 PR2-plot绘图
2.4.4 最优密码子确定 根据ENC值对2种扁核木属植物叶绿体基因组CDS进行排序,分别建立高、低表达基因库,计算表达库中密码子的RSCU值和ΔRSCU值。以既符合高表达密码子(ΔRSCU≥0.08)又符合高频密码子(RSCU>1)为条件来筛选最优密码子。结果如图7所示,扁核木含有高表达密码子29个,高频密码子24个,同时满足高表达、高频的最优密码子有15个,其中以U结尾的有9个,以A结尾的有6个;而蕤核含有高表达密码子25个,高频密码子28个,同时满足高表达、高频的最优密码子有15个,其中7个以U结尾,7个以A结尾,还有1个以G结尾。通过对最优密码子的进一步分析,发现了2种植物中共有6个最优密码子,分别是UGU、UUA、GAA、CAA、AUU、GCU。
2.5 核苷酸多态性分析
利用DnaSP6软件分析2种扁核木属植物叶绿体基因组序列的核苷酸变异程度。结果表明大部分变异发生在LSC区域,且与SSC和IR区相比表现出更高的核苷酸变异。核苷酸多态性指数>0.015的3个间隔区均位于LSC区,分别为GUG(1~1362 bp),UCCCAU(39 550~41 465 bp)和(74 082~75 529 bp)(图8)。
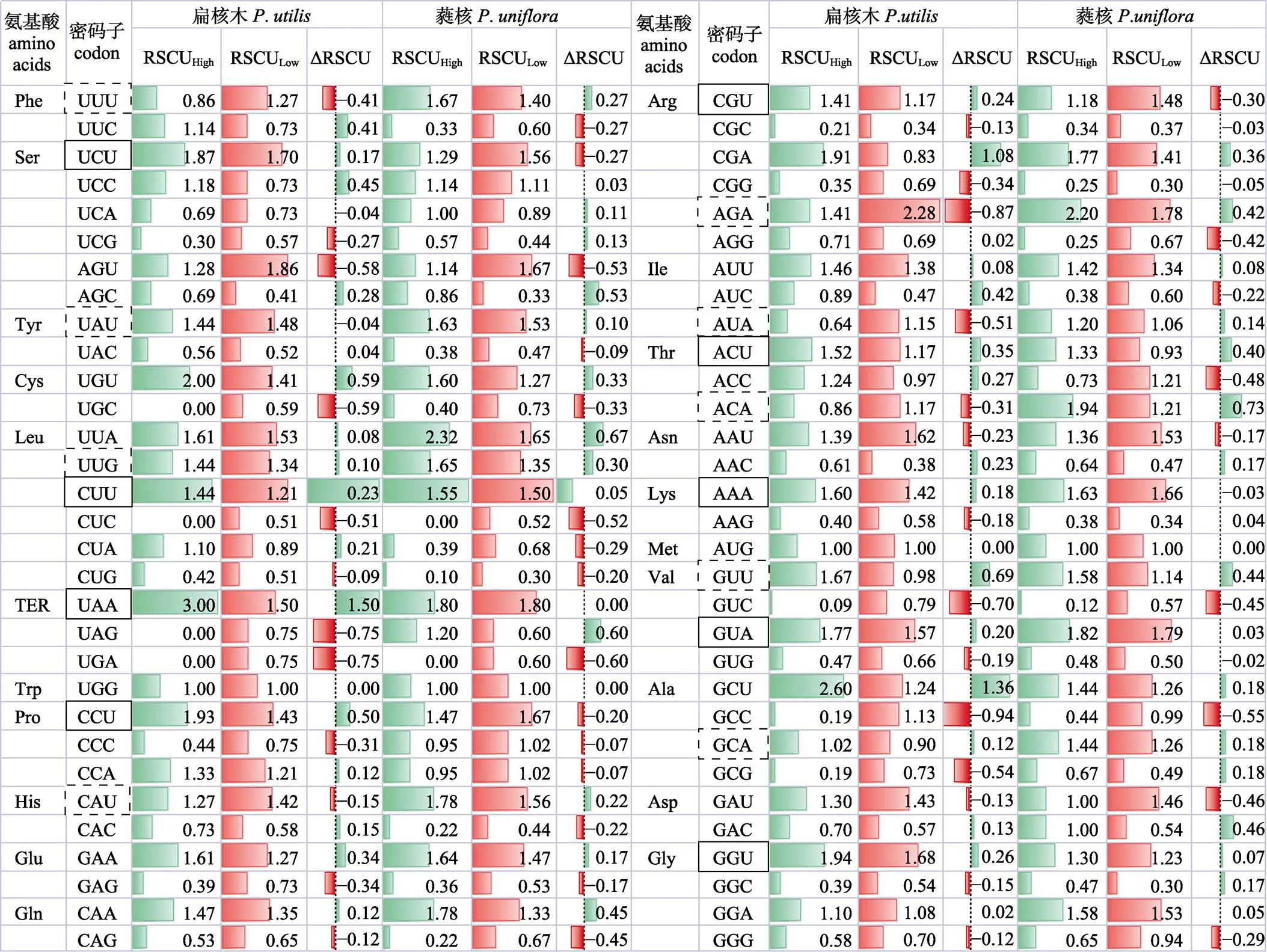
注:框选的密码子为扁核木最优密码子; 框选的密码子为蕤核最优密码子;加粗的密码子为2种植物共有的最优密码子。
2.6 系统发育分析
以黑刺李()为外类群,基于叶绿体基因组的间隔区序列构建扁核木属植物系统发育树。结果如图9所示,系统发育树以100%的支持率将4种扁核木属植物划分为2个小类,扁核木和台湾扁核木聚为一类,蕤核和东北扁核木聚为另一类,由此可以看出,扁核木属植物具有明显的南北系分化。

图8 扁核木属植物叶绿体基因核苷酸多态性
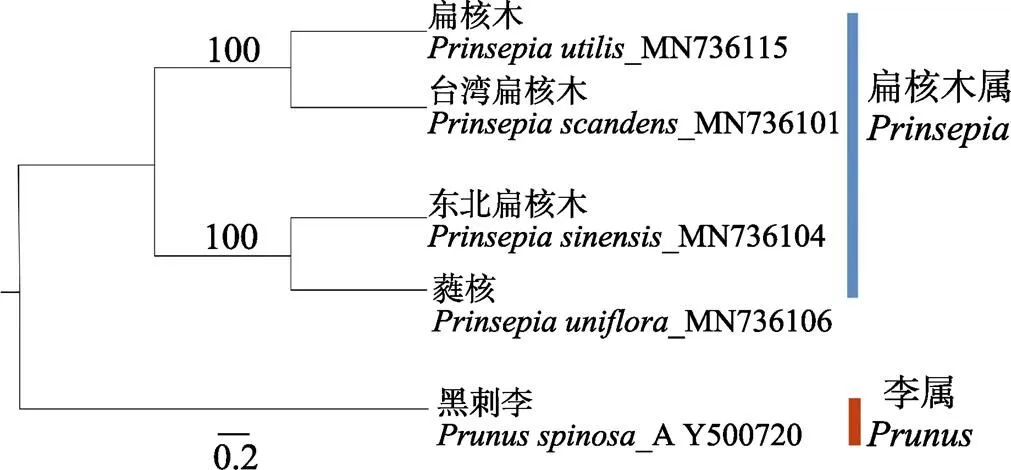
图9 基于叶绿体基因组的trnS-trnG间隔区序列构建的系统发育树
3 讨论
SSR现已作为一种高效的分子标记工具被广泛应用于遗传图谱构建、目的基因标定、种质资源鉴定等方面[29-30]。利用SSR分析从2种扁核木属植物叶绿体基因组中分别筛选出100个和84个SSR位点,其中多是以A/T碱基为主的单核苷酸重复类型,占总位点数的69.04%~77.00%;其次是二核苷酸、三核苷酸和四核苷酸,但占比均不到10%,说明随着核苷酸数的增加,占比会逐渐降低。这与在柴胡属植物叶绿体基因组得出的随着拷贝数目的增加SSRs数量明显减少这一结论相一致[31]。另外,二核苷酸及以上核苷酸重复单元也主要由A、T碱基组合而成,如二核苷酸重复(AT)、三核苷酸重复(AAT/ATT)、四核苷酸重复(AAAT/ATTT/AATT)等。蕤核叶绿体基因组中AT重复在二核苷酸重复类型中占到了100%,AAT/ATT重复在三核苷酸重复类型也占比100%,这进一步说明叶绿体基因组中的SSR序列组成碱基主要是A、T,较少有C、G串联重复[32]。筛选出来的这些SSR位点可以为扁核木属植物的种质鉴定及遗传多样性分析等研究提供分子标记基础。同时,核苷酸多样性分析发现,>0.015的高变异区有3个,包括了9个基因(-GUG、、、-UCC、-CAU、),其中UCCCAU和之前已经在不同植物中被检测到作为高度可变的区域,其中一些被用作DNA条形码[33-34]。
选用蕤核叶绿体基因组为参考,统计分析扁核木叶绿体基因组突变的数量、形式及特有基因。发现二者叶绿体基因组编码区存在大量的插入、缺失和SNP突变位点,从而导致扁核木和蕤核叶绿体基因组大小以及以和等为代表的编码基因序列存在较大差异,这些差异显著的基因将有助于后期的物种分类研究。WANG等[19]通过蔷薇科叶绿体基因组进化和突变模式的研究,发现在所有蛋白编码基因中序列的分化程度最高,相比其他基因更有助于物种区分,这与本研究结果一致。相关研究表明,第四纪以来多次气候(冰期与间冰期)的周期性波动很大程度上影响了现今植物种群分布和遗传格局[35]。刘家奇等[15]基于叶绿体基因组、和片段分析发现蕤核与我国北方多数植物类群一样,其遗传结构受气候及环境影响有着明显的遗传分化。而经基因类型比较,在蕤核叶绿体基因组中发现多个新编码基因,推测其可能与北方长期特殊的气候和环境响应有关。
叶绿体基因组间隔区序列不受基因功能限制,进化速率快。研究表明-间隔区能提供丰富的信息位点且引物通用,在种间及近亲属间的系统发育研究中应用广泛[28]。因此,本研究基于扁核木属叶绿体基因组的间隔区序列来构建系统发育树,将4种扁核木属植物按遗传距离及亲缘关系远近划分为南系(扁核木、台湾扁核木)和北系(蕤核、东北扁核木)两类。而陆玲娣[36]根据花蕊数量分类以及杨瑞林等[16]根据叶表皮角质膜及花粉外壁条脊排列形态进行聚类分析的研究结论与此一致。同时,通过核苷酸多态性分析发现,间隔区在> 0.015的高变区内,具有较显著的多态性,大小为1039 bp,进一步说明叶绿体基因组中的基因片段用于扁核木属植物的分类鉴定是可行的。
扁核木属植物叶绿体基因组同义密码子偏好以A/T结尾,这与多数陆生植物的研究结果一致,如:槲蕨属[37]、石斛属[38]植物等,并且扁核木叶绿体基因组中筛选出来的SSR序列也多是A、T串联重复,这可能与陆生植物叶绿体基因组富含A、T碱基且进化相对保守等有关。
同义密码子使用偏倚现象在绝大多数生物中均存在,但偏倚程度却因物种不同而异[39]。相关研究表明,造成密码子使用偏倚的原因并非单一因素所决定的,各物种在不同的环境中长期受选择压力、碱基突变、基因漂移等多因素影响,而密码子使用偏性就是在这个复杂的物种选择进化过程中形成的[40-41]。密码子第3位上的碱基突变不改变氨基酸类型,受到的选择压力较小[42]。因此,本研究基于GC3含量通过中性绘图分析发现自然选择是影响扁核木属植物密码子偏好性的主导因素。另外,从PR2-plot绘图可看出密码子第3位嘌呤(A、G)和嘧啶(C、T)的使用频率不尽相同,T>A、G>C,说明2种植物叶绿体基因组密码子偏好性除了受突变的影响,还与选择、碱基组成等其他因素有关。这与香花枇杷[10]、灯盏花[43]、杜梨[44]等植物叶绿体基因组密码子偏好性分析结果一致,说明选择和突变是造成植物密码子偏倚的主要动力来源。
[1] HENRY D, IN C S, YU M, CHANG W J. Chloroplast genomes: diversity, evolution and applications in genetic engineering[J]. Genome Biology, 2016, 17(1): 134.
[2] PARK I Y, SONG J H, YANG S Y, MOON B C. Comparative analysis ofchloroplast genomes and molecular marker development for the identification of authentic[J]. Plants, 2020, 9(2): 157.
[3] CLAUDIA L H, ABDULLAH, IBRAR A, MONICA M C, ALEJANDRO Z, THOMAS B C, MICHAEL R M. Evolutionary dynamics of chloroplast genomes in subfamily(Araceae)[J]. Genomics, 2020, 112(3): 2349-2360.
[4] 段义忠, 张 凯. 沙冬青属植物叶绿体基因组对比和系统发育分析[J]. 西北植物学报, 2020, 40(8): 1323-1332.
DUAN Y Z, ZHANG K. Comparative analysis and phylogenetic evolution of the complete chloroplast genome of[J]. Northwestern Journal of Botany, 2020, 40(8): 1323-1332. (in Chinese)
[5] 德 伟, 张一鸣. 生物化学与分子生物学[M]. 南京: 东南大学出版社, 2018.
DE W, ZHANG Y M. Biochemistry and molecular biology[M]. Nanjing: Southeast University Press, 2018. (in Chinese)
[6] 王 婧, 王天翼, 王罗云, 张建国, 曾艳飞. 沙枣叶绿体全基因组序列及其使用密码子偏性分析[J]. 西北植物学报, 2019, 39(9): 1559-1572.
WANG J, WANG T Y, WANG L Y, ZHANG J G, ZENG Y F. Assembling analysis of the whole chloroplast genome sequence ofand its codon usage bias[J]. Northwest Botany, 2019, 39(9): 1559-1572. (in Chinese)
[7] LIU S S, QIAO Z Q, WANG X M, ZENG H J, LI Y X, CAI N, CHEN Y. Analysis of codon usage patterns in “lonicerae flos” (Hand.-Mazz.) based on transcriptome data[J]. Gene, 2019(705): 127-132.
[8] 王占军, 丁 亮, 蔡倩文, 李 杰, 吴子琦, 姚怀桐, 王 悦, 焦春燕, 欧祖兰, 徐忠东, 施季森, 陈金慧. 3种木薯全基因组的密码子偏好性模式与变异来源比较[J]. 应用与环境生物学报, 2021, 27(4): 1013-1021.
WANG Z J, DING L, CAI Q W, LI J, WU Z Q, YAO H T, WANG Y, JIAO C Y, OU Z L, XU Z D, SHI J S, CHEN J H. Comparison of codon preference patterns and variation sources in cassavagenomes[J]. Journal of Application and Environmental Biology, 2021, 27(4): 1013-1021. (in Chinese)
[9] 辛雅萱, 黎若竹, 李 鑫, 陈丽琼, 唐军荣, 屈亚亚, 杨琳懿, 辛培尧, 李云芳. 杧果叶绿体基因组密码子使用偏好性分析[J]. 中南林业科技大学学报, 2021, 41(9): 148-156, 165.
XIN Y X, LI R Z, LI X, CHEN L Q, TANG J R, QU Y Y, YANG L Y, XIN P Y, LI Y F. Analysis on codon usage bias of chloroplast genome in[J]. Journal of Central South University of Forestry & Technology, 2021, 41(9): 148-156, 165. (in Chinese)
[10] 屈亚亚, 辛 静, 冯发玉, 董章宏, 瞿绍宏, 王海洋, 李 斌, 辛培尧. 香花枇杷质体基因组序列密码子偏性分析[J]. 西北林学院学报, 2021, 36(4): 138-144, 158.
QU Y Y, XIN J, FENG F Y, DONG Z H, QU S H, WANG H Y, LI B, XIN P Y. Codon usage bais in chloroplast genome ofChamp. ex Benth.[J]. Journal of Northwestern Forestry College, 2021, 36(4): 138-144, 158. (in Chinese)
[11] 原晓龙, 刘 音, 康洪梅, 陈中华, 李云琴, 王 毅. 蒜头果叶绿体基因组密码子偏好性分析[J]. 西南林业大学学报(自然科学), 2021, 41(3): 15-22.
YUAN X L, LIU Y, KANG H M, CHEN Z H, LI Y Q, WANG Y. Analysis of codon usage bias in chloroplast genome of[J]. Journal of Southwest Forestry University (Natural Sciences), 2021, 41(3): 15-22. (in Chinese)
[12] 周海燕, 杨峻山. 扁核木属植物化学成分及药理作用研究进展[J]. 中国现代中药, 2011, 13(5): 44-47.
ZHOU H Y, YANG J S. Progress in chemical composition and pharmacological action of[J]. Modern Chinese Medicine, 2011, 13(5): 44-47. (in Chinese)
[13] 解仲伯, 林华庆, 余楚钦, 王远苹. 青刺果各类提取物的药理作用研究进展[J]. 中成药, 2016, 38(12): 2651-2656.
XIE Z B, LIN H Q, YU C Q, WANG Y P. Progress in the pharmacological effects of various extracts of[J]. Chinese Patent Medicine, 2016, 38(12): 2651-2656. (in Chinese)
[14] 李新蕾, 李叶芳, 李凤荣, 关文灵. 干旱胁迫对扁核木种子萌发及幼苗生理特性的影响[J]. 云南农业大学学报(自然科学), 2020, 35(4): 682-687.
LI X L, LI Y F, LI F R, GUAN W L. Effects of drought stress on seed germination and seedling physiological characteristics of[J]. Journal of Yunnan Agricultural University (Natural Sciences), 2020, 35(4): 682-687. (in Chinese)
[15] 刘家奇, 付孟娇, 吴海洋, 田 斌. 基于叶绿体DNA的黄土高原特有植物蕤核的谱系地理学[J]. 广西植物, 2021, 41(3): 396-403.
LIU J Q, FU M J, WU H Y, TIAN B. Phylogeography of the Loess Plateau endemic plantinferred from the chloroplast DNA[J]. Guangxi Plants, 2021, 41 (3): 396-403. (in Chinese)
[16] 杨瑞林, 魏学智. 中国扁核木属植物花粉和叶表皮微形态研究[J]. 植物研究, 2015, 35(2): 172-178.
YANG R L, WEI X Z. Micromorphological observations on leaf epidermis and pollen ofRoyle in China[J]. Plant Research, 2015, 35(2): 172-178. (in Chinese)
[17] 叶俊伟, 田 斌. 中国西南地区重要木本油料植物扁核木的遗传结构及成因[J]. 生物多样性, 2021, 29(12): 1629-1637.
YE J W, TIAN B. Genetic structure and causes of an important woody oil plants in Southwest China of[J]. Biodiversity, 2021, 29(12): 1629-1637. (in Chinese)
[18] 范志远, 习学良, 欧阳和, 陈 武, 廖永坚, 邹伟烈. 青刺果的植物学特性及其人工栽培技术[J]. 西部林业科学, 2005(4): 47-52, 66.
FAN Z Y, XI X L, OU Y H, CHEN W, LIAO Y J, ZOU W L. Phyany properties ofand their artificial cultivation techniques[J]. Western Forestry Science, 2005(4): 47-52, 66. (in Chinese)
[19] WANG S, SHI C, GAO L Z. Plastid genome sequence of a wild woody oil species,, provides insights into evolutionary and mutational patterns of Rosaceae chloroplast genomes[J]. Public Library of Science, 2017, 8(9): 73946.
[20] WANG Y L, NIU Y, QIANG Z, TIAN Y, LI Y. The complete chloroplast genome of(Rosaceae), a medicinal plant found in northwest China[J]. Mitochondrial DNA Part B, 2021, 6(10): 3057-3058.
[21] KURTZ S, CHOUDHURI J V, OHLEBUSCH E, SCHLEIERMACHER C, STOYE J, GIEGERICH R. REPuter: the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Research, 2001(29): 4633-4642.
[22] BENSON G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 1999(27): 573- 580.
[23] 王 希, 陈 丽, 赵春雷. 利用MISA工具对不同类型序列进行SSR标记位点挖掘的探讨[J]. 中国农学通报, 2016, 32(10): 150-156.
WANG X, CHEN L, ZHAO C L. Exploration of SSR marker site mining for different types of sequences using the MISA tools[J]. China Agriculture Bulletin, 2016, 32(10): 150-156. (in Chinese)
[24] 傅建敏, 索玉静, 刘慧敏, 谭晓风. 柿属植物叶绿体蛋白质编码基因密码子用法[J]. 经济林研究, 2017, 35(2): 38-44.
FU J M, SUO Y J, LIU H M, TAN X F. Codon usage of chloroplast protein-coding genes in[J]. Economic Forest Study, 2017, 35(2): 38-44. (in Chinese)
[25] JIANG Y, DENG F, WANG H L, HU Z H. An extensive analysis on the global codon usage pattern of baculoviruses[J]. Archives of Virology, 2008, 153(12): 2273.
[26] YANG G F, SU K L, ZHAO Y R. Analysis of codon usage in the chloroplast genome of[J]. Acta Pharmacologica Sinica, 2015, 6(12): 171-179.
[27] 杜致辉, 杨 澜, 张朝君, 许红娟, 陈之林. 黑喉石斛叶绿体基因组特征及比较分析[J]. 热带作物学报, 2021, 42(11): 3111-3119.
DU Z H, YANG L, ZHANG C J, XU H J, CHEN Z L. Characteristics of the complete chloroplast genome ofand its comparative analysis[J]. Journal of Tropical Crops, 2021, 42(11): 3111-3119. (in Chinese)
[28] 董雷雷. 叶籽银杏序列分析及系统发育研究[D]. 济南: 山东农业大学, 2009.
DONG L L.sequence analysis and phylogenetic study ofL.[D]. Jinan: Shandong Agricultural University, 2009. (in Chinese)
[29] 耿树香, 杨生超, 宁德鲁, 李勇杰. 丽江油橄榄品种表型及SSR标记的多样性及聚类分析[J]. 西部林业科学, 2018, 47(6): 30-34.
GENG S X, YANG S C, NING D L, LI Y J. Diversity and cluster analysis ofphenotype and SSR markers in Lijiang[J]. Western Forestry Science, 2018, 47(6): 30-34. (in Chinese)
[30] CHMIELEWSKI M, MEYZA K, CHYBIKI I J, DZIALUK A, LITKOWIEC M, BURCZYK J. Chloroplast microsatellites as a tool for phylogeographic studies: the case of white oaks in Poland[J]. iForest Biogeosciences and Forestry, 2015(8): 765-771.
[31] 张明英, 张雨曲, 李依民, 高 静, 沈 霞, 杨新杰, 颜永刚, 王昌利, 张 岗. 北柴胡、紫花阔叶柴胡叶绿体全基因组解析及柴胡属药用植物叶绿体基因组比较与系统发育分析[J]. 药学学报, 2021, 56(2): 618-629.
ZHANG M Y, ZHANG Y Q, LI Y M, GAO J, SHEN X, YANG X J, YAN Y G, WANG C L, ZHANG G. Complete plastid genomes ofDC. andH. Wolff, with comparative and phylogenetic analyses of medicinalspecies[J]. Pharmaceutical Journal, 2021, 56(2): 618-629. (in Chinese)
[32] KUANG D Y, WU H, WANG Y L, GAO L M, ZHANG S Z, LU L. Complete chloroplast genome sequence of(Magnoliaceae): implication for DNA barcoding and population genetics[J]. Genome, 2011(54): 663-673.
[33] HU Y, WOESTE K E, ZHAO P. Completion of the chloroplast genomes of five Chineseand their contribution to chloroplast phylogeny[J]. Frontiers in Plant Science. 2016(7): 1955.
[34] LI X, YANG Y, HENRY R J, ROSSETTO M, WANG Y T, CHEN S L. Plant DNA barcoding: from gene to genome[J]. Biological Reviews of the Cambridge Philosophical Society, 2015, 90(1): 157-166.
[35] PROVAN J, BENNETT K D. Phylogeographic insights into cryptic glacial refugia[J]. Trends in Ecology and Evolution, 2008, 23(10): 564-571.
[36] 陆玲娣. 中国植物志: 第38卷[M]. 北京: 科学出版社, 1986.
LU L D. Chinese flora: Volume 38[M]. Beijing: Science Press, 1986. (in Chinese)
[37] 沈宗芳, 陆添权, 张志荣, 蔡传涛, 田 波. 槲蕨属叶绿体基因组密码子偏好性分析[J]. 广西植物, 2021, 41(2): 266-273.
SHEN Z F, LU T Q, ZHANG Z R, CAI Z T, TIAN B. Analysis of codon preference in the chloroplast genome of[J]. Guangxi Plants, 2021, 41(2): 266-273. (in Chinese)
[38] 王晓双, 杨 芳, 罗 茜, 刘映良, 朱 斌. 石斛属叶绿体基因组密码子使用偏性及系统发育分析[J]. 分子植物育种, 2021(3): 1-22.
WANG X S, YANG F, LUO Q, LIU Y L, ZHU B. Codon usage bias and phylogenetic analysis of chloroplast genome of[J]. Molecular Plant Breeding, 2021(3): 1-22. (in Chinese)
[39] LI G L, PAN Z L, GAO S C, HE Y Y, XIA Q Y, JIN Y, YAO H P. Analysis of synonymous codon usage of chloroplast genome in[J]. Genes and Genomics, 2019, 41(10): 1173-1181.
[40] XU C, CAI X M, CHEN Q Z, ZHOU H X, CAI Y, BEN A L. Factors affecting synonymous codon usage bias in chloroplast genome ofRamsey[J]. Evolutionary Bioinformatics, 2011(7): 271-278.
[41] TANG D F, WEI F, CAI Z Q, WEI Y Y, KHAN A, MIAO J H, WEI K H. Analysis of codon usage bias and evolution in the chloroplast genome ofBenth[J]. Development Genes and Evolution, 2020, 11(4): 670-679.
[42] 叶友菊, 倪州献, 白天道, 徐立安. 马尾松叶绿体基因组密码子偏好性分析[J]. 基因组学与应用生物学, 2018, 37(10): 4464-4471.
YE Y J, NI Z X, BAI T D, XU L A. Codon preference analysis ofchloroplast genome[J]. Genomics and Applied Biology, 2018, 37(10): 4464-4471. (in Chinese)
[43] 李显煌, 杨生超, 辛雅萱, 屈亚亚, 杨琳懿, 冯发玉, 范 伟, 辛培尧. 灯盏花叶绿体基因组密码子偏好性分析[J]. 云南农业大学学报(自然科学), 2021, 36(3): 384-392.
LI X H, YANG S C, XIN Y X, QU Y Y, YANG L Y, FENG F Y, FAN W, XIN P Y. Analysis of the codon usage bias of chloroplast genome in(Vant.) Hand- Mazz[J]. Journal of Yunnan Agricultural University (Natural Sciences), 2021, 36(3): 384-392. (in Chinese)
[44] 辛雅萱, 董章宏, 瞿绍宏, 刘 成, 叶 鹏, 辛培尧. 杜梨叶绿体基因组密码子偏好性分析[J]. 河北农业大学学报, 2020, 43(6): 51-59.
XIN Y X, DONG Z H, QU S H, LIU C, YE P, XIN P Y. Analysis on codon usage bias of chloroplast genome inBge.[J]. Journal of Hebei Agricultural University, 2020, 43(6): 51-59. (in Chinese)
Analysis of Chloroplast Genome Characteristics of
WANG Fei1,2, ZHAO Wenzhi1,2, DONG Zhanghong2, MA Luyao1,2, LI Weiying1,2, XIA Maotian1,2, WANG Zhengde1,2, XIN Peiyao2*
1. Southwest Research Center for Landscape Architecture Engineering, State Forestry and Grassland Administration, Southwest Forestry University, Kunming, Yunnan 650224, China; 2. Key Laboratory of National Forestry and Grassland Administration on Biodiversity Conservation in Southwest China, Southwest Forestry University, Kunming, Yunnan 650224, China
There are four species ofRoyle in China, which have high edible and medicinal value. In order to elucidate the chloroplast genome characteristics of, we analyzed the chloroplast genome structure, simple sequence repeats (SSR) loci, the preference of the codon, and sequence variation using bioinformatics methods based on the published chloroplast genome sequences of, and constructed a phylogenetic tree in. The results showed that the chloroplast genome sizes ofwere ranged from 159 179 bp to 168 206 bp, and the average GC content was 37.3%. In terms of the number of coding genes, only two tRNAs were different, while the number of protein-coding genes and rRNA were all consistent, showing a high level of conservation between species. A total of 49 dispersed repeats were detected in the chloroplast genomes ofand, including mainly forward and palindromic repeats, accounting for 73% to 82%, while the number of tandem repeats was 49 and 58, respectively. By the simple repetitive sequence (SSR) analysis, 100 and 84 SSRs loci were detected respectively in the chloroplast genome sequence ofand, and most of SSRs were single nucleotide repeats with A/T base. The codon bias analysis of chloroplast genome offound that the base content of GC3 was significantly lower than that of GC1 and GC2, indicating that the codon preference ended with A and U, and the effective number of codon (ENC) values was all greater than 48%, indicating that the codon bias was weak. Analysis of Neutral-plot and PR2-plot showed that natural selection was the main factor affecting the codon preference ofBy establishing high and low gene expression libraries and using RSCU values as reference, six optimal codons ofwere identified. Three highly variable regions-GUG-,UCCCAUandwere screened by the chloroplast genome sequence variation analysis with nucleotide polymorphism index Pi>0.015. Taking the chloroplast genome ofas a reference, a large number of insertions, deletions and SNP mutations were found in the coding region of chloroplast genome in, and several unique genes ofchloroplast genome were found. Based on the phylogenetic trees byspacers in the chloroplast genomes of, there were two groups in the four species of, one was southern (), and the other was northern (). The present study would provide a theoretical basis for study of the phylogeny, taxonomic identification and resource exploitation of the genus in.
Royle; chloroplast genome; characteristics analysis; phylogeny
Q732
A
10.3969/j.issn.1000-2561.2022.09.002
2022-02-15;
2022-03-09
云南省科技厅科技计划重点研发项目(No. 2018BB005).
王 飞(1996—),男,硕士研究生,研究方向:林木分子生物学与遗传改良。*通信作者(Corresponding author):辛培尧(XIN Peiyao),E-mail:xpytgyx@163.com。
