天料木属植物叶绿体基因组密码子使用偏性分析
2022-10-12尹为治李佳灵黄良鸿方正
尹为治,李佳灵,2,黄良鸿,方正
1.海南热带雨林国家公园管理局五指山分局,海南五指山 572299;2.海南大学生态环境学院,海南海口 570228
密码子不均衡使用的特性,被称为密码子使用偏性,所有已知物种的基因组都存在密码子偏好性,但偏好性各不相同[1]。对密码子使用偏性的研究有助于理解环境适应的分子机制、探讨物种间的亲缘关系[2]。目前,竹亚科[2]、槲蕨属[3]、柿属[4]、大戟科[5]等许多研究证实了密码子使用偏性对植物叶绿体基因组水平的适用性。
叶绿体基因组功能与光合作用联系密切[6],且具有基因组小,携带遗传信息多,在分子水平上差异大等特点[7]。被广泛应用于植物的物种鉴定[8-9]、遗传多样性分析[10]、系统发育学分析[11]等方面的研究。目前,有两千多种植物的叶绿体基因组已被测序,并在NCBI 发表。其中,包含了广南天料木(Homalium paniculiflorum)[12]、斯里兰卡天料木(Homalium ceylanicum)[12]、狭叶天料木 (Homalium stenophyllum)[12]、天料木(Homalium cochinchinense)[13]等天料木属植物的叶绿体基因组序列。
天料木属植物属于大风子科,中国有12 种和3个变种,主要分布于海南、广东、广西、云南等省的低海拔雨林中[14]。海南省原记载有7 种[15]。该属植物除较高的观赏和材用价值外,根、茎、叶等部位均有较高的药用价值[16-18]。近年来,天料木属植物叶绿体基因组数据不断增加。目前在密码子水平上对天料木属植物的叶绿体分子进化分析尚未报道。该文以NCBI 中登录的广南天料木、斯里兰卡天料木、狭叶天料木、天料木的叶绿体基因组为研究对象,揭示天料木属植物叶绿体基因组密码子偏性之间的差异、进化关系等,同时为更深入研究和应用提供支持。
1 数据来源与研究方法
1.1 数据来源
在NCBI 数据库获取广南天料木、斯里兰卡天料木、狭叶天料木、天料木的叶绿体基因组序列,登录号分别为:NC_045233、NC_045235、NC_045234、NC_045919。为避免抽样误差,筛选CDS(Coding sequence) 中序列长度≥300bp,以ATG 起始,TAA、TAG、TGA 终止,且序列中间不存在终止密码子的序列,排除重复序列[19]。分别获得了51、51、52、51 条CDS。
1.2 研究方法
将获得的CDS 的运用codonW1.4.2 分析软件计算ENC、CAI 及RSCU 等数值,运用Bioinformatics.org 的SMS(Sequence Manipulation Suite)在线工具箱计算密码子第1 位(GC1)、第2 位(GC2)、第3 位(GC3)点的GC 含量。分析时排除三个终止密码子及无偏性的蛋氨酸ATG、色氨酸TGG。运用R 语言和SPSS22 等软件分析制图。
1.2.1 中性绘图分析
根据曲线回归的斜率分析影响密码子偏性的主要因素,当斜率为零时,表示偏性不受定向突变压力的影响,而完全受选择性限制。斜率为1 时,表示密码子使用偏性完全受定向突变压力的影响[5]。取第1位碱基与第2 位碱基平均值GC12 和GC3 为纵横坐标作散点图。
1.2.2 PR2 绘图分析
PR2 分析(Parity Rule 2)用来研究密码子第3 位碱基A 与T 和C 与G 之间是否有突变不平衡现象[20]。分析时仅选取具有4 个同义密码子的氨基酸,其中丝氨酸只取TCA、TCC、TCG、TCT,亮氨酸只取CTA、CTC、CTG、CTT、精氨酸只取CGA、CGC、CGG、CGT,以G3/(G3+C3)|4、A3/(A3+T3)|4 作横纵坐标,做散点图[21]。
1.2.3 ENc 绘图分析
ENc 值(effective number of codons)解释密码子使用偏离随机选择的程度,ENC 值的范围在20~61 之间,越靠近20 偏性越强[22]。以ENc 值和GC3值为纵、横坐标做散点图,与ENc 期望值曲线{ENc期望值=2+GC3+29/(GC32+(1-GC3)2)}进行比较[21]。当突变压力对密码子使用模式起重要作用时,ENc 值位于期望曲线上或围绕期望曲线。当自然选择和其他因素的影响密码子偏性时,ENc 值远低于预期曲线[23]。
1.2.4 对应性分析
对应性分析用来描述对密码子偏性的潜在影响[24]。分析时排除无密码子偏性的ATG、TGG 和终止密码子。基于各基因密码子的RSCU 值(59 个)进行分析。将第1 轴(axis1)和第2 轴(axis2)作为横、纵坐标作散点图,并将第1 轴与密码子第3 位碱基(GC3)、密码子适应指数(CAI)、氨基酸总量(L_aa)等数据进行相关分析。
1.2.5 RSCU 分析
RSCU(相对同义密码子使用度)是该密码子的实际观察值与理论观察值之间的比值,如果RSCU=1,表示密码子使用无偏差,RSCU 值>1 时,密码子使用存在正偏差,使用频率高于其他密码子。相反,RSCU值<1 显示负偏差,使用频率低于其他密码子[25]。参照薛庆中[26]与尚明照[21]等的研究方法,以ENC 为偏性标准,两极各选10%的基因,分别建成高低偏性库,取两库△RSCU>0.08 的密码子为高表达密码子,同时满足叶绿体基因组RSCU 值>1 的为最优密码子。
1.2.6 基于RSCU 聚类
天料木属植物与海南大风子(Hydnocarpus hainanensis) ( 登 录 号:NC_042720)、 山 桂 花(Bennettiodendron leprosipes)(登录号:NC_045898)、短柄山桂花(Bennettiodendron brevipes)(登录号:NC_043885)、大叶刺篱木(Flacourtia rukam)(登录号:NC_045859)、山桐子(Idesia polycarpa)(登录号:NC_032060)、山羊角树(Carrierea calycina)(登录号:NC_043884)进行聚类分析。引入同为山茶亚目的坡垒(Hopea hainanensis)(登录号:NC_044642)和青梅(Vatica mangachapoi)(登录号:NC_041485) 作为外类群,分析时选取相同的基因,以同义密码子RSCU值为变量,采用组间联接法(欧式平方距离)进行密码子使用偏好性聚类分析。
2 结果与分析
2.1 密码子参数
天料木属4 种植物叶绿体基因组密码子参数的计算结果分析(表1),可见4 种植物叶绿体基因组的GC1、GC2、GC3 及GCa 值均小于0.5,说明均倾向于使用A/T 碱基。斯里兰卡天料木、广南天料木、狭叶天料木的各位点GC 含量及平均GC 含量更为接近,且ENc 值与CAI 值亦然。天料木的各参数较之其他3 种植物略有不同。统计得出,斯里兰卡天料木的ENc 值分别在36.76~56.32 之间、广南天料木和狭叶天料木的ENc 值均在36.04~56.32 之间,而天料木的ENc 值在37.08~55.09 之间。从ENc 值来看,天料木较其他3 种植物的叶绿体基因密码子偏性略强。对竹亚科、槲蕨属、柿属、大戟科以及樟树(Cinnamomum camphora)[27]、 马 尾 松 (Pinus massoniana)[28]、北美红杉(Sequoia sempervirens)[29]等多数文献的分析结果,均表明了叶绿体基因组密码子偏好使用A/T 碱基。可见,不论是单子叶植物、双子叶植物还是裸子植物,普遍偏好使用A/T 碱基。
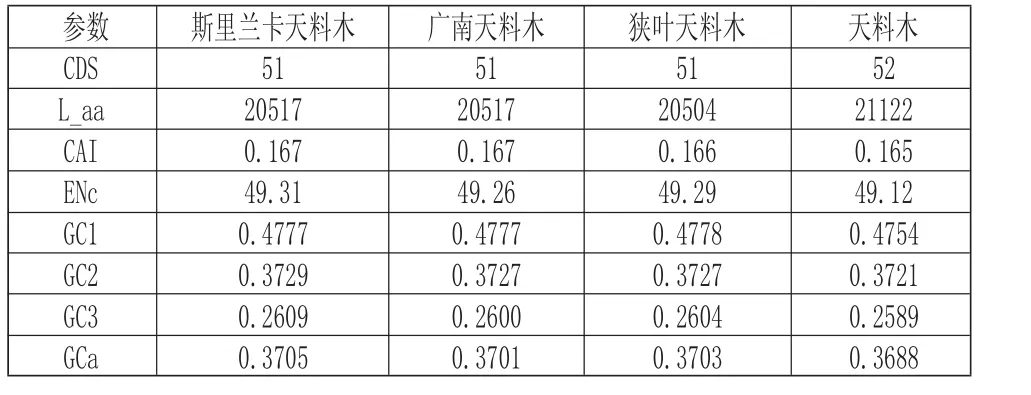
表1 四种天料木属植物叶绿体基因组特征Tab.1 Genomic Features of Chloroplast Genomes of Four Homalium Plant Species
2.2 中性绘图结果分析
中性绘图体现出GC12(0.3061~0.5564)和GC3(0.1619~0.3434) 的分布范围。4 种植物GC12 与GC3 双尾检验均呈显著相关(p<0.05),Pearson 相关系数分别为0.3270、0.3300、0.3460、0.3380。GC1与GC2 均表现极显著正相关性(p<0.01),系数分别为0.4710、0.4280、0.4540、0.4660。GC2 与GC3 均呈显著正相关 (p <0.05),系数分别为0.3010、0.2880、0.3230、0.3120。GC1 与GC3 均无显著相关性(p>0.05)。曲线斜率显示,4 种植物叶绿体基因组密码子影响因素中,自然选择等因素占53.36%~56.11%,突变压力占43.89%~46.64%。大戟科、竹亚科、槲蕨属、柿属、陆地棉(Gossypium hirsutum)、樟树、马尾松、灰毛浆果楝(Cipadessa cinerascens)等植物的研究结果:GC12 与GC3 均不显著相关,说明天料木属4 种植物叶绿体基因组密码子第3 位碱基组成与第1 位碱基、第2 位碱基较之其他植物更为近似,在密码子偏性影响因素中,突变压力的作用相对于其他物种来说影响更大。
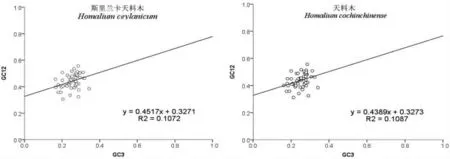

图1 四种天料木属植物中性绘图Fig.1 Neutrality Plot of Chloroplast Genomes Four Homalium Plant Species
2.3 ENc 绘图结果分析
实际ENc 值与ENc 期望值曲线相比较,绘制ENc 分析图(图2),4 种天料木属植物叶绿体基因组的ENc 分布极为相似。运用(ENc 期望-ENc 实际)/ENC 期望的公式,统计基因ENc 的分布组数(表2)。4 种植物均有超过60%的基因ENc 实际值位于在曲线上或围绕在曲线附近,表明突变压力在多数基因密码子使用模式的形成中起重要作用。该结果与槲蕨属、陆地棉等植物研究结果基本一致,与大戟科、樟树、马尾松、灰毛浆果楝等植物有较大差异。说明不同物种间叶绿体基因组密码子偏性存在差异。
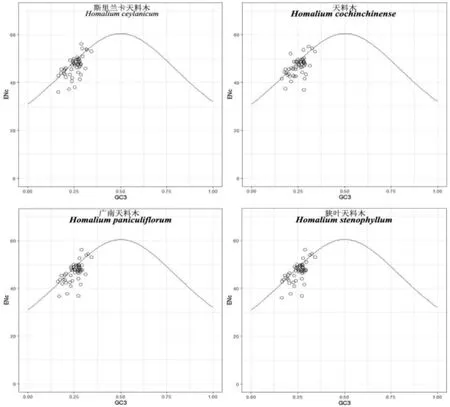
图2 四种天料木属植物ENc 绘图Fig.2 Enc Plot of Chloroplast Genomes Four Homalium Plant Species

表2 ENC 比值频率分布Tab.2 Distribution of Enc Frequency Ratio
2.4 PR2 绘图结果分析
PR2 绘图分析可解释叶绿体基因组同义密码子第3 位碱基的使用情况(图3)。天料木属4 种植物叶绿体基因组的多数基因分布在T/C 区域,嘧啶的使用高于嘌呤,密码子偏性除了突变压力因素外,更主要还是受自然选择等因素的影响。猫眼草(Euphorbia esula)[5]、陆地棉、美丽梧桐(Firmiana pulcherrima)和云南梧桐(Firmiana major)[31]等植物的结果一致,而同为大戟科的橡胶树(Hevea brasiliensis)、麻风树(Jatropha curcas)、木薯(Manihot esculenta)[5]等以及灰毛浆果楝[30]等植物则为密码子第3 位存在T/G 偏倚,再次说明叶绿体基因组密码子偏性在不同物种间甚至亲缘关系较近的物种间仍然可能存在较大差异。

图3 四种天料木属植物PR2 图Fig.3 PR2-plot of Chloroplast Genomes Four Homalium Plant Species
2.5 对应性结果分析
基于RSCU 对4 种天料木属植物进行对应性分析,前4 轴分别表现出35.57%、36.52%、35.76%、35.48%的差异。第1 轴的差异分别为:10.20%、10.20%、9.98%、10.07%,其余各因子轴的差异依次递减。第1 轴与CAI、L_aa、GC3、ENc 相关分析结果表明(表3),第1 轴与ENc、GC3、L_aa 均不显著相关(p>0.05),斯里兰卡天料木第1 轴与CAI 值呈显著负相关(p<0.05)。而天料木、广南天料木、狭叶天料木中第1 轴与CAI 值均呈极显著正相关(p<0.01)。推测基因表达水平相关因素,对天料木属4 种植物叶绿体基因组密码子偏性影响较大。对应性分析作图表明(图4),多数基因围绕原点分布,说明多数基因偏性较低。光合系统基因、遗传结构基因分布相对集中,该部分基因的密码子使用模式相对一致。尽管天料木属4 种植物有着大体相似的密码子使用模式,但种间基因仍有各自特殊的密码子使用特征。
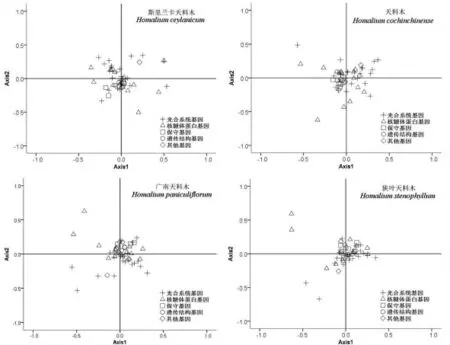
图4 四种天料木属植物对应性分析Fig.4 Corresponding Analysis of Chloroplast Genomes Four Homalium Plant Species
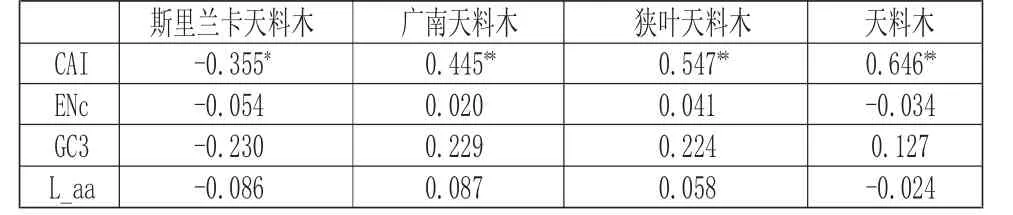
表3 四种天料木属第1 轴与密码子参数相关分析Tab.3 Correlation Analysis of Axis1 and Codon Usage Index of Chloroplast Genomes Four Homalium Plant Species
2.6 RSCU 结果分析
排除无密码子偏性的ATG、TGG 和终止密码子后,对叶绿体基因组RSCU 和高低表达库RSCU 差值ΔRSCU 分析得到(表4),4 种天料木属植物RSCU>1具有29 个相同的密码子,其中以A/T 结尾有28 个密码子,RSCU<1 的有30 个相同的密码子,其中以G/C 结尾有28 个。说明以A/T 结尾的密码子存在较强正偏差,使用频率较高。对ΔRSCU 值分析可知,4种植物均有13 个相同的最优密码子:UUU、UUA、UUG、AUU、GUA、CCU、ACU、GCU、CAA、AAA、GAA、UGU、GGU,但天料木多具有4 个最优密码子:GUU、UCU、CCA、AGU。其中12 个共同的最优密码子以A/T 结尾。
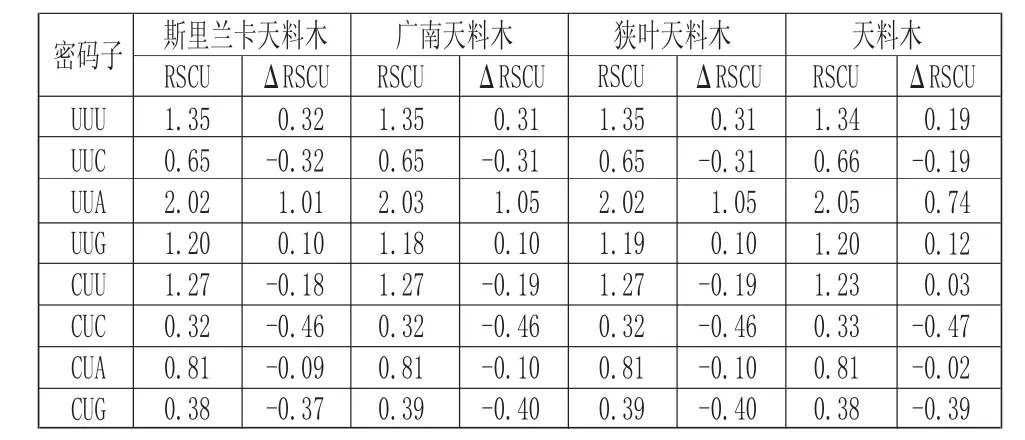
表4 四种天料木属植物叶绿体基因组氨基酸相对同义密码子使用度Tab.4 The RSCU of Chloroplast Genomes Four Homalium Plant Species

密码子 狭叶天料木斯里兰卡天料木广南天料木天料木RSCU ΔRSCU RSCU ΔRSCU RSCU ΔRSCU RSCU ΔRSCU AUU AUC AUA GUU GUC GUA GUG UCU UCC UCA UCG CCU CCC CCA CCG ACU ACC ACA ACG GCU GCC GCA GCG UAU UAC CAU CAC CAA CAG AAU AAC AAA AAG GAU GAC GAA GAG UGU UGC CGU CGC CGA CGG AGU AGC AGA AGG GGU GGC GGA GGG 1.52 0.54 0.94 1.46 0.50 1.48 0.56 1.68 0.95 1.22 0.51 1.60 0.76 1.14 0.51 1.60 0.75 1.27 0.38 1.81 0.62 1.12 0.46 1.65 0.35 1.50 0.50 1.56 0.44 1.55 0.45 1.53 0.47 1.57 0.43 1.52 0.48 1.42 0.58 1.35 0.44 1.35 0.36 1.27 0.37 1.85 0.64 1.28 0.47 1.56 0.69 0.53-0.20-0.32-0.27 0.03 0.91-0.67 0.00-0.19-0.30-0.39 0.63-0.38-0.08-0.17 0.87-0.15-0.46-0.26 0.56-0.24-0.40 0.09-0.22 0.22-0.28 0.28 0.36-0.36-0.27 0.27 0.34-0.34 0.00 0.00 0.28-0.28 0.71-0.71 1.89-0.29-0.18-0.29 0.77 0.09-0.57-0.57 0.84 0.07-0.27-0.64 1.52 0.54 0.94 1.46 0.50 1.49 0.55 1.67 0.96 1.22 0.51 1.60 0.77 1.13 0.51 1.60 0.74 1.28 0.38 1.80 0.62 1.11 0.47 1.65 0.35 1.50 0.50 1.57 0.43 1.56 0.44 1.54 0.46 1.57 0.43 1.52 0.48 1.42 0.58 1.36 0.45 1.33 0.36 1.27 0.37 1.87 0.63 1.28 0.48 1.55 0.68 0.52-0.22-0.3-0.21-0.03 0.91-0.67-0.03-0.10-0.30-0.40 0.50-0.35-0.02-0.14 0.79-0.09-0.44-0.27 0.59-0.30-0.39 0.09-0.18 0.18-0.29 0.29 0.34-0.34-0.27 0.27 0.34-0.34-0.02 0.02 0.28-0.28 0.67-0.67 1.89-0.32-0.17-0.29 0.76 0.08-0.56-0.56 0.79 0.06-0.25-0.60 1.52 0.54 0.94 1.46 0.50 1.49 0.56 1.68 0.95 1.22 0.51 1.59 0.77 1.13 0.51 1.60 0.74 1.28 0.38 1.81 0.62 1.11 0.47 1.65 0.35 1.50 0.50 1.57 0.43 1.56 0.44 1.54 0.46 1.57 0.43 1.52 0.48 1.42 0.58 1.35 0.45 1.35 0.36 1.27 0.37 1.86 0.63 1.28 0.48 1.55 0.69 0.57-0.25-0.32-0.27-0.03 0.97-0.67-0.03-0.10-0.30-0.40 0.54-0.33-0.07-0.13 0.79-0.09-0.44-0.27 0.59-0.30-0.39 0.09-0.18 0.18-0.29 0.29 0.34-0.34-0.27 0.27 0.34-0.34-0.02 0.02 0.28-0.28 0.67-0.67 1.89-0.32-0.17-0.29 0.76 0.08-0.56-0.56 0.79 0.06-0.25-0.60 1.50 0.54 0.96 1.47 0.49 1.47 0.57 1.71 0.95 1.22 0.50 1.59 0.74 1.16 0.52 1.60 0.75 1.30 0.35 1.80 0.63 1.12 0.44 1.66 0.34 1.51 0.49 1.57 0.43 1.56 0.44 1.52 0.48 1.57 0.43 1.54 0.46 1.41 0.59 1.31 0.44 1.38 0.37 1.27 0.35 1.86 0.64 1.27 0.46 1.60 0.67 0.47-0.17-0.29 0.12-0.27 0.78-0.65 0.36-0.18-0.11-0.52 0.42-0.46 0.12-0.08 0.72-0.02-0.24-0.46 0.34-0.21-0.08-0.05-0.24 0.24-0.14 0.14 0.39-0.39-0.17 0.17 0.37-0.37-0.01 0.01 0.25-0.25 0.71-0.71 1.17-0.04-0.10-0.36 0.22 0.23-0.15-0.52 0.86-0.08-0.18-0.61
2.7 基于RSCU 聚类分析
比较叶绿体基因组序列可以揭示近缘物种类群之间的系统进化关系,同时阐明植物的进化模式[32]。基于天料木属4 种植物、大风子科其他6 种植物以及2 种外类群植物RSCU 值的聚类结果显示(图5),斯里兰卡天料木、广南天料木和狭叶天料木距离最近,天料木与其他天料木属植物距离较远。天料木属植物、山桐子等4 种植物和大叶刺篱木聚为亚类,海南大风子与天料木等9 种植物聚为大类,坡垒、青梅组成另一大类。该聚类分析结果同植物亲缘关系基本一致。小麦等11 种植物psbA 基因[33]的分析得出,基于RSCU 的聚类并不能反映出植物之间的亲缘关系。但樟树、马尾松、小麦等7 种植物[34]以及水稻等9 种植物waxy 基因聚类分析[35]等结果与该研究一致。这可能是因为研究对象及统计方法的差异造成,但基于RSCU 的聚类仍能反映多数参考植物的亲缘关系。可以推断,斯里兰卡天料木、广南天料木和狭叶天料木叶绿体基因组具有极为相似的密码子偏性模式,而天料木略有不同。其他植物随聚类距离增大,而密码子偏性差异也随之增大。
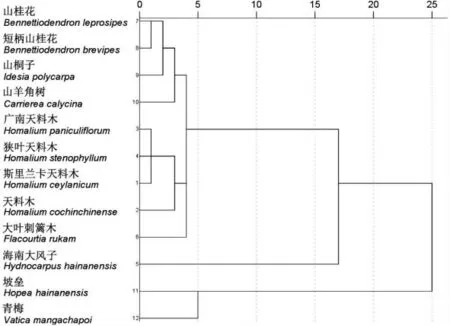
图5 四种天料木属植物基于RSCU 的聚类分析Fig.5 Cluster Tree Based on the Rscu of Chloroplast Genomes Four Homalium Plant Species
3 结论
天料木属4 种植物叶绿体基因组各密码子参数基本一致,且都偏好使用A/T 碱基,但天料木叶绿体基因组密码子偏性略强于其他3 种植物。结合上述分析结果,突变压力对天料木属4 种植物叶绿体基因组密码子偏性的影响较之其他参考物种影响更大,但自然选择等因素对其仍发挥着主要作用。天料木属4 种植物叶绿体基因组密码子第3 位碱基偏好使用嘧啶(T/C)。对应性分析得出,基因表达水平在天料木属4 种植物叶绿体基因组密码子偏性中影响较大,再一次证明自然选择在密码子偏性形成中起主要作用。光合系统基因和遗传结构基因的密码子使用模式相对一致。通过RSCU 分析,天料木属4 种植物叶绿体基因组以A/T 结尾的密码子使用频率较高。同时,确定了天料木属4 种植物13 个相同的 最 优 密 码 子:UUU、UUA、UUG、AUU、GUA、CCU、ACU、GCU、CAA、AAA、GAA、UGU、GGU,天料木多具有的4 个最优密码子GUU、UCU、CCA、AGU。最优密码子多以A/T结尾。基于RSCU 值的聚类结果表明,斯里兰卡天料木、广南天料木和狭叶天料木叶绿体基因组密码子偏性特征极为相似,而天料木与它们略有不同。该研究揭示了天料木属4 种植物叶绿体基因组密码子偏性特征、影响因素之间的比重以及与大风子科其他植物的系统进化关系,增加了植物叶绿体基因组密码子使用模式的了解,为其他大风子科植物系统发育分析提供了补充。
