巴西橡胶树胶乳中DUF1262结构域蛋白结构与基因表达分析
2022-10-12代龙军刘明洋阳江华郭冰冰王立丰
代龙军 刘明洋,2 阳江华 周 凯,2 郭冰冰 杨 洪 王立丰,2*
(1. 农业农村部橡胶树生物学与遗传资源利用重点实验室,省部共建国家重点实验室培育基地-海南省热带作物栽培生理学重点实验室,农业农村部儋州热带作物科学观测实验站,海南省高性能天然橡胶材料工程重点实验室,中国热带农业科学院橡胶研究所,海口 571101;2. 海南大学,海口 570228)
巴西橡胶树()是主要的产胶植物,天然橡胶合成核心步骤是异戊二烯焦磷酸单体聚合为天然橡胶。天然橡胶在巴西橡胶树乳管细胞中经类异戊二烯合成途径合成,是典型的植物次生代谢产物,受乙烯、茉莉酸等多种激素信号以及转录因子调控。研究表明天然橡胶转移酶复合体包括橡胶延伸因子(rubber elongation factor,REF)、小橡胶粒子蛋白(small rubber particle protein,SRPP)、顺式异戊二烯基转移酶(cis-prenyltransferase,CPT)、CPT 结合蛋白,也被称为CPT样蛋白、橡胶转移酶激活蛋白或HRT-REF 桥接蛋白,共4 个家族成员。异戊二烯焦磷酸单体聚合为天然橡胶的过程由橡胶转移酶催化,而橡胶转移酶是CPT 家族中一员,部分学者已将该家族的HRT1 和HRT2 作为橡胶转移酶的候选基因进行了研究。目前,橡胶转移酶的属性、异戊二烯焦磷酸单体聚合为天然橡胶的机制、天然橡胶分子质量大小的决定机制和橡胶粒子蛋白的作用是天然橡胶产业尚未解决的几个重要科学问题。
天然橡胶转移酶复合体的具体组成目前尚不完全清楚。Yamashita 等借助去垢剂洗涤过的橡胶粒子再添加REF、CPT(HRT1)和CPT 结合蛋白构建了离体天然橡胶合成体系,分析了添加这些蛋白质对合成体系的影响。研究者发现乳清中含有激活或抑制天然橡胶合成的蛋白质。例如,从乳清中纯化的真核蛋白翻译起始因子eIF-5A 能促进天然橡胶离体合成,橡胶树中多个eIF-5A异构体与GST 标签形成的融合蛋白能促进天然橡胶离体合成。eIF-5A 是真核细胞蛋白翻译的起始因子,而天然橡胶离体合成体系中不包括执行蛋白质翻译功能的核糖体,eIF-5A 非经核糖体起作用。乳清中也发现了一个抑制天然橡胶合成的蛋白,该蛋白与马铃薯块茎储藏蛋白patatin 同源具有脂酰基水解酶活性,对橡胶粒子有破坏作用,从而间接抑制天然橡胶合成。有研究者发现一个分子质量较大的241 kDa 蛋白、2 个分子量较小(3.65、1.60 kDa)蛋白质成分可能参与天然橡胶合成,但未对这些蛋白进行鉴定。因此,探索构成橡胶转移酶复合体的除REF、SRPP、CPT、CPT 结合蛋白以外的蛋白质组分具有重要的科学意义。
可见,未能确定橡胶转移酶复合体的全部蛋白组分是目前存在的主要问题。采用蛋白互作分析技术等筛选和鉴定全部参与天然橡胶生物合成的蛋白,解析这些蛋白的结构与功能,并在此基础上构建有功能的离体天然橡胶合成体系,为天然橡胶合成提供理论依据,并对橡胶树育种和品种改良提供支撑。
1 材料与方法
1.1 试验材料
以中国热带农业科学院橡胶研究所儋州基地种植的正常割胶的橡胶树品种热研73397,10年树龄正常割胶树的根、叶、树枝、花、树皮和胶乳为材料,进行基因克隆和表达分析;取胶乳用于制备橡胶粒子样本。试验所用的多克隆抗体委托南京金斯瑞生物科技有限公司制备。
参考樊松乐等和谢黎黎等的方法进行激素等叶面喷施处理、使用橡胶树品系热研73397株高为70~80 cm的组培苗;使用体积分数0.05%的乙醇溶液配制1 mmol·L水杨酸(SA)、200 μmol·L脱落酸(ABA)和体积分数2%过氧化氢(HO),对照组用体积分数0.05%的乙醇溶液,取样时间分别为0、2、6、10、24、48、72 h。干旱处理参照本课题组已发表的方法进行,取叶片提取总RNA。
1.2 橡胶粒子制备与橡胶粒子蛋白质提取
橡胶粒子制备参照本课题组已发表的方法进行,26 400×和4 ℃下离心1 h获得橡胶粒子,其中洗涤步骤在26 400×和4 ℃下离心10 min 收集橡胶粒子。橡胶粒子蛋白质提取过程是将洗涤过的橡胶粒子分散在5 倍体积的NP40 的裂解液(购自索来宝公司,主要成分为Tris(pH7.4),NaCl,1%NP-40,EDTA以及磷酸酶抑制剂)中,置于冰浴,使用摇床100 r·min震荡提取30 min。将提取混合物在26 400×和4 ℃下离心1 h,然后取出水相部分,在35 000 r·min(105 000×)和4 ℃下离心40 min,使液体澄清,小心移除液面的少量漂浮物,再取出水相部分即为蛋白质提取物。使用Bradford法测定蛋白质含量,将蛋白质提取物分装后保存于-80 ℃备用。
1.3 免疫共沉淀实验及捕获蛋白鉴定
免疫共沉淀实验使用的抗体为兔源多克隆抗体,系使用一个CPT序列(NCBI登录号AAM92881.1)的氨基酸序列片段(CSPPRELELKEAPGL)和CPT 结合蛋白(NCBI 登录号BAV89210.1)的2 个氨基酸序列片段(CDSKGVLKTNKKTIM 及MDLKPGAGGQRVNRC)作为免疫原分别制备,抗体经Protein A/G亲和纯化。
使用Pierce Co-Immunoprecipitation(Co-IP)Kit(货号26149),依照试剂盒说明书进行免疫共沉淀实验。实验规模为每个反应10 μg抗体和1 000 μg总橡胶粒子蛋白(体积为600 μL,使用试剂盒中的偶联缓冲液稀释)。正式进行免疫共沉淀实验前,总橡胶粒子蛋白样品先用10 μg 兔IgG(即无关抗体)偶联的琼脂糖树脂孵育,以减少非特异反应。
免疫共沉淀实验结束,洗脱的蛋白采用氯仿-甲醇法沉淀,然后对捕获的蛋白进行溶液内胰蛋白酶水解。捕获的蛋白经胰蛋白酶水解获得的肽段在U3000 RSLC nano-Qe plus 液质联用仪(Thermo Fisher)上分析,使用NCBI 的巴西橡胶树(taxid:3981)蛋白质数据库进行质谱数据——蛋白质数据匹配以实现蛋白质鉴定。获得鉴定但无明确功能描述的蛋白质,以其蛋白质序列为查询序列,blastP 检索SWISS-PROT 的经过人工审核的(reviewed)蛋白质数据库,若仍无法从匹配的蛋白质获得明确功能信息,则进一步检索blastP NCB未经过人工审核Ⅰ蛋白质数据库,以从匹配的蛋白质获取功能相关信息。经检索两个蛋白质数据库均无法获得明确功能信息的蛋白质则进一步分析。
1.4 基因克隆、荧光定量PCR和统计分析
试验使用的巴西橡胶树不同组织的mRNA 采用天根生物公司的植物总RNA提取试剂盒按说明书提供的方法提取。使用NanoDrop 2000 超微量核酸蛋白分析仪(Thermo Fisher)检测RNA 的浓度与纯度。通过LC-MS 鉴定的未知蛋白登录号为XP_021644636.1,对应的基因登录号为LOC110638365,据此设计克隆引物和荧光定量PCR 引物(见表1)。使用TaKaRa 反转录试剂盒从各组织mRNA 样本中获得cDNA,以cDNA 为模板进行PCR 扩增和基因克隆,对克隆的基因测序。用为内参基因(GenBank:HQ260674.1)进行PCR 荧光定量分析,使用2法计算相对表达量,使用SPSS 26.0 软件进行单因素ANOVA 检验分析处理与对照之间的差异显著性。
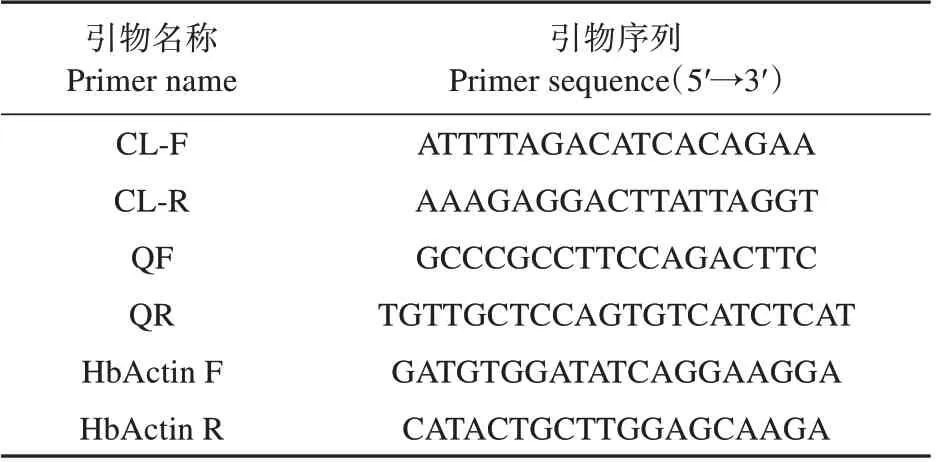
表1 LOC110638365基因ORF扩增和荧光定量引物序列Table 1 Primer sequences for cloning open reading frame and qRT-PCR analysis of LOC110638365
1.5 氨基酸序列同源性分析及互作蛋白分析
使用TMHMM-2.0 工具(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)分 析XP_021644636.1 的氨基酸序列形成跨膜结构域的可能性。对以下3 个来源的蛋白或核酸数据库进行检索:使用XP_021644636.1 的氨基酸序列对NCBI taxid 3981 蛋白质库进行blastP 检索,下载所有匹配的蛋白质序列;使用XP_021644636.1 的氨基酸序 列 对NCBI taxid 3981 TSA 库(Transcriptome Shotgun Assembly Sequence Database)进行tblastN检索,下载所有的核酸序列;使用Emboss getorf 工具(http://emboss. bioinformatics. nl/cgi-bin/emboss/getorf)将核酸序列翻译为氨基酸序列(保留长度大于250个氨基酸残基的序列,ORF区需具有完整的起始密码子和终止密码子);使用XP_021644636.1的氨基酸序列对中国热带农业科学院橡胶研究所基因组测序结果推导获得的蛋白质数据库进行blastP 检索后下载所有匹配的蛋白质序列;使用CDHIT 工 具(http://weizhong-lab. ucsd. edu/cdhitweb-server/cgi-bin/index.cgi?cmd=cd-hit)对这些序列进行聚类(cutoff=0.95,将一致性大于95%的序列归为一类)。使用MEGA 11 软件对这3 个来源的代表性蛋白质序列进行比对,并构建系统进化树,分析序列间的冗余情况及基因家族构成。
使用蛋白质相互作用分析网站(https://cn.string-db.org)提供的检索功能,搜索范围设定为大戟科(Euphorbiaceae)和杨属(),分别搜索XP_021644636.1 氨基酸序列的相似序列,并查看与相似序列互作的蛋白,从而了解目标蛋白质的功能。
2 结果与分析
2.1 免疫共沉淀技术鉴定DUF1262结构域蛋白
为了寻找参与或调节天然橡胶合成的未知蛋白质组分,使用抗CPT 抗体及抗CPT 结合蛋白抗体(CPT 和CPT 结合蛋白为天然橡胶合成的核心蛋白组分)对从橡胶粒子提取的蛋白进行了免疫共沉淀实验,通过液相色谱-质谱联用技术(LCMS)分析捕获的蛋白质。捕获的蛋白质使用NCBI橡胶树蛋白质数据库鉴定结果如下:使用CPT 抗体的实验捕获了111个蛋白,使用CPT结合蛋白抗体的实验捕获了54 个蛋白质。2 个试验中均捕获的与天然橡胶合成相关的蛋白质包括:3个CPT 家族成员(登录号:BAF98305.1、XP_021662710.1、XP_021657207.1);1 个CPT 结 合 蛋 白(XP_021659160.1);6 个 REF/SRPP 家 族 成 员CAA39880.1、 XP_021662186.1、 AMO43675.1、AMO43676.1、XP_021653593.1 和AHF95713.1,其中CAA39880.1 为橡胶粒子中丰度最高的REF 蛋白,即REF1;AHF95713.1 为橡胶粒子中丰度最高的SRPP 蛋白,即SRPP1。逐一分析了有功能描述的蛋白质与天然橡胶合成的相关性,排除与天然橡胶合成无关的有功能描述的蛋白。最后分析了无功能描述的蛋白质,以期从中发现与天然橡胶合成或天然橡胶合成调控相关的蛋白。使用NCBI 蛋白质库检索的情况下,使用CPT 抗体捕获的蛋白中有13 个为功能未知蛋白质;使用CPT 结合蛋白抗体捕获的蛋白中有6个为功能未知蛋白质;二者叠加共有15个功能未知蛋白质。这些未能通过质谱谱图—蛋白质数据库匹配直接获得功能描述的蛋白质,需进一步将其蛋白质序列先后blastP检索SWISS-PROT 经人工审核的蛋白数据库和NCBI nr 未经人工审核的蛋白质数据库,依据序列同源性获得功能描述。XP_021644636.1 是CPT 抗体捕获的蛋白质,图1 显示了该蛋白质一条肽段ELELKEAPGLDK 的二级质谱。

图1 LC-MS分析获得的XP_021644636.1一个多肽片段ELELKEAPGLDK 的二级质谱b1~b11和y1~y11分别代表肽键断裂后的N端碎片离子(B系列)和C端碎片离子(Y系列)Fig.1 MS spectrum of a polypeptide fragment ELELKEAPGLDK in XP_021644636.1obtained by LC-MS analysisb1-b11 and y1-y11 represented the N-terminal fragment ion(sB series)and C-terminal fragment ion(sY series)generated by peptide bond cleavage,respectively
首先用15 个功能未知蛋白序列对SWISSPROT蛋白质库进行blastP检索,其中有9个获得功能描述,6 个(XP_021644636.1、XP_021692246.1、XP_021689565.1、XP_021676080.1、XP_021647342.1、XP_021667550.1)未获得功能描述。然后以6个未获功能描述的蛋白质作为查询序列对NCBI nr 全库进行blastP 检索,其中5 个获得明确的功能描述,仅XP_021644636.1 没有明确功能描述。以XP_021644636.1 对NCBI nr 蛋白库进行blastP 检索,结果显示其AA 23~124 处为DUF1262 结构域(DUF:Domain of unknown function),其较疏水部分在DUF1262 结构域内(见图2A)。图2B 显示了XP_021644636.1 与NCBI pfam(蛋白质家族)数据库中其它物种同源蛋白的保守氨基酸残基。TMHMM 跨膜结构域分析显示,XP_021644636.1形成跨膜结构域的可能性低于13%(形成跨膜结构域可能性最高区段为AA 79-98)。XP_021644636.1的N 端存在小段弱疏水区的情况与橡胶延伸因子REF1 类似(见图2C)。但ProtParam 分析表明该蛋白为亲水蛋白质,总平均亲水性(Grand average of hydropathicity,GRAVY)值 为-0.487,而REF1 的GRAVY值为0.044,为疏水蛋白。

图2 XP_021644636.1的DUF1262结构域及形成跨膜结构的可能性A.NCBI blastP 结果中显示的DUF1262 结构域;B.DUF1262 结构域的保守氨基酸序列,XP_021644636.1 与其他5 个蛋白(NCBI pfam06880的前5 个代表性序列)的DUF1262 结构域序列比对,其中XP_009391265 来自wild Malaysian banana,XP_002892755 和XP_002892757 来自Arabidopsis lyrata subsp.Lyrata,XP_013677813 来自rape,OAP18071 来自thale cress。使用ClustalW2 软件进行序列比对,然后使用Genedoc软件绘制;C.TMHMM-2.0分析结果,箭头所示为2个蛋白(XP_021644636.1和橡胶延伸因子REF1)各自具有疏水倾向的区段Fig.2 DUF1262 domain of XP_021644636.1 and its propensity to form a transmembrane structureA.The DUF1262 domain shown in the blastP results;B.Conserved sequence of DUF1262 domain.XP_021644636.1 was aligned with the DUF1262 domain sequences of 5 other proteins(top five representative sequences of NCBI pfam06880),wherein XP_009391265 is from wild Malaysian banana,XP_002892755 and XP_002892757 are from Arabidopsis lyrata subsp.Lyrata,XP_013677813 was from rape,and OAP18071 was from thale cress(Arabidopsis thaliana).Sequence alignments were performed using ClustalW2 software and then plotted using Genedoc software;C.TMHMM-2.0 analysis results.Arrows indicated the hydrophobic regions of two proteins(XP_021644636.1 and rubber extension factor REF1)
2.2 DUF1262 结构域蛋白氨基酸序列同源性分析
以XP_021644636.1为查询序列对橡胶树相关蛋白质和核酸数据库进行检索,发现存在50 个类似序列,典型的氨基酸残基数量为392、410、381,这些蛋白的N端均含有DUF1262结构域(见图3)。以XP_021644636.1的氨基酸序列作为查询序列对NCBI taxid 3981 蛋白质数据库进行检索,匹配了15 个蛋白质序列,对NCBI taxid 3981 的TSA 库进行tblastN 检索,匹配了27 个核酸序列。以XP_021644636.1 的氨基酸序列作为查询序列对中国热带农业科学院橡胶研究所基因组测序推导得到的蛋白质数据库进行检索,匹配了12 个蛋白质序列,去除含有不明确氨基酸残基的4 个序列,仅保留8 个确定的序列。将TSA 序列使用EMBOSS getorf 工具转换为蛋白质序列。3 个来源叠加后共有50个有效蛋白质序列,为简洁起见,使用CDHIT工具对这些序列进行聚类(cutoff=0.95,将序列一致性大于95%的序列归为一类),使用MEGA 11软件对CDHIT选定的代表性蛋白序列进行比对和进化树构建,发现橡胶树DUF1262 家族蛋白主要集中于4 类(见图3)。其中,XP_021644636.1 即DUF1262蛋白单独聚为一类。

图3 橡胶树DUF1262家族蛋白序列聚类后代表序列的进化树蛋白质序列收集自NCBI taxid 3981的蛋白质数据库和TSA 库以及橡胶所基因组推导的蛋白质数据库;这些序列先经CDHIT聚类,然后在MEGA 11软件上用代表性蛋白质序列构建进化树Fig.3 Phylogenetic analysis of the representative sequences after clustering of the rubber tree DUF1262 family protein sequencesProtein sequences were collected from the NCBI tax id 3981 protein database and TSA library and the Rubber Institute of China’s genome-derived protein database;These sequences were first clustered by CDHIT,and then a phylogenetic tree was constructed with representative protein sequences on MEGA 11 software
2.3 蛋白相互作用网络分析
以XP_021644636.1 作为查询序列,在https://cn.string-db.org 网站数据库的大戟科范围内检索,相似度最高的蛋白质为N 端含DUF1262 结构域的木薯蛋白cassava4.1_031144m(),序列相似性为84.7%(e-value=3.6e)。与cassava4.1_031144m 相互作用的蛋白质包括含有双特异性磷酸酶(DSP,Dual specificity phosphatase)催化结构域的Protein-tyrosine-phosphatase mkp1-like蛋白和含螺旋-环-螺旋结构域转录因子org2 类似蛋白异构体x1 蛋白(Transcription factor org2-like isoform x1)。在https://cn.string-db.org 网站数据库杨属范围内检索,相似度最高的蛋白质为N 端含DUF1262 结构域的POPTR_0008s10880.1(),序列相似性(identity)为69.9%(e-value=1.1e)。与POPTR_0008s10880.1 相互作用的蛋白质包括含有VQ 基序的家族蛋白VQ(motifcontaining family protein),MAP 激酶磷酸酶1 家族蛋白(MAP kinase phosphatase 1 family protein),Kelch 重复f-box 家族蛋白(Kelch repeat-containing f-box family protein),有丝分裂原活化蛋白激酶磷酸酶1 样蛋白酪氨酸磷酸酶(Protein-tyrosine-phosphatase mkp1-like),胚胎发育晚期丰富蛋白(Late embryogenesis abundant protein)。以其他7 个代表性基因的氨基酸序列在https://cn.string-db.org 网站检索,结果与以XP_021644636.1 为查询序列类似,其相互作用蛋白均涉及信号转导过程或转录调控过程等。
2.4 基因克隆及荧光定量PCR分析
测序结果表明,使用克隆引物扩增的核酸序列与NCBI 数据库中登录号为LOC110638365 的基因吻合,该基因编码区1 179 bp,编码392 个氨基酸。编码的蛋白质分子质量为45.36 kDa,等电点为8.67。不同组织的荧光定量PCR 分析结果(见图4)表明,该基因的表达有明显的组织偏向性,在花中表达高于其他组织,树皮中次之,胶乳中最低。

图4 XP_021644636.1 蛋白基因(LOC110638365)在橡胶树不同组织中的表达水平Fig.4 Expression of XP_021644636.1 protein gene LOC110638365 in different tissues of rubber tree
组培苗叶片经水杨酸、脱落酸、过氧化氢外施处理6 h 后,叶片中XP_021644636.1 蛋白基因(LOC110638365)的转录本明显增加30~60 倍(见图5:A~C)。干旱处理诱导叶片中该基因上调表达最高到11 倍(见图5D)。这些结果显示该基因响应逆境胁迫上调表达。

图5 XP_021644636.1 蛋白基因(LOC110638365)在水杨酸(A)、过氧化氢(B)、脱落酸(C)和干旱(D)处理的橡胶树叶片中表达水平变化Fig.5 Changes of expression levels of XP_021644636.1 protein gene LOC110638365 in salicylic acid,abscisic acid,hydrogen peroxide and drought treated rubber leaves
3 讨论
天然橡胶在巴西橡胶树乳管细胞中经类异戊二烯合成途径合成,是典型的植物次生代谢产物,但橡胶树如何精细调控天然橡胶合成与植株抗逆的机制需持续研究。免疫共沉淀实验是一种有效筛选天然橡胶生物合成酶互作蛋白的有效方法。本项研究采用免疫共沉淀技术,从橡胶树胶乳蛋白中获得了与CPT 抗体互作的N 端含DUF1262 结构域蛋白。这一含DUF1262 结构域蛋白未被CPT结合蛋白抗体及无关抗体(兔IgG)捕获,表明XP_021644636.1蛋白与CPT存在相互作用。
蛋白相互作用网络分析表明DUF1262结构域蛋白(XP_021644636.1)可能与信号转导及转录调控过程有关,因为与之相似的木薯4.1_031144m蛋白与蛋白酪氨酸磷酸酶mkp1 类似蛋白及含螺旋-环-螺旋结构域转录因子org2 类似蛋白异构体x1蛋白等信号转导通路蛋白或转录因子有相互作用。其中有丝分裂原活化蛋白激酶磷酸酶1 样蛋白酪氨酸磷酸酶通过去磷酸化和抑制MPK6 和MPK3,并作为MPK6 和MPK3 信号传导的负调节剂调节防御反应等。考虑到XP_021644636.1蛋白基因在树皮有较高的表达量,推测该蛋白可能通过信号转导及转录过程影响树皮的生理活动。橡胶树树皮的重要生理活动之一是在韧皮部生产天然橡胶,天然橡胶由乳管细胞生产,但也需要乳管细胞周围的薄壁细胞等提供碳源物质与能量的支持,含DUF1262 结构域蛋白与胶乳再生的关系值得进一步研究。从TMHMM 跨膜结构域分析的结果看,XP_021644636.1 没有形成明显的跨膜区,但N 端(在DUF1262 结构域内)表现出一定的疏水倾向,这与REF/SRPP 家族成员存在小段疏水区类似。结合免疫共沉淀实验从橡胶粒子总蛋白中捕获XP_021644636.1 蛋白的事实,推测其可能是一个丰度极低的橡胶粒子膜结合蛋白并与橡胶粒子表面的蛋白质有较强的结合倾向,尽管从ProtParam的分析结果来看它比REF1更亲水。
DUF1262 属于未知功能结构域,目前仅有关于编码拟南芥()含DUF1262结构域蛋白基因的一些来自转录组学研究的报道。在不同的胁迫处理下,该基因的反应不尽相同。基因通常被甲基化抑制,外施水杨酸可诱导该基因发生去甲基化,其表达量增加。在本研究中,向叶片喷施水杨酸也使编码XP_021644636.1蛋白的基因上调表达。一个钙结合转录激活因子(CAMTA3)的突变引发了拟南芥叶片病变、数个防御相关基因及包含在内的数个功能未知基因上调表达。对拟南芥进行24 h 冷刺激激活了持续数天的应激记忆,导致许多原本受光胁迫/低温胁迫影响的基因表达解耦合,只有不受冷启动影响,冷启动处理后仍然在强光/低温分别作用下表现为耦合的下调表达。另外,查阅https://cn.string-db.org网站,发现除了与VQ motif 蛋白及Fbox 蛋白可能有相互作用,也可能与脱落酸和渗透胁迫诱导的受体类蛋白(Aba-and osmotic-stress-inducible receptor-like)及茉莉酸相关vq 基序基因1(Jasmonate-associated vq motif gene 1)蛋白等存在相互作用。这些关于拟南芥含DUF1262结构域蛋白基因的信息给橡胶树DUF1262 结构域蛋白的功能提供了参考,提示该蛋白在橡胶树中可能参与响应逆境刺激或激素信号传导引发的基因表达调节活动。
荧光定量PCR 实验结果显示该基因在橡胶树各组织中广泛表达,在胶乳中表达量较低,而花和树皮的表达量较高,而树皮是乳管分布丰富和产胶较多的组织。这表明XP_021644636.1蛋白非橡胶粒子或胶乳特有,在树皮的乳管细胞内表达较少,而在树皮中非乳管细胞中表达较多。荧光定量PCR结果也显示叶片中该基因响应多种逆境胁迫上调表达,表明该基因与橡胶树抗逆功能显著相关。
4 结论
通过免疫共沉淀实验和荧光定量技术,在巴西橡胶树橡胶粒子总蛋白中发现了一个功能未知的N 端含DUF1262 结构域的蛋白,该蛋白可能与CPT 蛋白相互作用,受水杨酸、脱落酸、过氧化氢及干旱处理诱导显著上调表达,表明其在橡胶树胶乳生物合成和抗逆中具有重要作用,为深入解析天然橡胶生物合成的理化机制奠定坚实基础。
