宣和猪PID1基因外显子3的SNP检测及其与肉质性状的关联分析
2022-09-30朱怡轩王孝义董新星严达伟李明丽
刘 梅,朱怡轩,王孝义,陈 强,董新星,严达伟,李明丽
(云南农业大学动物科学技术学院,昆明 650201)
【研究意义】宣和猪是针对优质猪肉市场和宣威火腿产业需求,以地方猪种乌金猪为基础培育素材、历经10余年选育而成的新品种,于2018年获得国家畜禽新品种证书。宣和猪不仅在繁殖、生长、胴体等主要生产性能上较乌金猪大幅改进,且乌金猪肉质优良、适应性强、耐粗饲、母性好等优点得以有效保持[1],优质高效特征明显,是优质猪肉及宣威火腿生产的理想品种。但目前,关于宣和猪主要的经济性状的具体分子遗传机制还不明确。通过候选基因等途径研究其肉质性状的遗传基础,揭示相关候选基因的遗传效应,对于进一步加快其遗传改良具有非常重要的意义。【前人研究进展】磷酸酪氨酸互作结构域1(Phosphotyrosine interaction domin containing 1,PID1)基因是Wang等[2-3]通过抑制消减杂交技术从肥胖人群腹膜后脂肪组织中筛选出的一个基因。据研究报道,PID1基因的主要生物学作用是降低胰岛素的敏感性,在脂肪、骨骼肌、心肌等胰岛素作用的靶组织中均呈较高表达。PID1是脂肪组织和肌肉中葡萄糖摄取的负调节因子,与脂蛋白相关受体蛋白1(Lipoprotein receptor-related protein 1, LRP1)相互作用以降低脂肪细胞的胰岛素敏感性[4],并经过降低胰岛素受体底物-1(Insulin receptor substrate, IRSs-1)/磷脂酰肌醇-3-激酶(Phosphatidylinositol-3-kinase, PI3K)/蛋白激酶B(protein kinase, AKT)信号通路[5-6]、减少葡萄糖转运子4(Glucose transporter, GLUT4)向包膜转位的机制,引发成熟脂肪细胞胰岛素刺激下葡萄糖摄取率的降低[7-8]。研究还发现肿瘤坏死因子α(Tumor necrosis Factor-α, TNF-α)不仅降低胰岛素敏感性,还显著上调3T3-L1成熟脂肪细胞中PID1基因的表达[9-10],TNF-α和PID1基因对胰岛素的调节都是通过IRSs-1/PI3K/AKT信号通路,而PID1蛋白作为信号分子在脂肪形成以及细胞生长过程中发挥作用,推测PID1基因可能作为重要的分子介质参与TNF-α诱导的胰岛素抵抗。并且PID1还促进3T3-L1成熟脂肪细胞中脂肪酸合成酶(Fatty acid synthase,FASN)基因mRNA的表达,FASN是脂肪酸从头合成的关键限速酶,促进长链饱和脂肪酸的合成[11],同时PID1基因表达过量能显著降低3T3-L1前体脂肪细胞的葡萄糖转换,促进3T3-L1前体脂肪细胞的增殖和脂肪细胞数目的增多[12],最终使脂肪沉积增多。曾勇庆等[13]研究发现,莱芜猪、鲁莱黑猪肌内脂肪含量明显高于大白猪,而钱源等[14]研究发现,在3个品种中,PID1基因mRNA表达量具有显著差异,表现为莱芜猪>鲁莱黑猪>大白猪,且肌内脂肪含量与PID1基因mRNA表达量呈显著正相关。此外,钱源等[15]还发现,在高、中、低3种肌内脂肪含量的莱芜猪中,PID1基因mRNA表达量越高,肌内脂肪含量也越高,由此推测PID1基因可能是影响包括肌内脂肪含量的肉质性状的候选基因。【本研究切入点】本研究以PID1基因作为候选基因研究宣和猪的肉质性状,基于外显子3的SNP突变位点多态性分析每个突变位点与猪肉质性状之间的关联,用来表明每个SNP位点的具体效应。【拟解决的关键问题】为实际育种中使用PID1基因标记改良宣和猪的肉质性状提供一些科学数据。
1 材料与方法
1.1 样品采集
试验中所用的119头宣和猪(♀76头,♂43头)都来自于云南省宣威市良种猪场,试验猪群生长健康状况均良好、系谱清晰,采用相同条件的饲养管理。每头试验猪采集耳组织样约1~2 g,保存于装有75%酒精的EP管中,储于-80 ℃冰箱中备用。
1.2 肉质测定
对119只试验猪的肉质性能测定在云南省宣威市屠宰场完成。在猪群日龄为165~183 d、体重为95~105 kg时对其进行屠宰,取其倒数第3~4肋间的背最长肌和腰大肌样品,按照《猪肌肉品质测定技术规范》(NY/T 821—2004)进行肉质测定。测定肌内脂肪、失水率、滴水损失、肉色、大理石纹、熟肉率等肉质评价指标。其中肌内脂肪含量(Intramuscular fat content, IMF)、滴水损失(Drip loss)、失水率(Water loss rate)、大理石纹(Marbling score)及肉色(Meat color)使用背最长肌进行数据测定,熟肉率(Cooked rate)数据的测定则使用腰大肌。
1.3 基因多态性测定
1.3.1 基因组DNA的提取和检测 采用DNA提取试剂盒(TSINGKE),于2019年12月在云南农业大学动物繁殖与育种实验室对猪耳组织进行DNA的提取。取5 μL待检的DNA原液与1~2 μL 6×loading buffer混合均匀,点样于1.0%琼脂糖凝胶中,110 V恒压电泳35 min,在全自动凝胶成像分析仪下观察DNA条带有无及亮度情况,以检测DNA提取质量。DNA检测合格后,在-20 ℃冰箱中保存备用。
1.3.2 引物合成与PCR扩增 根据Ensembl数据库中的猪PID1基因(序列号:ENSSSCG0007001 3396)的序列,利用 Primer Premier 5.0设计了6对引物,涵盖了PID1基因的第3外显子,在第5号引物(序列为F5:5′-CCCAGCAGAGGAAAGAAG-3′,R5:5′-CAAAGGCGTACACGGACA-3′,预期扩增片段为679 bp)的预扩增片段中检出SNP位点,其余5对引物都未检测到突变位点。引物均由北京擎科(昆明)生物技术有限公司制作合成,合成的引物放于-20 ℃ 冰箱储存备用。PCR扩增体系:DNA模板1.0 μL,2×PCR mix 12.5 μL,上游引物0.5 μL,下游引物0.5 μL,ddH2O 10.5 μL。扩增条件:94 ℃预变性5 min,94 ℃变性30 s,54.0 ℃下退火30 s,72 ℃下延伸30 s,共设置35个循环,最后72 ℃下延伸7 min。PCR所得产物使用1.0%的琼脂糖凝胶电泳进行实验室检测。
1.3.3 目标片段测序和基因型测定 经琼脂糖凝胶电泳检测合格的PCR产物由北京擎科(昆明)生物技术有限公司对其进行DNA正向测序。使用SeqMan、BioEdit等软件对试验样品DNA序列进行后续的分析比对,识别其多态位点,并经过碱基序列判断其个体的基因型。每个SNP位点的位置和基因型使用全基因组序列和相应的碱基名称对其进行正确的命名。
1.4 数据分析
1.4.1 群体遗传结构分析 (1)基因频率。基因频率是指在一个群体中某一等位基因的数量与占据同一基因座的全部等位基因总数的比例,即:
Pi= (2Nii+Nij)/2N
式中,Nii为ii基因型所有的个体数目,Nij为ij基因型所有的个体数目,N为所有检测的个体数目,Pi为i等位基因所占的频率。
(2)基因型频率。在检测个体基因型的基础上,进行各SNP位点基因型频率的计算,基因型频率按以下公式计算:
Pij=Nij/N
式中,Pij表示ij基因型的频率,Nij表示ij基因型的个体数,N表示检测个体数。
(3)多态信息含量。多态信息含量(Polymorphic information content,PIC)是用于估计SNP位点多态性高低的一个指标。当PIC<0.25时为低度多态,当0.25 ≤PIC<0.5时为中度多态,当PIC≥0.5时为高度多态,多态信息含量按以下公式计算:
式中,m表示等位基因数目,pi表示第i个等位基因所占的频率,pj表示第j个等位基因所占的频率。
(4)杂合度(Heterozygosity,H)是指某一SNP位点上杂合子所占的比例,按以下公式计算:
式中,m为某一SNP位点所具有的等位基因数;pi表示第i个等位基因的频率。
(5)Hardy-Weinberg平衡检验。对猪群某一SNP位点是否处于Hardy-Weinberg平衡状态的分析采用χ2检验。假设群体处于Hardy-Weinberg平衡状态,根据样本实际基因频率计算各种基因型的理论频率及理论个体数,然后计算χ2值:
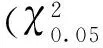
1.4.2 单倍型分析 对PID1基因的4个SNP位点使用Phase 2.0软件对其进行单倍型分析,得到PID1基因的单倍型和单倍型组合的类型,并根据该结果计算其频率。
1.4.3 不同的基因型和单倍型组合与肉质性状之间的关联分析 使用以下最小二乘模型对宣和猪PID1基因外显子3上的4个SNP位点的不同基因型和单倍型组合与肉质性状进行关联分析:
Yijl=μ+Gi+Sj+eijl
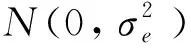
根据上述最小二乘模型[16],4个SNP位点不同的基因型和单倍型组合性状的平均数及标准差使用SAS 9.2[17]进行计算,结果表示形式为“平均数±标准差”,差异显著性根据Duncan法进行检验,差异显著以P<0.05表示,差异极显著以P<0.01表示。
2 结果与分析
2.1 PID1基因的PCR扩增与基因分型
2.1.1 PCR扩增结果 试验样品使用第5号引物经过PCR扩增以后,PCR扩增效果通过1.0%的琼脂糖凝胶电泳进行检测,检测结果见图1,扩增产物片段大小与预期扩增片段长度一致,符合进一步序列测序的要求。

图1 PID1基因 PCR扩增结果Fig.1 PCR amplified results of PID1 gene
2.1.2 基因多态性 在宣和猪PID1基因外显子3区域检测到4个SNP变异位点,分别为253 643 bp处的T→C(T253643C)、253 675 bp处的G→C(G253675C)、253 734 bp处的A→G(A253734G)和253 827 bp处的G→A(G253827A)。各位点均检测出3种基因型,即:T253643C位点的TT、TC和CC基因型(图2);G253675C位点的GG、GC和CC基因型(图3);A253734G位点的AA、AG和GG基因型(图4);G253827A位点的GG、GA和AA基因型(图5)。
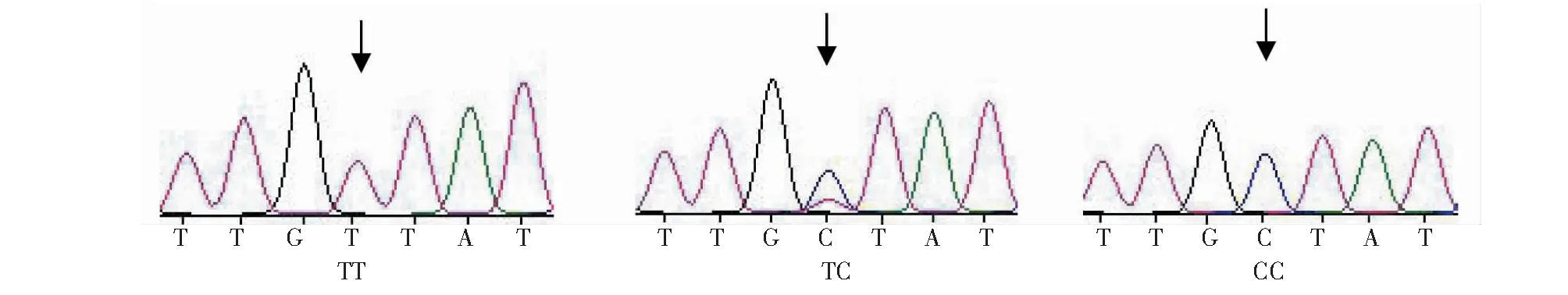
图2 PID1基因T253643C位点测序Fig.2 Sequenced of T253643C site in PID1 gene

图3 PID1基因G253675C位点测序Fig.3 Sequenced of G253675C site in PID1 gene
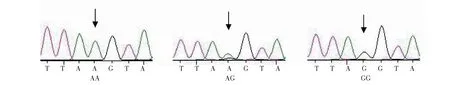
图4 PID1基因A253734G位点测序Fig.4 Sequenced of A253734G site in PID1 gene
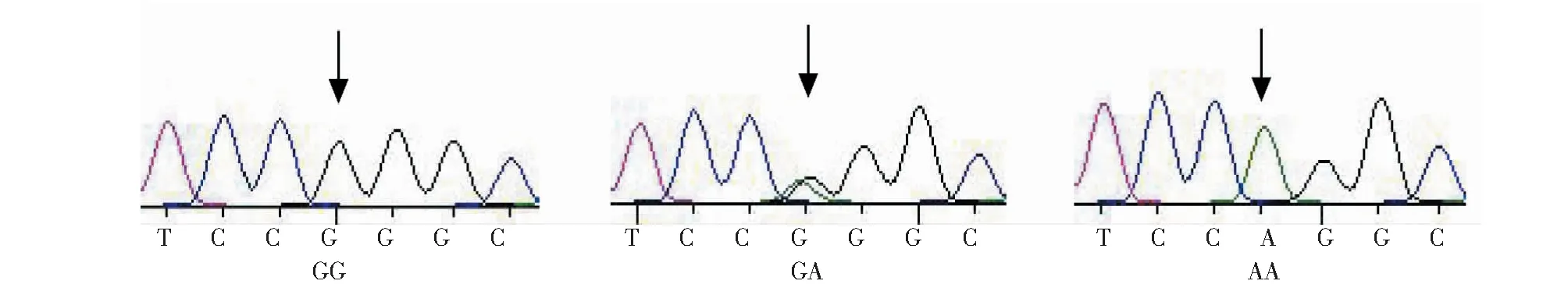
图5 PID1基因G253827A位点测序Fig.5 Sequenced of G253827A site in PID1 gene
2.2 各个位点的基因型与基因频率
宣和猪PID1基因外显子3的4个SNP位点的等位基因频率及基因型频率见表1。T253643C、G253675C、A253734G和G253827A位点分别以TT(0.4034)、GC(0.4790)、AG(0.4538)、AA(0.4454)基因型频率最高,以CC(0.2101)、CC(0.1597)、AA(0.2605)、GG(0.1261)基因型频率最低。从多态信息(PIC)和杂合度(H)和的数值看,4个位点遗传多样性较为丰富,都属于中度多态且均处于Hardy-Weinberg平衡状态(P<0.05)。

表1 宣和猪PID1基因各SNP位点的基因型及基因频率
2.3 4个位点单倍型及其频率
根据表2~3可知,宣和猪PID1基因上的4个SNP位点共有9种单倍型和24种单倍型组合,其中CGGA单倍型的频率(0.366)最高,CCGA单倍型的频率(0.008)最低;单倍型组合以CGGA/TCAA组合的频率(0.168)最高,CGGA/CGGA的频率(0.160)次之,7种单倍型组合CGGA/TGAA、CGGA/TCGA、CCGA/CCGA、CCAG/TGAG、CCAG/TCAA、TGAG/TCAG和TCGA/TCGA频率(0.008)最低且均只检出1个个体。

表2 单倍型及其频率
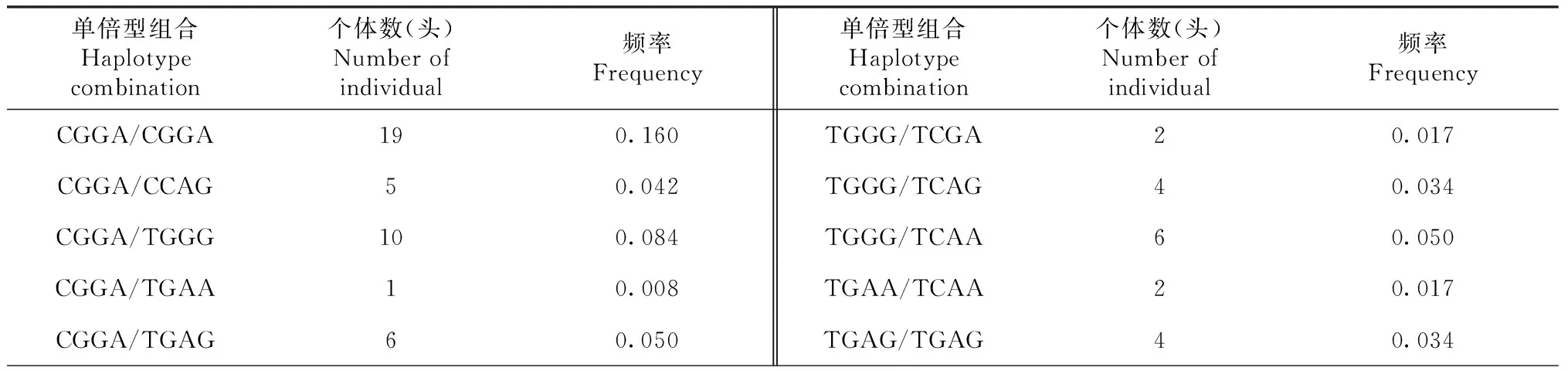
表3 单倍型组合及其频率
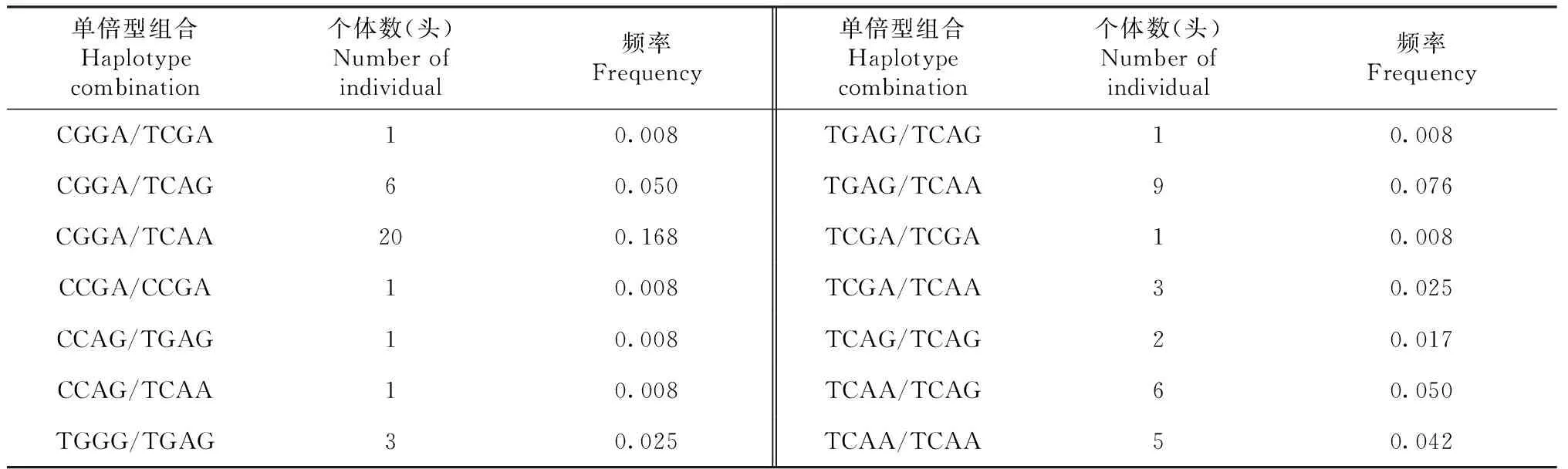
续表3 Continued table 3
2.4 PID1基因SNP位点与肉质性状的关联
2.4.1 各位点不同基因型的肉质性状差异 宣和猪PID1基因4个SNP位点不同基因型的肉质性状如表4所示。G253675C位点与大理石纹存在显著关联(P<0.05),A253734G位点与失水率和IMF含量存在显著关联(P<0.05),G253827A位点与失水率存在显著关联(P<0.05)。其中,T253643C位点的TC基因型个体除熟肉率低以外,其他5个性状均表现最佳,但与其它基因型都无显著性差异(P>0.05);G253675C位点GC基因型个体的大理石纹、失水率、熟肉率以及滴水损失均表现最佳,其大理石纹与CC基因型个体间存在显著差异(P<0.05),较CC基因型个体显著高0.43分;A253734G位点AG基因型个体的大理石纹和失水率表现最佳,其失水率与GG基因型个体间存在显著差异(P<0.05),较GG基因型低3.83%,GG和AA基因型个体的IMF含量存在显著差异(P<0.05),GG基因型较AA基因型高0.45%,AA基因型个体的肉色、熟肉率和滴水损失均表现最佳,但与其它基因型无显著差异;G253827A位点GG基因型个体的大理石纹、失水率和熟肉率均表现最佳,其失水率和AA基因型存在显著差异(P<0.05),较AA基因型低3.68%。
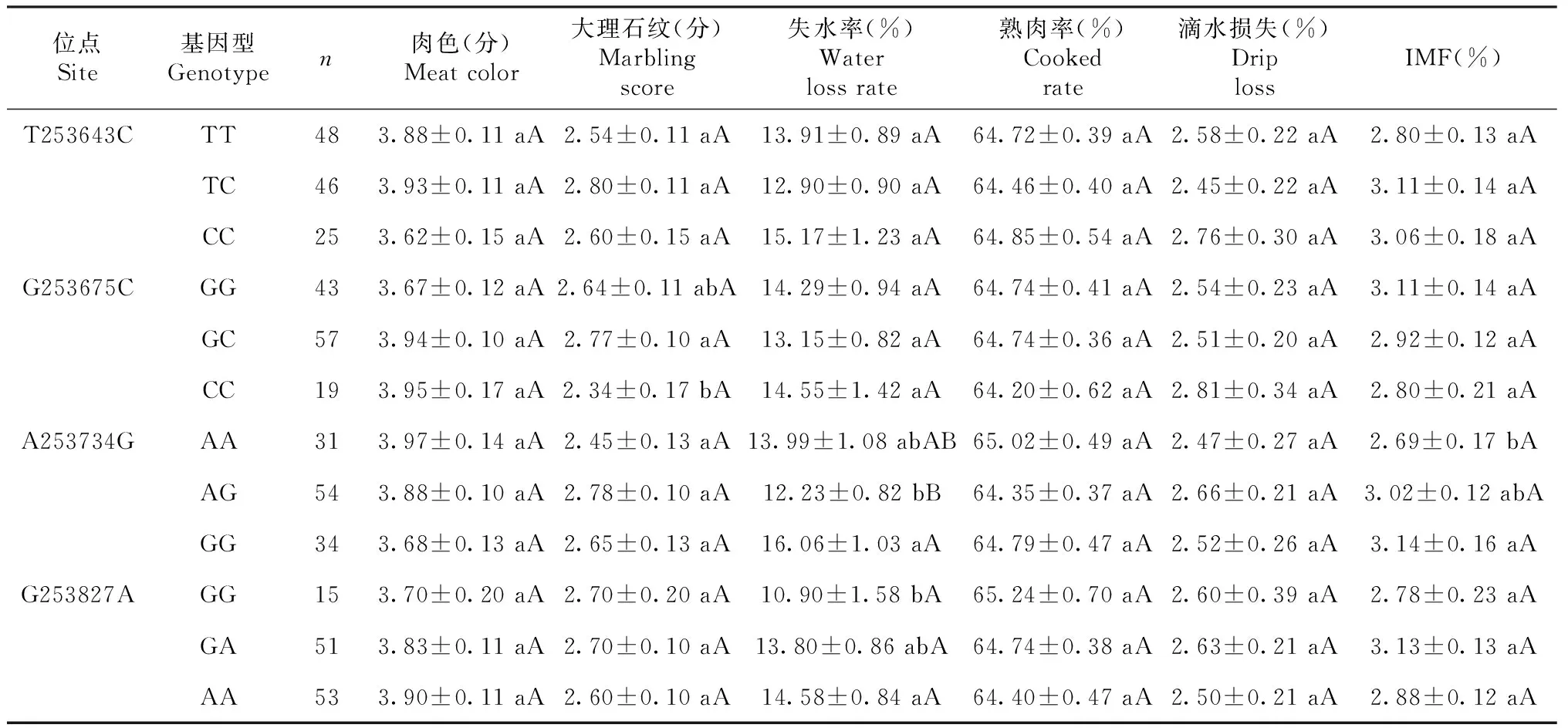
表4 宣和猪PID1基因SNP位点各基因型的肉质性状
2.4.2 4位点不同单倍型组合的肉质性状差异 由5表可知,在大理石纹、失水率、熟肉率和IMF含量这4个性状中,最优组合均含有CGGA单倍型,而性状最差的组合均含有TCAA单倍型。其中CGGA/CCAG组合的大理石纹评分(3.00分)最高,较最低组合TCAA/TCAA(2.30分)高0.7分;CGGA/TGAG组合的失水率(9.91%)最低,较最高组合TCAA/TCAA(17.51%)低7.6%;CGGA/TCAG组合的熟肉率(66.05%)最高,较最低组合TCAA/TCAA(63.07%)高2.98%;CGGA/TGGG组合的IMF含量最高(3.56%),较最低组合TCAA/TCAA(2.26%)高1.3%;TGAG/TCAA组合的滴水损失(1.91%)最低,较最高组合TCAA/TCAG(3.77%)低1.86%。其中CGGA/TGAG、CGGA/TCAA和TG AG/TCAA组合的滴水损失较TCAA/TCAG组合显著低(P<0.05);TCAA/TCAA组合的大理石纹评分、熟肉率和IMF含量均为最低,而失水率则为最高,因此,该组合具有明显降低肉质性状的效应。
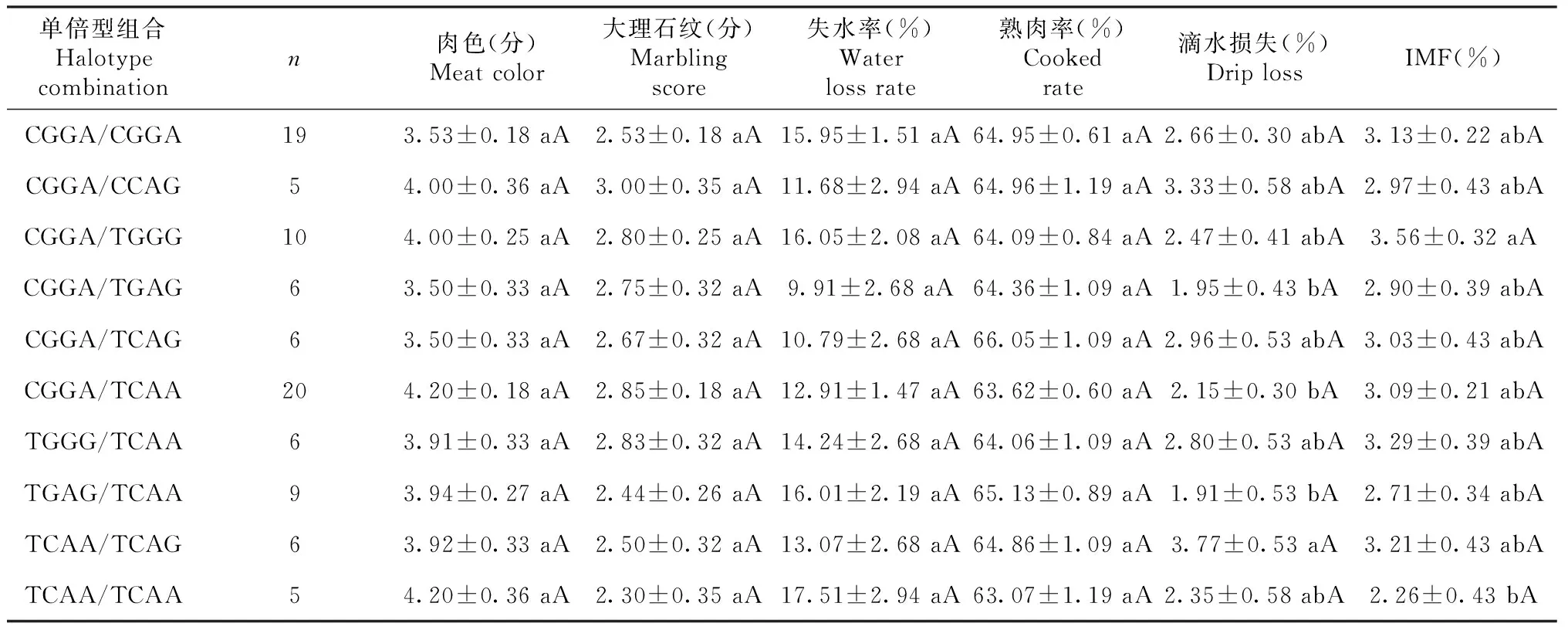
表5 宣和猪PID1基因SNPs位点各单倍型组合的肉质性状
3 讨 论
3.1 宣和猪PID1基因多态性
本试验在宣和猪PID1基因的外显子3区域检测到4个SNP变异位点,分别是T253643C、G253675C、A253734G和G253827A,且均为中度多态(PIC为0.3482~0.3748),杂合度在0.4490~0.4997,遗传多样性丰富。前人关于猪PID1基因的研究主要集中在CDS区的第267位点上的C→T突变,在莱芜猪和鲁莱黑猪中均表现为中度多态(0.25 不同位点各基因型的关联分析结果表明,除T253643C位点外,G253675C、A253734G和G2538 27A位点对宣和猪的肉质性状存在一定的影响。其中,G253675C位点对宣和猪的大理石纹评分有影响,以GC型个体的评分最高,表现出肉质优势;A253734G位点对宣和猪失水率和IMF含量有影响,以AG基因型的失水率最低,以GG基因型的IMF的含量最高;G253827A位点对宣和猪的失水率有影响,GG型个体的失水率最低,说明其肉质更为理想。进一步对上述4个位点不同单倍型组合分析结果显示,10种单倍型组合对宣和猪的滴水损失和IMF含量具有一定影响,CGGA/CCAG组合的大理石纹评分最高(3.00±0.35),CGGA/TGAG组合的失水率(9.91±2.68)最低,CGGA/TCAG组合的熟肉率最高(66.05±1.09),TGAG/TCAA组合的滴水损失(1.91±0.53)最低,CGGA/TGGG组合的IMF含量(3.56±0.32)最高,表明携带有CGGA单倍型个体的整体肉质水平较高。 IMF含量是重要的肉质性状之一,尤其对肉的风味及嫩度等有显著的协同效应,其受多基因控制,主效基因和微效基因之间通过形成复杂的网络和信号传导通路来影响肉的品质。但目前,主效基因仍未确定,研究多集中于与肌内脂肪沉积有关的候选基因来揭示其对肉质的影响,PID1基因就是其中之一。钱源等[14]研究结果显示,PID1基因在不同猪品种的肝脏中mRNA的表达量与IMF含量呈显著正相关。研究表明,肝脏不仅是机体脂肪代谢的重要器官,还是改造脂肪的主要器官,能调整外源性脂肪酸的碳链长短及饱和度,影响肌内脂肪酸的组成,进而影响猪肉的风味[20]。本研究中宣和猪的IMF含量(2.26~3.56)比较理想,但个体间无显著差异。徐正刚[18]的研究发现PID1基因对猪的肌内脂肪沉积性状有一定的影响,但检测到的突变位点与本研究不同,推测这可能是由于猪种不同所致。 目前,关于猪PID1基因的研究报道仍较少,主要集中在基因的克隆、组织表达规律以及其与肌内沉积性状关联分析方面,PID1基因能否作为猪肉质性状相关候选基因以及其影响机制还有待更进一步的研究分析。如果可以对PID1基因其他区域的SNP位点进行测定分析,并在不同的猪种中进行研究确认,将更有利于深入揭示PID1基因对猪肉质性状的真实效应,对表明PID1基因在育种实践中应用的可行性具有非常重要的意义。 本研究根据PCR产物直接测序法对119头宣和猪PID1基因外显子3的SNP位点进行了检测,共检测到4个SNP位点,结合肉质性状数据进行关联分析,发现G253675C位点与大理石纹评分存在显著关联、A253734G位点与失水率和IMF含量存在显著关联、G253827A位点与失水率存在显著关联(P<0.05),携带有CGGA单倍型个体的整体肉质较好,而带有TCAA单倍型个体的肉质则较差,结果显示PID1基因对宣和猪肉质性状有一定的影响。3.2 宣和猪PID1基因SNP位点对肉质性状的影响
3.3 宣和猪PID1基因未来研究展望
4 结 论
