基于众包标注的三代测序数据的纠错方法研究
2022-08-26戴道成
戴道成
(西安欧亚学院 金融学院,陕西 西安 710065)
0 引 言
众所周知,桑格(Sanger)测序法是人类历史上第一代DNA测序技术,因其成本高、通量低的缺点,而被以Roche公司的454技术、illumina公司的Solexa技术和ABI公司的Solid技术为代表的二代测序技术(Next Generation Sequencing, NGS)所取代。相较于Sanger测序,NGS测序的成本大大降低,同时实现了较高的测序精度,但read长度的极大缩短限制了NGS的广泛应用。以Pacific Biosciences公司SMRT技术和Oxford Nanopore Technologies公司纳米孔单分子技术为代表的三代测序技术(Third Generation Sequencing, TGS),不仅继承了NGS的优点,而且能产生长度大于10 kbp的长read,因而在序列组装、基因突变鉴定以及疾病诊断等诸多领域得到广泛的应用。但是错误随机分布的特性和15%的测序错误率是限制TGS大范围应用的主要瓶颈,由此产生两大类针对三代测序数据进行错误校正的方法。第一类是三代测序数据(long reads, LRs)的自校正方法,以HGAP和LoRMA为例,这些方法需要较高水平的覆盖度来确保质量,这无疑是增加了基因组计划的成本。第二类是混合校正方法,此方法需要同时比较LRs和二代测序数据(short reads, SRs),如proovread、LoRDEC和Jabba等,其经常需要计算和处理数百万个SRs到LRs的比对结果,这也是一个十分艰巨的挑战,需要消耗较多的资源和内存。为了避免以上问题,充分发挥已有纠错方法的优点,基于机器学习中的众包标注思想,通过计算高质量SRs集合的一致度、能力和准确度来对低质量LRs的位点进行标注,从而实现三代测序数据的精准纠错。
1 方法设计
1.1 概述
在基于众包标注的三代测序数据的纠错方法(Error Correction method of Third generation sequencing Data based on Crowdsourcing annotation, CTDC)中,将通过基因组仿真得到的三代测序数据视为待标注任务(Long Reads Assignment,LRA),将通过相同基因组仿真得到的二代测序数据视为标注工作者(Short Reads Worker, SRW),利用SRW对LRA进行标注,从而完成三代测序数据的纠错过程。
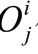
在CTDC中,对LRA中的每个位点进行众包标注的准确性主要取决于参与标注任务的SRW的能力,SRW能力越强,说明其可信度越大,相应位点的标签准确率越高。而SRW的能力受其对应LRA的位点任务难度以及比对质量等的影响,所以为了从众多标签中选出最准确的一个,需要将所有可能影响SRW能力的因素都考虑进去,包括LRA中每个位点的任务难度水平、SRW的准确度以及覆盖在此位点下的SRW之间的一致性。整个算法的设计过程如图1所示。

图1 众包标注的算法设计过程
1.2 SRs的数据预处理
通常来讲,对于一个待标注的LRs任务,初始采集到的来自同一样本的SRs包括三类:完全正确的SRs、具有测序误差噪声的SRs、比对错误而导致错位的SRs。因此,有必要对初始采集到的SRs进行数据预处理,过滤掉相关噪声数据,从而尽可能提高SRs的精度。
首先,由于二代测序数据的错误通常位于read的末端,在使用BLASR进行SRs到LRs的比对时将read尾部的5bp截掉,因此BLASR的参数为:blasr Sk Li --header --out result -m 5。
其次,保证SR与LR之间的相似度Similarity≥0.7,其中Similarity=numMatch/(numMatch + numSub + numIns +numDel),numMatch、numSub、numIns、numDel分别代表正确比对的位点数目、错误比对的位点数目、插入的位点数目和删除的位点数目。
同时,由于BLASR是一种高容错性的比对工具,每条read在M5格式中会产生多条比对结果,并且这些结果已经按照比对质量score值进行降序排序,因此选择最上面的比对结果。
1.3 SRW对LRA的众包标注
1.3.1 LRA难度的计算


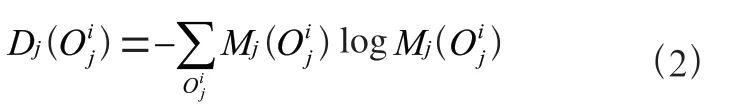
根据信息熵值衡量标签结果分布的平衡度,熵值越大,说明标签越分散,平衡度越高,任务难度越大;反之,标签越集中,平衡度越低,任务难度越小。
1.3.2 SRW能力的计算
在众包标注中,需要考虑SRW的三个要素,自身能力、标注一致度和标注准确度。下文分别进行分析和计算。
1.3.2.1 SRW一致度
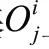
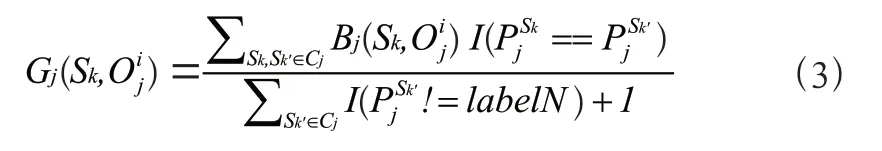

1.3.2.2 SRW准确度
SRW准确度通过其所标注任务的位点中正确标注的位点所占的比例来衡量。在SRW中,对于任意S,令A代表其准确度,计算公式为:

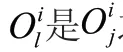
(1)根据LRs的测序错误率,为SRW中的每个S设置一个准确度初始值0.85;
(2)对于第1位碱基,以S的准确度初始值0.85计算相应的支持率,完成众包标注;
(3)对于第k位碱基,以前k-1位更新过的准确度值作为第k位的准确度初值,计算位点对象的支持率,从而实现位点的校正。同时根据校正结果重新计算第k位的准确度值,作为第k+1位的准确度初值。
(4)重复以上步骤,直至所有碱基都得到处理。
1.3.2.3 SRW能力
基于众包标注的已有研究,对于任意S,令B代表其在某一碱基 上的能力,计算公式为:

其中,λ为一个调和参数,用作权衡SRW准确度与一致度的权重比例,默认取值为0.5。
1.4 算法集成
综上,当算法对最终标签进行整合时,通常采用SRW的加权能力值进行以上过程,并且具有更高能力值的工作者对标签选择的贡献权重更大。因此,对式(1)的支持率M进行如下改进。

最大期望算法的计算过程为:

(2)根据式(6)求得所有待标注对象的各个标签的支持率,然后根据最大支持率更新相应标签;
(3)根据式(3)更新S的一致度G,同时根据式(4)更新S的准确度A,然后根据式(5)更新S的能力值B;

(5)迭代次数加1,如果迭代次数超过预设阈值,则算法转到(6),否则,算法转到(5);
(6)重复以上步骤,直至LRA中的所有碱基都得到标注。
2 实验分析
2.1 实验设计
2.1.1 数据说明
考虑到真实数据的巨大和难处理性,CTDC算法的性能验证实验中均使用仿真数据。首先,将人类基因组的chr19染色体作为参考序列,然后通过二代仿真工具TNsim生成长度为100 bp的SRs,并随机植入0.1%的变异位点,通过三代仿真工具PBSIM生成长度为15 000 bp、测序错误率为15%的LRs。
2.1.2 实验说明
本次实验主要验证CTDC算法对于三代测序数据的纠错能力。考虑到CTDC算法的纠错过程以及已有混合校正方法在测序覆盖度、资源消耗和运行内存上的不足,设计以下三组实验进行CTDC算法的性能验证。第一组实验:CTDC在SRs不同测序覆盖度下的精确度分析;第二组实验:CTDC与LoRDEC在SRs不同测序覆盖度下的纠错结果比较;第三组实验:CTDC、LoRDEC与proovread在SRs不同测序覆盖度下的运行时间和内存比较。
2.2 评估指标
TP:原位点有测序错误,且通过CTDC算法进行纠错;
TN:原位点无测序错误,且通过CTDC算法未进行纠错;
FP:原位点无测序错误,且通过CTDC算法进行纠错;
FN:原位点有测序错误,且通过CTDC算法未进行纠错;
Accuracy=(TP+TN)/(TP+TN+FN+FP),即通过CTDC算法成功纠错的位点数占位点总数的比例,通常来说,精确度越高,纠错效果越好。
2.3 结果分析
2.3.1 CTDC在SRs不同测序覆盖度下的精确度分析
对于SRs,设置生成的测序覆盖度分别为5×、10×、20×、30×、50×和100×,考虑到运行时间,将LRs的测序覆盖度设置为5×。经过CTDC纠错,对LRs的每一个位点进行统计分析,得到如图2所示的结果。
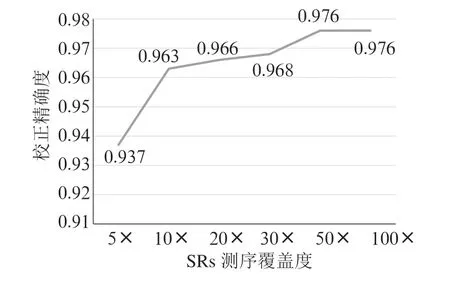
图2 SRs的不同测序覆盖度下CTDC对于三代测序数据的校正精确度
在图2中,随着SRs测序覆盖度的增加,越来越多的SRs参与众包标注,因此CTDC对于三代测序数据的校正性能越来越好。同时,当SRs的测序覆盖度达到50×以上时,CTDC的精确度达到最佳97.6%。
2.3.2 CTDC与LoRDEC在SRs不同测序覆盖度下的纠错结果比较
基于以上实验分析,设置如上的SRs与LRs,同时选择LoRDEC进行比对实验。经过纠错后的位点统计,得到如图3所示的结果。

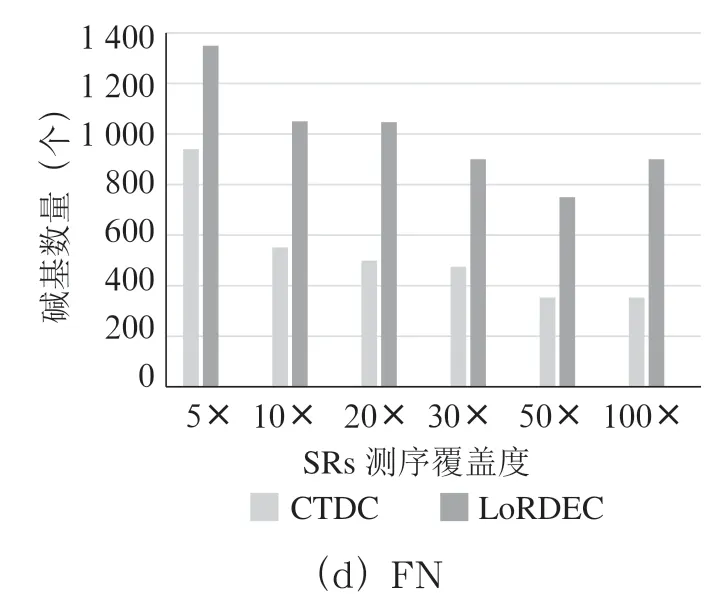
图3 SRs的不同测序覆盖度下CTDC与LoRDEC对于三代测序数据的校正结果
由图3可知,随着SRs测序覆盖度的增加,CTDC和LoRDEC对于三代测序数据的校正性能逐渐增强,但是在测序覆盖度相同时,CTDC的性能优于LoRDEC。为了便于比较CTDC和LoRDEC,绘制图4进行分析。

图4 SRs的不同测序覆盖度下CTDC与LoRDEC对于三代测序数据的校正精确度
总而言之,对于三代测序数据的校正,CTDC的性能优于LoRDEC。
2.3.3 CTDC、LoRDEC与proovread在SRs不同测序覆盖度下的运行时间和内存比较
为了验证CTDC在资源消耗和运行内存上的性能,同样设置如上的SRs与LRs,同时选择LoRDEC和proovread进行比对实验。经过对三代测序数据的纠错,所统计的实验结果如表1所示。

表1 SRs的不同测序覆盖度下CTDC、LoRDEC和Proovread的内存消耗比较
由表1可知,在低于20×的测序覆盖度下,对于三代测序数据的校正,CTDC的运行时间显著低于LoRDEC和proovread。随着SRs测序覆盖度的增加,LoRDEC与proovread的运行时间也明显增加,但均低于CTDC。同时,在运行内存上,CTDC由于计算上的优势,资源消耗明显低于LoRDEC和proovread。
3 结 论
针对测序数据进行有效的校正是获得高精度的基因序列的关键技术。针对现有三代测序数据纠错方法的不足,本文提出一种基于众包标注的三代测序数据纠错方法CTDC。CTDC使用经处理的高质量SRs对LRs的任务难度进行计算,结合其一致度、能力和准确度对当前低质量的各个位点对象分别进行众包标注,并使用最大期望算法完成纠错。根据不同测序覆盖度下的实验结果,当SRs的测序覆盖度为50×时,CTDC对于三代测序数据的校正精确度达到97.6%。同时,在运行时间和占用内存上,CTDC的性能明显优于已知的混合校正算法LORDEC和proovread。后续需要在众包标注算法中考虑LRs的重叠片段,以进一步提高三代测序数据的纠错性能,从而获取更高精度的测序数据,实现后续的基因分析乃至疾病的精准治疗。
