HS5-1增强子eRNA PEARL对原钙粘蛋白α基因簇的表达调控
2022-08-25徐思远寿佳吴强
徐思远,寿佳,吴强
增强子eRNA对原钙粘蛋白基因簇的表达调控
徐思远,寿佳,吴强
上海交通大学系统生物医学研究院比较生物医学研究中心,系统生物医学教育部重点实验室,上海 200240
远端增强子对关键靶基因的表达调控通常可以决定细胞的命运和功能,激活的增强子可以双向转录产生长非编码(long noncoding)增强子RNA (enhancer RNA, eRNA)调控靶基因表达,课题组前期研究发现增强子eRNA能够通过形成R环(R-loop)来促进增强子与靶基因的染色质远距离互作,引起局部三维基因组TAD (topologically associated domain)的改变。为了进一步探究eRNA在基因转录过程中的生物学功能,本研究选取原钙粘蛋白(protocadherin,)基因簇的增强子eRNA(eRNA associated with R-loop formation)作为研究对象,通过CRISPR (clustered regularly interspaced short palindromic repeats) DNA片段编辑技术、逆转录PCR、荧光定量PCR等遗传学和分子生物学实验,揭示了增强子eRNA对基因簇表达的促进作用。首先,本研究通过分析不同组织中增强子eRNA发现其表达具有组织特异性;其次,通过CRISPR诱导增强子内CTCF结合位点的反转或缺失会造成增强子eRNA转录水平降低至2%~10%,同时基因簇的转录水平也会降低至原来的13%~68%;最后,利用CRISPR DNA片段编辑技术删除eRNA双向转录起始位点或反转eRNA的转录起始位点后,基因簇的转录水平下降了约60%或40%,表明增强子eRNA在调控基因表达中发挥重要作用。以上研究结果进一步证实了增强子的转录产物可以调控基因簇的表达,为后续研究原钙粘蛋白基因簇在大脑中的表达调控机制提供了新的思路或方向。
增强子eRNA;簇状原钙粘蛋白;eRNA;基因表达;基因调控
多细胞生物中各种细胞类型的分化需要在发育过程中建立特定的基因表达模式。真核生物基因的转录调控主要由增强子完成,增强子是包含多种转录因子(transcription factor, TF)结合位点的DNA调控元件[1~3]。增强子最早发现于1981年,猿猴空泡病毒40 (simian virus 40, SV40)的重复序列能够通过质粒转染以不依赖自身序列方向的方式增强β珠蛋白基因的表达[4]。在之后的研究中,常将候选增强子与报告基因序列克隆到表达质粒中,通过检测报告基因活性和荧光强度,鉴定增强子元件的功能[5]。随着测序技术的发展,染色质免疫沉淀与高通量测序结合(chromatin immunoprecipitation high-throughput sequencing, ChIP–seq)的实验方法得到广泛应用,该方法通过特定的组蛋白修饰和表观遗传学标记识别候选增强子[6]。CRISPR (clustered regularly interspaced short palindromic repeats)基因编辑系统也可用于增强子的鉴定,对目标序列进行突变或删除,有助于筛选出潜在的调控元件[7]。但是这种方法会引起内源DNA双链断裂(double strand break,DSB),促发细胞的双链断裂修复机制,针对这种情况,研究者将失活后的Cas9蛋白(dead Cas9, dCas9)与抑制或者激活因子偶联靶向至增强子处,通过检测相邻基因的表达水平来识别增强子[8,9]。
根据转录因子结合情况的不同,增强子可分为沉默增强子和活性增强子[10,11],活性增强子在激活靶基因时会募集大量转录因子、RNA聚合酶II (RNA polymerase II, RNAPII)和表观遗传修饰酶,使自身处于开放状态[12]。使用氯化钾激活培养的神经细胞后,在全基因组范围内发现大量由刺激激活的增强子,这些增强子通过结合CBP招募RNAPII,在增强子区域双向转录产生增强子RNA (enhancer RNA, eRNA)[12]。增强子eRNA由活性增强子产生,大类上属于长链非编码RNA (long non-coding RNA, lncRNA)。增强子eRNA通常双向转录产生,是长度相对较短、丰度相对较低、易被RNA外切体(RNA exosome)降解的转录本[13~16]。增强子的转录是否发挥特定的生物学功能对研究增强子性质和基因调控具有重要意义[14]。早期的研究认为增强子的转录本是副产物,增强子仅起到募集转录因子的作用[17]。随着研究的深入,越来越多的证据表明eRNA在调控基因表达方面具有重要功能。增强子eRNA可能与Mediator蛋白复合物相互作用,促进基因转录[18,19];增强子eRNA可能与CTCF或黏连蛋白(cohesin)相互作用帮助建立稳定的增强子–启动子环[20,21];增强子eRNA可能招募组蛋白修饰酶来改变增强子区组蛋白甲基化或乙酰化水平[22,23]。基因组上不同的活性增强子通常可转录产生eRNA,但关注eRNA自身生物学功能的研究较少。
大脑能够通过轴突–树突间的突触连接形成特定的神经元回路,复杂神经元回路的组装需要细胞表面特定的分子标签,近60个簇状原钙粘蛋白(clustered protocadherin, c)基因赋予了细胞表面分子标签的多样性[24,25]。人类c基因位于5号染色体上,由53个串联排列的基因组成,形成3个紧密相连的原钙粘蛋白(,,)基因簇[26]。基因簇包含可变区和恒定区,由15个高度相似的基因组成,可变区外显子包括13个随机表达型外显子(-)和2个C型外显子(-),位于可变区下游的恒定区包含3个外显子。每个可变区外显子通过顺式剪接与下游的3个恒定区外显子相连形成成熟的原钙粘蛋白mRNA (图1A)[27,28]。在基因簇下游存在2个超敏位点(hypersensitive site, HS)增强子元件,分别被命名为和[29]。增强子主要调控后半部基因表达(除),增强子全局调控基因簇表达[30]。CTCF(CCCTC-binding factor)作为染色质架构蛋白在基因簇的表达中发挥重要作用,每个随机表达型外显子区域存在2个正向CTCF结合位点(CTCF binding site, CBS)[31~33]。在cohesin的协助下,外显子区域内正向的CBS与增强子内存在的2个反向的CBS元件(CBSa和CBSb)发生远距离染色质相互作用,促进基因表达[31,32,34]。当增强子内的CBS元件反转后,增强子与启动子间的远距离染色质相互作用减少,造成基因簇的表达水平显著下降[34~36]。RFX5、REST/NRSF等转录抑制因子在增强子处的结合会减弱增强子–启动子之间的远距离染色质相互作用[37,38]。另外,每个可变区外显子附近会反向转录产生长非编码lncRNA,这种转录活动引起基因启动子区DNA去甲基化,促进CTCF结合[39]。最近的研究发现远端调控元件增强子主要反向转录产生eRNA,并且在原位形成R环结构调控基因簇的选择性表达[40]。
本研究进一步探究活性增强子的转录产物eRNA在调控基因簇表达中发挥的生物学功能。本课题组最近的研究揭示了增强子元件的转录情况,发现增强子主要转录产生反向的eRNA[40]。然而增强子eRNA的正向转录产物是否具有功能,以及eRNA转录方向的改变是否会影响基因簇的表达还不清楚。本研究在此基础上,继续对增强子eRNA进行研究。首先,对增强子eRNA进行了组织特异性分析。其次,对本实验室之前构建的增强子CBS元件删除或反转的单克隆细胞株的eRNA和基因簇的表达情况进行定量分析;最后,利用CRISPR DNA片段编辑技术[41~44]在HEC-1-B细胞中构建增强子eRNA双向转录起始位点删除和反转的单克隆细胞株,检测其eRNA和基因簇的转录水平。这些结果为后续研究基因簇的表达调控机制和eRNA的生物学功能提供参考。
1 材料与方法
1.1 实验材料
人类子宫内膜瘤细胞系HEC-1-B从中科院上海细胞库采购;构建sgRNA所用质粒pGL3-U6- Puromycin来源于上海科技大学黄行许教授实验室,表达Cas9所用质粒pcDNA3.1-WT来源于北京大学席建忠教授实验室;高保真Phanta PCR扩增酶和逆转录试剂盒采购自南京诺唯赞生物科技有限公司;实时荧光定量工具酶采购自瑞士Roche公司;限制性内切酶I、T4 DNA连接酶采购自美国NEB公司。PCR产物纯化试剂盒采购自德国Qiagen公司,质粒小抽试剂盒采购自美国AXYGEN公司;细胞培养所用MEM培养基采购自美国Hyclone公司;胎牛血清采购自中国Excell Bio公司;10×PBS缓冲液和胰蛋白酶采购自美国Gibco公司;细胞培养所用青霉素–链霉素双抗、丙酮酸纳和谷氨酰胺,转染所用Lipofectamine 3000采购自美国Thermo公司;无水乙醇采购自国药集团;异丙醇、三氯甲烷采购自上海沪试实验室器材股份有限公司;RNA提取液TRIzol Reagent采购自美国Invitrogen公司;嘌呤霉素、氯化钠和SDS采购自美国Sigma公司;配制LB培养基所需胰蛋白胨、酵母提取物、琼脂采购自英国的OXOID公司;克隆载体采购自北京擎科生物科技有限公司;本研究所用引物由生工生物工程(上海)股份有限公司合成。
1.2 sgRNA质粒构建
合成实验所需sgRNA单链(表1),用ddH2O将引物浓度稀释至20 μmol/L备用。配制如下反应体系(总体积为20 μL)用于引物退火:2 μL 20 μmol/L sgRNA-F,2 μL 20 μmol/L sgRNA-R,2 μL 10× NEB buffer 2,14 μL ddH2O。用移液器吹打混匀后置于95℃水浴锅中5 min,关闭电源,盖上盖子冷却至室温,反应结束后短暂离心收集液体。
用限制性内切酶I将环形质粒pGL3-U6- Puromycin切开,切胶回收线性化载体。纯化后进行连接反应,用大肠杆菌感受态转化。次日挑取数个克隆进行摇菌培养,利用质粒小抽试剂盒抽提质粒,并送擎科生物上海公司进行一代测序。
1.3 细胞培养与转染
HEC-1-B细胞系的MEM完全培养基配方为87% MEM培养基、10%胎牛血清、1%青霉素–链霉素双抗、1%丙酮酸钠,1%谷氨酰胺。所有细胞均培养在37℃ 5% CO2的恒温培养箱中。
待6孔板细胞密度达到50%~60%时,用Lipofectamine 3000进行细胞转染。离心管中加入Lipo3000和MEM培养基,另外一个管中加入Cas9质粒,sgRNA质粒,p3000和MEM培养基。将液体吹打混匀,并转移至一个离心管中,室温孵育10 min后滴入到6孔板中,细胞放入37℃恒温培养箱中继续培养。
1.4 单克隆细胞株筛选
转染2天后,换用含有2 μg/mL的嘌呤霉素培养基进行4天的药物筛选,之后用正常培养基恢复培养2天。取部分细胞裂解其基因组,通过PCR扩增鉴定该混合物基因型,若混合物中含有目的片段删除的细胞,则对剩余细胞进行单克隆化铺板。在37℃细胞培养箱培养1周后,在倒置显微镜下对含有单个细胞团的孔进行标记,再培养1周后,对所有单克隆细胞株进行基因型鉴定。
用胰酶对细胞进行消化,通过碱裂解法裂解单克隆细胞基因组,进行PCR扩增。反应体系(总体积为20 μL)如下:10 μL 2 × Taq Mix,0.5 μL 10 μmol/L正向引物,0.5 μL 10 μmol/L反向引物,2 μL基因组模板,7 μL ddH2O。反应条件为94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸1 min,共40个循环;最终延伸5 min。通过琼脂糖凝胶电泳确定单克隆基因型,将阳性克隆的PCR产物进行送测。若测序结果为双峰,则将PCR产物纯化后通过TA克隆的方式鉴定单克隆细胞株的基因型。将PCR产物与克隆载体在室温条件下孵育5 min后,加入50 μL感受态细胞,冰上冷却30 min后进行热激,加入500 μL无抗LB进行摇菌,37℃恒温摇床培养1 h。离心后将菌液涂在含有氨苄抗性的平板上,过夜倒置培养。次日挑取10个单菌落进行摇菌,抽提质粒后送擎科生物公司进行一代测序。鉴定单克隆基因型所用引物见表1。

表1 引物序列
1.5 逆转录PCR和荧光定量PCR
待6孔板细胞达到90%密度时,弃尽细胞表面培养基。每个孔加入500 μL TRIzol消化细胞,并提取细胞总RNA。随后使用南京诺唯赞公司逆转录试剂盒进行逆转录实验。取1 μg总RNA,加入1 μL链特异性引物,用ddH2O补至12 μL,在PCR仪上65℃反应5 min将RNA模板变性,之后立即冰上冷却2 min;加入4×gDNA wiper Mix去除基因组DNA,42℃反应2 min;加入10×RT Mix和HiScript II Enzyme Mix各2 μL,经25℃ 5 min、50℃ 45 min、80℃ 2 min反应后得到cDNA文库。
将模板用ddH2O稀释至50 μL,配制荧光定量PCR反应体系。在PCR中依次加入以下溶液(总体积为10 μL):5 μL SYBR Green,0.3 μL 5μmol/L正反向引物混合物,2 μL cDNA模板,2.7 μL ddH2O,在荧光定量PCR仪上进行反应。以作为内参基因,采用2–ΔΔCt的方式计算基因相对表达量。荧光定量PCR所用引物见表1。
1.6 HS5-1增强子eRNA组织特异性分析
从ENCODE数据库中下载人类HEK293细胞[45]和小鼠肾脏组织[46]的启动子活性标记H3K4me3、增强子活性标记H3K27ac和染色质架构蛋白CTCF的ChIP-seq实验数据,并将其导入至UCSC浏览器中进行分析。将人类和小鼠基因簇的增强子区域进行放大,人类HEK293细胞的参考基因组为hg19,小鼠肾脏组织的参考基因组为mm10。FANTOM5数据库的CAGE数据[11]代表增强子eRNA的潜在转录起始位点,蓝色表示反向转录起始位点,红色表示正向转录起始位点。同时与之前发表的人类HEC-1-B细胞和小鼠大脑皮层组织的相关数据进行对照[40],以此揭示增强子eRNA的组织特异性。
2 结果与分析
2.1 原钙粘蛋白α基因簇HS5-1增强子eRNA表达具有组织特异性
增强子eRNA是由激活的增强子转录产生的,通常位于染色质开放区域,大量的转录因子和辅助因子会结合在增强子上,其周围的核小体也会发生相应的修饰[16]。全基因组分析表明,哺乳动物中的活性增强子双向转录产生的eRNA一般具有组织特异性[11]。本课题组最近的研究发现在人类HEC-1-B细胞和小鼠大脑皮层组织中的增强子是活性增强子,在eRNA转录的区域富集了RNAP II、H3K4me3、H3K27ac、CTCF和Rad21等信号,能够激活基因簇的表达[40]。为了进一步验证增强子eRNA的转录特征,本研究分析了人类HEK293细胞[45]和小鼠肾脏组织[46]的增强子eRNA表达谱。在HEK293细胞中,尽管增强子处有CTCF结合可以使增强子与启动子的空间距离相互靠近,但ChIP-seq数据显示增强子处的染色质组蛋白上没有发生甲基化和乙酰化修饰,推测该细胞系中增强子并未发生活化,也没有转录产生eRNA (图1B),这与文献中报道的eRNA的定量结果相一致[40]。和人类HEK293细胞类似,小鼠肾脏组织中增强子也并未活化(图1C),这与最近的一篇文章中揭示的小鼠肾脏组织的RNA-seq实验结果相一致,数据显示整个基因簇基因几乎不表达[39]。这些eRNA的转录特征分析表明增强子的转录具有组织特异性。
2.2 HS5-1增强子CTCF位点方向改变导致eRNA转录水平降低
本课题组最近的研究发现基因簇中的增强子eRNA通过在原位形成R环的方式介导增强子与基因簇启动子间的远距离染色质相互作用,进而激活基因簇的表达[40]。R环是指在转录过程中,新生的RNA链与DNA链进行碱基互补配对导致另一条单链DNA被置换,产生一种含有RNA-DNA杂合体的三链结构,这种R环结构在基因的表达调控和DNA修复中发挥了重要的功能[47,48]。本文利用荧光定量PCR对之前构建的增强子CBSa、CBSb位点基因编辑的细胞克隆与野生型细胞的和基因簇的转录水平进行定量分析。结果表明,在增强子CBS元件反转的细胞株中,增强子eRNA的转录水平降低至野生型克隆的5%,基因簇的转录水平下降至野生型克隆的15% (图2,A和B)。在增强子CBS元件一条等位基因反转、一条等位基因缺失的杂合子细胞株中,增强子eRNA和基因簇的转录水平都显著降低(图2,C和D),这表明CBS元件方向的改变会引起和基因簇的表达异常。在这两种情况中,增强子eRNA的转录方向发生原位反转破坏了原本的eRNA序列,由于R环倾向于在转录起始位点区域形成,因此可能破坏了R环结构的方向性,导致的转录水平降低。同时的转录方向在三维空间折叠后不再与基因簇的转录方向相同,由CBS元件介导的增强子–启动子环也被破坏,导致基因簇的表达下降。此外,这两种情况相比较,第一种单克隆细胞株中的和基因簇的表达受到的影响更大。在增强子CBSa位点反转的杂合子细胞株中,增强子eRNA和基因簇的转录水平分别下降90%和40% (图2,E和F)。在该细胞株中,CBSa位点方向的改变并不影响的长度,但是在一定程度上改变了的转录本序列,因此推测序列本身在调控基因簇的表达中发挥重要作用。虽然CBSa位点的原位反转没有破坏的转录起始区域,但这一反转不仅改变了CBSa位点自身的方向,导致增强子与启动子间的相互作用减弱;而且导致增强子内部的部分序列发生改变,这一改变可能破坏了增强子内原本的R环结构。这个结果表明CBSa元件方向的改变会引起的异常转录。在增强子CBSb位点删除的纯合子细胞株中,增强子eRNA的转录水平并没有发生明显变化(图2,G和H)。由于的转录起始位点位于两个CBS元件之间,因此对CBSb位点进行基因编辑没有破坏的转录起始区域和其序列本身。这个结果表明增强子CBSb元件作为基因簇的边界,它的缺失不会引起异常转录,但其删除后会破环TAD的边界,引起基因簇的表达降低(图2,G和H)。总之,增强子内CTCF结合位点方向的改变或缺失会引起eRNA的异常转录,同时影响基因簇的表达。

图1 原钙粘蛋白α基因簇HS5-1 eRNA的组织特异性分析
A:人类原钙粘蛋白基因簇()示意图。人类基因簇由15个可变区外显子和3个恒定区外显子组成,15个可变区外显子包括13个随机表达型外显子和2个C型外显子。每个基因的可变区外显子都携带自己的启动子,通过顺式剪接与下游3个恒定区外显子相连。B:ENCODE数据库中[45]人类HEK293细胞系中增强子处eRNA的特异性分析。依次为H3K4me3、H3K27ac和CTCF的ChIP-seq结果(橘色)。C:ENCODE数据库[46]小鼠肾脏组织(紫色)中增强子处eRNA的组织特异性分析。
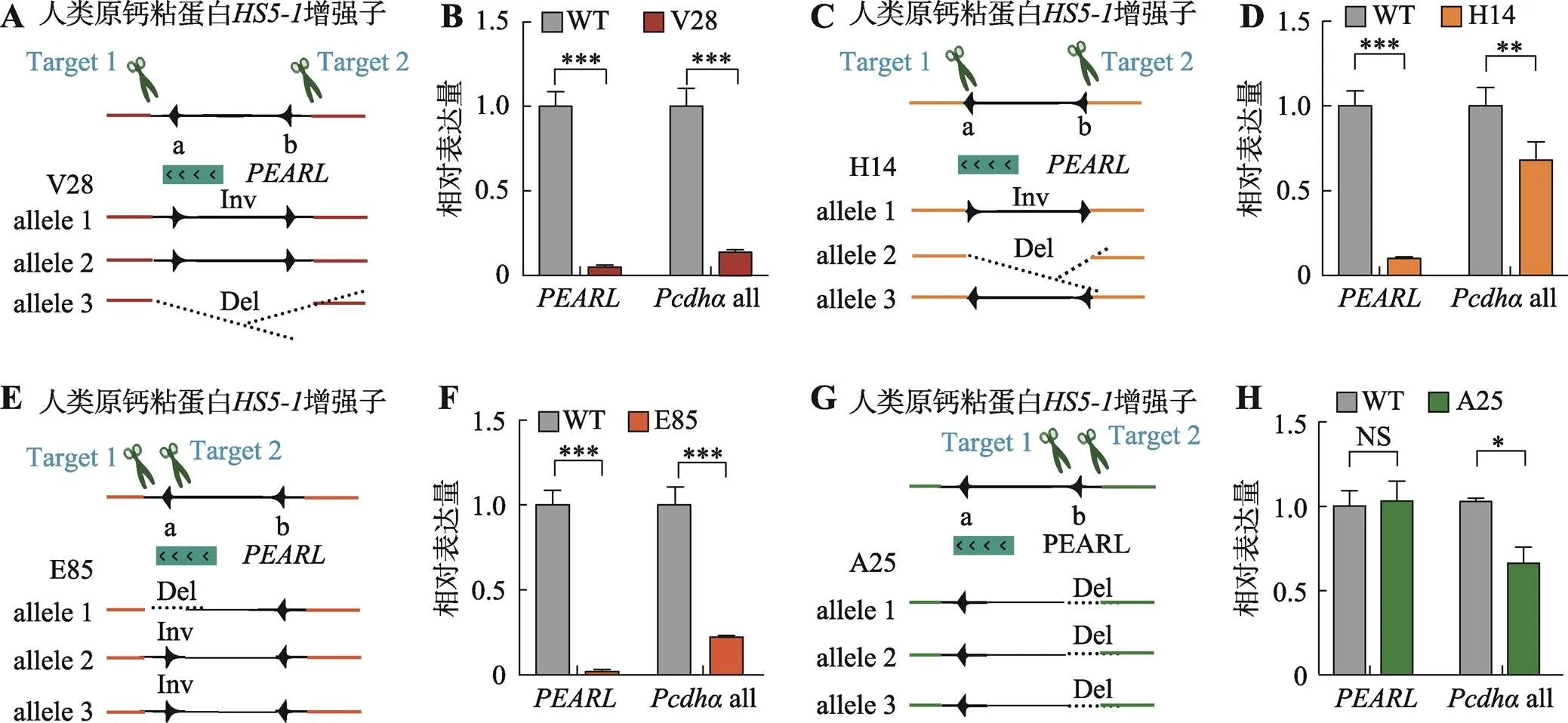
图2 HS5-1增强子CTCF位点方向改变导致eRNA转录水平降低
A:增强子CBS反转的单克隆细胞株基因型示意图。B:增强子eRNA和基因表达的定量分析。C:增强子CBS编辑后的单克隆细胞株基因型示意图。D:增强子eRNA和基因表达的定量结果。E:增强子CBSa反转的单克隆细胞株基因型示意图。F:增强子eRNA和基因表达的定量分析。G:增强子CBSb删除的单克隆细胞株基因型示意图。H:增强子eRNA和基因表达的定量结果。NS:没有显著性差异;*:<0.05;**:<0.01;***:<0.001。
2.3 HS5-1增强子eRNA对原钙粘蛋白α基因簇的激活功能
在此之前,已有的研究利用CRISPR DNA片段编辑技术删除了eRNA的转录起始位点来探究其生物学功能[40]。为了进一步研究增强子eRNA对基因簇的调控作用,同时排除CBS元件的影响,根据FANTOM5数据库揭示的增强子eRNA的转录起始位点,设计靶向正反向转录区域的成对sgRNA,利用CRISPR DNA片段编辑技术删除增强子eRNA分子的双向转录起始位点(图3A)。根据编辑区域设计成对引物对单克隆细胞株的基因型进行鉴定,凝胶电泳结果和一代测序结果表明,增强子eRNA双向转录起始位点被成功删除(图3,B和C)。通过荧光定量PCR实验对基因簇的转录水平进行定量分析发现,在缺失eRNA双向转录起始位点后,基因簇的转录下降了70% (图3D)。这个结果进一步说明增强子eRNA的双向转录过程甚至是转录本可以调控基因簇的表达。此外,CTCF蛋白敲低也造成增强子eRNA和基因簇的转录水平显著降低(图3,E和F)。增强子与可变型启动子间的远距离染色质相互作用不仅需要eRNA的转录,同时需要CTCF/cohesin等染色质架构蛋白的参与[40]。CTCF蛋白的减少,导致增强子–启动子之间的远距离染色质相互作用减弱,染色质环的破坏导致eRNA转录水平的下降。
增强子eRNA双向转录起始位点的删除,同时造成了增强子CBSa位点的缺失,由于CBSa位点具有重要的调控作用,因此基因簇的表达下降可能是由于CBSa位点被破坏所导致的。为了排除CBSa位点的干扰以及探究eRNA转录方向的改变是否会影响基因簇的表达,利用CRISPR DNA片段编辑技术将的转录起始位点进行原位反转(图4A)。通过对单克隆细胞株进行筛选,总共获得了两个转录起始位点反转的杂合子单克隆(图4,B和C)。荧光定量PCR实验结果表明,转录起始位点反转的单克隆细胞株中,增强子eRNA自身转录下降了80%左右;与此同时,其调控的基因簇的转录水平降至原来的一半(图4D)。转录起始位点的反转不仅改变了自身的转录方向,同时也破坏了其与基因簇转录方向的协同性。此外,转录方向的变化使得原本转录出的RNA序列随之改变,由于共转录R环主要在启动子和转录起始位点处形成,序列本身的改变会动摇、甚至是破坏R环结构,导致基因簇周围的染色质结构发生改变。原本由形成的R环结构所介导的增强子–启动子之间的远距离染色质相互作用也被破坏,导致基因簇的表达下调。这些结果表明原位反转的转录起始位点方向会影响基因簇的表达。

图3 使用CRISPR DNA片段编辑技术删除HS5-1增强子eRNA双向转录起始位点
A:敲除增强子eRNA转录起始区所用sgRNAs及单克隆基因型鉴定所用引物示意图。B:单克隆细胞株基因型鉴定凝胶电泳图。以野生型细胞作为对照,F1和R1的目的产物片段为1798 bp,删除转录起始区后产物片段为1079 bp,F1和R2的目的产物片段为826 bp。C:单克隆细胞株基因型鉴定一代测序图。红色碱基为PAM位点,红色箭头表示Cas9切割位点;粉色框内为上游接口处碱基,黄色框内为下游接口处碱基。D:野生型克隆、增强子eRNA转录起始区域删除的单克隆细胞株中基因表达的定量分析。E:敲低CTCF后的蛋白质免疫印记。F:CTCF敲低后的增强子eRNA和基因表达的定量分析。TSS:转录起始位点;WT:野生型;KD:敲低。**:<0.01;***:<0.001。

图4 使用CRISPR DNA片段编辑技术反转HS5-1增强子eRNA PEARL的转录起始位点
A:反转eRNA转录起始位点所用sgRNAs及单克隆基因型鉴定所用引物示意图。B:单克隆细胞株基因型鉴定凝胶电泳图。以野生型细胞作为对照,F1和R1的目的产物片段为1798 bp,杂合子中一条等位基因目的产物片段为1504 bp,F1和F2的目的产物片段为921 bp。C:单克隆细胞株基因型鉴定一代测序图。红色碱基为PAM位点,红色箭头表示Cas9切割位点;蓝色框内为上游接口处碱基,绿色框内为反转序列,黄色框内为下游接口处碱基。D:野生型克隆、转录起始位点反转的单克隆细胞株eRNA和基因表达的定量分析。TSS:转录起始位点;WT:野生型;inv:反转。**:<0.01;***:<0.001。
3 讨论
钙粘蛋白是Ca2+依赖性细胞粘附分子,钙粘蛋白之间特异的细胞间粘附在组织的形成和维护中发挥关键作用[49]。原钙粘蛋白属非经典钙粘蛋白,在神经元间的自身识别和非自身回避、神经元存活和迁移、轴突–树突间的连接等功能中发挥重要作用,同时在某些神经以及精神疾病中,原钙粘蛋白可以作为疾病诊断的标志物[24,25]。原钙粘蛋白基因簇下游的远距离顺式调控元件和在调控基因簇的表达中发挥重要功能[29]。此外,原钙粘蛋白基因的表达需要CTCF/cohesin复合物介导的增强子–启动子之间的远距离染色质相互作用[31,32,34]。
增强子eRNA转录是否具有功能一直是eRNA研究领域的重点问题[14]。最近的研究表明基因簇的增强子内主要反向转录产生eRNA来激活基因表达(图5A)[40]。本研究在此基础上进一步探究增强子eRNA的功能,对本实验室之前构建的增强子CTCF结合位点编辑的细胞株进行定量分析,发现CTCF结合位点方向的改变会引起的异常转录。由于eRNA的转录丰度较低,在对其克隆时并未获取其完整的序列,只得到了一个804 bp的分子序列[40]。增强子CTCF结合位点反转的细胞株(图2,A~D)转录的eRNA分子包括了上游CRISPR/Cas9切割位点,说明将CBS元件反转不仅改变了的转录方向,而且改变了原本的序列。在增强子CBSa位点反转的细胞株中(图2,E和F),由于CBSa位点的序列包含在804 bp的eRNA分子内,因此这一反转同样改变了原本的序列。这两种情况都可能破坏了原本的R环结构,造成和基因簇的转录水平显著降低,因此推测的序列具有调控基因簇基因表达的功能(图5B)。利用CRISPR DNA片段编辑技术删除eRNA的双向转录起始位点的实验表明,增强子的正向转录产物同样具有调控功能(图3)。尤其是当只反转的转录起始位点同样可以影响基因簇的表达,这一结果说明转录的方向性可能对基因簇具有调控作用(图4)。上述两个实验利用CRISPR系统编辑eRNA的转录起始位点区域,同样改变原有的转录本序列,转录本序列的变化将会影响原本稳定的R环结构(图5C)。
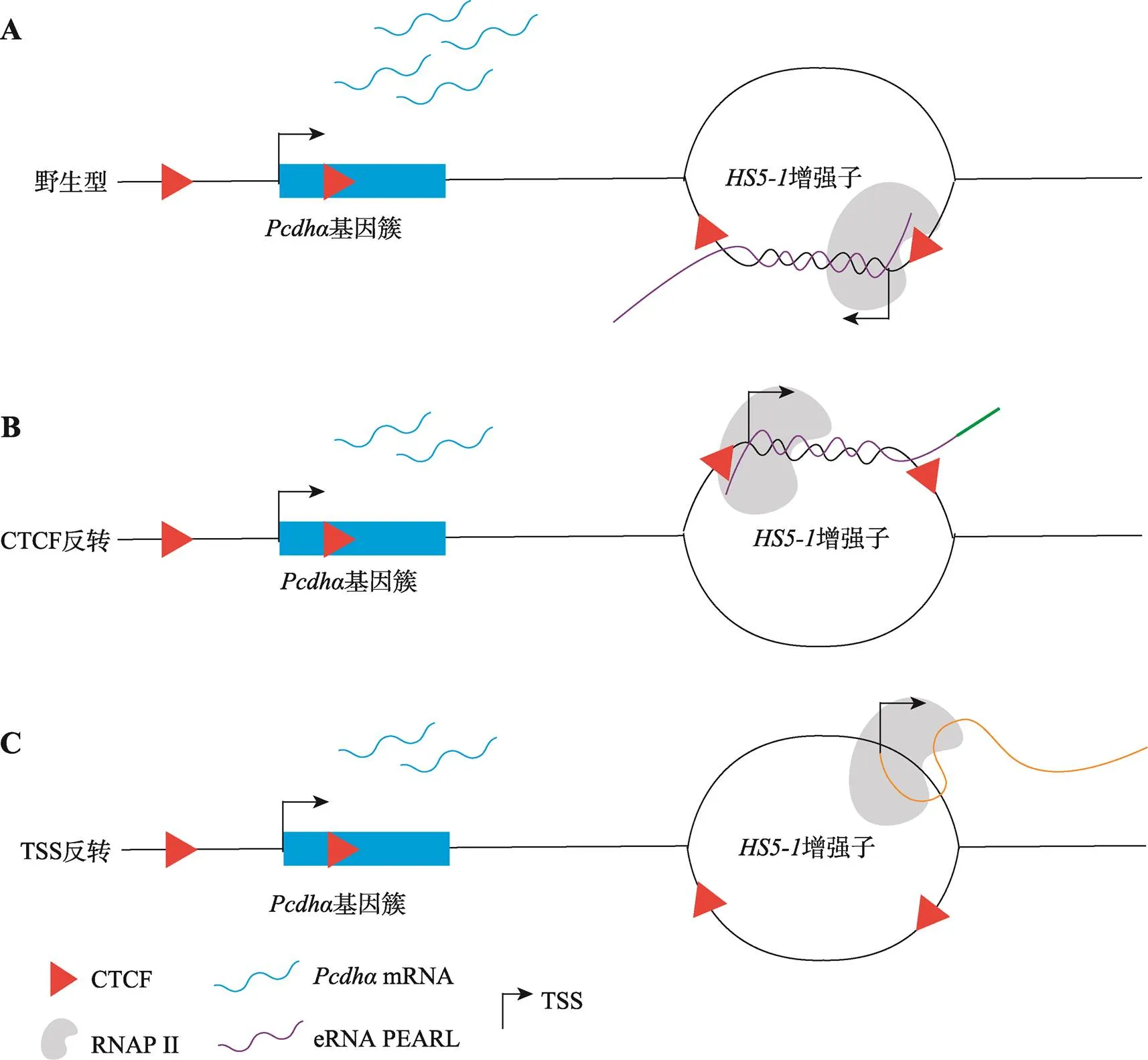
图5 增强子eRNA PEARL调控原钙粘蛋白α基因簇表达的示意图
A:在野生型细胞中,增强子eRNA(紫色)在增强子处形成R环激活基因簇表达。B:使用CRISPR DNA片段编辑系统反转增强子及其两边的CTCF位点,不会改变增强子eRNA转录本的5′端序列(紫色),但会改变eRNA转录本的3′端序列(绿色),因此可能会改变原本的R环结构,造成基因簇的表达降低。C:反转eRNA的转录起始位点会完全改变其自身序列(橙色),增强子内的R环结构可能遭到破坏,导致基因簇的表达降低。TSS:转录起始位点。
由于eRNA是由增强子DNA序列转录的,遗传学方法在一定程度上破坏了增强子区域。因此,在确定eRNA转录起始位点的前提下,可以通过定点突变的方式研究eRNA的功能。此外,一些辅助激活因子已被证实在调控eRNA转录,维持增强子活性和染色质结构方面发挥作用[22,50],关于辅助抑制因子的研究也开始逐步开展。例如,以炎症性巨噬细胞为模型,研究者发现GPS2和SMRT辅助抑制因子与辅助激活因子CBP和Mediator拮抗调控增强子活性[51]。辅助抑制因子的结合可以阻止增强子活化、抑制eRNA的转录,当辅助抑制因子被敲低后,增强子eRNA的转录被重新激活[51]。在基因簇中,增强子CBSb位点附近结合RFX5、REST/NRSF等转录抑制因子[37,38]。因此,这些转录因子可能作为eRNA转录辅助抑制因子阻止增强子的转录,去除这些因子后可能会增加增强子活性。
本研究主要讨论了基因簇下游增强子产生的eRNA在基因表达调控方面发挥的激活功能,对已经报道的增强子eRNA的生物学功能是一种补充。通过检测增强子CBS位点编辑的细胞株中eRNA和基因簇的转录水平,以及利用CRISPR系统编辑增强子eRNA的转录起始位点区域,发现的转录水平下降会导致基因簇的转录水平显著降低。这些结果表明,增强子eRNA在调控基因簇的表达中发挥重要作用,为进一步理解eRNA的生物学功能提供帮助。
[1] Ong CT, Corces VG. Enhancer function: new insights into the regulation of tissue-specific gene expression., 2011, 12(4): 283–293.
[2] Buenrostro JD, Giresi PG, Zaba LC, Chang HY, Greenleaf WJ. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position., 2013, 10(12): 1213–1218.
[3] Heinz S, Romanoski CE, Benner C, Glass CK. The selection and function of cell type-specific enhancers., 2015, 16(3): 144–154.
[4] Banerji J, Rusconi S, Schaffner W. Expression of a-globin gene is enhanced by remote SV40 DNA sequences., 1981, 27(2 Pt 1): 299–308.
[5] Andersson R, Sandelin A. Determinants of enhancer and promoter activities of regulatory elements., 2020, 21(2): 71–87.
[6] Furey TS. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions., 2012, 13(12): 840–852.
[7] Korkmaz G, Lopes R, Ugalde AP, Nevedomskaya E, Han RQ, Myacheva K, Zwart W, Elkon R, Agami R. Functional genetic screens for enhancer elements in the human genome using CRISPR-Cas9., 2016, 34(2): 192–198.
[8] Simeonov DR, Gowen BG, Boontanrart M, Roth TL, Gagnon JD, Mumbach MR, Satpathy AT, Lee YJ, Bray NL, Chan AY, Lituiev DS, Nguyen ML, Gate RE, Subramaniam M, Li ZM, Woo JM, Mitros T, Ray GJ, Curie GL, Naddaf N, Chu JS, Ma H, Boyer E, van Gool F, Huang HL, Liu RZ, Tobin VR, Schumann K, Daly MJ, Farh KK, Ansel KM, Ye CJ, Greenleaf WJ, Anderson MS, Bluestone JA, Chang HY, Corn JE, Marson A. Discovery of stimulation-responsive immune enhancers with CRISPR activation., 2017, 549(7670): 111–115.
[9] Klann TS, Black JB, Chellappan M, Safi A, Song LY, Hilton IB, Crawford GE, Reddy TE, Gersbach CA. CRISPR-Cas9 epigenome editing enables high-throughput screening for functional regulatory elements in the human genome., 2017, 35(6): 561–568.
[10] Calo E, Wysocka J. Modification of enhancer chromatin: what, how, and why?, 2013, 49(5): 825–837.
[11] Andersson R, Gebhard C, Miguel-Escalada I, Hoof I, Bornholdt J, Boyd M, Chen Y, Zhao XB, Schmidl C, Suzuki T, Ntini E, Arner E, Valen E, Li K, Schwarzfischer L, Glatz D, Raithel J, Lilje B, Rapin N, Bagger FO, Jørgensen M, Andersen PR, Bertin N, Rackham O, Burroughs AM, Baillie JK, Ishizu Y, Shimizu Y, Furuhata E, Maeda S, Negishi Y, Mungall CJ, Meehan TF, Lassmann T, Itoh M, Kawaji H, Kondo N, Kawai J, Lennartsson A, Daub CO, Heutink P, Hume DA, Jensen TH, Suzuki H, Hayashizaki Y, Müller F, Forrest ARR, Carninci P, Rehli M, Sandelin A. An atlas of active enhancers across human cell types and tissues., 2014, 507(7493): 455–461.
[12] Kim TK, Hemberg M, Gray JM, Costa AM, Bear DM, Wu J, Harmin DA, Laptewicz M, Barbara-Haley K, Kuersten S, Markenscoff-Papadimitriou E, Kuhl D, Bito H, Worley PF, Kreiman G, Greenberg ME. Widespread transcription at neuronal activity-regulated enhancers., 2010, 465(7295): 182–187.
[13] Pefanis E, Wang JG, Rothschild G, Lim J, Kazadi D, Sun JB, Federation A, Chao J, Elliott O, Liu ZP, Economides AN, Bradner JE, Rabadan R, Basu U. RNA exosome- regulated long non-coding RNA transcription controls super-enhancer activity., 2015, 161(4): 774–789.
[14] Li WB, Notani D, Rosenfeld MG. Enhancers as non- coding RNA transcription units: recent insights and future perspectives., 2016, 17(4): 207–223.
[15] Li X, Fu XD. Chromatin-associated RNAs as facilitators of functional genomic interactions., 2019, 20(9): 503–519.
[16] Sartorelli V, Lauberth SM. Enhancer RNAs are an important regulatory layer of the epigenome., 2020, 27(6): 521–528.
[17] Struhl K. Transcriptional noise and the fidelity of initiation by RNA polymerase Ⅱ., 2007, 14(2): 103–105.
[18] Lai F, Orom UA, Cesaroni M, Beringer M, Taatjes DJ, Blobel GA, Shiekhattar R. Activating RNAs associate with Mediator to enhance chromatin architecture and transcription., 2013, 494(7438): 497–501.
[19] Hsieh CL, Fei T, Chen YW, Li TT, Gao YF, Wang XD, Sun T, Sweeney CJ, Lee GSM, Chen SY, Balk SP, Liu XS, Brown M, Kantoff PW. Enhancer RNAs participate in androgen receptor-driven looping that selectively enhances gene activation., 2014, 111(20): 7319–7324.
[20] Li WB, Notani D, Ma Q, Tanasa B, Nunez E, Chen AY, Merkurjev D, Zhang J, Ohgi K, Song XY, Oh S, Kim HS, Glass CK, Rosenfeld MG. Functional roles of enhancer RNAs for oestrogen-dependent transcriptional activation., 2013, 498(7455): 516–520.
[21] Xiang JF, Yin QF, Chen T, Zhang Y, Zhang XO, Wu Z, Zhang SF, Wang HB, Ge JH, Lu XH, Yang L, Chen LL. Human colorectal cancer-specificlncRNA regulates long-range chromatin interactions at thelocus., 2014, 24(5): 513–531.
[22] Bose DA, Donahue G, Reinberg D, Shiekhattar R, Bonasio R, Berger SL. RNA binding to CBP stimulates histone acetylation and transcription., 2017, 168(1–2): 135– 149.e22.
[23] Dorighi KM, Swigut T, Henriques T, Bhanu NV, Scruggs BS, Nady N, Still CD, Garcia BA, Adelman K, Wysocka J. Mll3 and Mll4 facilitate enhancer RNA synthesis and transcription from promoters independently of H3K4 monomethylation., 2017, 66(4): 568–576.
[24] Jia ZL, Wu Q. Clustered protocadherins emerge as novel susceptibility loci for mental disorders., 2020, 14: 587819.
[25] Wu Q, Jia ZL. Wiring the brain by clustered protocadherin neural codes., 2021, 37(1): 117–131.
[26] Wu Q, Maniatis T. A striking organization of a large family of human neural cadherin-like cell adhesion genes., 1999, 97(6): 779–790.
[27] Tasic B, Nabholz CE, Baldwin KK, Kim Y, Rueckert EH, Ribich SA, Cramer P, Wu Q, Axel R, Maniatis T. Promoter choice determines splice site selection in protocadherin α and γ pre-mRNA splicing., 2002, 10(1): 21–33.
[28] Wang XZ, Su H, Bradley A. Molecular mechanisms governing-γ gene expression: evidence for a multiple promoter and-alternative splicing model., 2002, 16(15): 1890–1905.
[29] Ribich S, Tasic B, Maniatis T. Identification of long-range regulatory elements in the protocadherin-gene cluster., 2006, 103(52): 19719–19724.
[30] Kehayova P, Monahan K, Chen WS, Maniatis T. Regulatory elements required for the activation and repression of the protocadherin-gene cluster., 2011, 108(41): 17195–17200.
[31] Guo Y, Monahan K, Wu HY, Gertz J, Varley KE, Li W, Myers RM, Maniatis T, Wu Q. CTCF/cohesin-mediated DNA looping is required for protocadherin α promoter choice., 2012, 109(51): 21081– 21086.
[32] Monahan K, Rudnick ND, Kehayova PD, Pauli F, Newberry KM, Myers RM, Maniatis T. Role of CCCTC binding factor (CTCF) and cohesin in the generation of single-cell diversity of Protocadherin-α gene expression., 2012, 109(23): 9125–9130.
[33] Zhai YN, Xu Q, Guo Y, Wu Q. Characterization of a cluster of CTCF-binding sites in a protocadherin regulatory region., 2016, 38(4): 323–336.
翟亚男, 许泉, 郭亚, 吴强. 原钙粘蛋白基因簇调控区域中成簇的CTCF结合位点分析. 遗传, 2016, 38(4): 323–336.
[34] Guo Y, Xu Q, Canzio D, Shou J, Li JH, Gorkin DU, Jung I, Wu HY, Zhai YN, Tang YX, Lu YC, Wu YH, Jia ZL, Li W, Zhang MQ, Ren B, Krainer AR, Maniatis T, Wu Q. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function., 2015, 162(4): 900–910.
[35] Lu YJ, Shou J, Jia ZL, Wu YH, Li JH, Guo Y, Wu Q. Genetic evidence for asymmetric blocking of higher-order chromatin structure by CTCF/cohesin., 2019, 10(12): 914–920.
[36] Jia ZL, Li JW, Ge X, Wu YH, Guo Y, Wu Q. Tandem CTCF sites function as insulators to balance spatial chromatin contacts and topological enhancer-promoter selection., 2020, 21(1): 75.
[37] Wang N, Jia ZL, Wu Q. RFX5 regulates gene expression of thecluster., 2020, 42(8): 760–774.
王娜, 甲芝莲, 吴强. RFX5调控原钙粘蛋白基因簇的表达. 遗传, 2020, 42(8): 760–774.
[38] Tang YX, Jia ZL, Xu HL, Da LT, Wu Q. Mechanism of REST/NRSF regulation of clustered protocadherin α genes., 2021, 49(8): 4506–4521.
[39] Canzio D, Nwakeze CL, Horta A, Rajkumar SM, Coffey EL, Duffy EE, Duffié R, Monahan K, O'Keeffe S, Simon MD, Lomvardas S, Maniatis T. Antisense lncRNA transcription mediates DNA demethylation to drive stochastic protocadherin α promoter choice., 2019, 177(3): 639–653.e15.
[40] Zhou YX, Xu SY, Zhang M, Wu Q. Systematic functional characterization of antisense eRNA of protocadherin alpha composite enhancer., 2021, 35(19–20): 1383–1394.
[41] Li JH, Shou J, Wu Q. DNA fragment editing of genomes by CRISPR/Cas9., 2015, 37(10): 992–1002.
李金环, 寿佳, 吴强. CRISPR/Cas9系统在基因组DNA片段编辑中的应用. 遗传, 2015, 37(10): 992–1002.
[42] Shou J, Li JH, Liu YB, Wu Q. Precise and predictable CRISPR chromosomal rearrangements reveal principles of Cas9-mediated nucleotide insertion., 2018, 71(4): 498–509.e4.
[43] Liu PF, Wu Q. Probing 3D genome by CRISPR/Cas9., 2020, 42(1): 18–31.
刘沛峰, 吴强. CRISPR/Cas9基因编辑在三维基因组研究中的应用. 遗传, 2020, 42(1): 18–31.
[44] Wu Q, Shou J. Toward precise CRISPR DNA fragment editing and predictable 3D genome engineering., 2021, 12(11): 828–856.
[45] ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome., 2012, 489(7414): 57–74.
[46] Gorkin DU, Barozzi I, Zhao Y, Zhang YX, Huang H, Lee AY, Li B, Chiou J, Wildberg A, Ding B, Zhang B, Wang MC, Strattan JS, Davidson JM, Qiu YJ, Afzal V, Akiyama JA, Plajzer-Frick I, Novak CS, Kato M, Garvin TH, Pham QT, Harrington AN, Mannion BJ, Lee EA, Fukuda- Yuzawa Y, He YP, Preissl S, Chee S, Han JY, Williams BA, Trout D, Amrhein H, Yang HB, Cherry JM, Wang W, Gaulton K, Ecker JR, Shen Y, Dickel DE, Visel A, Pennacchio LA, Ren B. An atlas of dynamic chromatin landscapes in mouse fetal development., 2020, 583(7818): 744–751.
[47] García-Muse T, Aguiléra A. R loops: from physiological to pathological roles., 2019, 179(3): 604–618.
[48] Niehrs C, Luke B. Regulatory R-loops as facilitators of gene expression and genome stability., 2020, 21(3): 167–178.
[49] Suzuki ST. Protocadherins and diversity of the cadherin superfamily., 1996, 109(Pt 11): 2609–2611.
[50] Rahnamoun H, Lee J, Sun ZX, Lu HB, Ramsey KM, Komives EA, Lauberth SM. RNAs interact with BRD4 to promote enhanced chromatin engagement and transcription activation., 2018, 25(8): 687–697.
[51] Huang ZQ, Liang N, Goñi S, Damdimopoulos A, Wang C, Ballaire R, Jager J, Niskanen H, Han HY, Jakobsson T, Bracken AP, Aouadi M, Venteclef N, Kaikkonen MU, Fan RR, Treuter E. The corepressors GPS2 and SMRT control enhancer and silencer remodeling via eRNA transcription during inflammatory activation of macrophages., 2021, 81(5): 953–968.e9.
Additional evidence ofenhancer eRNAfor protocadherin alpha gene regulation
Siyuan Xu, Jia Shou, Qiang Wu
The regulation of target genes by distal enhancers usually determines the fate and function of cells. Active enhancers in specific regions of chromatin may transcribe bidirectionally to produce long non-coding enhancer RNA (eRNA) to regulate gene expression. We recently found that an antisense enhancer eRNA(eRNA associated with R-loop formation) regulates gene expression of members of theclusterR-loop formation. To further explore the biological function of eRNA, we performed additional genetic and molecular experiments such as CRISPR (clustered regularly interspaced short palindromic repeats) DNA-fragment editing, RT-PCR, and qPCR. First, we performed expression analyses of theeRNAand found that it was expressed in a tissue-specific manner. In addition, upon CRISPR DNA-fragment deletion or inversion of the CTCF sites in theenhancer region, the expressionof eRNAwas reduced to 2%–10% and the expressionofgene cluster was also reduced to 13%–68% of the original levels. Finally, deletion of the bidirectional transcription start site (TSS) ofeRNA or inversion of TSS of the eRNAresulted in approximately 60% or 40% decrease of levels ofgene expression. In summary, these data suggested a functional role of theeRNA in gene regulation of thecluster, providing a new direction for future researches on the regulatory mechanisms of clusteredgene expression in the brain.
enhancer RNA; clustered protocadherin; eRNA; gene expression; gene regulation
2022-05-15;
2022-06-30;
2022-07-08
国家自然科学基金项目(编号:31630039, 91940303)和上海市科学技术委员会项目(编号:19JC1412500, 21DZ2210200)资助[Supported by the National Nature Science Foundation of China (Nos. 31630039, 91940303) and the Science and Technology Commission of Shanghai Municipality Program (Nos. 19JC1412500, 21DZ2210200)]
徐思远,在读硕士研究生,专业方向:生物学。E-mail: xus1yuan@126.com
吴强,博士,教授,研究方向:三维基因组学。E-mail: qiangwu@sjtu.edu.cn
10.16288/j.yczz.22-160
(责任编委: 单革)
