基于随机森林的ERα生物活性预测研究
2022-08-18马双宝
何 毅,马双宝,孙 彪
基于随机森林的生物活性预测研究
何 毅,马双宝*,孙 彪
(武汉纺织大学 机械工程与自动化学院,湖北 武汉 430200)
针对生物活性的检测速度慢且需耗费大量人力物力的问题,本文提出了基于随机森林的ERα生物活性预测模型。首先,对生物活性数据集进行数据清洗,使用3σ准则去除异常值。其次,利用随机森林重要变量重要度筛选出前20个对生物活性影响大的变量。随后,基于随机森林对筛选出的变量进行ERα生物活性预测。结果表明,所建立的模型的均方误差为0.017,具有良好的预测性能。
抗乳腺癌药物;生物活性;3σ准则;随机森林
乳腺癌是女性最常见的恶行肿瘤,其发病率与死亡率在女性癌症中均位居第一[1]。在对基因缺失小鼠的实验结果表明,确实在乳腺发育过程中扮演了重要的角色。目前,抗激素治疗常用于表达的乳腺癌患者,其通过调节雌激素受体活性来控制体内雌激素水平。因此,被认为是治疗乳腺癌的重要靶标,能够拮抗活性的化合物可能是治疗乳腺癌的候选药物。
如今在药物数量剧增的情况下,最经济合理的研究方式是利用计算机辅助的人工智能算法对药物活性进行预测分析[2]。谭露露[7]提出一种基于注意力机制的多特性融合方案,并结合边注意的图卷积网络,对不同种类的生物活性进行预测,但由于模型结构复杂,导致预测结果存在一定的过拟合问题。谢良旭[8]通过平均法、堆叠法融合浅层神经网络的模型融合方法来对药物分子进行预测。许美贤[9]提出一种基于PSO优化BP神经网络的生物活性预测模型,结果表明,所建立模型的预测准确度相较于优化前有所提升,但预测精度较低。综上所述,由于影响生物活性的特征因素较为繁杂,导致现有生物活性预测算法存在模型复杂、精度较低、泛化性能差等问题,针对这些问题,本文提出基于随机森林的生物活性预测模型,首先通过随机森林算法提取与生物活性相关性较高的特征因子,减少冗余因子,其次采用筛选后的高相关性样本数据对模型进行训练,进一步在保证检测精度的同时降低模型复杂度,使得模型具有良好的泛化性能。
1 ERα生物活性模型
1.1 随机森林算法
随机森林对噪声和异常值不敏感,容忍度较高,能够在不需要降维条件下处理具有高维特征的输入样本,同时随机森林可以评估各个特征在分类问题上的重要性,具有良好的可扩展性和并行性。
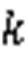
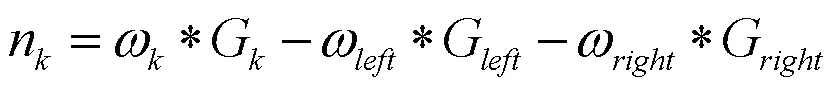
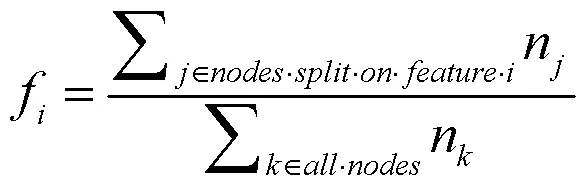
将对每一个特征重要性进行归一化处理以此来保证所有的特征处于同一量纲,归一化处理的公式如式(3)所示:
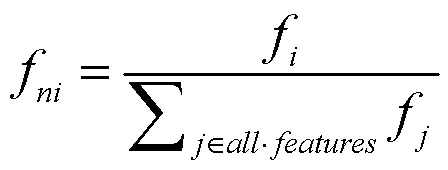
通过算法将数据处理好的362个变量进行贡献度排名,选出排名靠前的前20个变量作为模型的特征变量。贡献度排名情况如图1所示。
根据随机森林得出的变量贡献度排名可知不同变量对化合物的影响程度不同。为了确保研究的准确性,应该剔除不重要的变量和保留排名靠前的变量,从而保证所选取变量与因变量之间的相关性。同时可根据所提取的特征变量之间的距离相关系数来判断变量之间的相关程度,计算结果表明,20个变量之间相关性较低,独立性较好,从而不存在变量之间相互干扰的情况,保证了研究的客观性,为后续研究做好重要基础。
1.2 数据收集与处理
本文采用加拿大阿尔伯塔大学药物分子数据库中1974种化合物对的生物活性进行研究。但获取到的数据多而凌乱,无法开展研究,因此需要对数据进行处理,使其变成需要的样本数据。处理过程分为下四个步骤:
(1)如果一位点只含有少部分数据,大部分数据是残缺的,对于这种数据,应该舍弃;
(2)由第一步可得,在729个样本中只有小部分数据保留,而大部分残缺的数据将删除;
(3)被删去的那些数据,将用其前后两个小时数据的平均值代替;
(4)根据拉依达准则(3σ准则)筛选出异常值,然后去除。
拉依达准则又称为3σ准则,其基本原理是先假设一组数据只有随机误差,在此基础上,对它进行计算处理来得到其标准偏差,再按照一定的概率确定一个范围,在这个范围内的误差属于随机误差,不在此范围之内的数据就不属于随机误差,将在随机误差范围内的数据留下,将不在随机误差范围内的数据删除掉。
在正态分布里面,σ表示的标准差,µ表示均值,x=µ是图像的对称轴。3σ准则为:
数值分布在(µ-σ,µ+σ)的概率是0.6826;数值分布在(µ-2σ,µ+2σ)的概率是0.9544;数值分布在(µ-3σ,µ+3σ)的概率是0.9974。Y的取值有99.94%集中在(µ-3σ,µ+3σ)范围内,不在(µ-3σ,µ+3σ)区间内的可能性小于0.03%。
1.3 基于ERα生物活性预测模型的建立
整个模型的建立分为以下步骤:首先读取20个主要指标作为输入,1974个化合物作为输入,再对样本数据进行划分,80%作为测试集,20%作为训练集。然后采用Min-max标准化的方法对数据进行标准化,调整随机森林回归模型内置参数对训练集进行训练,得出基本模型。再用训练得出的模型对测试集的特征进行预测,得出生物活性的预测值,再将得出的生物活性预测值与原数据表中相对应的进行相减,得出两者差值平均值的绝对值,以及预测的准确率。最后,对训练的模型进行评估,得出随机森林回归算法的默认评估值。
Nestimators指定了弱分类器的个数,虽然设置的值越大,模型的准确度越高,但模型的训练时间也就越长。经过多次参数的调整,最终将指定值定为120时训练的效果最好。当random_state为0时,每次构建的模型不同;当random_state为3时,每次生成的数据集不一样;当random_state为42时,每次拆分出的训练集和测试集是不同的,对于本次模型的训练,偏向于设置为42,这是反复训练不同的测试集和训练集得出最好的结果。
2 结果分析及验证
模型预测完成后,将预测的生物活性预测值PIC50输出到表格中,与原表中相对应的生物活性实际值进行比较,如表1所示。
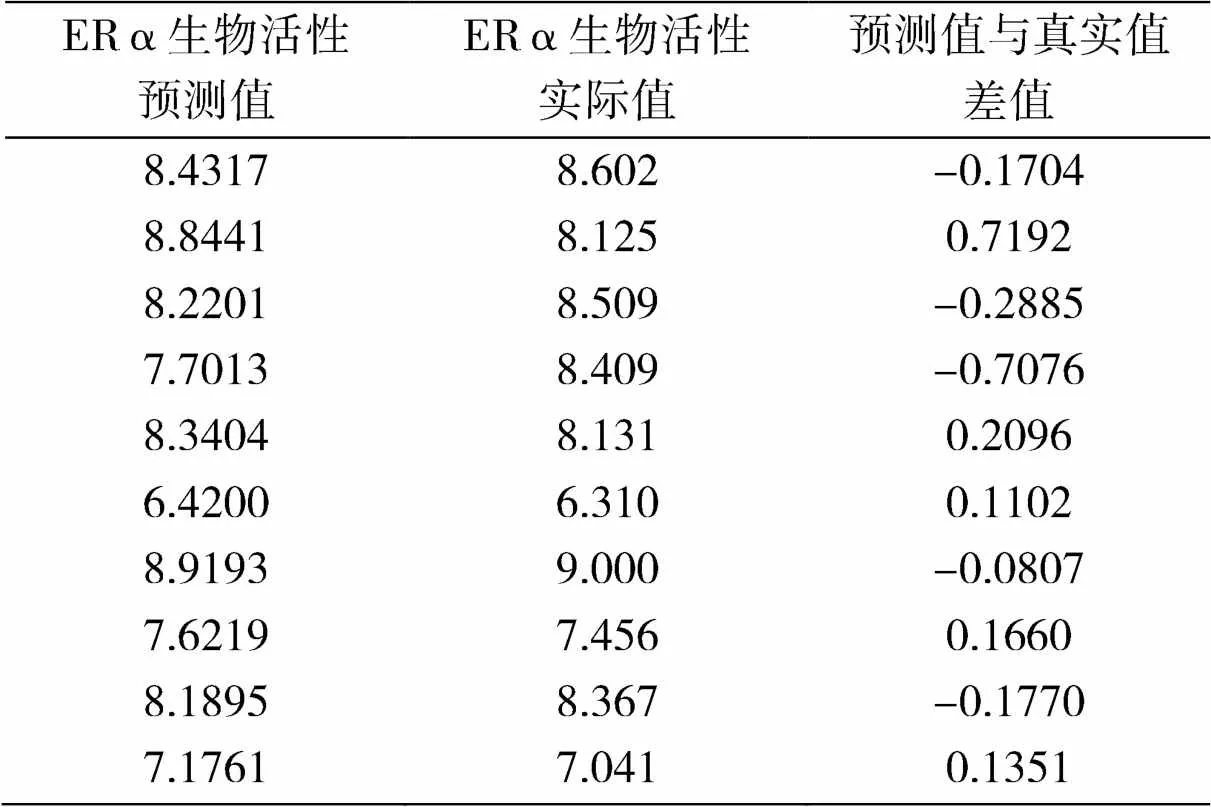
表1 部分预测值与真实值比较
均方误差即实际值与预测值差的平方和的平均值,即均方误差EMS为:
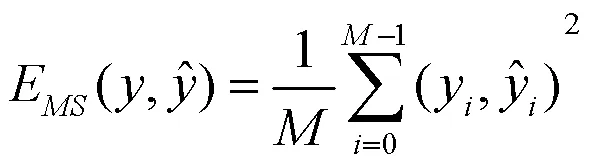
3 结论
本文首先通过数据清洗,筛选出异常值,使得样本更加合理。其次利用随机森林将影响生物活性贡献度排名前20的变量作为模型的特征变量,使得模型特征提取的效果更好,其次通过比较预测值和实际值,并对差值做散点图,得到模型的均方误差为0.017,说明模型具有很好的预测精度。由于本文使用的数据集偏小,预测结果不具有一定的泛化性,后面将采用更大的数据集,并优化算法,将其扩展到其他生物活性预测任务上。
[1] Pecero ML, Salvador-Bofill J, Molina-Pinelo S. Long non-coding RNAs as monitoring tools and the ERα peutic targets in breast cancer[J]. Cell Oncol, 2019, 42(1):1-12.
[2] 苏敏仪, 刘慧思, 林海霞, 等. 应用机器学习方法构建药物分子解离速率常数的预测模型[J]. 物理化学学报, 2020, 36(1): 179-187.
[3] Ding JJ, Xu Z, Zhang YY, et al. Exosome-mediated miR-222 transferring: an insight intoNF-κB-mediated breast cancer metastasis[J]. Exp Cell Res, 2018, 369(1): 129-138.
[4] 汤井田, 曹扬, 肖嘉莹, 等. 基于粒子群优化支持向量机的瑞芬太尼血药浓度预测模型[J]. 中国药学杂志, 2013, 48(16): 1394-1399.
[5] 刘雅琴, 王成, 章鲁. 基于神经网络的乳腺癌生存预测模型[J]. 中国生物医学工报, 2009, 28(2): 221-227.
[6] 袁仙琴. 基于基因表达数据的化合物肝毒性SVM预测模型研究[D]. 镇江: 江苏大学, 2018.
[7] 谭露露, 张鑫鑫, 周银座. 多特性融合图卷积方法的分子生物活性预测[J]. 电子科技大学学报, 2021, 50(06): 921-929.
[8] 谢良旭, 李峰, 谢建平, 等. 基于融合神经网络模型的药物分子性质预测[J]. 计算机科学, 2021, 48(09): 251- 256.
[9] 许美贤, 郑琰, 李炎举. 基于PSO-BP神经网络与PSO-SVM的抗乳腺癌药物性质预测[J/OL]. 南京信息工程大学学报(自然科学版), https://kns.cnki.net/kcms /detail/ 32.1801.N.20220117.1819.002.html.
Prediction of Bioactivity of ERα based on Random Forest
HE Yi, MA Shuang-bao, SUN Biao
(School of Mechanical Engineering and Automation, Wuhan Textile University, Wuhan Hubei 430200, China)
Aiming at the problem that the detection speed of ERα bioactivity is slow and requires a lot of manpower and material resources, this paper proposed a prediction model of ERα bioactivity based on random forest. First, the ERα bioactivity dataset was cleaned and outliers were removed using the 3σ criterion. Secondly, the importance of random forest important variables was used to screen out the top 20 variables with great influence on biological activity. Then, ERα bioactivity of selected variables was predicted based on random forest. The results show that the mean-square error of the model is 0.017, and it has good prediction performance.
anti breast cancer drugs; biological activity; 3σ criteria; random forest
TP311
A
2095-414X(2022)04-0054-03
通讯作者:马双宝(1979-),男,副教授,博士,研究方向:微弱信息检测与电力控制系统设计.
