大菱鲆源荧光假单胞菌群体感应淬灭酶基因的克隆及生物信息学分析
2022-08-17崔方超李婷婷王当丰刘景云谭茜倩吕欣然励建荣
申 悦,崔方超*,李婷婷,王当丰,刘景云,谭茜倩,吕欣然,励建荣
(1 渤海大学食品科学与工程学院 生鲜农产品贮藏加工及安全控制技术国家地方联合工程研究中心 辽宁锦州 121013 2 大连民族大学生命科学学院 辽宁大连 116600)
群体感应是细菌间进行信息交流的一种方式,通过分泌信号分子来达到交流的目的,从而调控相应功能基因的表达,使细菌表现出一些表型特征,如毒力因子的分泌、生物被膜的形成、细菌运动性和扩散性的增强等[1]。这些行为一方面能够增强细菌自身的防御能力,提高生存能力,另一方面会导致一些病原菌毒力因子和致病基因的表达[2]。群体感应系统存在于许多水产腐败菌中,能够参与并调控水产品的腐败进程,如胞外蛋白酶的产生、生物被膜的形成、黏液的分泌等,对水产品的品质产生严重的影响。温和气单胞菌是鱼类等水产品低温贮藏过程中常见的腐败菌之一,能够产生AHLs 来调控其生物被膜的产生,蛋白酶的表达和群集泳动[3]。大量化学保鲜剂的使用使细菌出现耐药性,增加了水产品食用的危险。寻找安全的群体感应抑制剂(Quorum sensing inhibitors,QSIs)来干扰或破坏其参与调控的生物学功能,可在非致死条件下阻断依赖QS 的细菌侵染,并抑制相关致腐因子的分泌,从而为解决细菌耐药性,控制水产品品质变化,延长水产品的货架期提供新的途径[4]。
群体感应淬灭(Quorum quenching,QQ)通过抑制信号分子的合成和积累,或对信号分子进行降解和修饰,以此破坏细菌群体感应系统,从而阻断腐败菌或致病菌带来的不利影响[5]。群体感应淬灭酶(Quorum quenching enzymes)能利用群体感应中的信号分子AHLs 作为底物,通过酶促反应使AHLs 分解[6]。根据淬灭酶催化机制的不同,可分为内酯酶、酰化酶和氧化还原酶。内酯酶水解AHLs 内酯环的酯键,酰化酶水解酰基骨架,氧化还原酶可以催化氧化或还原AHLs。其中AHL 酰化酶能以不可逆的方式破坏高丝氨酸内酯部分和酰基链之间的酰胺键,释放出游离高丝氨酸内酯(HSL)和相应的脂肪酸[7]。AHL 酰化酶广泛存在于各种细菌中,包括革兰氏阴性菌和革兰氏阳性菌,如铜绿假单胞菌 (Pseudomonas aeruginosa)、Anabaena sp.PCC7120和青枯菌(Ralstonia solanacearum),然而其底物特异性因AHL 的不同酰基链取代而不同。据报道铜绿假单胞菌PAO1中存在2 种酰基转移酶PvdQ 和QuiP,两者都可以编码AHL 酰化酶,并降解3O-oxo-C12-HSL[8]。Rodrigo 等[9]发现薰衣草链霉菌(Streptomyces lavendulae)产生的AHL 酰化酶SlPA,对含有较长酰基链的AHLs 催化效率更高。Ivanova 等[10]发现酰化酶能够限制铜绿假单胞菌的生物膜形成。在活体动物模型中对该酶进行评估,结果该酶可将生物膜的形成延迟7 d。Kusada 等[11]从嗜酸菌MRS7 中分离并鉴定一种新的AHL 酰化酶(MacQ),该酶具有降解多种AHLs 的能力,包括C6到C14侧链,并且内酯环上3 号碳原子的氧取代不影响该酶的降解能力。
荧光假单胞菌 (Pseudomonas fluorescens)是水产品贮藏过程中常见的嗜冷菌,属于好氧或兼性厌氧的革兰氏阴性细菌。由于其适应性强,生长周期短,胞外酶活性高,被公认为动物和水产品中的主要腐败菌[12]。本实验室从腐败大菱鲆中分离出一株荧光假单胞菌。在该菌株中发现pf-1240基因编码的蛋白序列与QuiP 蛋白的氨基酸序列一致性达65.82%。有文献报道,QuiP 可降解AHLs,推测荧光假单胞菌PF-1240 蛋白可能也具有降解AHLs 的活性,可抑制细菌间的群体感应现象,从而抑制腐败菌的腐败特性,达到延缓水产品腐败的效果。迄今为止,还没有关于从荧光假单胞菌中克隆AHL 降解酶的报道。本研究以荧光假单胞菌为研究对象,从其全基因组中克隆pf-1240基因,并分析其生物信息学特征,以期为后续揭示该酶淬灭AHLs,抑制腐败菌群体感应现象提供理论参考。
1 材料与方法
1.1 菌株来源
供试菌:荧光假单胞菌 (Pseudomonas fluorescens)PF08 从腐败大菱鲆中分离、鉴定,保藏于渤海大学食品科学学院水产品贮藏与加工研究所。
1.2 主要试剂和仪器
LB 肉汤,青岛海博生物技术有限公司;50×TAE 缓冲液、琼脂糖、DNA 分子量标准Maker(100~5 000 bp)、DiaSpin 柱式PCR 产物纯化试剂盒、Gold view 核酸染色剂,上海生工生物工程有限公司;异丙基硫代半乳糖苷(IPTG)、X-Gal、氨苄青霉素 (Amp),北京索莱宝科技有限公司;PrimeSTARRMax DNA Polymerase,北京宝生物生物技术有限公司;Mighty TA-cloning Reagent Set for PrimeSTARR试剂盒,大连Takara 公司。
DL-CJ-2N 超级洁净工作台,北京市东联哈尔仪器制造有限公司;ABI stepone plus PCR 仪,德国Eppendorf 公司;DYY-8C 电泳仪,北京市六一仪器厂;Quantity One 凝胶成像系统,美国Bio-Rad 公司;LDZX-50KB 立式压力蒸汽灭菌器,上海申安医疗器械厂;PE Victor X3 酶标仪,美国Perkin Elmer 公司。
1.3 试验方法
1.3.1 基因组DNA 的提取及目的基因的扩增 将PF08 从-80 ℃超低温冰箱中取出,按1∶100 的体积比接种于新鲜LB 肉汤液体培养基中,在28℃,160 r/min 条件下,过夜培养12~16 h,继续用相同的方法对菌株进行传代,待菌株生长到OD595nm为0.6 时备用。
采用加热法快速提取DNA,取1 mL 菌液加入1.5 mL 离心管中8 000×g 室温离心5 min 后,弃上清,将菌体溶解于100 μL 无菌水中,95 ℃加热10 min,再次离心,所用的DNA 即溶解于上清中。
根据NCBI 中报道的该菌株的青霉素酰化酶基因序列(WP_017137176.1 AYG06728.1)利用生工在线工具(http://www.sangon.com/newPrimerDesign)进行扩增引物的设计,并送至上海生工生物工程股份有限公司合成。

表1 荧光假单胞菌pf-1240 基因引物序列Table 1 The primers of pf-1240 gene of Pseudomonas fluorescens
PCR 扩增反应体系为:DNA 模板1.0 μL,PrimeSTARRMax DNA Polymerase 25 μL,ddH2O 22.4 μL,上游引物0.8 μL,下游引物0.8 μL,总体积50 μL。PCR 扩增反应程序:95 ℃,预变性4 min;98 ℃,变性10 s;65 ℃,退火18 s;72 ℃,延伸2 min;72 ℃,保持5 min;循环25 次,4 ℃保温。
PCR 产物经1.0%琼脂糖凝胶电泳,观察凝胶成像系统的扩增效果并照相。
1.3.2 目的基因的纯化及TA 克隆测序 利用DiaSpin 柱式PCR 产物纯化试剂盒,对扩增的目的基因进行纯化,产物经1.0%琼脂糖凝胶电泳。确定纯化成功后,进行加“A”反应,反应液立即与pMD20-T 载体连接,而后转化到E.coli JM109感受态细胞中,在SOC 培养基中37 ℃培养1 h后,涂布到选择培养基上,挑选白色菌落进行阳性鉴定,具体步骤参照梅永超[13]的方法,鉴定成功后,送去北京天一辉公司进行测序。
1.4 pf-1240 基因序列生物信息学分析
利用纽普生物在线DNA 序列翻译成氨基酸序列的网站(https://www.novopro.cn/tools/translate.html)。将pf-1240 基因序列上传到网站中,选择阅读框1 为翻译起始位置,序列顺序为正向,选用standard(1)模式进行翻译,翻译结果用于下一步分析。将氨基酸序列导入ProtParam 在线软件(https://web.expasy.org/protparam/) 对pf-1240 编码的蛋白进行理化性质预测和氨基酸组成分析,得出该蛋白的分子质量、分子式、等电点、氨基酸组成等理化性质。利用ProtScale(https://web.expasy.org/protscale/)分析PF-1240 蛋白亲/疏水性,打开网址后,将蛋白序列输入对话框内,选择Hphob./ Kyte &Doolittle 即可分析出蛋白的亲/疏水性;利用Net NGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc/)对蛋白的N-糖基化位点进行预测,用NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)对蛋白的磷酸化位点进行预测;采用TMHMM Server v.2.0 预测蛋白跨膜区域;用CDD 工具选择CD-Search(https://www.ncbi.nlm.nih.gov/cdd/) 分析蛋白的结构域[14];采用SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html) 和COILS server (https://embnet.vital-it.ch/software/COILS_form.html) 对蛋白二级结构及卷曲螺旋结构进行分析[15-16];利用Protcomp 9.0 软件对蛋白的亚细胞定位进行分析[17];利用Discovery Studio(DS)对PF-1240 蛋白进行三级结构建模[18]。利用NCBI 在线软件比对功能 (BLAST:Basic Local Alignment Search Tool)对PF-1240 蛋白的氨基酸序列进行蛋白质同源性分析。之后选择同源性高的9 个氨基酸序列在MEGAX 软件运用最小邻距离法 (Neighbor-Joining,NJ) 并选用自长支持率(Bootstrap)1 000 次重复构建系统进化树,对PF-1240 蛋白进行同源性分析。
2 结果与分析
2.1 荧光假单胞菌PF08 的pf-1240 基因PCR扩增、克隆
以PF08 的全基因组DNA 为模板,PCR 扩增产物的琼脂糖凝胶电泳如图1a 所示。其中泳道1为扩增产物,可以看出扩增出的条带在2 000~3 000 bp 之间,与目的基因的大小相符,初步确定扩增出了与预期大小一致的片段。将上述PCR 产物进行纯化,纯化结果见图1a 中泳道2,能够看出与纯化前的条带大小一致,可以用于下一步试验。
将转化后的转化子涂布于选择性培养平板上,长出的菌落如图1b 所示,可观察到其分为蓝色和白色,其中蓝色菌落应为空载体转化子,而白色菌落应为重组载体转化子即阳性克隆子。随机挑选10 个阳性克隆子,进行PCR 扩增验证,结果如图1c 显示。10 个扩增条带大小符合预期且清晰无杂带,说明挑取的阳性克隆子全部成功导入了目的基因,下一步即可将阳性克隆子送去测序。
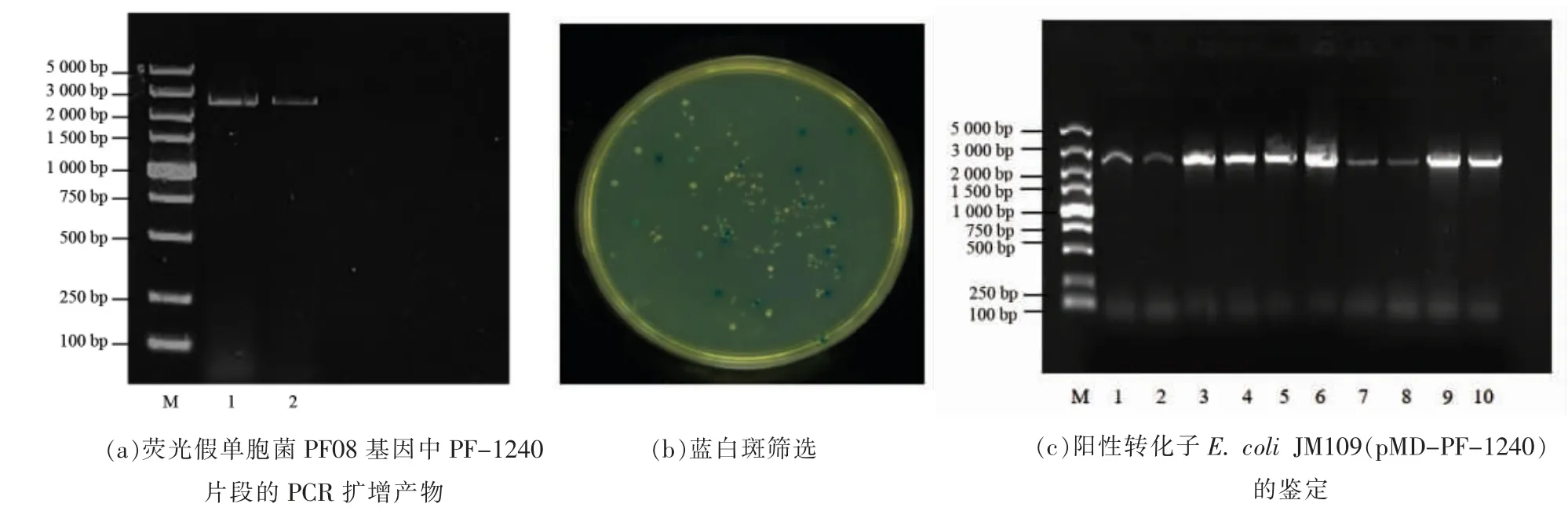
图1 目的基因的克隆Fig.1 Cloning of target gene
2.2 测序结果及序列比对
通过TA 克隆测序,得到了2 389 bp 的pf-1240 基因(WP_120731947.1)全长。与NCBI 数据库中荧光假单胞菌青霉素酰化酶家族基因(Gene ID:61637474)的相似性为83.90%。
2.3 pf-1240 基因的序列分析
蛋白质的理化性质是蛋白质研究的基础。经ExPaSy PortParam 在线软件预测显示荧光假单胞菌pf-1240 基因共编码795 个氨基酸,推测对应的蛋白分子式为C3867H6010N1084O1160S19,相对分子质量为86 855.96,理论等电点(pI)为7.81,不稳定系数为35.29,说明该蛋白质是一个稳定的蛋白(>40为不稳定蛋白),脂肪指数为79.65,总平均亲水性为-0.344,蛋白总平均亲水系数为负值,属于亲水蛋白。氨基酸疏水性是氨基酸的一种基本性质,疏水性氨基酸位于蛋白质内部,由于其疏水性的相互作用,故在保持蛋白质三级结构上起作用。图3中的横坐标表示氨基酸位置,纵坐标表示得分,Score 值小于0 表示亲水,大于0 表示疏水。其氨基酸最大疏水性位置为163 位亮氨酸,最大值为2.400,第153 位脯氨酸出现最小值为-2.633。氨基酸组成如表2所示,其中含量最高的是丙氨酸(Ala),占11.84%。
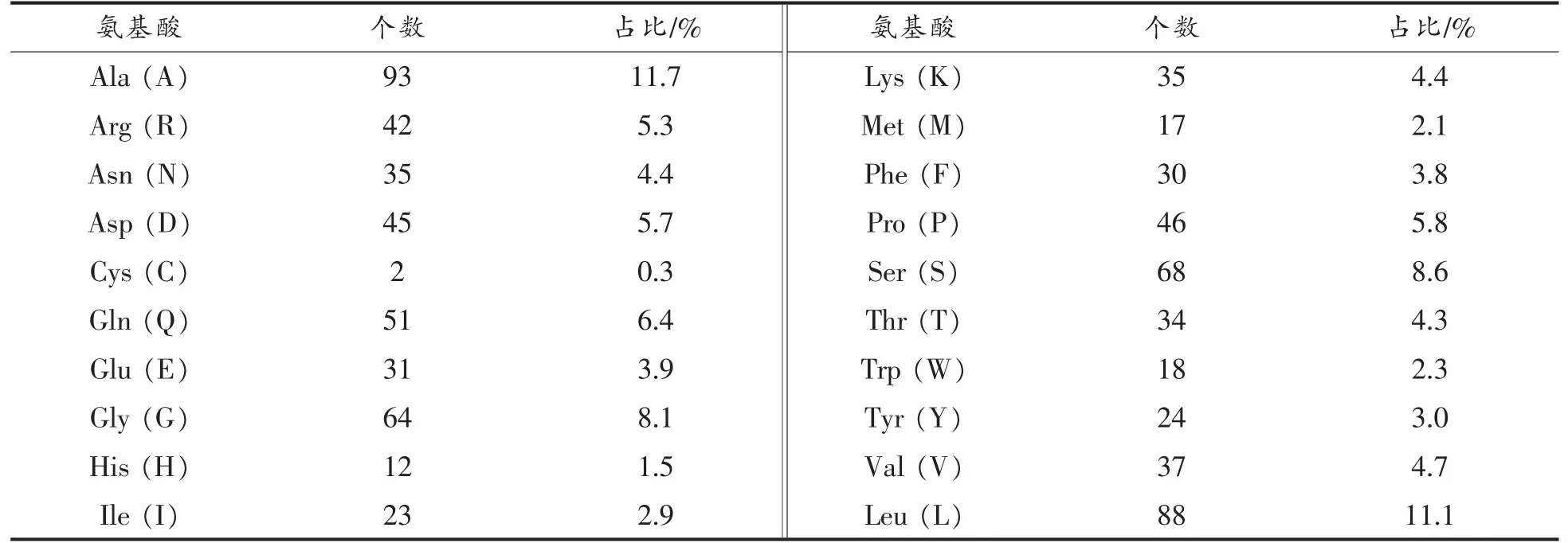
表2 荧光假单胞菌PF-1240 蛋白的氨基酸组成Table 2 Amino acid composition of Pseudomonas fluorescens PF-1240 protein
图2 pf-1240 基因测序结果Fig.2 Sequencing results of pf-1240 gene
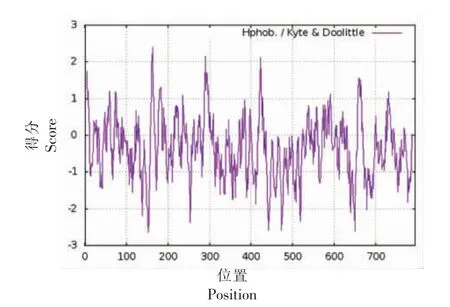
图3 氨基酸亲疏水性分析Fig.3 Hydrophilic-hydrophobic property analysis of amino acid
2.4 PF-1240 蛋白信号肽和跨膜结构的预测
通过SignalP-5.0 Server 分析PF-1240 氨基酸序列预测信号肽。如图4所示,其中SP 表示由Sec 易位子转运并被信号肽酶I(Lep)切割的“标准”分泌信号肽;TAT 表示由双精氨酸转Tat 易位子转运并被信号肽酶I(Lep)切割的Tat 信号肽;LIPO 表示由Sec 易位子转运并被信号肽酶II(Lsp) 切割的脂蛋白信号肽。预测结果显示PF-1240 蛋白可能存在脂蛋白信号肽,存在的概率为0.4521,裂解位点在第9 和第10 个氨基酸之间,这与已有的研究有所不同。已有的研究中表明青霉素酰化酶(Penicillin G acylase,PGA)翻译后利用双精氨酸易位(Tat)机械将前体易位至周质膜,然后进行自催化分子内肽键裂解[19]。跨膜结构域是指蛋白质序列中跨越细胞膜的区域,通常为螺旋结构,含20~25 个氨基酸残基,构成其蛋白质的氨基酸大多数为疏水性氨基酸,运用TMHMM Server.v.2.0 对荧光假单胞菌PF-1240 蛋白跨膜结构进行预测,结果如图5所示,整条多肽链均处于细胞膜外,说明该蛋白不存在跨膜结构。这与目前已有研究的鱼腥藻AiiC 酰化酶相似,该酶是由2 544 bp 基因编码847 个氨基酸的蛋白质,N 末端区域存在1 个信号肽和1 个假定的跨膜结构域,表明AiiC 定位在周质中,其酰化酶的活性主要在菌体裂解后的细胞碎片中发挥作用[20]。
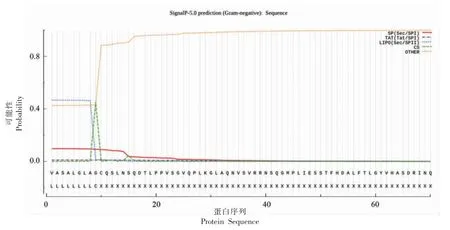
图4 PF-1240 蛋白信号肽预测Fig.4 Prediction of signal peptide of PF-1240 protein
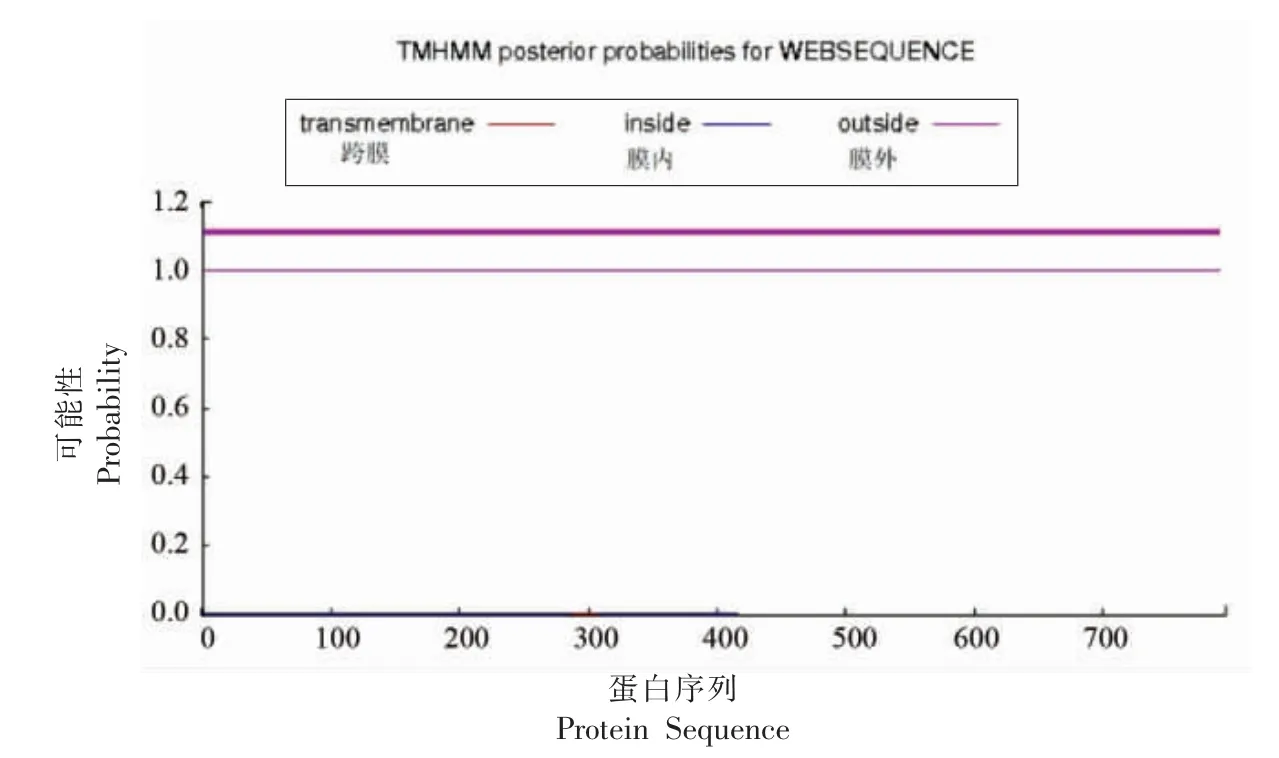
图5 PF-1240 蛋白跨膜区预测Fig.5 Prediction of protein transmembrane regions of PF-1240 protein
2.5 PF-1240 蛋白翻译后修饰
蛋白质的翻译后修饰 (Post-translational modification,PTM)是指对翻译后的蛋白质进行共价加工的过程,通过在1 个或多个氨基酸残基加上修饰基团,可以改变蛋白质的理化性质,进而影响蛋白质的空间构象和活性状态、亚细胞定位、折叠及其稳定性以及蛋白质-蛋白质相互作用,是调节蛋白质功能的重要方式[21]。蛋白质翻译后修饰使得蛋白具有特定功能,常见的蛋白质翻译后修饰包括泛素化、磷酸化、糖基化、甲基化、乙酰化、酯基化等。糖基化(Glycosylation)是指蛋白质或脂质在酶的作用下被连接上糖链的过程。作为生物体内最为重要的蛋白质翻译后修饰形式之一,糖基化调控了蛋白质在组织和细胞中的定位、功能、活性、寿命和多样性[22]。在此,运用NetNGlyc 1.0 对PF-1240 蛋白糖基化位点进行分析,结果见图6。PF-1240 蛋白N-糖基化位点有5 个,分别位于第34,111,166,346,623 位氨基酸。
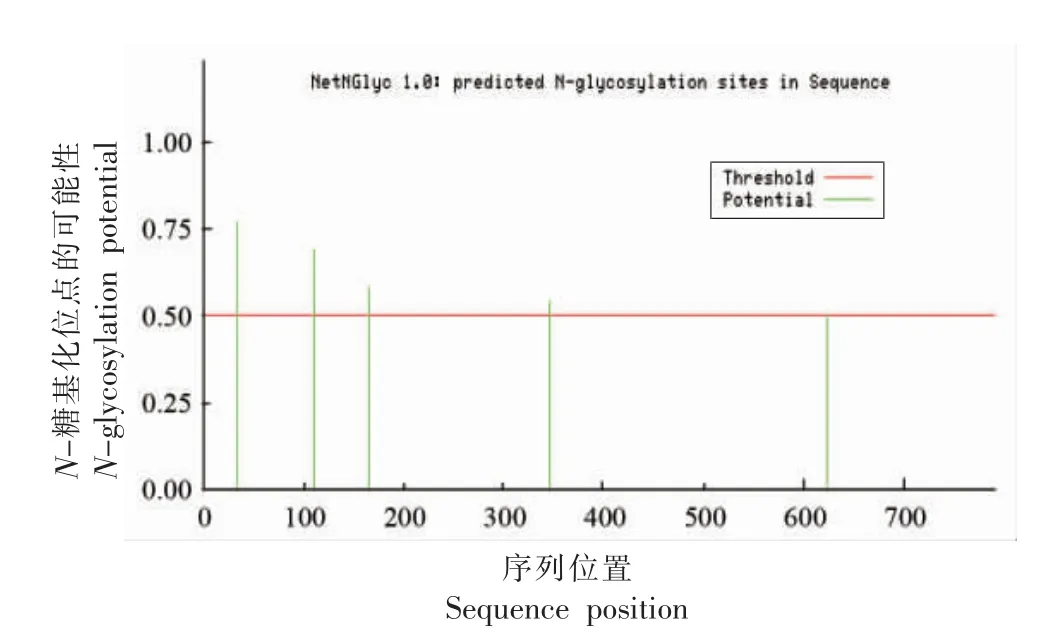
图6 PF-1240 蛋白糖基化位点分析Fig.6 Protein glycosylation site analysis of PF-1240 protein
运用NetPhos 3.1 Server 在线软件预测蛋白磷酸化位点,结果如图7所示。该蛋白序列中有79 个潜在磷酸化位点,其中50 个丝氨酸磷酸化位点,19 个苏氨酸磷酸化位点,10 个酪氨酸磷酸化位点。磷酸化是蛋白质最重要的翻译后修饰之一,对蛋白质发挥信号传导作用,具有重要意义[23]。
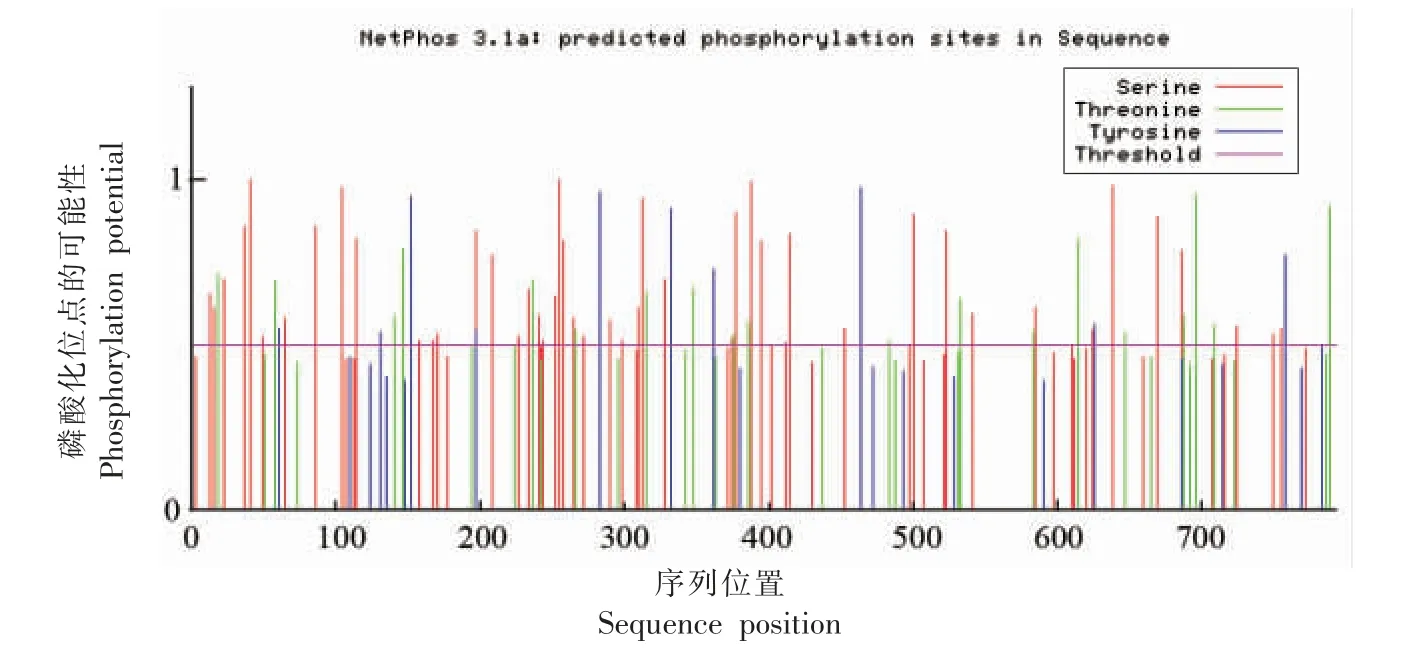
图7 PF-1240 蛋白磷酸化的分析Fig.7 Protein phosphorylation site analysis of PF-1240 protein
2.6 PF-1240 蛋白亚细胞定位
亚细胞定位是指某种蛋白或表达产物在细胞内的具体存在部位。例如:在核内、胞质内或者细胞膜上存在。亚细胞定位对于蛋白质功能非常重要,蛋白质必须处于合适的亚细胞定位才能行使其功能[24]。通过在线软件PSORTb v.3.0 分析预测PF-1240 蛋白的亚细胞定位结果如表3,该蛋白主要分布在细胞周质。因此,该蛋白主要在细胞周质发挥作用。与其它酰化酶类似,通过翻译表达到胞外,与周围环境中的AHLs 发生作用。

表3 蛋白亚细胞定位预测Table 3 Predicted of subcellular location of protein
2.7 PF-1240 蛋白开放阅读框及保守结构域分析
通过https://www.ncbi.nlm.nih.gov/orffinder/在线软件,预测了PF-1240 蛋白的开放阅读框,如图8 所示。该开放阅读框由791 个氨基酸组成,与荧光假单胞PF01 的QuiP 序列(Genebank ID:Q3KH00.1)的一致性为100%,证明该蛋白属于群体感应信号分子淬灭酶的一种,与QuiP 具有类似的功能。
图8 PF-1240 蛋白开放阅读框预测Fig.8 Prediction of PF-1240 protein open reading frame
AHL 酰化酶是一种AHL 降解酶,通过水解酰基侧链和HSL 部分之间的酰胺键来切割AHL 分子,其方式与青霉素酰化酶的作用方式非常相似。保守结构域在基因功能中发挥重要作用。NCBI 的结构域预测(图9)显示该肽链中含有1 个保守结构域,氨基酸第35~779 位为Penicil_amidase 结构域,E-value 为0,证明其结构预测可靠,说明在这段序列中有一个完整的青霉素酰胺酶结构域,在这个结构域中只有1 个丝氨酸催化中心。通过在线程序InterPro 对蛋白保守结构域进行分析,有6个蛋白条目与目的蛋白匹配成功(图10)。其中匹配最成功的条目是IPR002692:Penicillin/GL-7-ACA/AHL/aculeacin-A acylase。数据库定义这些家族中的蛋白代表了一些酰化酶,它们都属于MEROPS 肽酶家族S45,是PGA 前体和头孢菌素酰化酶前体,也是一类自发形成成熟酰化酶异二聚体的自裂解蛋白质。该家族包括AHL 酰化酶PvdQ 和QuiP,以及已证明具有青霉素酰化酶活性的ACULACIN-A 酰化酶[25]。目的蛋白从第5 个到792 个氨基酸与IPR002692 蛋白相一致,第31 个到788 个氨基酸与IPR029055 (N 端亲核氨基水解酶)同源超家族蛋白序列相匹配,该家族中的蛋白利用氨基末端残基的侧链,结合在β 折叠上,作为亲核分子在羰基碳上的催化攻击。这一家族的一个重要成员嗜黄菌的PGA 被证明能够降解链长为6~8 个碳的AHLs[26]。通过详细的结构研究,已经证实AHL 酰化酶与所有其它青霉素酰化酶属于同一个Ntn 水解酶超家族[27],根据保守结构域的分析结果,可以推测出PF-1240 蛋白属于PGA,具有酰化酶的功能。
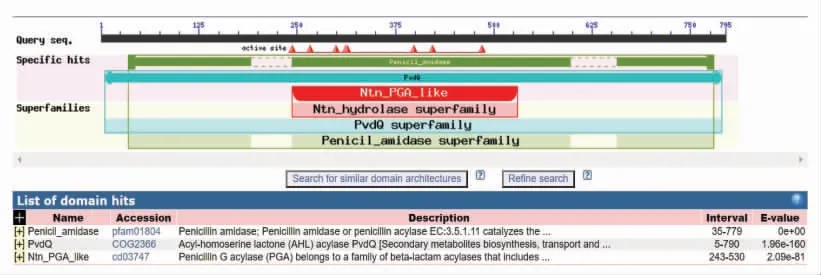
图9 PF-1240 蛋白的结构域预测Fig.9 Structural domain prediction of PF-1240 protein
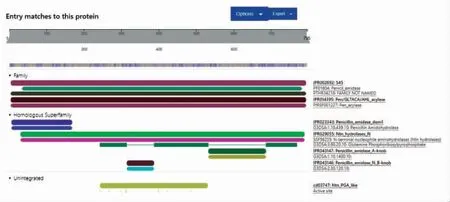
图10 PF-1240 蛋白保守结构域匹配序列Fig.10 PF-1240 Protein conserved domain matching sequence
2.8 PF-1240 蛋白二级结构特征
通过SOPM 程序预测蛋白的二级结构,如图11所示。其中α-螺旋区域有344 个氨基酸,占42.01%,β 折叠有70 个氨基酸,占8.81%,无规则卷曲有290 个氨基酸,占36.48%,延伸链区有101个氨基酸,占12.7%。从蛋白质的整体结构来看,无规则卷曲和α-螺旋是PF-1240 蛋白的主要结构元件,分散于整条多肽链;而延伸链和β-折叠相对较少,其中2 个明显的α-螺旋分别位于两端。使用COILS server 预测了蛋白的卷曲螺旋结构,结果如图12,显示了在 3 种窗口宽度下的检测值均较低,表明蛋白中可能不存在卷曲螺旋结构。

图11 PF-1240 蛋白二级结构预测Fig.11 Prediction of PF-1240 protein secondary structure
图12 PF-1240 蛋白卷曲螺旋结构预测Fig.12 Prediction of PF-1240 protein coil structure
2.9 PF-1240 蛋白的生物进化分析
依据PF08 的pf-1240 基因编码的蛋白序列以及对其潜在功能的预测,在NCBI 中进行Blast同源性检索,获取多个菌株的PF-1240 同源蛋白序列进行多序列比对,包括链霉菌(Strepyomyces sp.)的 AhlM、铜绿假单胞菌的PvdQ、PA0305 和QuiP、丁香假单胞菌 (Pseudomonas syringae)的Psyr_1971 和Psyr_4858、希瓦氏菌(Shewanella sp.)的Aac,苍白杆菌(Bacillus pallidus)A44 编码aiiO 的AHL 酰化酶,R.alstonia sp.XJ12B 的aiiD,并用MEGA-X 软件邻位相连法 (Neighborjoining)构建系统进化树,结果证明荧光假单胞菌PF-1240 蛋白与铜绿假单胞菌的QuiP 氨基酸序列同源性较高,为65.82%,进化关系最为接近,且在其它物种中也具有很高的保守性。
2.10 PF-1240 蛋白的三级结构预测建模
通过 Discovery Studio (DS) 的 Homology Modeling 方法自动预测蛋白质的三级结构,选用4e56(间隔区缩短型头孢菌素酰化酶突变体)为模板,通过DS 序列的比对,对序列进行适当修改,构建出PF-1240 蛋白的三级结构,如图14所示。青霉素酰化酶家族蛋白的前体包含了信号肽、中间肽、α 亚基和β 亚基。成熟的蛋白是由α 亚基和β亚基组成的二聚体,β 亚基的第1 个氨基酸丝氨酸是1 个保守的活性位点。从图中可看出PF-1240 是由2 个不同大小的亚基组成,黄色部分为保守的丝氨酸活性位点。

图13 PF-1240 蛋白系统进化树Fig.13 Phylogenetic tree of PF-1240 protein
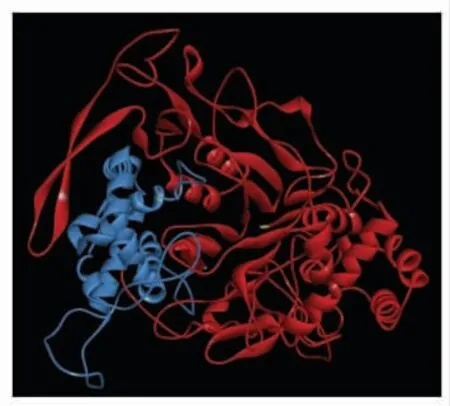
图14 PF-1240 蛋白三级结构Fig.14 The tertiary structure of PF-1240 protein
3 结论
本研究从1 株大菱鲆源荧光假单胞菌中成功克隆得到全长2 235 bp 的pf-1240 基因,又通过生物信息学分析进一步确定了该蛋白的理化性质。PF-1240 蛋白序列包含795 个氨基酸,预测氨基酸相对分子质量为86 855.96,理论等电点pI 为7.81,α-螺旋占42.01%,β-折叠占8.81%。第35 个氨基酸到779 个为Penicil_amidase 结构域,说明该蛋白属于青霉素酰化酶家族蛋白,具有水解酰胺键的活性,能够催化N-酰基高丝氨酸内酯和酰基链之间的酰胺键断裂。铜绿假单胞菌产生的QuiP 是铜绿假单胞菌基因PA1032 的产物,作为第2 个AHL 酰化酶,能够灭活铜绿假单胞菌的群体信号分子[28],与PF-1240 蛋白的相似性为65.82%。Huang 等[29]发现QuiP 在铜绿假单胞菌PAO1 中的表达后,导致菌株PAO1 中3-O-C12-HSL 信号分子的积累减少,表明该酶对长链AHL具有活性。
由于群体感应是存在于微生物中独特的调控机制,从而使其成为人为调控微生物行为的潜在靶点,能够在食品保鲜、生物发酵等领域显现出应用潜力。随着细菌群体感应现象研究的不断深入,大量的群体感应淬灭酶被发现,越来越多的淬灭酶结构和功能被解析出来。通过DS 同源建模,能够对蛋白三维空间有初步了解,为后续的试验研究提供指导性意见。AHL 酰化酶通过降解革兰氏阴性菌群体感应AHL,从而调控毒力因子的形成。通过不断的对其进行深入研究和分析,对人为控制细菌群体感应造成的水产品腐败具有非常诱人的应用前景。
