中国美利奴羊(新疆型)羊毛长度与产毛量的全基因组关联分析
2022-08-09徐新明哈妮克孜吐拉甫刘剑锋付雪峰于丽娟拉扎特艾尼瓦尔
徐新明,哈妮克孜·吐拉甫,刘剑锋,石 刚,付雪峰,于丽娟,拉扎特·艾尼瓦尔,狄 江
(1新疆畜牧科学院,新疆 乌鲁木齐 830000;2中国农业大学 动物科技学院,北京 100193)
羊毛性状,如羊毛产量、毛纤维长度、毛丛强力和纤维直径,是毛用羊非常重要的选育指标[1],受遗传和环境因素的影响。羊毛的生长过程与毛囊的发育[2]和毛囊生长周期[3-4]及毛囊细胞分化[5]有关。羊毛的生长发育在分子水平上受相关基因表达的调控,重要基因的突变会影响羊毛的生长,从而造成羊毛性状的差异。从遗传学角度分析,筛选与羊毛性状相关的候选基因是毛用羊分子育种的关键步骤[6]。然而,在最新的绵羊数量性状基因座数据库(http://www.animalgenome.org/cgi-bin/QTLdb/OA/index)中,只有54个数量性状基因座(quantitative trait locus,QTL)与羊毛性状相关。此外,QTL的置信区间较长,很难用其来确定影响目标性状的特定基因。因此,在绵羊的育种研究中,迫切需要从基因组水平挖掘更多与羊毛性状密切相关的单核苷酸多态性(single nucleotide polymorphism,SNP)位点,并对SNP进行更为精确的QTL定位,以丰富对于羊毛性状遗传结构的理解。
目前基于高通量SNP技术的全基因组关联分析(genome-wide association studies,GWAS),正在取代传统的QTL分析而成为更强大的基因检测技术[7-8]。已有学者将GWAS成功应用于绵羊重要经济性状的QTL定位研究[9-12],并在多个绵羊品种中鉴定出一批羊毛性状的候选基因和QTL[13-14]。中国美利奴羊是我国主要的细毛羊品种,由我国自主培育,分为新疆型、军垦型、科尔沁型和吉林型4个品系[15],其遗传结构和群体生产性能存在差异。中国美利奴羊(新疆型)由澳大利亚美利奴羊和新疆细毛羊杂交育成[16],是新疆主要的细毛羊品种,也是我国重要的毛用羊品种。目前,有关中国美利奴羊(新疆型)羊毛性状候选基因和QTL的研究尚未见报道。为此,本研究测定了中国美利奴羊(新疆型)周岁(16月龄)和成年(30月龄)的羊毛长度与产毛量(污毛质量),利用GWAS鉴定羊毛性状的候选基因和QTL位点,旨在探索中国美利奴羊(新疆型)羊毛性状发育的候选基因和常见的潜在遗传变异,为其育种提供有价值的分子标记。
1 材料与方法
1.1 试验羊群体概况与羊毛性状测定
试验样本来源于新疆巩乃斯种羊场的中国美利奴羊(新疆型)核心育种群2009-2012年出生的366只周岁母羊(33只公羊后代)。试验年4月中旬到11月中旬,采取牧场和舍内饲养相结合的方式饲养,其他时间(冬季)人工喂养。初配母羊在18月龄用试情公羊试情,发情后母羊采用人工授精方法进行配种。羔羊出生后称体质量,并记录每只羔羊的生产日期、性别和出生类型(单羔、双羔)。测定周岁羊(16月龄)和成年羊(30月龄)剪毛前后的体质量(校正产毛量用)、产毛量,同时采集体侧部位的毛样并在实验室测定羊毛长度,并采集耳组织样品低温保存待用。
1.2 试验羊DNA的SNP分型
采用常规苯酚/氯仿方法提取耳组织基因组DNA。利用Illumina OvineSNP50 BeadChip中密度芯片(包括54 241个SNP位点)在Infinium Ⅱ multisample assay (Illumina)平台上检测全部试验羊DNA样品SNP。使用iScan(Illumina)扫描芯片检测结果,并利用GenomeStudio软件对原始数据进行分析,从而得到每个样品的SNP分型数据。
将全部SNP分型结果进行质控分析,去除不符合条件的SNP。去除条件为:杂交阳性率(call rate)<90%,最小等位基因频率(MAF)<3%,显著偏离哈迪-温伯格平衡(Hardy-Weinberg equilibrium,HWE),χ2显著性检验P<0.001),位于性染色体上。
1.3 试验羊羊毛性状全基因组关联分析
采用single-locus回归方法,通过PLINK软件进行羊毛性状全基因组关联分析,模型如下:
y=μ+Xf+bm+Zs+e。
式中:y为表型值;μ为羊毛性状总平均;f为固定效应,对于周岁羊,固定效应包括群体(6个水平)、出生类型(单羔或双羔)、年份(2009,2010,2011,2012)、母羊年龄(初产、经产)等,对于成年羊,固定效应包括群体(6个水平)和年份(2010,2011和2012);b为表型值对SNP基因型的回归系数;m为SNP基因型向量(设2为次等位基因,0、1和 2代表SNP基因型11、12和 22);s为家系效应;X、Z为f和s的指数矩阵;e为残差效应。
采用permutation方法对关联分析结果进行校正,通过permutation 10 000次获得2个性状的基因组水平显著性阈值,以-lg (P)表示。
1.4 试验羊SNP的基因注释
OvineSNP50 BeadChip中的SNP主要是基于绵羊早期基因组(序列版本:Ovis_aries_v1.0)确定,绵羊早期基因组序列并不完整,部分序列之间存在缺口,需要使用最新的绵羊基因组数据重新进行定位,以获得准确的SNP信息。因此,本试验首先将OvineSNP50 BeadChip基因组注释文件在最新的绵羊基因组数据库(序列版本:Ovis_aries_v3.1,http://www.livestockgenomics.csiro.au/sheep/oarv3/Oarv3.150kSNP.position.gff3)中进行SNP的重新定位。然后,利用NCBI中的Ovisaries Annotation Release 100工具(http://www.ncbi.nlm.nih.gov/projects /mapview/map_search.cgi?taxid=9940&build =100.0)鉴定与羊毛性状显著关联SNP对应或邻近的基因。同时,基于绵羊数量性状位点数据库(Sheep QTLdb,http://www.animalgenome.org/cgi-bin/QTLdb/OA/index),利用Ovisar-ies Annotation Release 100工具对羊毛性状显著关联SNP进行比对、验证。
2 结果与分析
2.1 试验羊羊毛性状的描述性统计
由表1可知,周岁羊羊毛长度平均值为10.17 cm,成年羊羊毛长度平均值是9.84 cm。周岁羊产毛量平均为3.84 kg/只,略轻于成年羊的4.45 kg/只。试验羊群体羊毛性状的平均值均在正常范围(羊毛长度5.0~20.0 cm,产毛量2.0~10.0 kg/只)内,数据均在正态分布范围内,符合全基因组关联分析的要求。

表1 中国美利奴羊(新疆型)羊毛性状的描述性统计
2.2 试验羊基因组SNP的筛选
由表2可知,从试验羊群体中共筛选出44 798个SNP,每条染色体上的SNP数量为672~5 150个,SNP间的距离为49.97~65.59 kb 。

表2 中国美利奴羊(新疆型)基因组SNP在染色体上的分布和平均距离
2.3 试验羊SNP与羊毛性状的全基因组关联分析及基因注释结果
由图1和表3可知,本试验共检测出21个与羊毛长度和产毛量显著关联的SNP,分布在14条染色体上,其中1、2、4、7、8、12、13、14、15、25号染色体各有1个,6号和16号染色体各有2个,3号染色体有3个,10号染色体有4个。21个SNP中,4个SNP位于已知功能基因中,17个SNP位于已知功能基因附近。
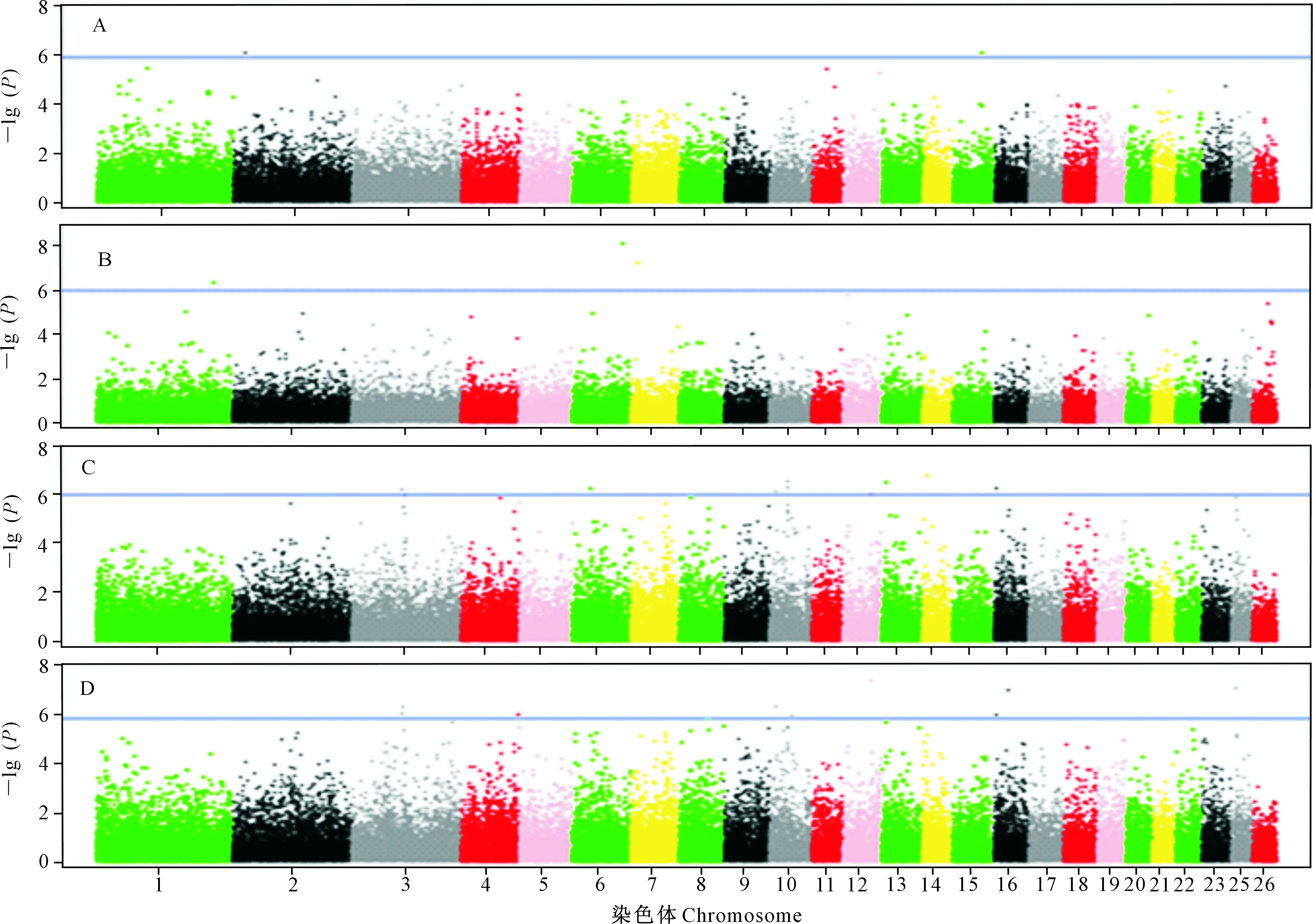
A.周岁羊羊毛长度;B.周岁羊产毛量;C.成年羊羊毛长度;D.成年羊产毛量。基因组显著性以图中的水平线为限
由表3可知,对于周岁羊,有5个SNP与羊毛性状显著相关,其中2个(OAR2_26198532.1、OAR15_61161801.1)与羊毛长度相关,3个(OAR6_111912224.1、OAR7_15117952.1、OAR1_258037376.1)与产毛量相关,这5个SNP的注释基因位于9 162~143 316 bp。
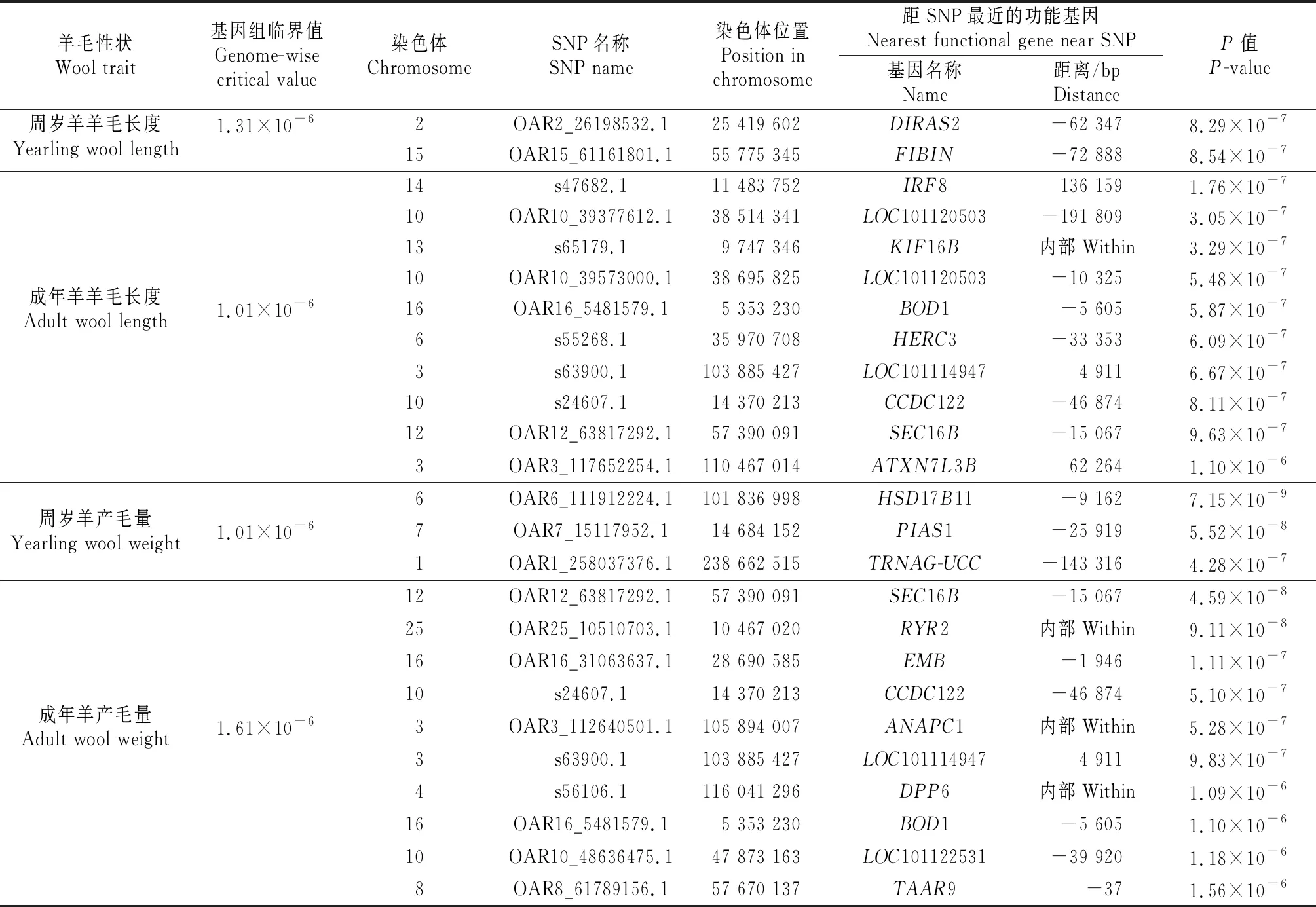
表3 中国美利奴羊(新疆型)SNP与羊毛性状的全基因组关联分析及基因注释结果
对于成年羊,有16个SNP与羊毛性状显著相关,其中6个(s47682.1、OAR10_39377612.1、s65179.1、OAR10_39573000.1、s55268.1、OAR3_117652254.1)与羊毛长度显著相关,6个(OAR25_10510703.1、OAR16_31063637.1、OAR3_112640501.1、s56106.1、OAR10_48636475.1、OAR8_61789156.1)与产毛量显著相关,4个(OAR16_5481579.1、s63900.1、s24607.1、OAR12_63817292.1)与羊毛长度和产毛量均显著相关。s65179.1、OAR25_10510703.1、OAR3_112640501.1和s56106.1 4个SNP分别位于驱动蛋白家庭成员16b(kinesin family member 16B,KIF16B)、利阿诺定受体2(ryanodine receptor 2,RYR2)、后期促进复合亚基1(anaphase promoting complex subunit 1,ANAPC1)和二肽基肽酶6(dipeptidyl peptidase 6,DPP6)基因内,并且这4个SNP位置均位于基因编码区。
2.4 试验羊羊毛性状显著相关SNP与已知羊毛性状相关QTL的比较
由表4可以看出,与绵羊QTLdb比较,有5个SNP位于已报道的与羊毛纤维长度和产毛量相关的QTL中,分别是s63900.1、OAR3_112640501.1、OAR7_15117952.1、OAR15_61161801.1和OAR25_10510703.1。有2个SNP位于已报道的与纤维直径均值相关的QTL中,分别是s55268.1和OAR6_111912224.1。OAR25_10510703.1位于与污毛质量相关的QTL中,OAR12_63817292.1位于与污毛平均日增重相关的QTL中。OAR25_10510703.1定位到2个与纤维长度相关的QTL(ID为14016和12919)和1个与污毛质量相关的QTL(ID为12913),OAR7_15117952.1定位到1个与纤维长度相关的QTL(ID为14015 )和1个与初级纤维直径变异系数相关的QTL(ID为14020),结果表明这2个位点对中国美利奴羊(新疆型)羊毛长度和产毛量具有显著性作用。

表4 中国美利奴羊(新疆型)羊毛性状显著相关SNP分析
3 讨 论
近年来,GWAS已经成为动物育种领域挖掘基因的有效方法,有学者基于GWAS技术已经在绵羊上发现了多个与体质量、抗病、肉质和毛用性状相关的遗传变异位点[17-18],但目前基于GWAS的中国美利奴羊(新疆型)羊毛性状的SNP信息尚不清楚。本研究利用Illumina OvineSNP50 BeadChip中密度芯片对中国美利奴羊(新疆型)群体的羊毛长度和产毛量进行了GWAS分析,从基因组水平鉴定出21个显著性SNP位点。研究表明,羊毛产量与年龄之间存在较低的遗传相关性[19-20]。本试验将周岁羊和成年羊羊毛性状作为独立性状进行分析,并未发现两者存在重叠的SNP,也未观察到SNP的多效性现象,表明不同基因在羊毛发育的不同时期可能发挥不同作用,仅从单一时期获取数据进行遗传分析,不足以挖掘出真实有效的遗传变异信息,这要求人们在绵羊选育中应将年龄作为重要因素进行分析。
研究表明,FIBIN是斑马鱼胸鳍芽发育所必需的一种分泌因子[21];在脊椎动物中,FIBIN可调控胎儿皮肤成纤维细胞的形成与发育[22]。本试验所鉴定的21个显著性SNP位点中,OAR15_61161801.1位点与周岁羊羊毛纤维长度显著相关,且定位到羊毛纤维长度QTL(ID:12917),该位点位于FIBIN基因附近,这提示FIBIN基因可能是影响羊毛长度性状的重要功能基因。研究表明,雄激素可促进身体不同部位的毛发生长[23-24]。HSD17B11是雄酮合成酶,在雄激素代谢中发挥重要功能[25]。目前对于HSD17B11是否直接调控毛发生长尚未见相关报道。本研究结果表明,邻近HSD17B11基因的OAR6_111912224.1位点与周岁羊产毛量显著相关,提示HSD17B11可能通过影响雄激素代谢而调控羊毛生长。
本研究发现,有16个SNP与成年羊毛性状显著相关,其中位于染色体13上的s65179.1位点与纤维长度相关,该位点位于KIF16B基因中。KIF16B在功能上与表皮生长因子(epidermal growth factor,EGF)、纤维生长因子(fiber growth factor,FGF)和Wnt信号通路有关,而EGF、FGF和Wnt信号通路被证明在羊毛毛囊发育和纤维生长中起着重要作用[26-28]。Wang等[29]发现,中国美利奴羊(军垦型)KIF16B也存在与纤维直径变异系数(coefficient of variation of fiber diameter,FDCV)显著相关的SNP,这表明该基因可能存在多效性现象。有研究证明,RYR2与皮肤角质形成和毛囊细胞分化相关[30]。本研究发现,OAR25_10510703.1位点位于RYR2基因中,与纤维长度和污毛质量显著相关,这提示RYR2可能是中国美利奴羊(新疆型)羊毛纤维长度和羊毛产量的重要候选基因。
本试验对中国美利奴羊(新疆型)的羊毛长度和产毛量进行了GWAS分析,筛选到了相关候选基因和SNP位点,为该品种绵羊的育种研究提供了羊毛性状初步候选区域和基因,为解析绵羊羊毛长度和产毛性状的遗传变异奠定了基础。
