桑树叶绿体psbA基因的生物信息学预测及分析
2022-07-19杨贵明
王 敬 王 鹏 王 晖 杨贵明 周 玲*
(1.承德医学院蚕业研究所/河北省高校特产蚕桑技术应用研发中心,河北承德 067000; 2.承德应用技术职业学院,河北承德 067000)
psbA基因存在于桑属植物的叶绿体,碱基序列位于叶绿体外显子全部碱基序列中的第333—1 394位,全长1 062 bp。高等植物的叶绿体基因在遗传中为母系遗传,序列具有极高度的保守性[1]。这使其在植物的进化过程中变异率低,因此,psbA成为研究植物进化过程的重要标记基因之一[2]。郭亮亮对桑属植物叶绿体psbA基因序列和进化进行分析[3],林琳对29种桃金娘目植物psbA基因进行密码子偏好及聚类分析[4],发现结合RSCU的值和CDS序列的聚类分析可更好地对物种进行高精度的分类,获得psbA基因的进化的内在规律。当下,尚无对桑树psbA基因进行生物信息学分析的报道,本研究拟对其氨基酸序列的基础生物信息学进行分析及预测,为基于psbA基因研究植物的分类提供理论基础。
1 材料及方法
psbA基因(登录号:NC_008359.1,333-1394),其氨基酸序列(登录号:YP_762241.1)。使用开放阅读框OFR Finder(https://www.ncbi.nlm.nih.gov/orffinder/)对桑树psbA基因进行分析;对桑树氨基酸psbA序列的理化性质使用Prot Param(https://www.expasy.org/resources/protparam)推断;用Prot Scale(https://web.expasy.org/protscale/)对其氨基酸序列的疏水性/亲水性预测;使用SignalP-5.0(https://services.healthtech.dtu.dk/service.php?SignalP-5.0)对其氨基酸序列信号肽分析;TMHMM Server.v.2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)分析氨基酸序列的跨膜结构域;PBIL(https://npsa-pbil.ibcp.fr/)分析氨基酸序列的二级结构;通过使用PredictProtein(https://predictprotein.org/)对其进行亚细胞定位、聚类分析、靶位点预测,并使用构建同源树的DNAman对桑、白果树、箭毒木、榕树、腾构等11个物种进行同源树构建。
2 结果及分析
2.1 psbA基因开放阅读框特征
DNA序列中所具备编码蛋白质潜质的序列被称为开放阅读框。用OFR Finder在线软件对其基因序列分析得出(图1)psbA基因有7条开放阅读框,这7条开放阅读框的长度依次为:1 062 bp、87 bp、90 bp、123 bp、129 bp、111 bp和84 bp;psbA基因的起始密码子及终止密码子的位置依次为:1 bp处、182 bp处、93 bp处、1 011 bp处、756 bp处、869 bp处、575 bp处及1 062 bp处、268 bp处、182 bp处、889 bp处、628 bp处、759 bp处和492 bp处;所翻译的氨基酸数目依次是353个、28个、29个、40个、42个、36个和27个。

图1 psbA基因的开放阅读框特征
2.2 psbA氨基酸序列的理化性质推断
经Prot Param在线软件分析得出,psbA序列由353个氨基酸构成,根据其所含元素的数量推算其分子式C1789H2676N456O492S14,分子量38892.53,等电点为5.21,由于其不稳定指数为34.66(34.66<40),因此属稳定蛋白。N末端为甲硫氨酸。其所含的氨基酸中,甘氨酸(Gly)占比最高为9.6%,其次为亮氨酸(Leu)、异亮氨酸(Ile)、丝氨酸(Ser),谷氨酰胺(Gln)含量最少(图2)。
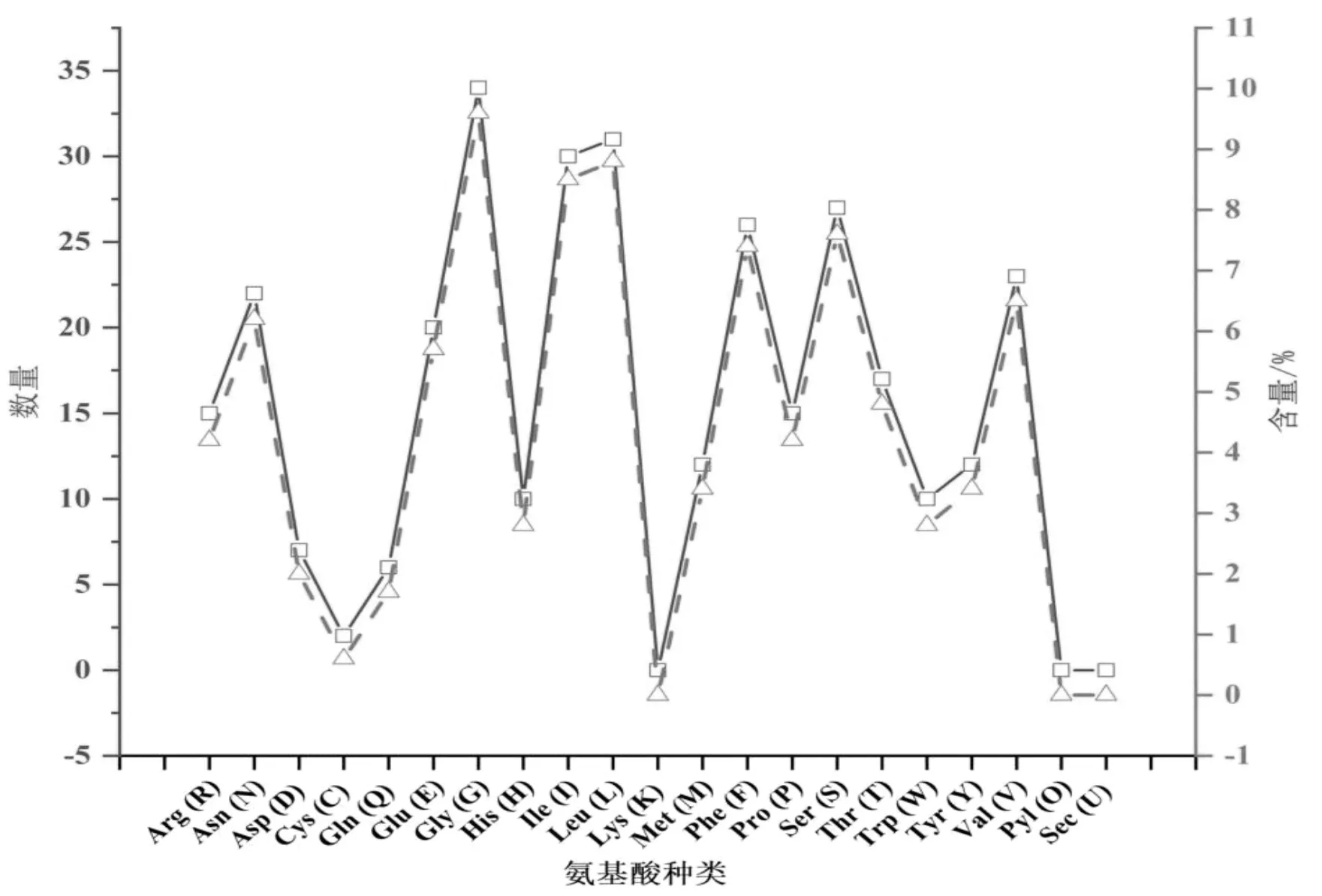
图2 psbA基因各氨基酸的种类及含量推断
2.3 psbA的氨基酸序列疏水性/亲水性预测
经软件Prot Scale分析得出(图3),由图可看出负值峰少于正值峰,表现为疏水性。

图3 psbA氨基酸序列的疏水性/亲水性预测
2.4 psbA氨基酸序列的信号肽分析
经软件SignalP-5.0预测得出图4。由图4可看出信号肽存在的几率为0.002 1,趋近于0,不存在信号肽,可推断为非分泌蛋白。
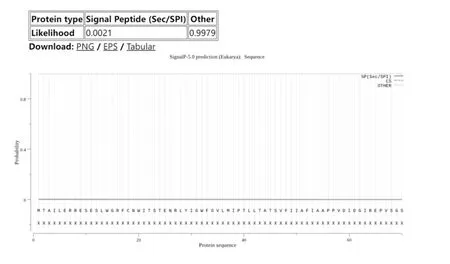
图4 桑树psbA氨基酸序列的信号肽分析
2.5 psbA氨基酸序列跨膜结构域的推测
经TMHMM Server.v.2.0分析得出膜内区域的概率趋近于0%,膜外的概率接近于100%,不存在跨膜结构域(图5)。
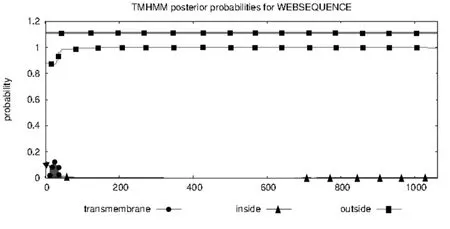
表示为跨膜区域;表示为膜内区域;表示膜外区域图5 桑树psbA氨基酸序列跨膜结构域的推测
2.6 psbA基因氨基酸序列的二级结构预判
经PBIL分析的结果显示,氨基酸序列的组成中包括:12.75%的延伸链、58.64%的无规则卷曲和28.61%螺旋(图6)。

图6 psbA氨基酸序列二级结构的预判
2.7 psbA氨基酸序列的亚细胞定位、GO功能、结合区的推测
经PredictProtein分析得出:psbA氨基酸序列存在真核生物细胞的叶绿体中,与当前文献的查询结果相同。蛋白结合位点为13个,依次位于:59-64、129-134、188-192、193-193、194-197、251-251、253-253、267-270、297-302、317-321、323-324、327-327、329-340位(图7)。

图7 psbA氨基酸序列二级结构的预判
psbA氨基酸序列的GO功能注释包括细胞组分、生物学进程、分子功能。细胞组成包括膜的整体组成、叶绿体类囊体膜、类囊体、叶绿体、光系统II;生物学进程有光合作用、蛋白质-发色团连锁、对除草剂反应、光合电子传输系统II;分子功能包括辅酶绑定、铁离子结合、氧化还原酶活性、阴离子结合和电子传送器(图8)。

图8 psbA氨基酸序列GO功能
2.8 桑等物种psbA基因序列的亲缘关系推测
经使用软件DNAMAN对桑、白果树、箭毒木等共11个物种构建同源树,(图9)所示。桑与其余10个物种的psbA氨基酸序列相似度极高,其最低相似度是98%,说明psbA氨基酸序列保守性极高。
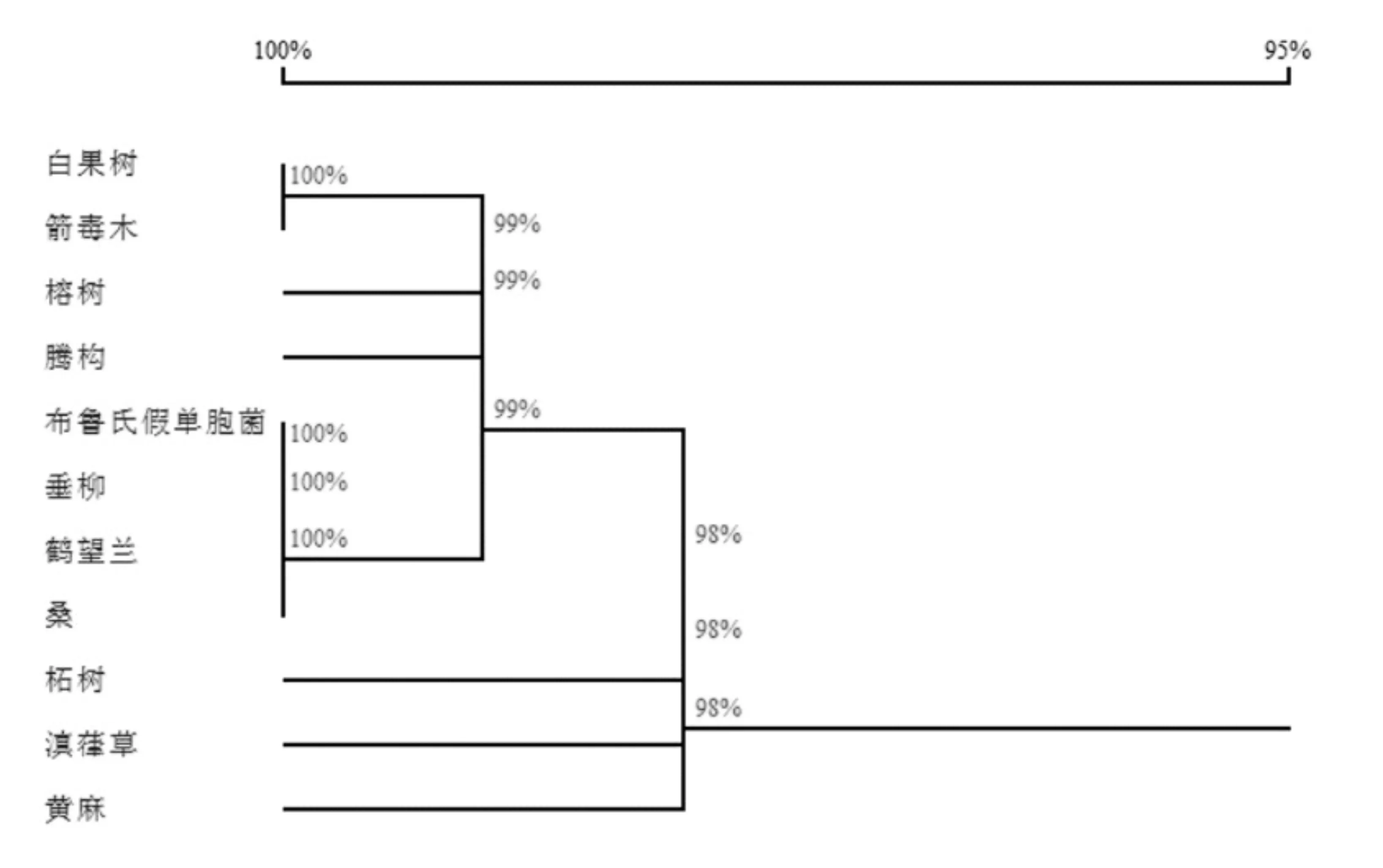
图9 11个物种psbA氨基酸序列同源树
3 结论及讨论
psbA作为叶绿体中的关键调控基因,在光合作用中起传递电子到编码光合系统 II 反应蛋白的作用[5]。高等植物叶绿体psbA基因的启动子作为叶绿体基因工程中常用的启动子[6],经常用于分子发育关系的比较[7]。由于叶绿体基因为母系遗传,变异程度低,常被运用于物种的发育及进化领域研究[8]。
通过利用生物信息学对桑树psbA基因进行预测,psbA基因存在于真核生物细胞的叶绿体中,和侯世昌[9]等人的研究相同。psbA基因存在7条开放阅读框,含有353个氨基酸。氨基酸序列具有疏水性,无信号肽。psbA的结构以无规则卷曲为主要方式。GO功能分为三类,序列分布有13个蛋白结合位点。桑与白果树、箭毒木等11个物种的psbA氨基酸序列具有极高的同源性,说明psbA在生物进化中保守性极高。本文使用生物信息学手段对桑树psbA基因进行预测分析,与常见的对于桑属植物的分类研究方式略有不同。对于桑属植物的分类研究还有更多的方法,本文仅对psbA基因进行了初步的生物信息学分析,对于psbA基因功能的探索还需要进一步进行实验研究。
