无刺龙舌兰叶绿体基因组特征及密码子偏好性分析
2022-07-14王飞辛雅萱董章宏赵文植李卫英马路遥夏茂甜辛培尧
王飞 辛雅萱 董章宏 赵文植 李卫英 马路遥 夏茂甜 辛培尧




摘要:【目的】分析無刺龙舌兰叶绿体基因组特征及密码子偏好性,为无刺龙舌兰叶绿体相关基因的表达、修饰和物种进化研究提供参考。【方法】对无刺龙舌兰的叶绿体基因组进行测序、组装和注释,分析密码子偏好性及其影响因素,并通过建立高、低基因表达库,筛选出最优密码子。基于20个已发表的龙舌兰科植物叶绿体基因组数据构建系统发育进化树。【结果】无刺龙舌兰叶绿体基因组总长157579 bp,大单拷贝区(LSC)、小单拷贝区(SSC)和2个反向重复区(IRa和IRb)的长度分别为85940、18279和26680 bp,GC含量为37.8%,包括135个基因(85个蛋白编码基因、38个tRNA基因、8个rRNA基因及4个未知功能的基因),从中筛选出51个长度大于300 bp的基因编码区(CDS)序列,其有效密码子数(ENC)均大于41.0。GC1、GC2、GC3和GC3s含量分别为46.75%、39.61%、29.19%和26.06%,说明密码子第3位多以A/T结尾。GCall与GC1、GC2和GC3均呈极显著相关(P<0.01,下同),但GC3与GC1和GC2均无显著相关性(P>0.05,下同),表明密码子第1、2位的碱基组成相似,但与第3位的相似度不高。选择和突变是导致叶绿体基因组密码子偏好性的主要因素。筛选出14个多以A/U结尾的最优密码子。无刺龙舌兰与克雷塔罗丝兰和西地格丝兰为姊妹关系,自荐值为100%。【结论】无刺龙舌兰叶绿体基因组为保守的四分体结构,叶绿体基因组密码子偏好性较弱,主要受选择和突变等多因素影响。基于植物叶绿体基因组构建系统发育进化树在物种的分类鉴定及确定各物种间系统发育关系的研究中是一种准确、可靠的方法。
关键词:无刺龙舌兰;叶绿体基因组;密码子偏好性;系统发育分析
中图分类号:S563.8 文献标志码: A 文章编号:2095-1191(2022)04-1030-10
Chloroplast genome characteristics and codon preference analysis of Yucca treculeana
WANG Fei, XIN Ya-xuan, DONG Zhang-hong, ZHAO Wen-zhi,
LI Wei-ying, MA Lu-yao, XIA Mao-tian, XIN Pei-yao
(Southwest Landscape Architecture Engineering Technology Research Center of National Forestry and Grassland Administration, Southwest Forestry University/Key Laboratory of Biodiversity Conservation in Southwest China of National Forestry and Grassland Administration, Southwest Forestry University, Kunming, Yunnan 650224, China)
Abstract:【Objective】To analyze chloroplast genome characteristics and codon preference of Yucca treculeana, so as to provide reference for the study of chloroplast-related gene expression,modification and species evolution. 【Method】Chloroplast genome of Y. treculeana was sequenced, assembled and annotated to analyze codon preference and its influe-ncing factors, and the optimal codon was screened by establishing high and low gene expression libraries. Phylogenetic tree was constructed based on 20 published chloroplast genome data of Agavaceae plants. 【Result】The total length of Y. treculeana chloroplast genome was 157579 bp, and the length of the large single copy region(LSC), small single copy region(SSC), and two reverse repeat regions(IR),namely IRa and IRb regions were 85940, 18279 and 26680 bp, respectively. The total GC content was 37.8%. Among 135 genes (85 protein-coding gene, 38 tRNA, 8 rRNA and 4 unknown functional genes), 51 coding DNA sequences(CDS) with length greater than 300 bp were screened out, and the ENC was greater than 41.0. The contents of GC1, GC2, GC3 and GC3s were 46.75%, 39.61%, 29.19% and 26.06%, respectively, indicating that the third codon most ended in A/T. GCall was significantly correlated with GC1, GC2 and GC3 (P<0.01, the same below), but GC3 was not significantly correlated with GC1 and GC2 (P>0.05, the same below), suggesting that the base composition of the first and second codon of chloroplast genome was basically similar,but the similarity with the third codon was not high. Selection and mutation were the main causes of chloroplast genome codon preference. 14 optimal codons ending in A/U were screened out. Y. treculeana was sisterly with Y. queretaroensis and Y. schidigera,and the bootstrap values was 100%. 【Conclusion】The chloroplast genome of Y. treculeana has a conserved tetrad structure, and the codon bias is weak, which is mainly affected by multiple factors such as selection and mutation. It is an accurate and reliable method to construct phylogenetic tree based on chloroplast genome in studying the taxonomic identification and phylogenetic relationship among plant species.
Key words: Yucca treculeana; chloroplast genome; codon preference; phylogeny analysis
Foundation items:Yunnan Science and Technology Department Science and Technology Plan Development Key Research Project (2018BB005); Southwest Forestry University Science and Technology Innovation Fund Project (KY21034)
0 引言
【研究意义】无刺龙舌兰(Yucca treculeana)为丝兰属(Yucca)多年生植物,原产于北美洲,多生于降雨稀少的沙漠地区,适应性强,剑形叶,花茎高挺,整体形态优美,具有极高的观赏价值,且其根和茎分别用作啤酒发泡剂、除臭剂的生产原料,叶纤维强韧亦可用于制作绳缆(Irish,2000)。据调查,丝兰属内约有40个种。截至2021年7月,NCBI数据库中已确定发表叶绿体全基因组的丝兰属植物有6种。目前,丝兰属植物在不同分类系统存在争议,根据哈钦松系统和克朗奎斯特系统将其划分为龙舌兰科(Agavaceae);而APG Ⅲ分类系统则将其定位至天门冬科(Asparagaceae)(http://www.iplant.cn/info/Yucca)。造成分类差异的原因不仅与分类系统本身有关,还与丝兰属植物形态特征相似度较高密切相关。因此,在利用传统形态学方法分类存在争议的情况下,借助无刺龙舌兰叶绿体基因组构建的系统发育进化树能直观反映物种间遗传关系,同时有助于对其进行分类(Kai et al.,2018)。此外,通过分析无刺龙舌兰叶绿体基因组密码子偏性,可优化基因密码子,提高目的基因表达水平,对选育优良品种具有重要意义。【前人研究进展】叶绿体是绿色植物和部分藻类用于光合作用的细胞器(Jia-Yee et al.,2015),也是除线粒体外的另一个含有遗传信息的细胞器。与含有庞大遗传信息的核基因组相比,叶绿体基因组长度较小,仅为160 kb左右,且碱基含量和基因数量均较少,但因叶绿体内基因所在位置的特殊性,其具有结构完整性和序列保守性(Parks et al.,2009;Jansen and Uhlman,2012)。叶绿体基因组为单亲遗传,碱基很少发生替换,使其在植物分类及系统进化研究中具有独特的优势(Tao et al.,2017)。在解析植物叶绿体基因组特征的基础上,结合系统发育分析探究物种间进化关系已有相关报道。高亚芳等(2019)通过对药用植物金铁锁叶绿体基因组的测序、组装和注释,明确了其基因组成,进而利用叶绿体全基因组构建系统发育进化树,解析了石竹科属间的系统发育关系。段义忠和张凯(2021)对2种沙冬青属植物叶绿体基因组的反向重复区(IR)进行比对分析,并基于叶绿体基因组构建了系统发育进化树,为该属的物种鉴别、种群动态研究打下基础。此外,作为编码氨基酸的遗传密码子在基因功能和蛋白质表达的研究中意义重大。肽链合成过程中每种氨基酸对应1~6种密码子(即同义密码子)(牛元等,2018),但密码子的使用偏好于一种或几种特定密码子,称为密码子偏好性(Li et al.,2019)。只有无选择压或基因突变的情况下,才不存在密码子使用偏好性(Long et al.,2018)。但实际上,生物体是自然选择的结果,均存在密码子使用偏好性(Li et al.,2019)。密码子使用偏好性存在于各生物体中,自然选择、碱基组成、基因的长度和表达水平、RNA丰度和二级结构、氨基酸保守性等均可能造成密码子使用偏好性(Xu et al.,2011)。目前,不同植物密码子偏好性分析已有相关报道。金刚等(2018)揭示了同为龙舌兰科植物剑麻的叶绿体基因组密码子偏好性,并指出密码子使用偏好性受选择和突变等多重因素影响。另外,香花枇杷(屈亚亚等,2021)、杧果(辛雅萱等,2021)、永椿香槐(李江飞等,2021)和蒜头(原晓龙等,2021)等叶绿体基因组密码子偏好性分析均是在测序获得叶绿体全基因组的基础上,应用统计学方法分析其密码子使用模式及形成原因。【本研究切入点】无刺龙舌在植物分类中仍存在一定的争议,利用其叶绿体基因组构建的系统发育进化树有助于其分类研究,但目前未见丝兰属植物叶绿体基因组特征及密码子偏好性的相关研究报道。【拟解决的关键问题】对无刺龙舌兰的叶绿体基因组进行测序、组装和注释,分析密码子偏好性及其影响因素,并通过建立高、低基因表达库,筛选出最优密码子;基于20个已发表的龙舌兰科植物叶绿体基因组数据构建系统发育进化树,为无刺龙舌兰乃至丝兰属植物叶绿体相关基因的表达、修饰和物种进化研究提供理论依据。
1 材料与方法
1. 1 试验材料
供试材料为新鲜无刺龙舌兰叶片,采自于中国科学院西双版纳热带植物园,将采集好的样叶装入自封袋放入液氮中速冻,带回实验室后保存于-80 ℃的超低温冰箱备用。主要设备仪器:超微量紫外分光光度计(DN2000)、核酸蛋白测定仪(NanoDrop 2000 Thermo Scientific)、琼脂糖凝胶电泳仪(北京六一DYCP-31DN)和凝胶成像系统(Gene Company Limited,GBOX.E3)。
1. 2 DNA提取及测序
采用改良CTAB法提取无刺龙舌兰的全基因组DNA(Windarti et al.,2021),并利用琼脂糖凝胶电泳及NanoDrop2000对其完整性、质量和浓度进行检测。将检测合格的DNA样品送至北京诺禾致源科技股份有限公司建库测序。
1. 3 叶绿体基因组组装、注释及编码区(CDS)序列挑选
以克雷塔罗丝兰(Yucca queretaroensis)(登录号KX931468)为参考序列,使用GetOrganelle进行基因组序列组装(Jin et al.,2020),采用GeSeq(https:// chlorobox.mpimp-golm.mpg.de/geseq.html)對无刺龙舌兰叶绿体基因组进行注释(Tillich et al.,2017),再用Geneious 8.1.3手动校正(Kearse et al.,2012)。将注释好的无刺龙舌兰叶绿体基因组数据(登录号OL912952)上传至NCBI数据库(https://www.ncbi.nlm.nih.gov/)。利用OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)(Lohse et al.,2013)绘制无刺龙舌兰叶绿体基因组物理图谱。
利用Geneious 8.1.3、BioEdit Sequence Alignment Editor和ORFfinder(https://www.ncbi. nlm.nih.gov/orffinder/)对无刺龙舌兰叶绿体基因组进行分析,共获得85个基因的CDS序列。为了降低误差,剔除长度小于300 bp、内部含有终止密码子和重复基因的序列,最终筛选出51个以ATG为起始密码子的CDS用作后续分析。
1. 4 密码子相关参数计算
使用CodonW 1.4.2和CUSP(https://bioinforma-tics.nl/emboss-explorer/)统计上述筛选出的51个CDS序列的有效密码子数(Effective number of codon,ENC)、同义密码子相对使用度(Relative synonymous codon usage,RSCU)、各CDS序列总GC含量(用GCall表示),以及密码子第1、2、3位上的GC含量(分别用GC1、GC2和GC3表示)。
1. 5 中性绘图分析
利用中性绘图直观反映影响密码子使用偏好性因素,即利用Excel 2010以CDS的GC3值为横坐标、GC12(GC1和GC2的均值)为纵坐标绘制散点图,用于分析GC12和GC3的相关性,进而判断密码子偏好性影响因素(Jiang et al.,2008)。若GC12与GC3相关性显著,回归系数接近1,说明突变是其主要影响因素;反之,则说明GC12与GC3差异大,自然选择是主要影响因素(Wei et al.,2014)。
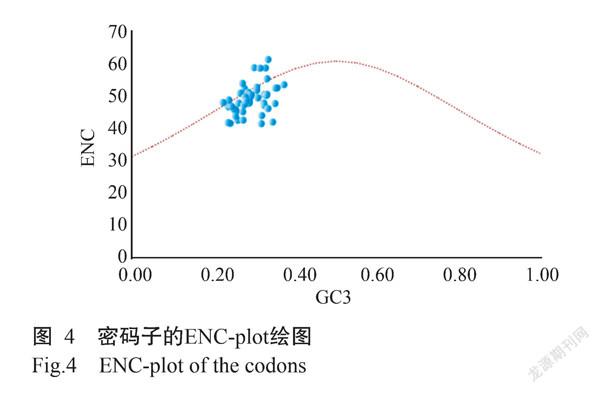
1. 6 ENC-plot分析
利用Excel 2010以GC3为横坐标、ENC为纵坐标,利用两者的实际值来构建二维散点图,并在图中添加ENC=2+GC3+29/[GC2+(1-GC3)2]的标准曲线,根据散点在标准曲线周围的分散情况,再结合ENC比值频率,进而判断影响密码子偏好性的原因(Sueoka,2017)。若代表CDS的散点落在曲线附近,则密码子偏好性受突变影响;而落在曲线下方较远的位置,则受自然选择影响(Wang et al.,2018)。
1. 7 PR2-plot分析
利用Excel 2010以G3/(G3+C3)为横坐标、A3/(A3 +T3)为纵坐标进行绘图,用于分析密码子第3位上的碱基组成。图的中心点表示A=T且G=C,即无偏性的密码子状态,由中心点向其余点发出的矢量表示偏性方向和程度(Yang et al.,2015)。
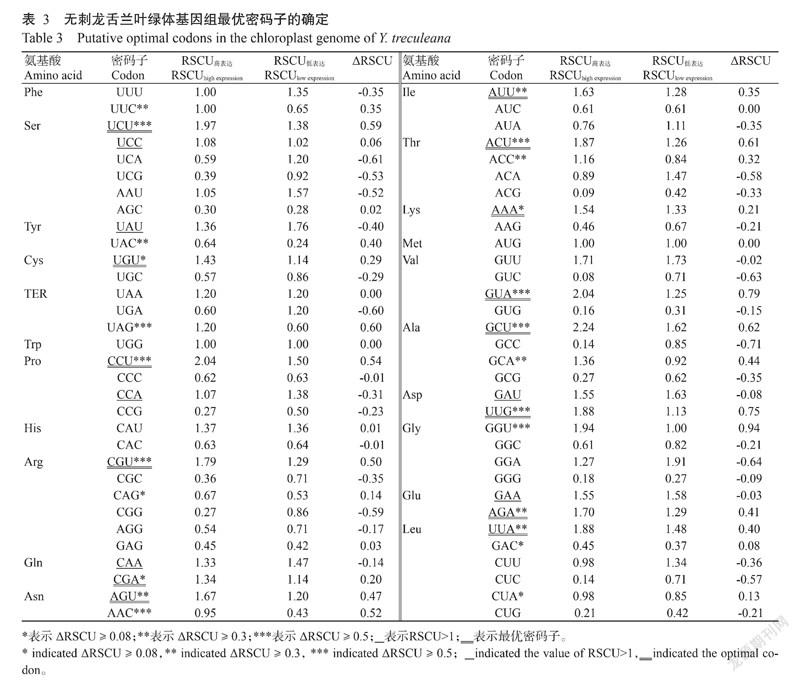
1. 8 最优密码子分析
同义密码子相对使用度(RSCU)作为选择最优密码子的条件之一,RSCU=1时,密码子无偏好性;RSCU>1时,密码子使用频率偏高;反之,表示密码子出现频率低(Wu et al.,2019;惠小涵等,2020)。最优密码子选择时,以ENC为标准,对51个CDS序列的ENC进行排序,分别从ENC最高和最低两端选取10%的基因,建立高、低表达库。计算ΔRSCU值(ΔRSCU=RSCU高表达-RSCU低表达),将ΔRSCU≥0.08,且RSCU>1的密码子定为最优密码子。
1. 9 系统发育分析
为了确定无刺龙舌兰在龙舌兰科中的系统发育位置,从NCBI数据库下载20个已发表的龙舌兰科植物叶绿体基因组与外类群绵枣儿(Barnardia japonica),结合无刺龙舌兰叶绿体基因组进行系统发育分析。将22个叶绿体基因组序列用MAFFT 7比对,BioEdit手动调整,用最大似然法(ML)对系统发育关系进行分析,使用RAxML 8.2.12中的HPC2 on XSEDE模型构建系统发育进化树(Stamatakis et al.,2008;Katoh and Standley,2013),最后用FigTree 1.4.0进行树图美化。
2 结果与分析
2. 1 叶绿体基因组基本特征分析结果
无刺龙舌兰叶绿体基因组大小为157579 bp,呈反向平行的双链环状四分体结构,大单拷贝区(LSC)、小单拷贝区(SSC)和2个IR区(IRa和IRb)的大小分别为85940、18279和26680 bp,平均GC含量为37.8%。另外,叶绿体基因组共有135个基因(85个蛋白编码基因、38个tRNA基因、8个rRNA基因及4个未知功能的基因),絕大多数存在于LSC 区(图1)。4个未知功能的基因中,ycf3和ycf4位于LSC区,ycf2位于IR区并有一个拷贝,ycf1基因则位于SSC区和IRb区的交界位置。值得注意的是,LSC区的atpB基因出现了1个突变位点和1个缺失位点,导致该基因序列不能正常翻译,基因功能丧失,致使假基因化。
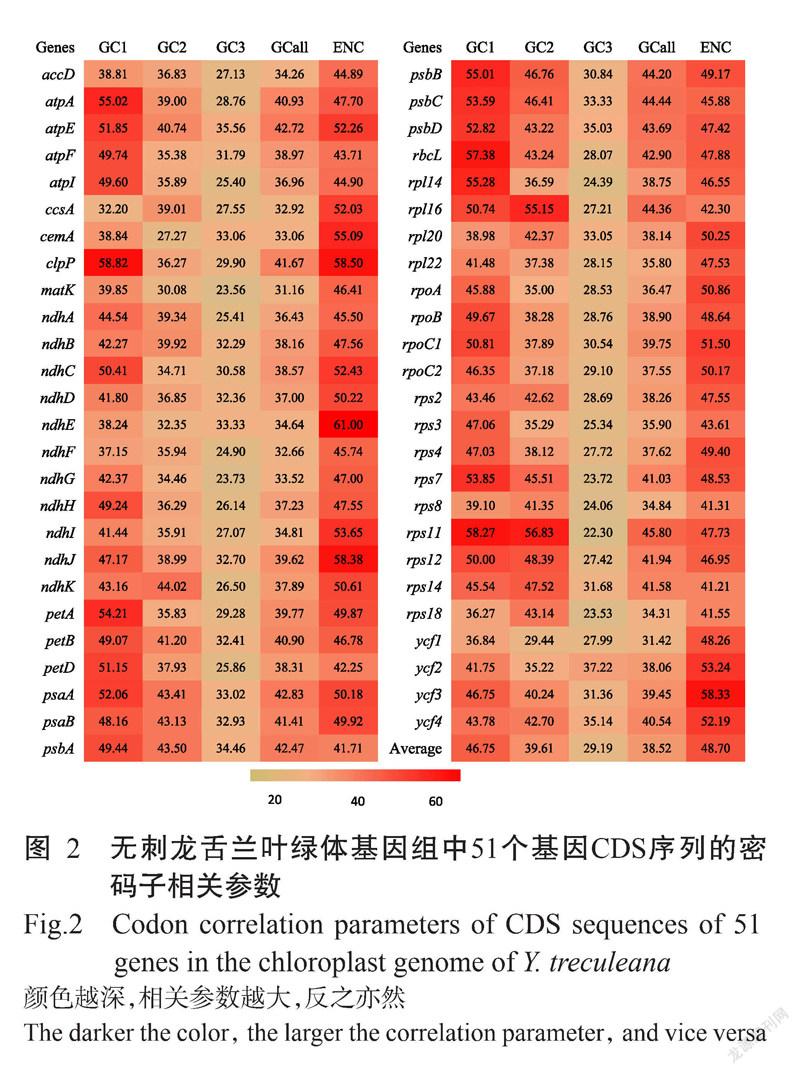
2. 2 密码子组成及使用度分析结果
无刺龙舌兰叶绿体基因组密码子组成如图2所示。GC1、GC2和GC3含量分别为46.75%、39.61%和29.19%。密码子第1、2、3位的GC分布并不均匀,第3位上的GC含量明显低于前两位,分布趋势为GC1>GC2>GC3,GC3s含量为26.06%,表明密码子第3位多以A/T 结尾。
ENC理论取值范围为20.0~61.0,与同义密码子的偏性呈负相关。无刺龙舌兰51个CDS序列的ENC为41.2~61.0,均>41.0,说明无刺龙舌兰叶绿体基因组密码子偏好性较弱。其中,ndhE基因的ENC为理论取值范围的上限(即61.0),说明该基因密码子使用无偏好性。
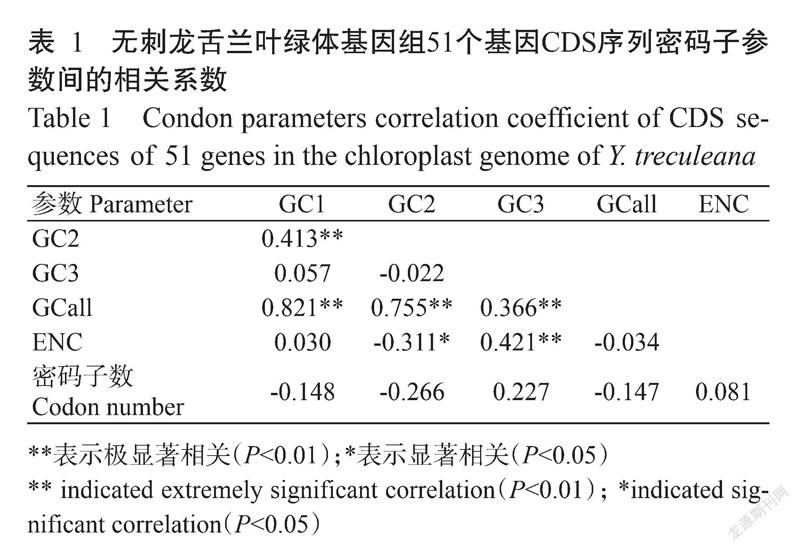
从密码子各参数的相关分析结果(表1)可看出,GCall与GC1、GC2和GC3均呈极显著相关(P<0.01,下同),且GC1与GC2及ENC值与GC3均呈极显著相关,但GC3与GC1和GC2均无显著相关性(P>0.05,下同),表明叶绿体基因组密码子第1、2位的碱基组成基本类似,但与第3位的碱基相似度不高。而叶绿体基因组密码子数与各GC含量和ENC均无显著相关性。
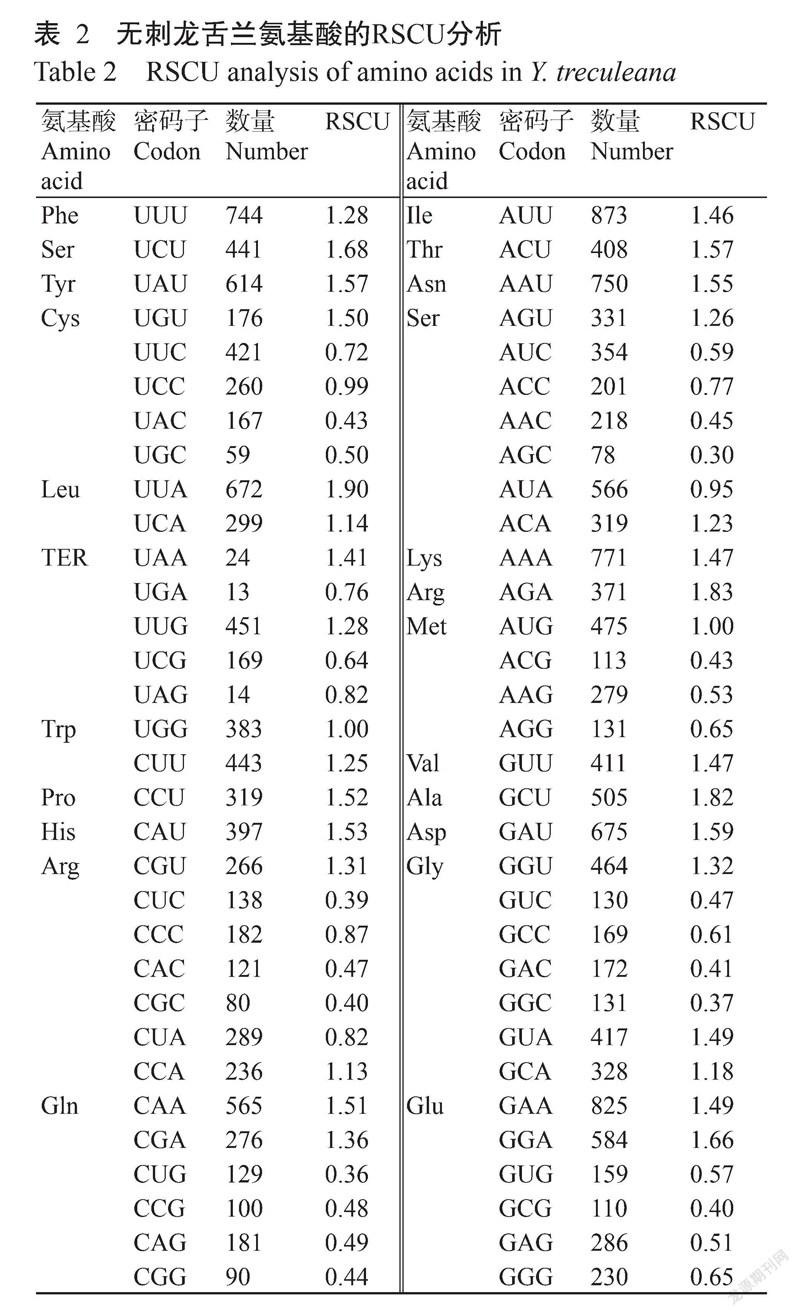
从无刺龙舌兰各氨基酸RSCU分析(表2)可看出,RSCU>1的密码子共30个,多为A/U结尾,而以G/C结尾的密码子RSCU多数都小于1,表明无刺龙舌兰叶绿体基因组密码子偏好以A/U结尾。
2. 3 中性绘图分析结果
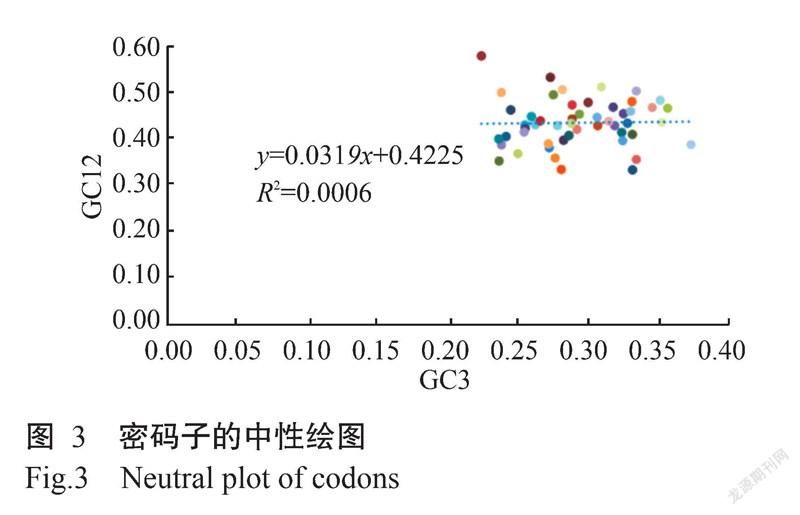
从图3可看出,GC12和GC3的取值范围分别为0.33~0.51和0.22~0.35,回归系数(即斜率)为0.0391,GC12与GC3的相关系数(R)为0.024,二者相关性弱,表明密码子第1、2位与第3位之间碱基的相关性不强,可初步判断出自然选择是影响密码子使用偏好性的主要因素之一。
2. 4 ENC-plot分析结果
从图4中可看出,多数基因CDS序列处于标准曲线下方。再结合图5可知,ENC比值分布在-0.05~0.05区间的基因CDS序列有15个,其接近标准曲线,实际ENC与预期ENC接近,说明其偏好性主要受突变影响;而其余36个基因CDS序列的ENC比值分布在 -0.05~0.05区间之外,位于标准曲线较远的位置,表示与预期ENC相差较大,即这部分基因密码子偏好性与GC3含量有关。因此,除了突变的影响,自然选择等其他因素很大程度上也会影响密码子的偏好性。
2. 5 PR2-plot分析结果
PR2-plot绘图是通过分析密码子第3位碱基类型,从而揭示密码子使用偏性影响因素。由图6可知,51个基因CDS序列在图中4个区域分布并不均匀,其中,左下方分布较多。T>A、C>G,即嘧啶使用频率高于嘌呤,进一步说明突变并不是影响无刺龙舌兰叶绿体基因组密码子使用偏好性的唯一因素,选择、碱基组成等其他因素对其密码子使用偏好性也有一定影响。
2. 6 最优密码子的确定
对ENC进行排序,从两端各选5个基因来构建高、低表达库,分别计算△RSCU,结果如表3所示。以△RSCU≥0.08为标准,再结合叶绿体基因的RSCU>1确定最优密码子,共14个,分别是UCU、UGU、UUA、UUG、UAG、CCU、CGU、CGA、AUU、ACU、AGU、ACC、AAA、AGA、GCU、GGU、GUA和GCA。其中8个以U结尾,5个以A结尾,1个以G结尾,说明最优密码子多以A/U结尾。
2. 7 无刺龙舌兰叶绿体基因组系统发育分析
基于叶绿体基因组序列,以天门冬科(Asparagaceae)绵枣儿属(Barnardia)植物绵枣儿(B. japonica)为外类群,构建无刺龙舌兰在内的21种龙舌兰科植物的系统发育进化树,如图7所示。除外类群绵枣儿之外,龙舌兰科15个属的植物按进化关系远近可分为7个分支,其中丝兰属(Yucca)(Clade I)、玉簪属(Hosta)(Clade IV)和知母属(Anemarrhena)(Clade VI)植物各单独分为一支,与其他属的亲缘关系较远;龙舌兰属(Agave)、龙荟兰属(Beschorneria)和龙香玉属(Manfreda)植物聚为一支(Clade II);皂百合属(Chlorogalum)、糠米百合属(Camassia)、夕丽花属(Hesperocallis)、西丝兰属(Hesperoyucca)和草丝兰属(Hesperaloe)植物聚为一支(Clade III);拟菝葜属(Behnia)和吊兰属(Chlorophytum)植物聚为一支(Clade V),虎尾兰属(Sansevieria)和龙血树属(Dracaena)植物聚为一支(Clade VII),说明聚为一支的各属植物之间亲缘关较近。在7种丝兰属植物中,系统发育进化树以100%的自荐值(Bootstrap values)支持无刺龙舌兰(Y. treculeana)与克雷塔罗丝兰(Y. queretaroensis)和西地格丝兰(Y. schidigera)聚为一小支,互为姊妹关系,说明三者的遗传距离较小,共同起源较接近。
3 讨论
研究表明,被子植物叶绿体基因组长度一般为120~220 kb,包含大约130个基因,LSC、SSC和IR区的长度分别为81~90、18~20和20~29 kb(Zhang et al.,2012)。本研究无刺龙舌兰叶绿体全基因组长度为157579 bp,总基因数为135个,共分为LSC、SSC和IR(包括IRa和IRb)的长度分别为85940、18279和26680 bp,与被子植物叶绿体基因组特征(Zhang et al.,2012)相似。假基因(通常用符号ψ表示)是基因组中与CDS相似的非功能基因组DNA拷贝,多存在于真核生物,于1977年在研究爪蟾核糖体5S RNA基因时被首次发现(Xie et al.,2019)。本研究发现,无刺龙舌兰叶绿体基因组LSC区的atpB基因第111位点的C突变为G,第462位点缺失1个A,导致atpB基因无法正常翻译成氨基酸序列,造成了atpB基因的假基因化。关于该基因的假基因化并未见相关报道,仅Malinova(2021)在研究月见草(Oenothera)质体突变体I-iota时发现atpB基因编码区的第11位点有一个携带腺嘌呤A的插入,而叶绿体基因中的翻译重编码可纠正atpB基因的移码突变。
碱基组成的差异会引起同义密码子的非均衡使用现象(胡晓艳等,2019)。研究表明,密码子第3位上的碱基突变不改变氨基酸类型且受选择压较小,因此,GC3常被用作分析密码子使用模式的重要依据(Tang et al.,2020)。本研究PR2-plot分析結果显示,无刺龙舌兰叶绿体CDS在密码子第3位上,T>A,C>G,与酸枣(胡晓艳等,2019)、向日葵(Helianthus annuus)(Chen et al., 2021)等植物的研究结果一致,但与苏铁(Cycas revoluta)(Zhou et al.,2008)、灯盏花(李显煌等,2021)等植物的研究结果存在差异,即苏铁和灯盏花叶绿体CDS序列在密码子第3位上出现了T>A,C>G且A、T与 G、C的使用频率相当的情况,存在差异的原因可能与植物进化速率及环境型有关。对多数植物而言,自然选择和突变是造成其密码子偏好性的主要动力(Musto, 2016)。植物叶绿体基因组密码子使用偏好性虽受选择、突变等多重因素的影响,但主要影响因素只有1~2种(Zhang et al.,2018)。本研究利用中性绘图、ENC-plot和PR2-plot对造成无刺龙舌兰叶绿体基因组密码子偏好性的原因进行统计分析,结果发现突变和选择是导致无刺龙舌兰叶绿体基因组密码子偏性的主要影响因素,与龙舌兰属植物剑麻(金刚等,2018)的研究结论一致,说明选择和突变对密码子偏好性的影响在亲缘关系较近的植物中较一致,可能与物种的遗传进化有关。沈宗芳等(2021)在3种槲蕨属植物叶绿体基因组密码子偏好性分析研究中也得出相同结论。另外,关于物种遗传距离远近与密码子使用偏好性之间是否存在一定关系的问题,各学者所持观点不同(Tang et al.,2021),如Somaratne等(2019)通过对2种亲缘关系较近的胡枝子属(Desmodieae)植物叶绿体基因组进行序列差异及密码子使用模式分析,结果发现亲缘关系远近与密码子使用模式相关,即亲缘关系越近,其密码子使用模式也会越相似。目前,有关丝兰属植物密码子使用偏好性的研究较少,探究密码子使用模式与物种的亲缘关系是否存在相关性还需要对属内各物种的密码子偏性进行深入研究。
大量研究表明,植物叶绿体基因组具有结构完整性和序列保守性,因此,在植物分类、系统进化的研究中优势突出(Parks et al.,2009)。目前,直接利用叶绿体基因组构建龙舌兰科植物的系统发育进化树的研究报道较少,仅Kate和Mark(2013)对蛇纹石土壤上特有植物进行分类研究时,首次利用叶绿体基因组对龙舌兰科部分植物进行系统进化关系探究。另外,在龙舌兰科植物自花授粉起源的研究中,McKain等(2016)基于叶绿体基因组构建了最大似然(ML)树和贝叶斯(Bayes)樹,初步明确了属间的授粉起源关系,所构建的系统发育进化树显示西地格丝兰和克雷塔罗丝兰为姊妹关系,且同与短叶丝兰聚为丝兰属一支,无论是丝兰属的4种植物间,还是其余各属之间,其分支结果均与本研究的系统发育进化树分支结果一致。因此,在物种的分类鉴定及确定各物种间系统发育关系的研究中,基于植物叶绿体基因组构建系统发育进化树是一种准确、可靠的方法。
4 结论
无刺龙舌兰叶绿体基因组为保守的四分体结构,叶绿体基因组密码子偏好性较弱,主要受选择和突变等多因素影响。基于植物叶绿体基因组构建系统发育进化树在物种的分类鉴定及确定各物种间系统发育关系的研究中是一种准确、可靠的方法。
参考文献:
段义忠,张凯. 2020. 沙冬青属植物叶绿体基因组对比和系统发育分析[J]. 西北植物学报,40(8):1323-1332. [Duan Y Z,Zhang K. 2020. Comparative analysis and phylogenetic evolution of the complete chloroplast genome of Ammopiptanthus[J]. Northwestern Journal of Botany,40(8):1323-1332.]doi:10.7606/j.issn.1000-4025.2020.08. 1323.
高亚芳,刘莹莹,杨从卫,李国栋,钱子刚. 2019. 金铁锁叶绿体基因组序列及其系统发育分析[J]. 中草药,50(22):5532-5536. [Gao Y F,Liu Y Y,Yang C W,Li G D,Qian Z G. 2019. Analysis on structure and phylogeny of complete chloroplast genomes in Psammosilene tunicoides[J]. Chinese Herbal Medicine,50 (22):5532-5536.]doi:10.7501/j.issn.0253-2670.2019.22.022.
惠小涵,程婷婷,柯卫东,郭宏波. 2020. 莲藕PPO基因密码子偏好性特征分析[J]. 江苏农业学报,36(2):438-446. [ Hui X H,Cheng T T,Ke W D,Guo H B. 2020. Analysis on codon preference of PPO gene in lotus root[J]. Jiangsu Journal of Agricultural Sciences,36(2):438-446.]doi:10.3969/j.issn.1000-4440.2020.02.026
胡晓艳,许艳秋,韩有志,杜淑辉. 2019. 酸枣叶绿体基因组密码子使用偏性分析[J]. 森林与环境学报,39(6):621-628. [Hu X Y,Xu Y Q,Han Y Z,Du S H. 2019. Codon usage bias analysis of the chloroplast genome of Ziziphus jujuba var. Spinosa[J]. Journal of Forest and Environment,39(6):621-628.] doi:10.13324/j.cnki.jfcf.2019. 06.009.
金刚,覃旭,龙凌云,王丽萍,覃剑峰,危丹妮,陈涛,蔡中全. 2018. 剑麻叶绿体基因组编码序列密码子的使用特征[J]. 福建农林大学学报(自然科学版),47(6):705-710. [Jin G,Qin X,Long L Y,Wang L P,Qin J F,Wei D N,Chen T,Cai Z Q. 2018. Characteristics of codon usage in the chloroplast protein-coding genes of Agave hybrid No.11648[J]. Journal of Fujian Agriculture & Forestry University (Natural Science Edition),47(6):705-710.] doi:10.13323/j.cnki.j.fafu(nat.sci.).2018.06.011.
李江飞,原晓龙,李熙颜,王瑜,蔡年辉,陈诗,许玉兰. 2021. 永椿香槐叶绿体基因组密码子偏好性分析[J]. 分子植物育种,https://kns.cnki.net/kcms/detail/46.1068.S.2021 0630.1627.017.html. [Li J F,Yuan X L,Li X Y,Wang Y,Cai N H,Chen S,Xu Y L. 2021. Analysis on codon usage bias of chloroplast genome in Cladrastis yungchunii[J]. Molecular Plant Breeding,https://kns.cnki.net/kcms/detail/46.1068.S.20210630.1627.017.html.]
李显煌,杨生超,辛雅萱,屈亚亚,杨琳懿,冯发玉,范伟,辛培尧. 2021. 灯盏花叶绿体基因组密码子偏好性分析[J]. 云南农业大学学报(自然科学),36(3):384-392. [Li X H,Yang S C,Xin Y X,Qu Y Y,Yang L Y,Feng F Y,Fan W,Xin P Y. 2021. Analysis of the codon usage bias of chloroplast genome in Erigeron breviscapus(Vant.) Hand-Mazz[J]. Journal of Yunnan Agricultural University(Natural Science),36 (3):384-392.] doi:10.12101/j.issn. 1004-390X(n).202010007.
牛元,徐瓊,王嵛德,代立兰,庄健,赵亚兰. 2018. 大花香水月季叶绿体基因组密码子使用偏性分析[J]. 西北林学院学报,33(3):123-130. [Niu Y,Xu Q,Wang Y D,Dai L L,Zhuang J,Zhao Y L. 2018. An analysis on codon usage bias of the chloroplast genome of Rosa odorata var. Gigantea[J]. Journal of Northwest Forest College,33 (3):123-130.] doi:10.3969/j.issn.1001-7461.2018.03.19.
屈亚亚,辛静,冯发玉,董章宏,瞿绍宏,王海洋,李斌,辛培尧. 2021. 香花枇杷质体基因组序列密码子偏性分析[J]. 西北林学院学报,5(4):1-8. [Qu Y Y,Xin J,Feng F Y,Dong Z H,Qu S H,Wang H Y,Li B,Xin P Y. 2021. Condon usage bais in chloroplast genome of Eriobotrya fragrans Champ. Ex Benth[J]. Journal of Northwest Forest College,5(4):1-8.] doi:10.3969/j.issn.1001-7461.2021.04.20.
沈宗芳,陆添权,张志荣, 蔡传涛,田波. 2021. 槲蕨属叶绿体基因组密码子偏好性分析[J]. 广西植物,41(2):266-273. [Shen Z F,Lu T Q,Zhang Z R,Cai Z T,Tian B. 2021. Condon bias of chloroplast genome of Drynaria[J]. Guangxi Plant,41(2):266-273.] doi:10.11931/guihaia.gxzw201904013.
辛雅萱,黎若竹,李鑫,陈丽琼,唐军荣,屈亚亚,杨琳懿,辛培尧,李云芳. 2021. 杧果叶绿体基因组密码子使用偏好性分析[J]. 中南林业科技大学学报,(9):148-156. [Xin Y X,Li R Z,Li X,Chen L Q,Tang J R,Qu Y Y,Yang L Y,Xin P Y,Li Y F. 2021. Analysis on codon usage bias of chloroplast genome in Mangifera indica[J]. Journal of Central South University of Forestry and Technology,(9):148-156.] doi:10.14067/j.cnki.1673-923x.2021.09.016.
原晓龙,刘音,康洪梅,陈中华,李云琴,王毅. 2021. 蒜头果叶绿体基因组密码子偏好性分析[J]. 西南林业大学学报(自然科学),41(3):15-22. [Yuan X L,Liu Y,Kang H M,Chen Z H,Li Y Q,Wang Y. 2021. Analysis of codon usage bias in chloroplast genome of Malania oleifera[J]. Journal of Southwest Forestry University (Natural Sciences),41(3):15-22.] doi:10.11929/j.swfu.2020 01047.
Chen S Y,Zhang H,Wang X,Zhang Y H,Ruan G H,Ma J. 2021. Analysis of codon usage bias in the chloroplast genome of J-01[J]. IOP Conference Series:Earth and Environmental Science,792(1):1755. doi:10.1088/1755-1315/792/1/012009.
Irish G. 2000. Agaves,Yuccas,and related plants:A gardener’s guide[M]. Portland :Timber Press.
Jansen R K,Uhlman R. 2012. Plastid genomes of seed plants. genomics of chloroplasts and mitochondria[M]. Dordrecht:Springer.
Jiang Y,Deng F,Wang H L,Hu Z H. 2008. An extensive analysis on the global codon usage pattern of baculoviruses[J]. Archives of Virology,153(12):2273. doi:10. 1007/s00705-008-0260-1.
Jia-Yee S,Thore R,Abigail G,Hannah M P,Wendy G,Annie Y,Marc R,Sven K D. 2015. Complete chloroplast genome of the Wollemi Pine (Wollemia nobilis ):Structure and evolution[J]. Public Library of Science,10(6):126-128. doi:10.1371/journal.pone.0128126.
Jin J J,Yu W B,Yang J B,Song Y,Yi T S,Li D Z. 2020. GetOrganelle:A fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology,21(1):241. doi:10.1186/s13059-020-02154-5.
Kai J,Song X Q,Chen C G. 2020. Coons for the chloroplast genome of the Magnoliaceae use trait analysis[J]. China Agricultural Science and Technology Herald,22(11):52-62.
Kate H,Mark F. 2013. A chloroplast phylogeny of agavaceae subfamily Chlorogaloideae:Implications for the Tempo of Evolution on Serpentine Soils[J]. Systematic Botany,38(4):674. doi:10.1600/036364413X674850.
Katoh K,Standley D M. 2013. MAFFT multiple sequence alignment software version 7:Improvements in performance and usability[J]. Molecular Biology and Evolution,30(4):772-780. doi:10.1093/molbev/mst010.
Kearse M,Moir R,Wilson A. 2012. Geneious Basic:An integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics,28(12):1647-1649. doi:10.1093/bioinformatics/bts199.
Li G L,Pan Z L,Gao S C,He Y Y,Xia Q Y,Jin Y,Yao H P. 2019. Analysis of synonymous codon usage of chloroplast genome in Porphyra umbilicalis[J]. Genes Geno-mics,10(41):1173-1181. doi:10.1007/s13258-019-00847-1.
Lohse M,Drechsel O,Kahlau S. 2013. Organellar genome DRAW—A suite of tools for generating physical maps of plastid and mitochondrial genomes and visualizing expression data sets[J]. Nucleic Acids Research,(41):575-581. doi:10.1093/nar/gkt289.
Long S,Yao H,Wu Q,Li G L. 2018. Analysis of compositional bias and codon usage pattern of the coding sequence in Banna virus genome[J]. Virus Research,(10):1016. doi:10.1016/j.virusres.2018.10.006.
Malinova I,Zupok A,Massouh A,Meyer E H,Greiner S. 2021. Correction of frameshift mutations in the atpB gene by translational recoding in chloroplasts of Oenothera and tobacco[J]. The Plant Cell,33(5):1-24. doi:10.1093/PLCELL/KOAB050.
McKain M R,McNeal J R,Kellar P R. 2016. Timing of rapid diversification and convergent origins of active pollination within Agavoideae(Asparagaceae)[J]. American Journal of Botany,103(10):1717-1729.
Musto H. 2016,What we know and what we should know about codon usage[J]. Journal of Molecular Evolution,82(6):245-246. doi:10.1007/s00239-016-9742-z.
Parks M,Cronn R,Liston A. 2009. Increasing phylogenetic resolution at low taxonomic levels using massively parallel sequencing of chloroplast genomes[J]. BMC Developmental Biology,12(7):84.
Somaratne Y,Guan D L,Wang W Q,Zhao L,Xu S Q. 2019. The complete chloroplast genomes of two Lespedeza species:Insights into codon usage bias,RNA editing sites,and phylogenetic relationships in Desmodieae (Fabaceae:Papilionoideae)[J]. Plants,9(1):3390. doi:10.3390/plants 9010051.
Stamatakis A,Hoover P,Rougemont J. 2008. A rapid bootstrap algorithm for the RAxML web servers[J]. Systema-tic Biology,57(5):758-771. doi:10.1080/1063515080242 9642.
Sueoka N. 2017. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phylogenetic analyses[J]. Journal of Molecular Evolution,19(53):469-476.
Tang D F,Wei F,Cai Z Q,Wei Y Y,Khan A,Miao J H,Wei K H. 2020. Analysis of codon usage bias and evolution in the chloroplast genome of Mesona chinensis Benth[J].Development Genes and Evolution,(10):1007. doi:10. 1007/s00427-020-00670-9.
Tang Y J,Zhao Y,Huang G D. 2021. Mangifera indica L. chloroplast genome codon using preference analysis[J]. Journal of Tropical Crop Science,7:1-11.
Tao X L,Ma L C,Zhang Z S,Liu W X,Liu Z P. 2017. Characterization of the complete chloroplast genome of alfalfa (Medicago sativa) (Leguminosae)[J]. Gene Reports,(6):1-6. doi:10.1016/j.genrep.2016.12.006.
Tillich M,Lehwark P,Pellizzer T,Ulbricht-Jones E S,Fischer A,Bock R,Greiner S. 2017. GeSeq-versatile and accurate annotation of organelle genomes[J]. Nucleic Acids Research,45(7):6-11. doi:10.1093/nar/gkx391.
Wang H,Meng T,Wei W. 2018. Analysis of synonymous codon usage bias in helicase gene from Autographa californica multiple nucleopolyhedrovirus[J]. Genes & Genomi-cs,(40):767-780. doi:10.1007/s13258-018-0689-x.
Wei L,He J,Jia X,Qi Q. 2014. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution[J]. BMC Evolutionary Biology,(14):262. doi:10.1186/s12862-014-0262-4.
Windarti C K,Maryanto A E,Salamah A. 2021. The study of heat shock protein 70 (Hsp70) gene expression on Agera-tum conyzoides L. and Synedrella nodiflora L. in Universitas Indonesia,Depok and Cibodas Botanical Garden[J]. Journal of Physics:Conference Series,1725(1):012059. doi:10.1088/1742-6596/1725/1/012059.
Wu M L,Chen S P,Chen H. 2019. Condon preference of chloroplast genome of Bambusoideae[J]. Journal of Environmental Quality,(1):9-14. doi:10.13324/j.cnki.jfcf.2019.01.002.
Xie J B,Li Y,Liu X M,Zhao Y Y,Li B L,Ingvarsson P K,Zhang D Q. 2019. Evolutionary origins of pseudogenes and their association with regulatory sequences in plants[J]. The Plant Cell,31(3):601-619. doi:10.1105/tpc. 18.00601.
Xu C,Cai X,Chen Q. 2011. Factors affecting synonymous codon usage bias in chloroplast genome of Oncidium gower ramsey[J]. Evolutionary Bioinformatics,10(7):271-278. doi:10.4137/EBO.S8092.
Yang G F,Su K L,Zhao Y R. 2015. Analysis of codon usage in the chloroplast genome of Medicago truncatula[J]. Acta Pharmacologica Sinica,6(12):171-179.
Zhang R,Zhang L,Wang W. 2018. Differences in codon usage bias between photosynthesis related genes and genetic system-related genes of chloroplast genomes in cultivated and wild Solanum species[J]. International Journal of Molecular Sciences,(19):3142. doi:10.3390/ijms 19103142.
Zhang T W,Fang Y J,Wang X M,Deng X,Zhang X W,Hu S N,Yu J. 2012. The complete chloroplast and mitochondrial genome sequences of Boea hygrometrica:Insights into the evolution of plant organellar genomes[J]. Public Library of Science,7(1):e30531. doi:10.1371/journal.pone.0030531.
Zhou M,Long W,Li X. 2008. Patterns of synonymous codon usage bias in chloroplast genomes of seed plants[J]. Fore-stry Studies in China,10(4):235-242. doi:10.1007/s11632-008-0047-1.
收稿日期:2021-08-24
基金項目:云南省科技厅科技计划重点研发项目(2018BB005);西南林业大学科技创新基金项目(KY21034)
通讯作者:辛培尧(1975-),https://orcid.org/0000-0001-8512-7083,教授,主要从事植物遗传育种与快繁研究研究工作,E-mail:xpytgyx@163.com
第一作者:王飞(1996-),https://orcid.org/ORCID:0000-0002-9117-6620,主要从事林木分子生物学与遗传改良研究,E-mail:1767785429@qq.com
