河南省审定小麦品种旗叶性状全基因组关联分析
2022-06-24曹廷杰周艳杰杨剑张玉娥胡卫国王西成赵虹
曹廷杰 周艳杰 杨剑 张玉娥 胡卫国 王西成 赵虹






摘要:为解析河南省育成小麦品种旗叶性状的遗传基础,以河南省审定的96个小麦品种为材料,2019年在河南省南阳市、安阳市、新乡市原阳县3个环境下对旗叶长(FLL)、旗叶宽(FLW)和旗叶长宽比(FLFW)进行鉴定,并利用小麦90K 单核苷酸多态性(SNP)芯片进行全基因组关联分析。结果表明,旗叶长和旗叶长宽比在3个环境中均呈极显著正相关关系(r值分别为0.800、0.799、0.729);旗叶宽与旗叶长宽比之间呈极显著负相关关系(r值分别为 -0.334、-0.597、-0.606)。筛选到6个旗叶较宽(开麦21、中育9398、豫农202、花培1号、郑育麦9987、郑麦583)和7个旗叶较长(温9629、郑农16、豫农201、新麦9号、平麦998、豫麦34、豫农202)的小麦品种。检测到59个与旗叶显著关联的SNPs位点,其中与旗叶长显著关联的SNPs有8个,分别位于2B、2D、4A、4B、7B染色体上,可解释11.96%~15.43%的表型变异;与旗叶宽显著关联的SNPs有41个,分别位于1A、2A、2B、3B、3D、4B、5A、5B、6A、6B、6D、7B染色体上,可解释12.18%~21.57%的表型变异;与旗叶长宽比显著关联的SNPs有10个,分别位于2B、4A、6B染色体上,可解释12.85%~15.60%的表型变异;6B染色体上的Ku_c32100_105位点同时与旗叶宽和旗叶长宽比显著关联。经比较发现被定位在2B染色体上的位点可能是1个新的位点,研究结果为从遗传水平揭示小麦旗叶发育提供了重要的参考。
关键词:小麦品种;旗叶性状;相关性;全基因组关联分析;河南省
中图分类号:S512.103.2 文献标志码:A
文章编号:1002-1302(2022)11-0053-10
收稿日期:2021-08-13
基金项目:河南省小麦产业技术体系项目(编号:S2010-01-G03)。
作者简介:曹廷杰(1977—),男,河南南阳人,博士,副研究员,主要从事小麦育种及重要性状遗传分析研究。E-mail:caotingeji893@163.com。
小麦旗叶产生的同化物是籽粒灌浆期积累干物质的主要来源,对小麦产量起着决定性作用。旗叶为植株最上层的叶片,与其他叶片相比,旗叶叶绿体细胞最多,叶绿体中基粒类囊体数量多,光合作用强度最高。此外,旗叶作为源器官,距离穗部(库)最近,因而物质运输效率最高],对小麦穗粒数和千粒质量贡献最大。小麦旗叶长和宽与产量呈现极显著正相关关系,因此,改良旗叶性状对提升小麦产量具有重要意义。小麦旗叶性状受数量性状位点(QTL)控制,前人已定位了多个控制旗叶性状的QTLs。姚俭昕等利用90K小麦SNP芯片对小偃81、西农1376构建的重组自交系群体进行旗叶长QTL定位,检测到2个QTLs均位于5A染色体上。Tu等利用20828/SY95-71重组自交系群体进行QTL定位,定位到控制旗叶长和旗叶宽的QTLs分别位于2B(2个)、5B和2B、2D染色体上,且位于2B染色体上的2个QTLs不重叠。Liu等利用ND3331和Zang1817的重组自交系群体进行定位,分别定位到6个控制旗叶长和2个控制旗叶宽的QTLs,可解释4.62%~14.70%的表型变异,其中QFLW-4B.1和QFLW-4B.2同时控制2个性状。
小麦基因组大小约为15 GB,且含有3套部分同源染色体组,利用重组自交系进行QTL定位所需时间较长,且能获得的重组频率非常低,导致定位精度较低。全基因组关联分析是一种基于连锁不平衡的定位方法,该方法直接利用自然群体材料进行基因型检测、表型鉴定以及遗传分析,省时省力且定位更精准,目前已开发了9K、90K、660K等多种小麦SNP芯片,显著提高了全基因组关联分析的分辨率。全基因组关联分析已广泛应用于解析小麦复杂数量性状的遗传机制,如籽粒性状、穗发芽抗性、小花育性相关性状、抗逆性、抗病性等,而目前利用全基因组关联分析解析旗叶性状的报道较少。河南省是我国小麦主产区,小麦育种和小麦生产在全国均占有重要地位,小麦种植区域广泛,小麦类型多样。本研究以2000—2010年河南省审定的96个小麦品种为材料进行旗叶性状的全基因组关联分析,解析其遗传机制,为小麦旗叶性状的遗传改良提供依据。
1 材料与方法
1.1 试验材料
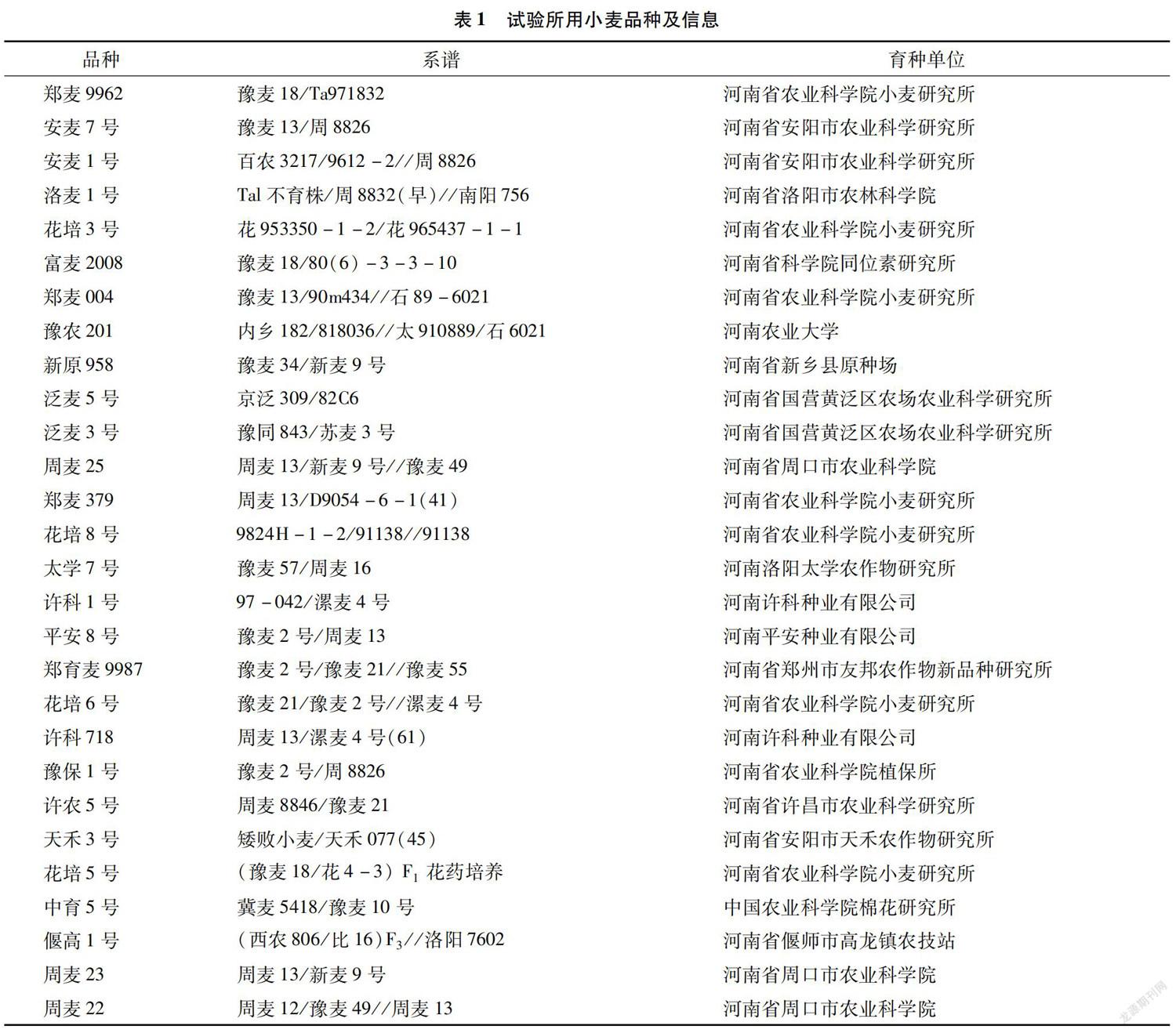
材料为河南省审定的96个小麦品种,其中包括26个弱春性品种和70个半冬性品种(表1),2019年分别于河南省南阳市(NY)、安阳市(AY)、新乡市原阳县(YY)3个环境下种植,试验采取随机区組设计,每小区种植2行,行长为2 m,行距为0.3 m,株距为0.3 m,重复2次,按照国家区域试验标准进行田间试验管理。
1.2 表型鉴定
在小麦灌浆初期,每小区随机选取10个单株主茎测量旗叶长(FLL)和旗叶宽(FLW),同时计算旗叶长宽比(旗叶长/旗叶宽,FLFW),取平均值作为每个小区的观测值。
1.3 表型数据的统计分析
将环境作为随机效应,利用Lme4软件包混合线性模型在多环境下进行最佳线性无偏预测(BLUP)。
Y=G+L+G×L+R/L+e。
其中:Y为表型观测值;G是品种效应;L是环境效应;R表示区组效应;e表示残差。
估计各个效应的方差组分,用于计算各个性状的广义遗传力(H),计算公式如下:
H=σσ+σl+σrl×100%。
其中:σ表示基因型方差;σ是基因型与环境间互作的方差;σ是残差组分;r是重复次数;l是环境数。
1.4 全基因组关联分析
使用TASSEL软件的混合线性(Q+K)模型(compressed mixed linear model,简称MLM)对3个性状的BLUP值进行全基因组关联分析,基因分型结果、分子标记的物理位置、群体结构、亲缘关系矩阵K值以及群体结构Q值均已在之前的研究中进行了分析。
在计算时对每个标记的效应进行估计,标记阈值-lg P≥3.0作为显著位点。
2 结果与分析
2.1 表型数据分析
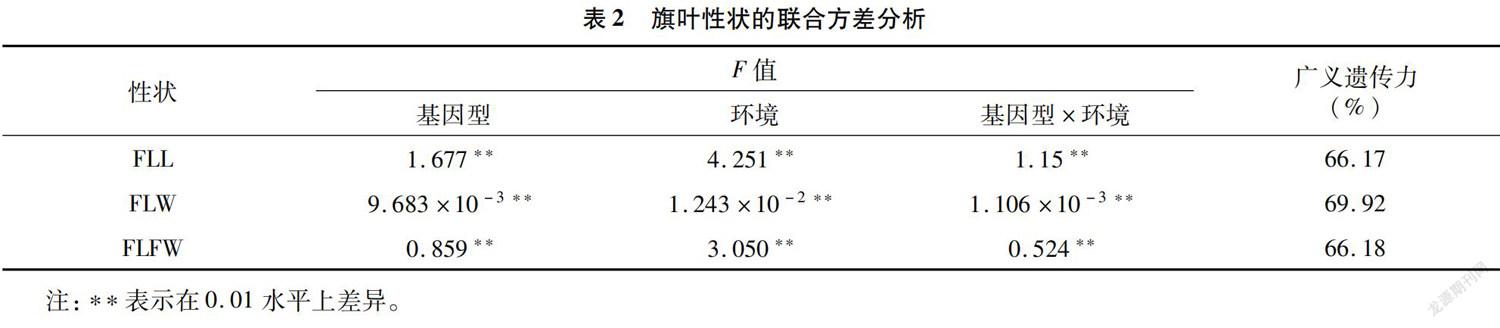
利用混合线性模型计算出3个性状(旗叶长、旗叶宽和旗叶长宽比)中基因型、环境及相互之间互作的变异(表2),经计算3个性状的广义遗传力基本相当,分别为66.17%、69.92%、66.18%。
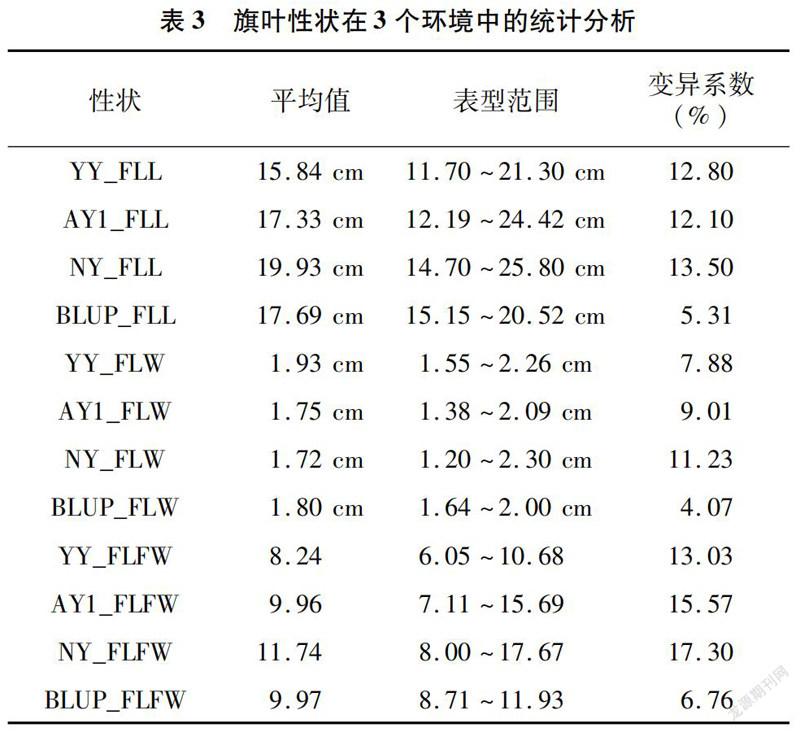
由表3可知,96个供试小麦品种的旗叶长、旗叶宽和长宽比在不同环境下均存在差异,其中南阳试验点旗叶长和长宽比平均值最高,分别为19.93 cm、11.74,且变异系数最大,分别为13.5%、17.30%,原阳试验点旗叶宽平均值最高,为1.93 cm。3个性状均为数量性状,在不同环境中均呈正态或近似正态的连续分布(图1)。
在供试小麦材料中,开麦21、中育9398、豫农202、花培1号、郑育麦9987和郑麦583号的旗叶较宽,平均值均大于2 cm(分别为2.15、2.08、2.03、2.02、2.02、2.01 cm)。温9629、郑农16、豫农201、新麦9号、平麦998、豫麦34和豫农202旗叶较长,平均值均大于20 cm(分别为22.69、21.50、21.22、20.96、20.56、20.27、20.05 cm)。
由表4可知,旗叶长、旗叶长宽比在原阳和安阳试验点之间、安阳和南阳试验点之间呈极显著正相关关系,旗叶宽在3个试验点之间均呈显著正相关关系。原阳试验点中每2个性状之间均达到极显著相关性,旗叶长和旗叶宽、旗叶长宽比之间呈极显著正相关关系,旗叶宽和旗叶长宽比之间呈极显著负相关关系;在安阳和南阳试验点中,旗叶长和旗叶宽之间相关性均不显著,旗叶长和旗叶长宽比之间均呈极显著正相關关系,旗叶宽和旗叶长宽比之间均呈极显著负相关关系。
2.2 全基因组关联分析
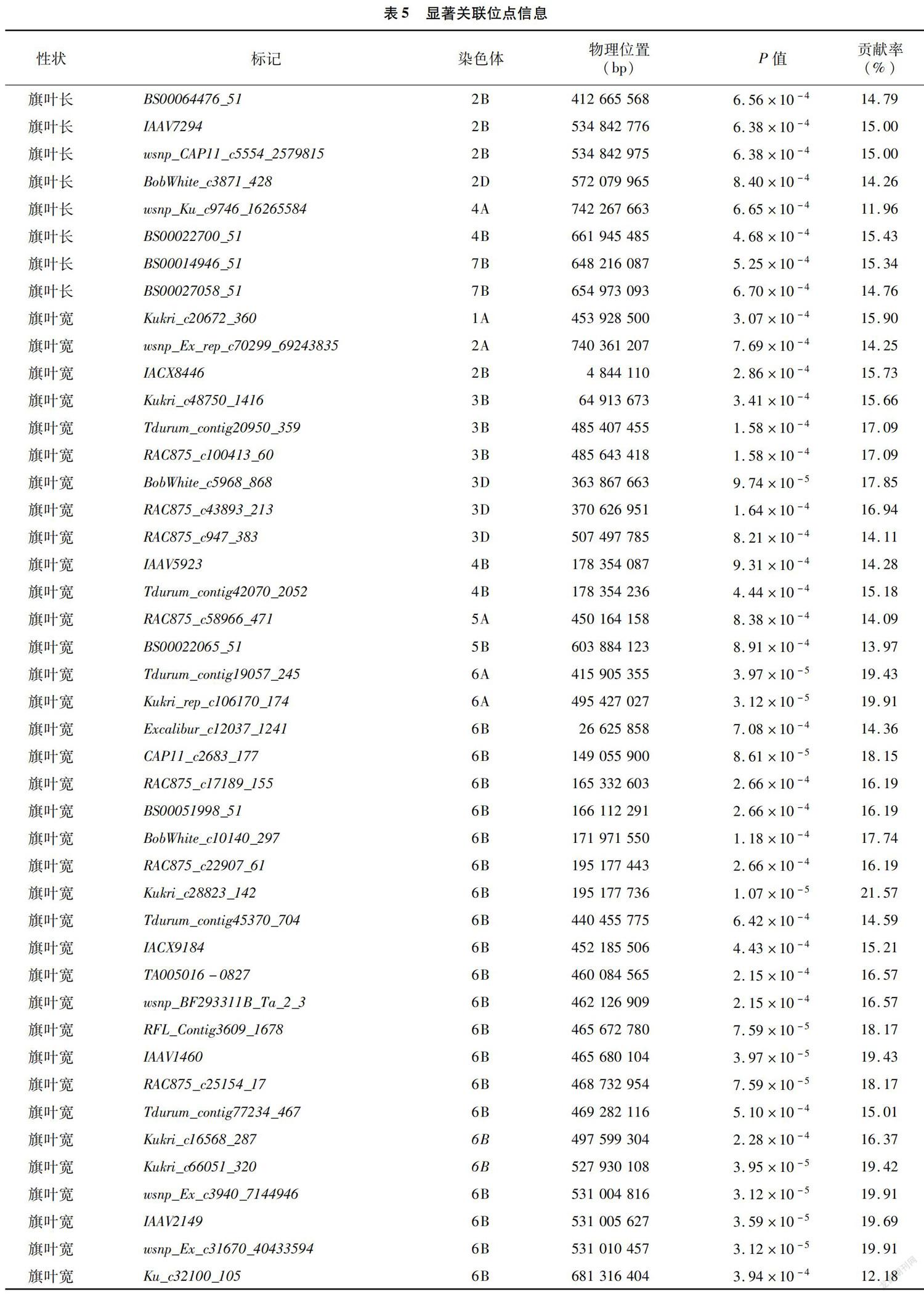
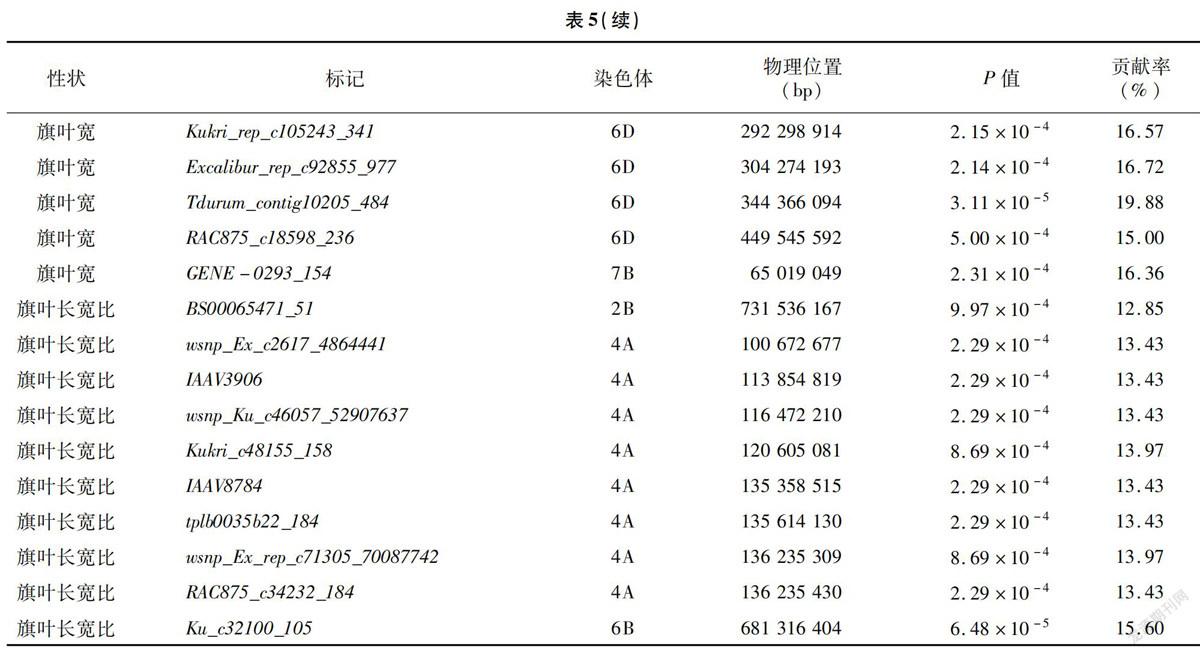
由表5可知,利用小麦90K SNP芯片对3个性状进行全基因组关联分析,分别检测到8、41、10个分别与旗叶长、旗叶宽和旗叶长宽比显著关联的SNPs(P<0.001)。其中,在2B、2D、4A、4B、7B染色体上分别检测到3、1、1、1、2个SNPs与旗叶长显著关联,可解释11.96%~15.43%的表型变异(图2-a)。检测到与旗叶宽显著关联的SNPs分别位于1A(1个)、2A(1个)、2B(1个)、3B(3个)、3D(3个)、4B(2个)、5A(1个)、5B(1个)、6A(2个)、6B(21个)、6D(4个)和7B(1个)染色体上,可解释12.18%~21.57%的表型变异(图2-b)。与旗叶长宽比显著关联的SNPs分别位于2B(1个)、4A(8个)和6B(1个)染色体上,可解释12.85%~15.60%的表型变异(图2-c)。
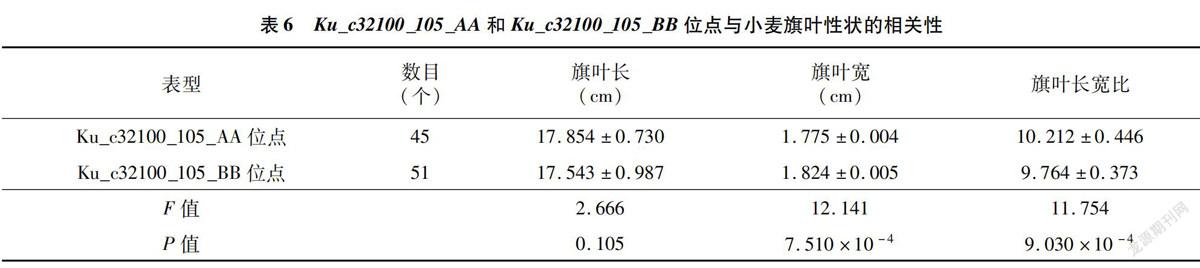
在检测到的显著关联的分子标记中,位于6B染色体上的Ku_c32100_105位点同时与旗叶宽和旗叶长宽比显著关联,Ku_c32100_105_BB基因型品种的旗叶比Ku_c32100_105_AA基因型的更宽,但Ku_c32100_105_AA基因型品种的旗叶长宽比值比Ku_c32100_105_BB基因型的更大(表6)。
3 结论与讨论
河南小麦在由低产向高产的转变过程中呈现旗叶变宽、长宽比变小的趋势。本研究对96份河南小麦品种进行研究发现旗叶长和旗叶宽表型分布广泛,这表明河南小麦品种的旗叶存在广泛的多样性,且部分品种仍存在改良的空间。本研究筛选出的多份旗叶性状表现优异的小麦品种,其中豫农202的旗叶长和旗叶宽在供试材料中均表现突出,具有用于小麦旗叶遗传改良的潜力。
Ma等利用双亲分离群体分析表明旗叶长和旗叶宽的遗传力分别为60%、66%,本研究在自然群体中的计算结果与之基本一致,说明尽管旗叶性状受到环境的影响,但是遗传因素仍为主要因素。此外,旗叶宽和旗叶长宽比之间、旗叶长和旗叶长宽比之间存在显著的相关性,这与前人的研究结果相吻合。但在本研究中旗叶长与旗叶宽之间
不存在显著的相关性,说明可以对旗叶形态进行相对独立的遗传改良。
复杂农艺性状的遗传解析是现代遗传学的热点,长久以来利用双亲分离群体定位到了大量的QTLs,但受限于亲本狭窄的遗传背景,难以完成复杂背景下广泛位点的鉴定。全基因组关联分析利用了搜集的自然群体作为遗传材料,在特定遗传结构的群体下可以检测出所有与性状相关的位点。
本研究中检测到8个与旗叶长显著关联的SNPs,其中位于2D染色体上的BobWhite_c3871_428可能与Liu等在2DL染色体臂上定位到的QTL位于同一染色体区段内。此外,在2B染色体上的3个SNPs位于412.67~534.84 Mb物理区间内,Yan等利用关联分析也在2BL染色体臂上检测到1个显著性位点,但其位置与本研究中显著关联的SNPs相距约100 Mb,因此推测二者并不是同一个QTL。
控制小麦旗叶宽的QTL主要位于小麦第2和第5同源群上,姚俭昕等定位到的旗叶宽QTLs侧翼分子标记对应于中国春5A染色体上约500 Mb的位置,与本研究中检测到位于5A染色体上的位点相距约50 Mb,检测到定位于2A染色体上的旗叶宽QTL与NAL1基因在小麦中的直系同源基因相距约80 Mb,NAL1基因在水稻中已表明参与调控旗叶宽,由于受限于关联群体的分辨率,尚难以辨别是否为同一个QTL。5B染色体上发掘的控制旗叶宽的位点位于中国春参考基因组约604 Mb的位置,连俊方等在河南小麦骨干亲本周8425B中定位到位于5B染色体上控制旗叶宽的QTLQflw-5B,该QTL位于分子标记wsnp_Ku_c3869_7094615和BS00078572_51之间,对应于中国春437.6~698.2Mb的物理区间,刘朦朦等也在5B染色体相似区段鉴定到控制旗叶宽的QTL,其优势单倍型在河南省小麦中选择运用较高,这2个QTLs与本研究中5B染色体上控制旗叶宽的位点位置接近,而本研究中亚群3和亚群4为周麦13(周8425B/周麦9号)或周麦16(周麦9号/周8425B)的后代,因此推测河南小麦旗叶宽遗传并选择应用了周8425B的Qflw-5B位点。此外,笔者在7B染色体短臂65.02 Mb处定位到1个旗叶宽相关的显著SNP位点,经比对该SNP与之前该群体定位到的1个与产量相关的QTL物理位置(61.82 Mb)接近,该位点可能通过调控旗叶宽来提升小麦产量。目前在2B染色体上所定位的旗叶宽QTLs均位于长臂上,本研究中检测到与旗叶宽显著关联的SNP位点IACX8446位于2B染色体短臂末端,推测可能是1个新的位点。
本研究在6B染色体上检测到1个同时调控旗叶宽和旗叶长宽比的位点Ku_c32100_105,该位点在调控2个性状中具有相反的效应,因此,通过选择该位点以增加旗叶宽的同时可以有效降低旗叶长宽比,进一步图位克隆该基因对解析小麦旗叶发育具有重要意義。
参考文献:
[1]李春喜,韩 蕊,邵 云,等. 小麦开花期旗叶光合特性与地上部干物质量的相关和通径分析[J]. 江苏农业科学,2019,47(6):66-70.
[2]姜思彤,苏 娜, 傅兆麟. 小麦旗叶叶绿素含量的时空差异性分析[J]. 黑龙江农业科学,2018(10):22-26.
[3]钮力亚,王伟伟,王 伟,等. 小麦功能叶对产量及其构成因素的影响[J]. 作物研究,2018,32(4):295-298.
[4]傅兆麟,马宝珍,王光杰,等. 小麦旗叶与穗粒重关系的研究[J]. 麦类作物学报,2001,21(1):92-94.
[5]姚俭昕,张传量,宋晓朋,等. 基于90K芯片的小麦穗长和旗叶长QTL分析[J]. 麦类作物学报,2020,40(11):1283-1289.
[6]Tu Y,Liu H,Liu J J,et al. QTL mapping and validation of bread wheat flag leaf morphology across multiple environments in different genetic backgrounds[J]. Theoretical and Applied Genetics,2021,134(1):261-278.
[7]Liu K Y,Xu H,Liu G,et al. QTL mapping of flag leaf-related traits in wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics,2018,131(4):839-849.
[8]Consortium I W G S,Investigators I R P,Appels R,et al. Shifting the limits in wheat research and breeding using a fully annotated reference genome[J]. Science,2018,361(6403):7191.
[9]Cavanagh C R,Chao S,Wang S C,et al.Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars[J]. Proceedings of the National Academy of Sciences of the United States of America,2013,110(20):8057-8062.
[10]Wang S C,Wong D,Forrest K,et al. Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array[J]. Plant Biotechnology Journal,2014,12(6):787-796.
[11]Rasheed A,Xia X C,Ogbonnaya F,et al. Genome-wide association for grain morphology in synthetic hexaploid wheats using digital imaging analysis[J]. BMC Plant Biology,2014,14:128.
[12]Zhou Y,Tang H,Cheng M P,et al. Genome-wide association study for pre-harvest sprouting resistance in a large germplasm collection of Chinese wheat landraces[J]. Frontiers in Plant Science,2017,8:401.
[13]Guo Z F,Chen D J,Alqudah A M,et al. Genome-wide association analyses of 54 traits identified multiple loci for the determination of floret fertility in wheat[J]. New Phytologist,2017,214(1):257-270.
[14]時晓磊,严勇亮,石书兵,等. 小麦根部耐盐性状全基因组关联分析[J]. 植物遗传资源学报,2021,22(1):57-73.
[15]朱展望. 利用全基因组连锁分析和关联分析定位小麦赤霉病抗性基因及分子标记开发[D]. 北京:中国农业科学院,2020.
[16]Bates D,Mächler M,Bolker B,et al. Fitting linear mixed-effects models using lme4[J]. Journal of Statistical Software,2015,67(1):1-48.
[17]Piepho H P,Möhring J. Computing heritability and selection response from unbalanced plant breeding trials[J]. Genetics,2007,177(3):1881-1888.
[18]Bradbury P J,Zhang Z W,Kroon D E,et al. TASSEL:software for association mapping of complex traits in diverse samples[J]. Bioinformatics,2007,23(19):2633-2635.
[19]Yang J,Zhou Y J,Hu W G,et al. Unlocking the relationships among population structure,plant architecture,growing season,and environmental adaptation in Henan wheat cultivars[J]. BMC Plant Biology,2020,20:469.
[20]雷振生,林作楫. 黄淮麦区冬小麦合理株型结构研究[J]. 华北农学报,1994,9(4):27-32.
[21]Ma J A,Tu Y,Zhu J,et al.Flag leaf size and posture of bread wheat:genetic dissection,QTL validation and their relationships with yield-related traits[J]. Theoretical and Applied Genetics,2020,133(1):297-315.
[22]Huang X H,Han B.Natural variations and genome-wide association studies in crop plants[J]. Annual Review of Plant Biology,2014,65:531-551.
[23]Liu Y X,Tao Y,Wang Z Q,et al. Identification of QTL for flag leaf length in common wheat and their pleiotropic effects[J]. Molecular Breeding,2017,38(1):1-11.
[24]Yan X F,Zhao L,Ren Y,et al. Identification of genetic loci and a candidate gene related to flag leaf traits in common wheat by genome-wide association study and linkage mapping[J]. Molecular Breeding,2020,40(6):1-15.
[25]王 芳. 控制我国小麦旗叶性状的遗传位点及候选基因的功能分析[D]. 郑州:河南农业大学,2016.
[26]Fan X L,Cui F,Zhao C H,et al. QTLs for flag leaf size and their influence on yield-related traits in wheat (Triticum aestivum L.)[J]. Molecular Breeding,2015,35(1):1-16.
[27]常 鑫,李法計,张兆萍,等. 小麦旗叶长、宽及面积的QTL分析[J]. 西北植物学报,2014,34(5):896-901.
[28]王 芳,宋艳红,阳文龙,等. 小麦旗叶宽相关基因TaNAL1-5的克隆与功能分析[J]. 分子植物育种,2016,14(5):1037-1048.
[29]Fujita D,Trijatmiko K R,Tagle A G,et al. NAL1 allele from a rice Landrace greatly increases yield in modern indica cultivars[J]. PNAS,2013,110(51):20431-20436.
[30]Zhang G H,Li S Y,Wang L,et al. LSCHL4 from Japonica cultivar,which is allelic to NAL1,increases yield of indica super rice 93-11[J]. Molecular Plant,2014,7(8):1350-1364.
[31]连俊方,张德强,武炳瑾,等. 利用90K基因芯片进行小麦旗叶相关性状的QTL定位[J]. 麦类作物学报,2016,36(6):689-698.
[32]刘朦朦,张萌娜,张倩倩,等. 小麦旗叶宽主效QTL QFlw-5B遗传效应解析[J]. 麦类作物学报,2019,39(12):1399-1405.
[33]王 盈,赵 磊,董中东,等. 小麦株高和旗叶相关性状的QTL定位[J]. 麦类作物学报,2019,39(7):761-767.
