抗体基因点突变、插入和缺失体外检测方法的建立及应用
2022-06-15罗思敏叶菱秀
罗思敏,叶菱秀,郝 茜
上海交通大学基础医学院免疫学与微生物学系,上海市免疫学研究所,上海 200025
抗体是由浆细胞分泌的免疫球蛋白,拥有2 条完全相同的重链和轻链。重链蛋白由可变(variable,V)、多样(diversity,D)、连接(joining,J)、恒定(constant,C)区的基因编码构成,其中胚系基因众多的V、D、J 片段需通过V(D)J 重排方可行使功能,如形成由低亲和力、单一类型抗体组成的初级抗体库。当初始B细胞受到外界抗原刺激时,其可从骨髓迁出进入脾脏、淋巴结和扁桃体等次级淋巴组织,经历体细胞高频突变(somatic hypermutation,SHM)和类别转换(class switch recombination,CSR),进一步产生由高亲和力、不同类型抗体组成的次级抗体库[1-2]。在SHM 过程中,激活诱导胞苷脱氨酶(activation-induced cytidine deaminase,AID)可作用于抗体可变区的基因序列,将序列中的胞苷残基脱氨变成尿苷,引发突变事件。研究[3]发现,AID 倾向于作用在RGYW(R=A/G,Y=C/T,W=A/T)基序上,特别是AGCT基序。与此同时,机体可通过碱基切除修复(base excision repair,BER)和错配修复(mismatch repair,MMR)途径对DNA 进行修复,而此过程中一些损伤修复因子可能会使DNA 断裂末端非模板碱基发生插入或缺失,从而使抗体基因产生更多的插入和缺失片段。
广谱中和抗体(broadly neutralizing antibodies,bnAbs)是由B 细胞产生的能够中和多种病毒毒株的抗体[4];和普通中和抗体相比,bnAbs具有较长的抗体重链互补决定区(complementarity determining regions,CDRs),大量的点突变、抗体基因片段插入 和 缺 失(insertions and deletions,indels) 等 特征[5-6]。虽然基因片段的插入和缺失是抗体亲和力成熟过程中的小概率事件,但其对抗体的广谱中和能力发挥着关键作用[7-8]。研究[8]发现,在已报道的人类免疫缺陷病毒1 型(human immunodeficiency virus type 1,HIV-1)的广谱中和抗体中,约有40%存在indels。目前,由于缺乏合适的研究体系,这些indels 的产生机制尚不明确;且本课题组的前期工作发现,长度为1 bp 和大于1 bp 的indels 的产生来源亦不尽相同。
有研究[9-10]显示,众多的细胞作用因子参与了由AID 介导的SHM 过程中的DNA 损伤修复。其中,DNA 聚合酶β(polymerase β,Polβ)由基因Polb编码(小鼠中),参与了填补单个碱基的短片段BER、引入多个碱基的长片段BER 过程,被认为可能是AID 介导SHM 过程中影响抗体基因突变事件(包括点突变、插入和缺失)的重要因子[11]。由于Polβ 在DNA 复制中发挥了重要作用,完全敲除Polb基因会导致小鼠死亡[12],因此可考虑利用Polβ 抑制剂来分析Polβ 对抗体基因点突变、插入和缺失的影响。相关研究发现,扎西他滨(2',3'-dideoxycytidine,DDC)[13]和5- 甲 氧 基 黄 酮(5-methoxyflavone,5-MF)[14]为Polβ 的抑制剂,能有效抑制Polβ 的功能。因此,本研究猜测,是否可使用DDC 和5-MF来观察AID 介导SHM 过程中Polβ 对抗体基因突变事件的发生频率存在的影响,目前此类研究尚未见报道。
在小鼠B 淋巴瘤细胞CH12F3 中,抗体重链(heavy chain,H)由2 条等位基因编码,一条为有功能等位基因,另一条为无功能等位基因,其中有功能等位基因中的VDJ 序列由VH1-53、DH1-1、JH2 组成[15-16],且固定该序列有利于建库测序过程中的PCR引物设计。本研究利用细胞生物学技术和高通量测序技术,在过表达AID 的CH12F3 细胞中建立体外检测抗体基因突变的方法,并利用该方法初步探究Polβ抑制剂对抗体基因突变频率的影响。
1 材料与方法
1.1 实验材料
1.1.1 细胞与质粒 小鼠B 淋巴瘤细胞株CH12F3、慢病毒包装质粒pCL-10A1、表达质粒pMX-AID 均由中国科学院分子细胞科学卓越创新中心孟飞龙课题组馈赠。
1.1.2 主要试剂与仪器 DDC、0.4%台盼蓝染色液、β-巯基乙醇(Sigma,美国),5-MF(MCE,美国),Polβ 抗 体(Abcam,美 国),AICDA (activationinduced cytidine deaminase)抗体、β 微管蛋白(βtubulin)抗体(武汉爱博泰克生物科技有限公司),山羊抗兔IgG(Proteintech,美国),DMEM 培养基(Corning,美国),RPMI-1640 培养基、青霉素-链霉素、L-谷氨酰胺、嘌呤霉素(Gibco,美国),胎牛血清(上海吉泰依科赛生物科技有限公司),DNA 纯化回收试剂盒[天根生化科技(北京)有限公司],VE-186转移电泳槽(上海天能科技有限公司)。
1.2 实验方法
1.2.1 细胞培养 CH12F3 细胞(悬浮细胞)采用含10%胎牛血清、100 U/mL 青霉素和100 μg/mL 链霉素、2 mmol/L L-谷氨酰胺以及5×10-5mol/L β-巯基乙醇的RPMI-1640 培养基进行培养。293T 细胞(贴壁细胞)采用含10%胎牛血清的DMEM 培养基进行培养。所有细胞的培养条件均为37 ℃、5%CO2,取对数生长期的细胞用于后续实验。
1.2.2 慢病毒包装 转染前24 h,用胰蛋白酶消化293T 细胞;随后,用含10%胎牛血清的DMEM 培养基将该细胞密度调整为8×105/mL,并将其接种于10 cm培养皿中;转染前2 h 进行细胞换液。将30 μg pMXAID 质 粒、6 μg pCL-10A1 质 粒、62 μL 2 mol/L CaCl2、500 μL 2×4-羟 乙 基 哌 嗪 乙 磺 酸 缓 冲 盐 水(HEPES buffer saline,HBS)、双蒸水制成1 mL 转染混合液,静置20 min 后将该混合液均匀滴加至上述培养皿中,于37 ℃、5%CO2条件下培养12 h 后进行细胞换液,加入5.5 mL 含10%胎牛血清的DMEM 培养基继续培养。于转染后48 h,用10 mL 注射器和0.45 μm 过滤器收集慢病毒上清液,再次加入5.5 mL含10%胎牛血清的DMEM 培养基进行培养,于转染后72 h再次过滤并收集慢病毒上清液,混合后立即使用或于4 ℃存放备用。
1.2.3 慢病毒感染 将CH12F3细胞(5×105/mL)接种于24 孔板中,每孔加入1 mL 慢病毒上清液(“1.2.2”中获得),混匀后静置20 min。将细胞培养板用封口膜密封后,于32 ℃、1 200×g条件下离心1.5 h,取出培养板并去除封口膜后,将其置于37 ℃、5% CO2恒温培养箱中培养12 h,更换为“1.2.1”中培养CH12F3 细胞的培养基后继续培养12 h,再次进行慢病毒感染(步骤同前)。每组设置5 个复孔,于第2 次感染36 h 后,加入1 μg/mL 嘌呤霉素进行48 h筛选。收集经慢病毒感染的CH12F3 细胞(处理组)和感染前的CH12F3 细胞(对照组),于300 ×g离心5 min 后弃去上清液,用于后续蛋白质印迹法(Western blotting)检测和高通量测序文库的构建。
1.2.4 Western blotting检测 分别向“1.2.3”收集的处理组和对照组细胞中加入细胞裂解液,冰上裂解15 min 后于4 ℃、15 000×g离心5 min,吸取上清液转移至新的离心管中,加入2×SDS 上样缓冲液,于100 ℃下加热10 min 获得总蛋白。经SDS-PAGE 凝胶电泳、硝酸纤维素膜印迹后,用含5%脱脂奶粉的PBST封闭1 h,加入对应的一抗,包括AID抗体(1∶1 000)、Polβ 抗体(1∶1 000)、β-tubulin 抗体(1∶1 000),4 ℃孵育过夜后用PBST洗膜3次,再加入二抗(1∶5 000)。室温孵育1 h,经PBST 洗膜3 次后,加入化学发光显影液进行曝光。
1.2.5 高通量测序文库的构建、数据处理及分析从“1.2.3”收集的处理组和对照组CH12F3 细胞中提取基因组DNA,通过3 轮PCR 进行文库构建(图1)。第1 轮PCR 反应中,以抽提的基因组DNA 为模板,使用抗体基因VDJ 片段的特异性引物进行扩增,反应体系为:基因组DNA 2 μg、dNTP 200 μmol/L、正向/反向引物各0.2 μmol/L、Q5 反应液10 μL、Q5®高保真DNA 聚合酶1 U,反应条件为:98 ℃30 s,98 ℃10 s、62 ℃30 s、72 ℃30 s (20 个循环),72 ℃2 min。第2 轮PCR 反应中,以2 μL 第1 轮扩增产物为模板,引物扩增片段覆盖VDJ 区的CDR2 和CDR3,反应体系与第一轮PCR 相同(模板除外),反应条件亦与第一轮PCR 相同(除需将循环数降至16)。第3 轮PCR 反应中,以1 μL 第2 轮扩增产物为模板,以Illumina P5 和P7 作为引物,反应体系与第一轮PCR 相同(模板除外),反应条件亦与第一轮PCR 相同(除需将循环数降至15)。将最终的PCR 扩增产物(350~450 bp)进行琼脂糖凝胶电泳,再使用DNA 纯化回收试剂盒回收DNA 片段,于Illumina Hiseq 测序仪进行高通量测序,测序模式为2×150。3轮PCR所用的引物序列参照文献[17]。
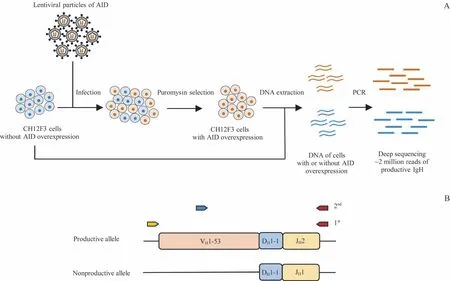
图1 高通量测序样品准备流程图及PCR策略Fig 1 Flowchart of preparation for high-throughput sequencing and PCR strategies
随后,我们对高通量测序数据进行处理。使用课题组自定义脚本拆解数据,并用Bowtie2 软件[18]将其与参考序列进行比对。同时,使用SHM 流程代码[3]对点突变、缺失和插入进行定义:如果1 个核苷酸与参考序列不同,且其Illumina 测序质量分数大于20,则将其视为点突变;如果相对于参考序列,缺口两侧的4 个连续核苷酸的Illumina 测序质量分数均大于20,则该缺口被定义为缺失;如果相对于参考序列,仅存在单个插入且插入序列中2 个连续核苷酸的Illumina 测序质量分数均大于20,则被定义为插入。
而后,我们对测序结果进行分析,具体如下:①点突变分析。绘制单碱基突变图谱(即由每个位点的核苷酸所含点突变的读长占总测序读长的百分比构成)、总体突变频率图(即由包含点突变的序列读长占所有测序读长的百分比构成),以观察突变模式和发生频率。②indels 分析。绘制indels 发生的频率图(即由indels 总数占所有测序读长的百分比构成)和indels 的分布图(即由每20 个碱基中缺失的起点或插入的位点总数占所有测序读长的百分比构成),以观察indels的发生频率和分布情况。
1.2.6 台盼蓝拒染法检测 取“1.2.1”中培养的CH12F3细胞(5×105/mL)接种于24孔板中,每孔接种的细胞总量为106,每组设置3个复孔。采用不同浓度的DDC(12.5、25、50、100、200、400、800 μmol/L)和等体积的0.9%NaCl分别处理CH12F3细胞,采用不同浓度的5-MF(12.5、25、50、100、200 μmol/L)和等体积的DMSO分别处理CH12F3细胞,经24、48、72 h后分别将上述细胞悬液与台盼蓝溶液以1∶1的比例均匀混合。取10 μL细胞混合液加入计数板中,于显微镜下进行计数,统计存活(未染色)的细胞数量及活细胞率。最终,分别选择该2种抑制剂对细胞增殖影响较小的最大浓度作为后续的实验浓度。
1.2.7 DDC 和5-MF 对抗体基因VDJ 片段点突变、插入和缺失的检测 分别以“1.2.6”中获得的DDC浓度、5-MF 浓度处理CH12F3 细胞(即实验组),以等体积的0.9%NaCl处理的细胞、等体积的DMSO 处理的细胞依次记为上述实验组的对照组,每组设置5 个重复样品。将上述4 组细胞分别由慢病毒包装和感染后,收集感染前、后的细胞行Western blotting,检测其AID 和Polβ 的表达;同时,提取细胞的基因组DNA,并行高通量测序文库的构建及分析。具体方法同“1.2.2”—“1.2.5”。
1.3 统计学方法
使用GraphPad Prism 8.0 软件进行统计分析。定量资料以±s表示,组间比较使用独立样本t检验。P<0.05表示差异具有统计学意义。
2 结果
2.1 CH12F3 细胞处理组和对照组中抗体基因VDJ片段的突变分析
采用Western blotting 检测CH12F3 处理组和对照组中AID的表达,结果(图2A)显示,CH12F3细胞处理组中AID的表达较对照组有显著增加,即慢病毒感染成功。
通过对高通量测序的结果进行分析,我们发现CH12F3 细胞处理组抗体基因VDJ 片段上存在大量的点突变,这些点突变主要集中在CDR2 和CDR3 区且突变的频率均较高(图2B),同时该突变模式与体内生发中心B 细胞的突变模式[3]相一致。此外,通过对indels 的发生频率进行分析,发现和对照组相比,CH12F3 细胞处理组的indels 的发生频率较高(均P=0.000,图2C、2D);且该组中片段长度大于1 bp 的indels 的发生频率亦较高(均P=0.000,图2F、2G)。通过对上述indels 事件在抗体基因上的分布进行分析,结果(图2E、2H)显示,CDR2和CDR3区域的突变频率高于VDJ 外显子上的其他区域,即indels 发生的区域与高频碱基突变的位置相对应。
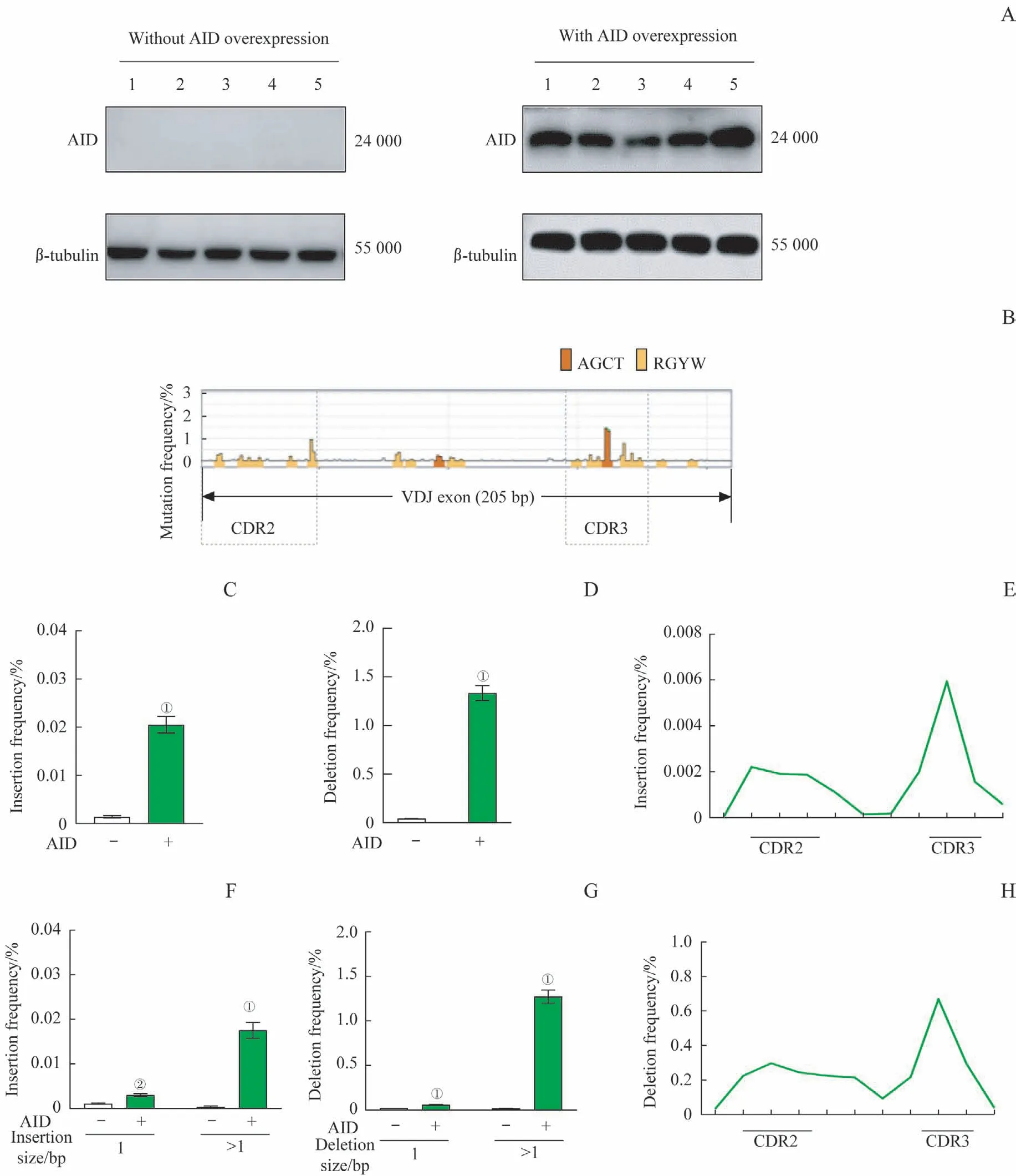
图2 CH12F3细胞处理组和对照组中,AID的表达及抗体基因VDJ片段的点突变、插入和缺失频率的分析Fig 2 Analysis of point mutation, insertion and deletion frequencies in the VDJ regions of antibody genes and AID expression in CH12F3 cells with or without AID overexpression
2.2 Polβ 抑制剂——DDC 对抗体基因VDJ 片段的突变分析
本研究通过台盼蓝拒染法检测不同浓度的DDC处理CH12F3 细胞后对其增殖能力的影响,结果(图3A)显示,和经0.9% NaCl 处理的CH12F3 细胞(对照组)相比,分别经200、400、800 μmol/L DDC 处理的CH12F3 细胞在48、72 h 时的细胞数量均有显著下 降(均P<0.05),而100 μmol/L DDC 处 理 的CH12F3 细胞则无明显差异,因此该浓度被认为是DDC 不影响细胞增殖的最大浓度。随后,采用100 μmol/L DDC 处理细胞(实验组),再通过Western blotting 检测慢病毒感染后的实验组和对照组细胞中AID 和Polβ 的表达,结果(图3B)显示该2组细胞的AID 均有表达,即慢病毒感染成功;同时,实验组细胞中Polβ 的表达量与对照组间差异无统计学意义(P=0.267),即100 μmol/L DDC 不影响Polβ的表达。
而后,对慢病毒感染前、后的实验组和对照组共4 组细胞的基因组DNA 进行文库构建及高通量测序。对抗体基因VDJ 片段的点突变进行分析,结果(图3C)显示,慢病毒感染后的实验组和对照组相比,其点突变频率较低(P=0.000);单碱基突变图谱(图3D)显示,感染后的实验组和对照组细胞中抗体基因VDJ 序列的突变图谱高度相似,且点突变主要分布在VDJ 片段的CDR2 和CDR3 区。对抗体基因VDJ片段的插入频率进行分析,结果显示,感染后的实验组和对照组相比,长度为1 bp及大于1 bp的插入频率间差异均无统计学意义(图3E),且这些插入大多发生在CDR2 和CDR3 区域(图3F)。对抗体基因VDJ片段的缺失频率进行分析,结果显示,感染后的实验组和对照组相比,其长度为1 bp 的缺失频率较低(P=0.009,图3G),且这些缺失主要发生在CDR2 和CDR3区域(图3H)。
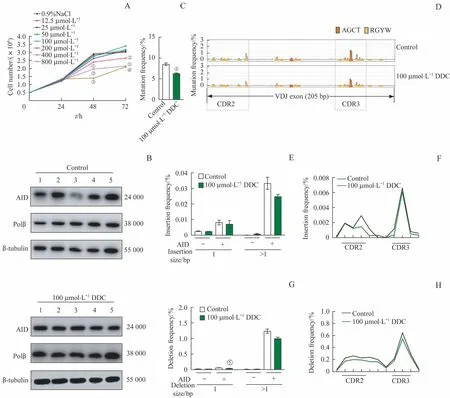
图3 DDC对CH12F3细胞增殖及对抗体基因VDJ片段的点突变、插入和缺失频率的影响Fig 3 Effects of DDC on the proliferation of CH12F3 cells and the frequencies of point mutation,insertion and deletion in the VDJ regions of antibody genes
2.3 Polβ抑制剂——5-MF对抗体基因VDJ片段的突变分析
通过台盼蓝拒染法检测不同浓度的5-MF 处理CH12F3 细胞后对其增殖能力的影响,结果(图4A)显示,和经DMSO 处理的CH12F3 细胞(对照组)相比,分别经50、100、200 μmol/L 5-MF处理的CH12F3细胞在24、48、72 h 的细胞计数上显著下降(均P<0.05),而25 μmol/L 5-MF 处理的CH12F3 细胞影响较小,因此该浓度被认为是5-MF 对细胞增殖影响较小的最大浓度。随后,采用25 μmol/L 5-MF 处理细胞(实验组),再通过Western blotting 检测慢病毒感染后的实验组和对照组细胞中AID 和Polβ 的表达,结果(图4B)显示该2 组细胞的AID 均有表达,即慢病毒感染成功;同时,实验组细胞中Polβ 的表达量与对照组间差异无统计学意义(P=0.115),即25 μmol/L 5-MF不影响Polβ的表达。
而后,对慢病毒感染前、后的实验组和对照组细胞的基因组DNA 进行文库构建及高通量测序分析。对抗体基因VDJ 片段的点突变进行分析,结果(图4C)显示,慢病毒感染后的实验组和对照组相比,其点突变频率较低(P=0.000);单碱基突变图谱(图4D)显示,感染后的实验组和对照组的点突变主要分布在抗体基因VDJ 片段CDR2 和CDR3 区。对抗体基因VDJ 片段的插入频率进行分析,结果显示,感染后的实验组和对照组相比,其长度大于1 bp的插入频率较低(P=0.000,图4E),且这些插入大多发生在CDR2 和CDR3 区域(图4F)。对抗体基因VDJ 片段的缺失频率进行分析,结果显示,感染后的实验组和对照组相比,其长度大于1 bp 的缺失频率亦较低(P=0.000,图4G),且这些缺失亦主要发生在CDR2和CDR3区域(图4H)。
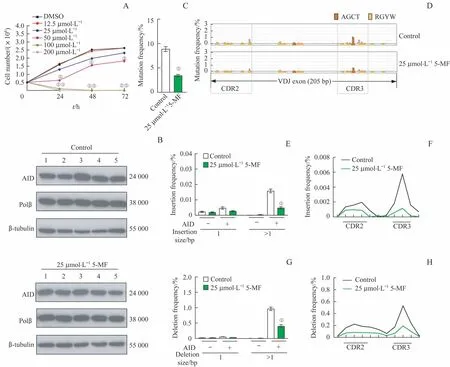
图4 5-MF对CH12F3细胞增殖及对抗体基因VDJ片段的点突变、插入和缺失频率的影响Fig 4 Effects of 5-MF on the proliferation of CH12F3 cells and the frequencies of point mutation, insertion and deletion in the VDJ regions of antibody genes
3 讨论
在因病毒感染导致的疾病预防和治疗过程中,疫苗研制常受到毒株突变的限制。近年来,研究人员发现,少数个体在病毒慢性感染阶段可产生广谱中和抗体,以识别并广泛中和多种类型的毒株[19]。目前,已 有 报 道 从 被HIV[6,20-21]、流 感 病 毒[22]、丙 肝 病毒[23]、埃博拉病毒[24]、寨卡病毒[25]和疟原虫[26]等感染的患者体内分离得到bnAbs,这些发现从体液免疫角度给疫苗研制带来了新的思路。但bnAbs的产生时间较长且难以被诱导,因此如何快速获得bnAbs成为了亟待解决的问题。和普通的中和抗体相比,bnAbs 具有更高频率的点突变、缺失和插入等,且这些突变事件的发生不利于疫苗的设计,具体机制亦不明晰。因此,在抗体进化过程中,确定某一个或某一些影响抗体基因点突变、插入和缺失的作用因子至关重要。
抗体基因的突变事件发生频率极低,用传统的实验手段很难在细胞系中进行检测,因此无法对其产生的机制进行深入研究。为解决这一难题,本研究通过在CH12F3B 细胞系中过表达AID 的方式,结合高通量测序对该细胞中抗体基因VDJ 片段进行深度测序,成功捕获到了大量的基因点突变、插入和缺失事件。我们发现,在体外细胞系中检测到抗体基因VDJ 片段上CDR2 和CDR3 区的点突变的频率较高且分布较集中,这种由AID诱导产生的细胞内高频突变模式与体内生发中心B 细胞的突变模式一致[3]。此外,插入和缺失发生的位置也主要在抗体基因VDJ 片段上CDR2 和CDR3 区,且抗体基因缺失的分布规律与小鼠体内生发中心B 细胞的研究[3]结果亦相一致。上述结果表明,我们在体外CH12F3 细胞系中构建的AID过表达的研究方法可有效检测抗体基因VDJ片段突变事件的产生,进而有望利用该方法去寻找影响其突变事件发生的关键因子。
随后,我们利用已建立的方法进一步探索参与DNA 损伤修复的细胞作用因子——Polβ 的抑制剂DDC 和5-MF 对抗体基因点突变、缺失和插入的影响。通过对慢病毒感染后的实验组和对照组的突变频率进行分析,我们发现,经100 μmol/L DDC 处理的CH12F3 细胞与对照组相比,点突变频率、长度为1 bp的缺失频率均较低,而大于1 bp的插入和缺失频率亦有下降但差异无统计学意义;而经25 μmol/L 5-MF 处理的CH12F3 细胞与对照组比较,点突变频率、长度大于1 bp的插入和缺失频率均有降低。这些结果均表明,DDC和5-MF能够通过抑制Polβ的活性来影响抗体基因VDJ 序列上点突变、插入和缺失的发生频率,因此Polβ 或是影响抗体基因点突变、插入和缺失的关键因子。
本研究在体外建立利用小鼠B 淋巴瘤细胞系CH12F3 研究抗体基因VDJ 片段点突变、插入和缺失的方法具有以下优势:①方便快捷,实验周期较短。②便于通过基因敲除、蛋白过表达或抑制剂处理等方式处理细胞,进行高通量筛选,初步找出可能影响突变事件的关键因子。③投入成本较低,实验设计相对简单。尽管如此,该方法也存在一定的局限性:①细胞环境较为单一,而机体内部环境复杂多样,CH12F3 细胞过表达AID 并不能完全模拟生发中心B细胞经历SHM 的过程。②单次只能检测1 个基因对抗体基因VDJ 序列产生的影响。③对实验细胞内的点突变、插入和缺失事件的检测数少于体内实验,所得实验结论推导到体内未必相符。因此,通过本研究方法筛选得到的影响抗体基因突变事件的关键因子还需通过体内实验加以验证。
综上所述,本研究建立了通过细胞系研究抗体基因VDJ 片段点突变、插入和缺失的检测方法,或将为体外筛选影响抗体基因突变事件发生的关键因子提供高效的方法。近年来,伴随着高通量测序分析抗体序列的不断发展,设计的测序引物不能涵盖所有抗体基因片段、抗体重链与轻链的配对信息难以检测、对测序过程中可能引入的碱基插入和缺失进行分析等问题仍将是解析抗体基因序列中突变事件产生机制的难点。未来,本研究还将继续深入探究在AID 介导的SHM 过程中抗体基因VDJ序列突变事件的产生机制,以期为bnAbs的筛选和诱导提供新的线索。
利益冲突声明/Conflict of Interests
所有作者声明不存在利益冲突。
All authors disclose no relevant conflict of interests.
作者贡献/Authors'Contributions
郝茜和叶菱秀参与了实验设计,罗思敏和郝茜参与了数据分析和整理,罗思敏、郝茜和叶菱秀参与了论文的写作和修改。所有作者均阅读并同意了最终稿件的提交。
The study was designed by HAO Qian and YEAP Lengsiew. The data was collected and analyzed by LUO Simin and HAO Qian. The manuscript was drafted and revised by LUO Simin, HAO Qian and YEAP Lengsiew. All the authors have read the last version of paper and consented for submission.
·Received:2021-12-31
·Accepted:2022-03-25
·Published online:2022-04-28
