中国西南地区3个隔离人群遗传亚结构分析
2022-05-24王飞王萌张兴华宇克莉郑连斌杨亚军
王飞,王萌,张兴华,宇克莉,郑连斌,杨亚军
研究报告
中国西南地区3个隔离人群遗传亚结构分析
王飞1,王萌1,张兴华2,宇克莉2,郑连斌2,杨亚军1
1. 复旦大学生命科学学院,上海 200438 2. 天津师范大学生命科学学院,天津 300387
中国西南地区分布着众多少数民族,同时也包含许多地理和遗传隔离的人群,然而这些族群的遗传结构,特别是父系遗传结构缺乏深度解析。本研究采用Y染色体捕获及Illumina测序技术,对西南地区3个隔离人群男性无关个体样本,包括四川平武县的白马人、石棉县的木雅人及云南景洪市空格人,通过定点复合扩增检测,直接计数法计算相关单倍群频率,应用Past3.0软件进行PCA (principle component analysis, PCA)主成分分析并绘制群体聚类树,研究了3个族群的父系遗传结构,并探讨其遗传亚结构关系。本研究共观察到云南空格人三种Y染色体单倍群、四川白马人四种Y染色体单倍群以及木雅人五种Y染色体单倍群。结果表明,空格人与佤族的父系遗传关系最近;而白马人和木雅人主要集中在D单倍群及其下游,研究提示着白马人和四川羌族的父系遗传关系最近;木雅人与昌都地区藏族和林芝地区藏族父系遗传关系最近。本文对不同族群遗传结构的分析,丰富了我国隔离人群遗传关系的研究成果。
Y染色体;群体遗传学;隔离人群;遗传亚结构
我国的西南地区民族众多,少数民族多样性极为丰富,同时也存在着一些隔离人群[1],其中包括分布于甘肃、四川交界深山中的白马人、分布在云南西双版纳州的空格人以及分布在四川石棉县一带的木雅人[2]。上述各族群中存在较为普遍的族内通婚、分布集中的现象,这使得他们具有良好的遗传学研究背景。白马人群居住在川、甘交界处,最新数据显示,白马人的总人口约20,000多人。然而白马人在语言、生活习俗、宗教等方面与周围的藏族、羌族等其他少数民族都具有很大差异。白马人的族属划分在学术界一直具有较大的争议,20世纪50年代,白马人被划分为藏族的一个支系,语言学研究显示白马藏语是藏族安多方言与康巴方言之间的一种次方言[3]。孙宏开先生认为白马人最大可能是白马氐的后裔[4];张海国教授以肤纹做聚类分析,认为白马人是古氐人的后裔[5];也有学者认为“白马”是古藏语,白马语与某些地区的藏族语言极为相似,故将白马人视为是藏族的支系[6]。上述文献报道都基于历史学和民族学的角度来讨论白马人的族源问题。空格人主要聚居在云南西双版纳傣族自治州景洪市勐养镇北部(西双版纳最大的国家自然保护区的南缘),分布于7个村寨。到1997年底,他们共有278户,人口总数为1435人,空格语属于南亚语系孟高棉语族瓦德昂语支[7];木雅人主要分布在四川省康定县、九龙县、石棉县一带,以农业为主兼有畜牧业,木雅语属于汉藏语系藏缅语族[8]。关于上述两个族群的文献报道甚少,目前仅见天津师范大学郑连斌课题组从体质人类学角度去探讨木雅人和空格人的族源关系[9]。
单核苷酸多态性(single nucleotide polymorphisms, SNPs)广泛存在于人类基因组中,具有突变率低、易于分型等优势,可以准确反映族群间遗传结构和个体间遗传差异。其中Y染色体的非重组区域以单倍型形式呈父系遗传,有效群体小,对遗传漂变敏感,包含更多的父系群体历史信息[10]。通过分析Y-DNA单倍型,可揭示某些族群和地区人群的父系遗传结构。本研究从分子生物学和遗传学的角度来探讨白马人、空格人、木雅人的遗传结构关系,从父系遗传的角度研究白马人、空格人以及木雅人的遗传多态性,不仅丰富我国少数民族的群体遗传资料,有助于进一步研究族群的起源,同时也为我国族群间亲缘关系研究提供新的遗传学上的线索。
1 材料与方法
1.1 样本收集和DNA提取
本研究共收集四川省平武县白马人33例,加上本课题组先前未发表的白马人群数据一共167例,四川雅安市石棉县一带的木雅人74例,云南空格人共48例。所选对象均为正常个体,3代内无族外通婚现象,彼此间无亲缘关系。本研究样本来源于2015年科技部基础专项中所收集的中国少数民族遗传物质,上述样本提供者均签署知情同意书,本研究也通过复旦大学伦理委员会审查。所有样本采用JP-96唾液 DNA 提取试剂盒(上海莱枫生物科技有限公司)进行DNA提取。使用NanoDrop 8000 (美国Thermo Fisher Scientific公司)进行DNA定量。
1.2 Y-SNP基因分型
采用Y染色体捕获及Illumina测序技术对中国西南地区3个隔离人群的男性无关个体样本进行定点复合扩增检测。使用9700型PCR仪(美国Applied Biosystems公司)进行PCR扩增,用ABI 3730xl型遗传分析仪(美国Applied Biosystems公司)进行毛细管电泳检测。利用GeneMapperTMID-X v3.2软件分析样本分型。选取Y染色体谱系树上主要D单倍群及主要的下游分支见图1,包括M174、F5993、M15、M55、P99、N1、F1531、P47、F738、F1635。
1.3 数据分析
本研究中所确定的云南和四川等地的白马人、空格人、木雅人的Y染色体单倍群频率包含本次实验所得数据和文献报道数据,其中云南空格人的14个民族的参考数据来自于已报道文献[11],包括佤族、侗族、黎族、毛南族、仡佬族、侗族、苗族、白族、哈尼族、景颇族、纳西族、土家族、羌族、彝族。根据Y-SNPs位点的分型基于国际Y染色体协会(Y chromosome consortium, YCC)的单倍群命名原则[12]划分样本所属单倍群,采用直接计数法统计各单倍群频率。同时对Y-SNPs单倍群相同的样本,基于15个Y-STRs基因座分型使用Network v10.0软件进行网络图分析。同时基于本次研究的白马人、木雅人及文献[13~19]报道的22个民族群体的Y-SNP单倍群频率数据,选取分布频率较高的D单倍群及其下游分支数据。依据各单倍群的基因频率并使用Past3.0软件进行主成分分析,来探讨白马人、木雅人和空格人3个隔离人群的遗传亚结构。
2 结果与分析
2.1 父系遗传结构
2.1.1 Y-SNPs单倍群在各族群间的分布
白马人、木雅人与参考族群的主要单倍群频率分布表见表1;云南空格人和14个参考族群的单倍群频率见表2。结果显示,各单倍群频率在藏族人群之间存在显著差异,在167例男性白马人样本中,单倍型主要集中在D单倍型及其下游分支,为61.1%。在D的下游分支单倍群中,以单倍群D1a1a-M15 (占比41.9%)为主。而在74例男性木雅人样本中,D单倍型及其下游分支占比59.5%,其下游分支类型则以D1a1b1-P47 (占比51.4%)为主。在云南空格人男性样本中,共观察到了3种单倍型C、N、O。其中,N单倍群分布频率最高,为54.16%;其次是O单倍群,为35.41%。

图1 基于Y-SNP位点构成的单倍群谱系树
2.1.2 主成分分析
基于参考族群和测试族群单倍群频率进行PCA分析,结果见图2,从图中可以看出,各族群的分布总体上较为集中。从遗传距离的角度分析,本研究中云南空格人与纳西族、佤族的关系较为接近,而与苗族、侗族和毛南族的关系较远。从语系的角度来看,大部分仡台语族(侗族、仡佬族、黎族、毛南族)之间遗传距离较近,大部分藏缅语族(羌族、纳西族、哈尼族、羌族)遗传距离也较近,但语系相近的景颇族与羌族遗传距离较远。
2.2 群体聚类分析
2.2.1 空格人的群体聚类树分析
云南空格人与其他14个民族的群体聚类树,树型结果见图3。从图中可以看出聚类树呈现出两大分支,第一大分支为羌族和空格人、佤族以及纳西族,其余11个族群为第二分支。其次,依据聚类树的分支结构,第二分支中的各族群又可以区分为2个亚类,第一个亚类为彝族、哈尼族、土家族和侗族,而第二亚类为黎族、景颇族、白族、仡佬族、毛南族、广西侗族和苗族。结合语言学分类来看,各族群呈现小范围中相同语系族群聚集的特征:例如,仡台语系中侗族、仡佬族、毛南族、黎族遗传关系较近,藏缅语族中白族、景颇族、哈尼族和土家族遗传关系较近,与纳西族遗传关系较远。聚类树与PCA结果基本一致,均提示着云南空格人与纳西族和佤族的遗传距离较近。
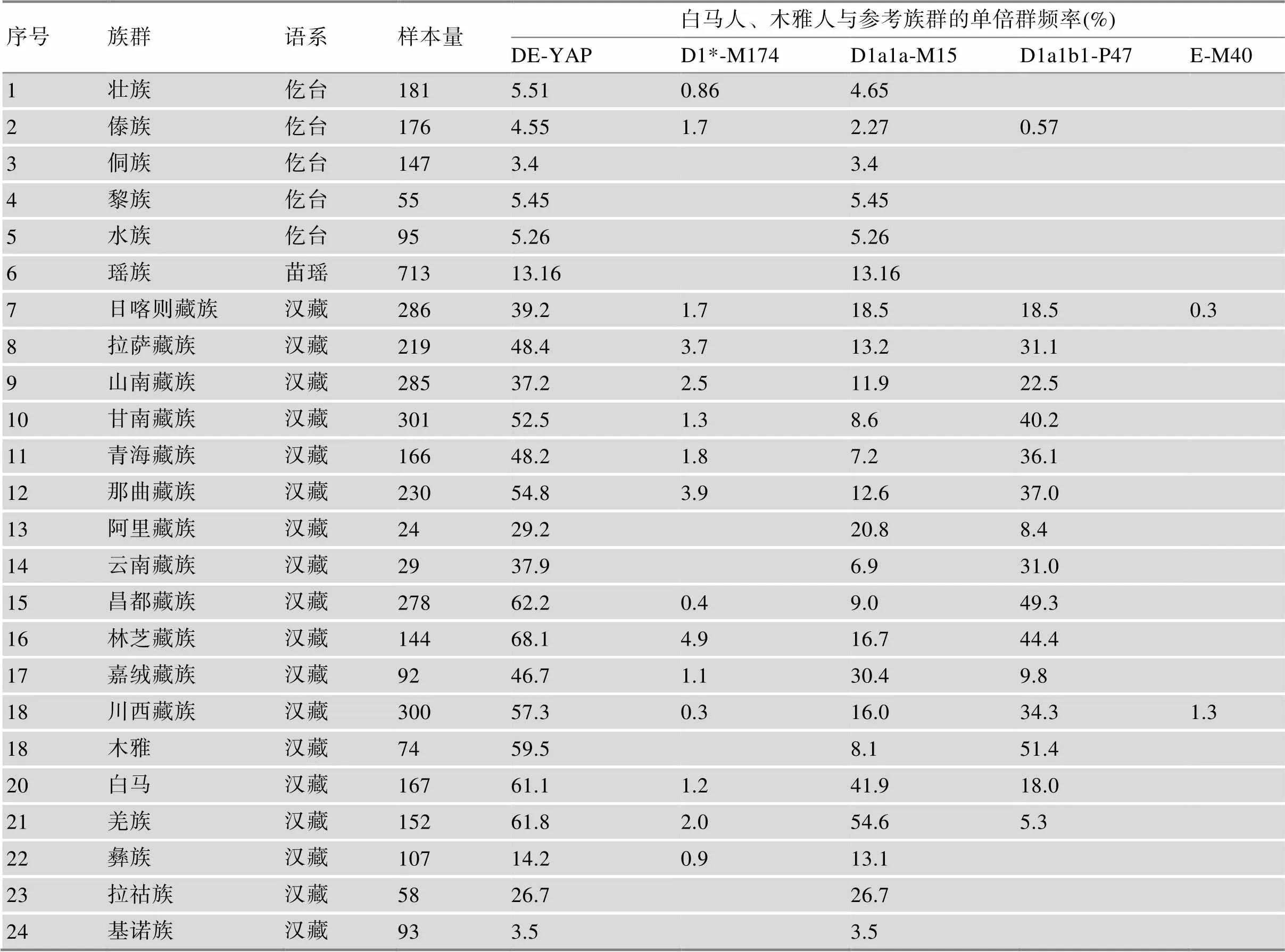
表1 D1*-M174次级系谱在各族群中的单倍群分布表
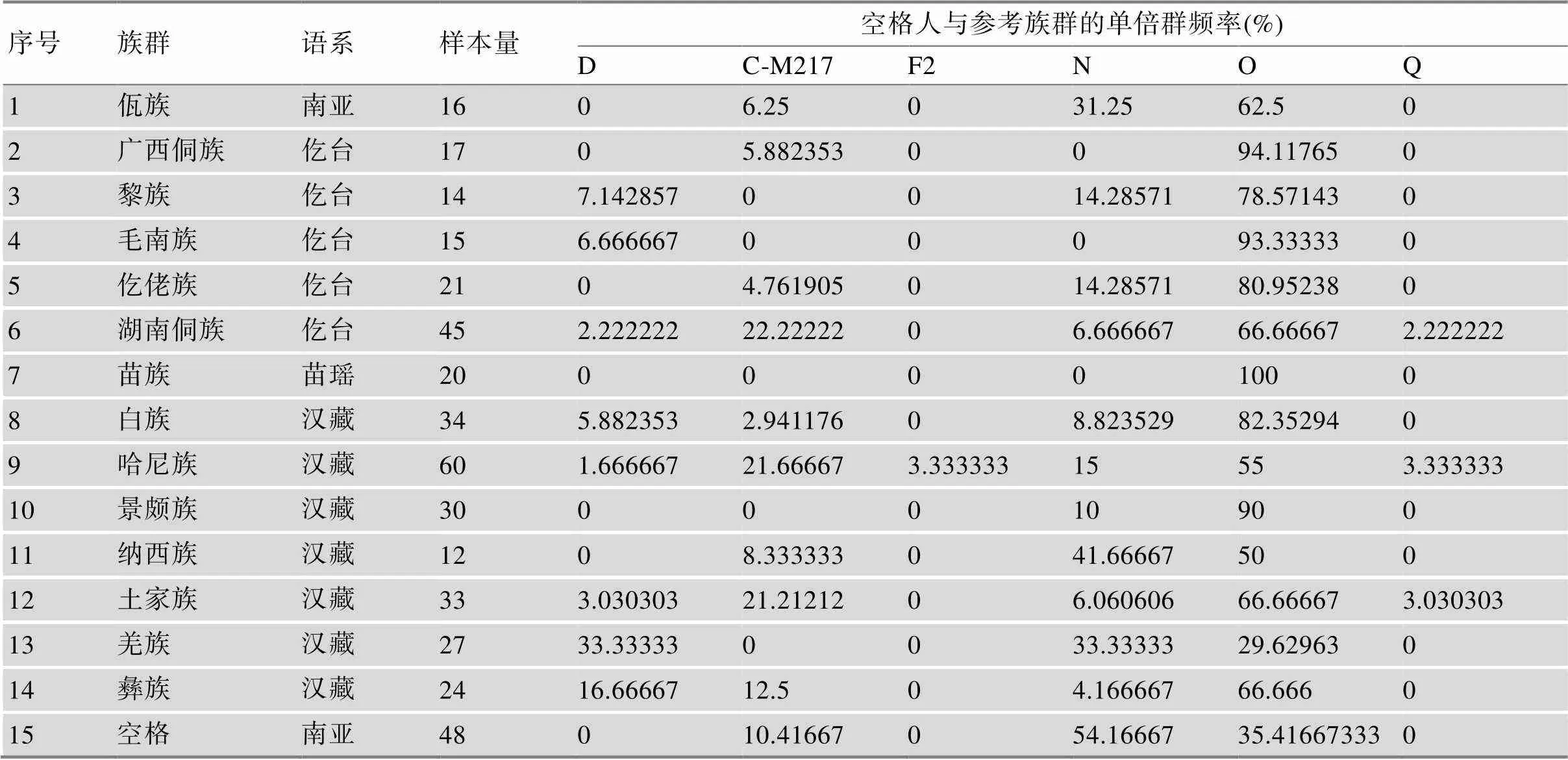
表2 14个参考族群与空格人的单倍群频率分布表

图2 云南空格人与其他14个民族的PCA图

图3 云南空格人与其他14个民族的群体聚类树
2.2.2 白马人和木雅人群体聚类树分析
白马人和木雅人与其他测试族群的群体聚类树结果见图4。从图中可以看出聚类树呈现出两大分支,相同语系的各族群彼此间呈现小范围的聚集。我们发现,在第一分支内部白马人和羌族聚集在一起,为单独的一个亚支,提示着白马人在遗传结构上与羌族较为接近;其次,依据聚类树的分支结构,第二分支在总体上可以区分为2个小亚类,而汉藏语系的各族群彼此间相互聚集,其中与木雅人在遗传关系上最为接近的是昌都地区藏族。在语言学上,汉藏语系中不同地区间的藏族之间也呈现小范围聚集。聚类树与语言学分析结果均提示,木雅人与昌都地区藏族的遗传关系最近,白马人与羌族的遗传关系最近。

图4 四川白马人和木雅人与其他22个民族的群体聚类树
2.2.3 白马人和木雅人与藏族群体和北方汉族群体遗传关系网络图分析
进一步观察白马人、木雅人与中国不同地区藏族分支人群和北方汉族之间的遗传关系,选择在上述藏族人群中分布频率较高的D1*-M174及其下游的群体,均为典型的东亚群体,包括日喀则地区藏族、昌都地区藏族、林芝地区藏族、阿里地区藏族等典型的藏族群体,根据15个Y-STRs基因座组成的单倍型绘制network网络图见图5。Network结果显示,各地区藏族与我国北方汉族、白马人、木雅人之间在单倍群D1*-M174及其下游分支中均存在不同人群共享Y-STRs单倍型。其中,白马人主要集中在靠近根部的饼图上,并且占有较大份额,与多个民族存在共享单倍型,而北方少数民族、藏缅群体则主要分布在网络外边缘,说明他们的D型可能是由白马人从古老类型演变而出许多新的类型,并传递到了北方藏族,同时也验证了历史学、民族学上认为白马人可能为东亚人群中最为古老单倍群代表的观点。
3 讨论
本研究对四川白马人、木雅人以及云南空格人等男性无关个体的Y-SNP位点进行分型,观察到白马人和木雅人的单倍群类型主要集中在D单倍群及其下游,分别占比了61.1%和59.5%,这与我国藏族人群的Y染色体单倍群分布频率大体一致[20],其中昌都、林芝地区藏族群体中D单倍群所占比例最高达到62.2%和68.1%。D1a1a-M15分布广泛,是藏族和其他人群之间出现频率很高的共享单倍群,而D1a1b1-P47作为另一主要分支,也广泛出现在藏族人群当中。Y-SNPs单倍群在世界人群的分布呈现明显的地理特异性,D单倍群起源于非洲地区,经长时间的扩散演变在东亚大陆上逐渐分化出3个分支,其中D2单倍群主要分布于日本群岛,D单倍群分支下属的单倍群D1*-M174主要分布于青藏高原及东缘区的人群中[21~23],大多数族群的D单倍群比例较低(如壮族、傣族、侗族、黎族、水族)[24]。而在研究白马人和木雅人数据的同时,我们选出西南地区几个D1单倍群比例较高(60%~70%)的族群——昌都藏族、林芝藏族、阿里藏族等族群进行分析比较。

图5 基于15个Y-STRs基因座分型的藏族人群网络图分析结果
PCA和Network网络图均表明不同地区的藏族族群都拥有较多的共享单倍群,同时均拥有一定比例的D1a1a-M15与D1a1b1-P47单倍群。对四川白马人和木雅人的主要单倍群进行分析,可以看出,相同语系间的族群以小范围的相互聚集在一起,结合白马人和木雅人以及测试族群的群体聚类树结构和PCA主成分分析结果来看,白马人与羌族的遗传关系最近,这与张海国从肤纹表型得出的白马人与羌族最为接近的结论有相似之处[5];数据研究还显示,木雅人在遗传结构上与昌都地区藏族和林芝地区藏族更为接近。本研究中,白马人主要集中在D1*-M174及其下游,共占61.1%;与其他族群相比,白马人和羌族在D1*-M174单倍群下游位置D1a1a- M15单倍群频率所占比例最高,分别为41.9%和51.4%;同时拥有D1*-M174的木雅人的D单倍群下游分支则主要集中在D1a1b1-P47,占51.4%。D1*-M174及其下游D1a1a-M15与D1a1b-P47是较为古老的单倍群代表,基于各族群的单倍群频率分布提示着白马人和羌族相比较其他藏族族群,拥有更古老的单倍群。
与此同时,本研究对云南空格人48例男性无关样本进行单倍群分型,共观察到3种单倍群,主要集中在N-M231单倍群(54.16%)和O-M175 (35.41%)。对云南空格人与其他14个民族的Y-SNP主要单倍群频率进行比较,可以看出,不同语系之间存在较大差异,相同语系间均呈现小范围的互相聚集。从各语系间聚类结果来看,部分族群的聚类结果并不明显,这可能是由于所选择的参考族群大部分为西南地区,地理位置较为接近,各族群间的基因交流频繁所致。本研究基于各族群的单倍群频率,通过PCA和群体聚类树的分析结果均提示着云南空格人与佤族遗传关系最近,从语系上来看,佤族和空格人均属于南亚语系,同时佤族主要分布在云南的西南部地区,这可能是导致佤族与空格人遗传关系较近的原因。
本研究从父系遗传Y染色体角度,对白马人、木雅人和空格人隔离人群的男性无关样本的Y-SNP位点的检测和分析。从各族群的单倍群频率分布和主成分分析结果均提示着白马人和羌族的父系遗传关系最近,木雅人与昌都地区藏族和林芝地区藏族父系遗传关系最近,同时各族群的单倍型频率和网络图结果均提示着白马人是我国最为古老的父系单倍群代表。考虑到样本量与采集样本随机性的影响,未来将增加对中国西南地区各地域内白马人、木雅人、空格人等族群的样本采集与数据扩充,以减少样本采集地选择与样本量偏少所带来的误差。本研究中对中国西南地区部分隔离人群的研究,既为阐明我国西南地区族群间的遗传结构提供了基础数据,同时也丰富了我国少数民族的人群遗传多样性。
[1] Shifman S, Darvasi A. The value of isolated populations., 2001, 28(4): 309–310.
[2] Jia YL, Zhang XH, Yu KL, et al. Anthropologic study of 5 tongue moving types of Kongge and Bajia., 2018, 41(1): 102–107.
贾亚兰, 张兴华, 宇克莉, 等. 空格人与八甲人5项舌运动类型的人类学分析. 南京师大学报(自然科学版), 2018, 41(1): 102–107.
[3] Huang Y. Analysis on the origin of “Baima Tibetan”., 2002, 30(4): 62–69.
黄英. “白马藏人”族源探析. 兰州大学学报(社会科学版), 2002, 30(4): 62–69.
[4] Sun HK. Di ethnic group in history and Baima in Sichuan and Gansu region: a preliminary study on the Baima., 1980(3): 33–43.
孙宏开. 历史上的氐族和川甘地区的白马人–白马人族属初探. 民族研究, 1980(3): 33–43.
[5] Zhang HG. Identification of ethnic genus of Baima Tibetan by skin pattern., 1989(6): 431–435.
张海国. 肤纹识别白马藏人的族属. 泸州医学院学报, 1989(6): 431–435.
[6] Mo Chao. Review of Baima culture research., 2017(2): 224–228.
莫超. 白马人文化研究述评. 甘肃社会科学, 2017(2): 224–228.
[7] Liu G. Investigation on the Kongge in Yunnan: a case study of the Kongge in Manbayue Village., 2003(2): 44–50.
刘刚. 云南空格人调查——以曼坝约村空格人为例. 民族研究, 2003(2): 44–50.
[8] Han LS, Wu M, Wang ZS, Yu KL, Zhang XH, Zheng LB. Skinfold thickness of Muya, Ersu, Bajia and Kongge adults in China.,, 2019(6): 71–76.
韩立森, 武敏, 王子善, 宇克莉, 张兴华, 郑连斌. 中国木雅人、尔苏人、八甲人与空格人的皮褶厚度. 天津师范大学学报(自然科学版), 2019(6): 71–76.
[9] Wei Y, Zhang XH, Yu KL, Yan S, Bao JP, Jia YL, Wang ZS, Zheng LB. Somatotyping characteristics of Jino, Muya, Ersu, Bajia and Kongge in China., 2017(5): 605–609.
魏榆, 张兴华, 宇克莉, 严实, 包金萍, 贾亚兰, 王子善, 郑连斌. 中国基诺族、木雅人、尔苏人、八甲人与空格人5个族群的体型特征. 解剖学报, 2017(5): 605–609.
[10] Wang SJ, Qian EF, Li Y, Song ZY, Zhao H, Xie HX, Li CX, Huang J, Jiang L. A genetic sub-structure study of the Tibetan population in southwest China., 2020, 42(6): 565–576.
王小娟, 钱恩芳, 李悦, 宋正阳, 赵慧, 谢何鑫, 李彩霞, 黄江, 江丽. 中国西南地区藏族人群遗传亚结构研究. 遗传, 2020, 42(6): 565–576.
[11] Qi XB, Cui CY, Peng Y, Zhang XM, Yang ZH, Zhong H, Zhang H, Xiang K, Cao XY, Wang Y, Ouzhuluobu, Basang, Ciwangsangbu, Bianba, Gonggalanzi, Wu TY, Chen H, Shi H, Su B. Genetic evidence of paleolithic colonization and neolithic expansion of modern humans on the tibetan plateau., 2013, 30(8): 1761–1778.
[12] Karafet TM, Mendez FL, Meilerman MB, Underhill PA, Zegura SL, Hammer MF. New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree., 2008, 18(5): 830–838.
[13] Chu JY, Huang W, Kuang SQ, Wang JM, Xu JJ, Chu ZT, Yang ZQ, Lin KQ, Li P, Wu M, Geng ZC, Tan CC, Du RF, Jin L. Genetic relationship of populations in China., 1998, 95(20): 11763–11768.
[14] Jin L, Su B. Natives or immigrants: modern human origin in east Asia., 2000, 1(2): 126–133.
[15] Karafet T, Xu L, Du R, Wang W, Feng S, Wells RS, Redd AJ, Zegura SL, Hammer MF. Paternal population history of East Asia: sources, patterns, and microevolutionary processes., 2001, 69(3): 615–628.
[16] Ke Y, Su B, Song X, Lu D, Chen L, Li H, Qi C, Marzuki S, Deka R, Underhill P, Xiao C, Shriver M, Lell J, Wallace D, Wells RS, Seielstad M, Oefner P, Zhu D, Jin J, Huang W, Chakraborty R, Chen Z, Jin L. African origin of modern humans in East Asia: a tale of 12,000 Y chromosomes.,2001, 292(5519): 1151–1153.
[17] Kivisild T, Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, Metspalu E, Adojaan M, Tolk HV, Stepanov V, Gölge M, Usanga E, Papiha SS, Cinnioğlu C, King R, Cavalli-Sforza L, Underhill PA, Villems R. The genetic heritage of the earliest settlers persists both in Indian tribal and caste populations., 2003, 72(2): 313–332.
[18] Lell JT, Sukernik RI, Starikovskaya YB, Su B, Jin L, Schurr TG, Underhill PA, Wallace DC. The dual origin and Siberian affinities of native American Y chromosomes., 2002, 70(1): 192–206.
[19] Qian Y, Qian B, Su B, Yu J, Ke Y, Chu Z, Shi L, Lu D, Chu J, Jin L. Multiple origins of Tibetan Y chromosomes., 2000, 106(4): 453–454.
[20] Feng QD, Lu Y, Ni XM, Yuan K, Yang YJ, Yang X, Liu C, Lou HY, Ning ZL, Wang YC, Lu DS, Zhang C, Zhou Y, Shi M, Tian L, Wang XJ, Zhang X, Li J, Khan A, Guan YQ, Tang K, Wang SJ, Xu SH. Genetic history of Xinjiang's Uyghurs suggests bronze age multiple-way contacts in Eurasia., 2017, 34(10): 2572–2582.
[21] Hammer MF, Karafet TM, Park H, Omoto K, Harihara S, Stoneking M, Horai S. Dual origins of the Japanese: common ground for hunter-gatherer and farmer Y chromosomes., 2006, 51(1): 47–58.
[22] Thangaraj K, Singh L, Reddy AG, Rao VR, Sehgal SC, Underhill PA, Pierson M, Frame IG, Hagelberg E. Genetic affinities of the Andaman Islanders, a vanishing human population., 2003, 13(2): 86–93.
[23] Jiang L, Peng JX, Huang MS, Liu J, Wang L, Ma Q, Zhao H, Yang X, Ji AQ, Li CX. Differentiation analysis for estimating individual ancestry from the Tibetan Plateau by an archaic altitude adaptation EPAS1 haplotype among East Asian populations.,2018, 132(6): 1527–1535.
[24] Huang MS, Ma Q, Wang L, Ma X, Li CX, Jiang L. The study of Tibetan ancestry informative SNPs on high altitude adaptive genes., 2017, 32(6): 588–599.
Genetic substructure analysis of three isolated populations in southwest China
Fei Wang1, Meng Wang1, Xinghua Zhang2, Keli Yu2, Lianbin Zheng2, Yajun Yang1
Southwest China is home to numerous ethnic minorities, as well as many geographically and genetically isolated groups. However, the genetic substructure of these ethnic groups, especially the paternal genetic structure between groups, has not been comprehensively analyzed. In this study, we used Y chromosome capture and Illumina sequencing technologies to investigate the paternal genetic structure of three isolated groups of male unrelated individuals, including Baima in Pingwu, Sichuan Province, Muya in Shimian, Sichuan Province, and Kongge in Jinghong, Yunnan Province. We calculated the frequencies of related haplogroups by the fixed-point compound amplification method and direct counting method, and used the Past3.0 software to perform principal component analysis to draw a population clustering tree. we observed that Kongge had 3 Y chromosome haplogroups, Baima had 4 Y chromosome haplogroups, and Muya had 5 Y chromosome haplogroups. The results showed that Kongge was most closely related to the Wa, and the Y chromosome types of the Baima and Muya were mainly concentrated in the D haplogroup and its lower reaches. It has the closest relationship with the Tibetans in Qamdo and Nyingchi. The study on the genetic structure of different ethnic groups has enriched the genetic relationship of isolated populations and provided a new perspective for understanding Chinese ethnic groups.
Y chromosome; population genetics; isolated populations; genetic sub-structure
2022-01-13;
2022-03-20;
2022-03-29
科技基础性工作专项(编号:2015FY111700),国家自然科学基金项目(编号:31271283,31401022),国家重点研发计划(编号:2016YFF0202301)资助[Supported by the Special for basic work of science and technology (No. 2015FY111700), the National Natural Science Foundation of China (Nos. 31271283,31401022), and the National Key Research and Development Program of the Ministry of Science and Technology (No. 2016YFF0202301)]
王飞,在读硕士研究生,专业方向:群体遗传学。E-mail: 19210700153@fudan.edu.cn
杨亚军,博士,教授,研究方向:群体遗传学。E-mail: yyj229@263.net
10.16288/j.yczz.22-013
(责任编委: 谢小冬)