玉米生育期相关性状QTL定位
2022-05-13陈俊宇卢峰陈婉莹何永辉刘欢欢印志同
陈俊宇 卢峰 陈婉莹 何永辉 刘欢欢 印志同
摘要:生育期是玉米重要的性状之一,其中抽穗期、散粉期和吐丝期是3个最重要的指标。以玉米自交系LDC-1和YS501构建的186个重组自交系为材料,在2个大田种植环境下对抽穗期、散粉期和吐丝期3个生育期相关性状进行QTL解析。基于Maize10k芯片数据构建总长为2 569.89 cM的高密度遗传图谱,共包含2 624个bin,相邻标记间的平均距离为0.98 cM。共检测到32个生育期相关性状QTLs,似然函数比值对数值(logarithm of odds,简称LOD)为2.52~10.86,贡献了3.33%~17.19%的表型变异。其中qDTT9-1、qDTS9-2在不同环境中稳定表达且效应较大,在育种中可能具有较大利用价值。本研究结果可为进一步精细定位生育期相关性状QTLs和利用分子标记辅助选择开展品种改良提供参考。
关键词:玉米;生育期;遗传图谱;QTL定位
中图分类号: S513.03 文献标志码: A
文章编号:1002-1302(2022)08-0063-06
生育期是玉米重要的性状之一,不同玉米品种生育期存在较大差异。在生产中,需要依据种植地区与播种时间选择生育期适宜的玉米品种以保证产量的稳定,因此通过育种手段选育适宜生育期品种就显得尤为重要。而抽穗期、散粉期、吐丝期作为3个最重要的生育指标,在玉米生长发育进程中占据重要地位,不仅与营养生长向生殖生长的转变有关,还与花期协调和籽粒产量密切相关[1-2]。
抽穗期、散粉期、吐丝期等性状均为典型的数量性状,由多基因控制。目前,国内外研究者对玉米生育期相关性状进行了较多的QTL定位分析[3-6]。公强等以CN165×单330(早熟种质)构建F2 ∶3家系对花期相关性状进行QTL鉴定,在3个环境检测到26个生育期相关性状的QTLs,可以解释的表型变异为7.1%~26.5%,3个环境中均于第8染色体上的phi060~umc2401区间检测到了与抽穗时间相关的QTL,其余QTL并未在3个环境重复检测到[7]。李玉玲等利用同样来源F2 ∶3和BC2S1群体对玉米生育期相关性状进行初步定位与分析,发现在2类群体中QTL检测结果存在较大差异,其中F2 ∶3群体检测出的QTL数目和贡献率均高于BC2S1,同时指出遗传背景差异对生育期主效QTL的检测影响较大[8]。Buckler等利用1组5 000个重组自交系在8个环境条件下对植株进行表型鉴定,检测到控制开花期、吐丝期等性状的位点均超过50个,未检测到单一大效应的QTL,只有7个等位基因对开花时间的影响超过1 d,该结果说明开花期性状是由多个微效基因所控制的[9]。Coles等通过温带玉米自交系和热带自交系构建了4个群体,利用多群体联合分析鉴定出了控制开花时间的基因组区域[10]。目前,有关生育期相关基因的克隆研究方面也取得了一定进展,已经克隆的基因包括第1染色体上的DWARF8(D8)和INDETERMINATE GROWTH1(ID1)、第7染色体的DELAYED FLOWERING 1(dlf1)以及位于第8染色体的VEGETATIVE TOGENERATIVE TRANSITION1(VGT1)[11-15],这些基因通过自主调控玉米开花转换、介导开花诱导信号的传递和调控营养生长向生殖生长转变等途径调节玉米生育期。尽管国内外学者在生育期相关基因定位和克隆方面取得了一些进展,但是由于不同研究中群体类型、遗传背景、群体规模、统计方法等因素的差异,检测的QTL结果不尽相同,在不同环境中能稳定检测到的QTL仍较少。除此以外,大多数定位和克隆的基因或位点的利用在育种实践中存在困难或局限,因此仍需开发新的位点。
本研究以玉米自交系LDC-1和YS501所衍生的重组自交系,构建高密度遗传连锁图谱,对生育期相关性状进行QTL定位,探寻能够在多个环境条件下均能稳定表达的QTL,旨在为解析玉米生育期的遗传机制、挖掘生育期有关基因及开展玉米分子育种研究提供参考。
1 材料与方法
1.1 材料
材料為玉米商用杂交种天玉88的2个亲本自交系LDC-1和YS501,以及该杂交种连续自交7代衍生的186个重组自交系。
1.2 田间试验设计
试验材料在2020、2021年2年分别种植于2个环境中:2020年夏种植在扬州大学扬子津校区试验田(E1,32.4°N、119.4°E);2021年夏种植在镇江世业洲试验田(E2,32.2°N、119.5°E)。田间采用随机区组设计,2次重复,每行10株,行长2 m,行距为60 cm,株距为20 cm,田间管理同一般大田。
1.3 性状调查
在玉米整个生长阶段调查玉米从播种到抽穗、散粉、吐丝的时间作为生育期相关指标,其中抽穗期(days to tasseling,简称DTT)记为从播种到抽出雄穗的时间,散粉期(days to pollen-shedding,简称DTP)记为从播种到开花散粉的时间,吐丝期(days to silking,简称DTS)记为从播种到雌穗吐丝的时间。以上数据以家系为单位进行统计。
1.4 数据处理
基于SPSS23.0软件对表型数据进行方差分析、相关分析和描述统计,利用Microsoft Excel 2016处理基因型数据。
1.5 连锁图谱构建与QTL分析
由北京中玉金标记生物技术股份有限公司对亲本及其衍生的重组自交系群体进行Mazie10K基因型检测。使用Microsoft Excel 2016软件按照以下步骤处理获得的基因型数据:(1)删除亲本基因型杂合与亲本间无多态性的标记;(2)删掉杂合率和缺失率大于20%的SNP标记;(3)筛选出等位基因频率大于0.05的标记;(4)利用binmap R包对获得的SNP数据进行纠错。遗传图谱基于R/qtl构建,通过Kosambi函数计算标记间距离,遗传图距的单位为厘摩(centimorgan,简称cM)。
各参数的QTLs检测采用Windows QTL Cartographer 2.5软件完成,使用Kosambi函数转化重组率为图距,作图步长為1.00 cM;采用复合区间作图法检测QTL位点,通过1 000次重排获得QTL显著的LOD阈值(P<0.05)。同一条染色体上的同一性状LOD相距10 cM左右;置信区间出现重叠的位点被认为是同一QTL[16]。
本研究中QTL命名参考McCouch等的方法[17],规则如下:生育期相关性状的QTL以qDTT2-1为例,q表示QTL,DTT为性状英文字母缩写,2-1表示在2号染色体检测到的第1个QTL。
2 结果与分析
2.1 遗传图谱构建
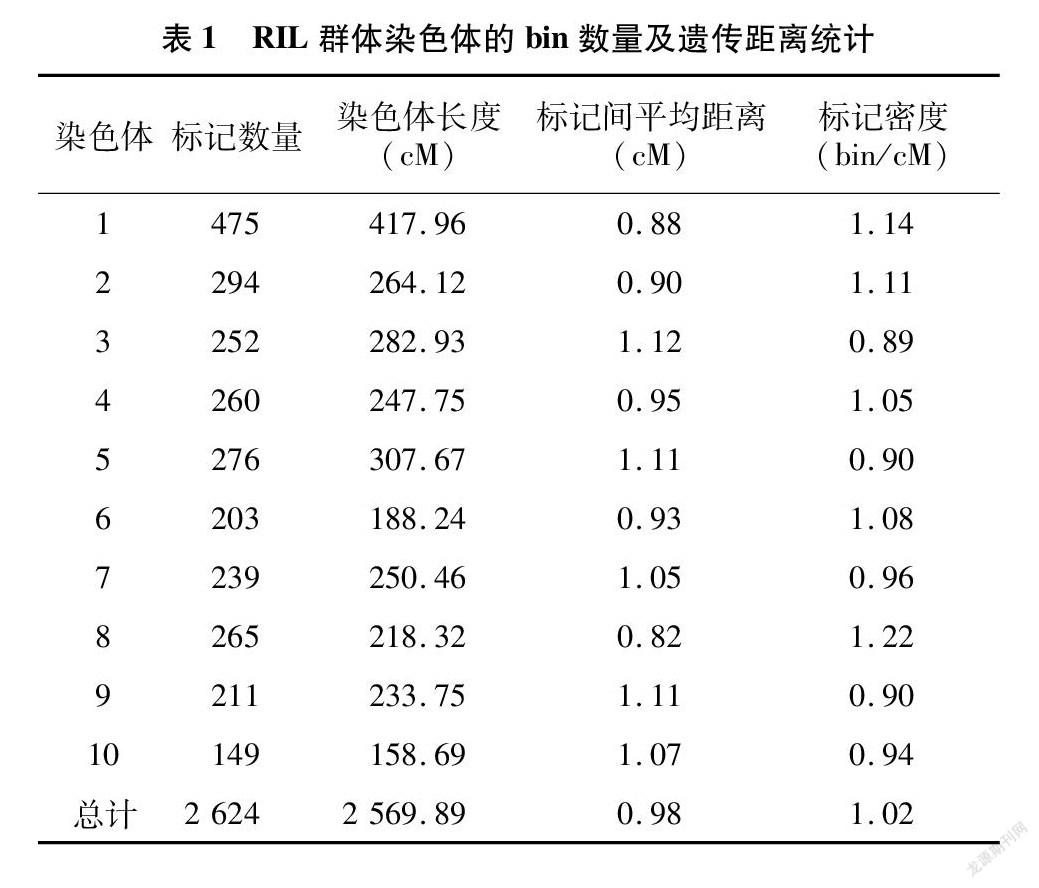
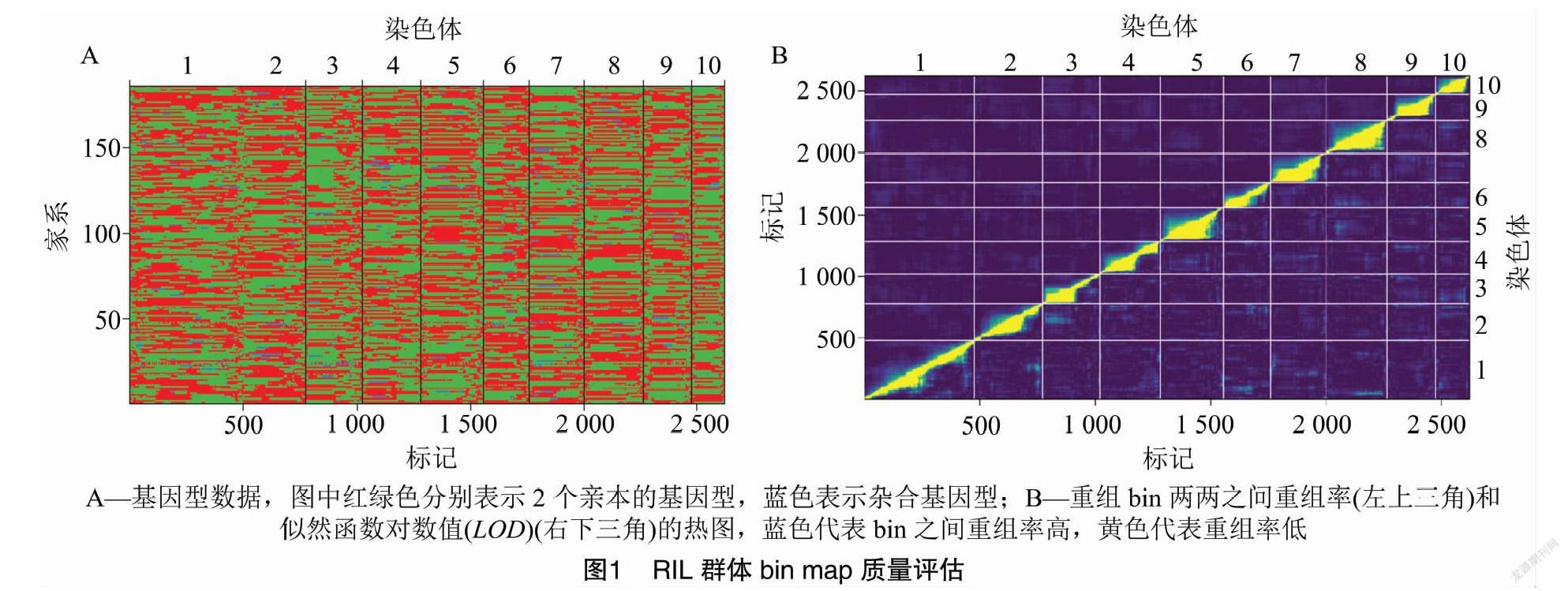
利用10K的SNP标记检测RIL群体的基因型,采用滑动窗口法,将连续多个标记基因型结果相同的SNP视为1个bin,排除基因型错误的SNP标记。经过筛选后bin map共包含2 624个bin(表1、图1),构建bin map遗传图谱,全长为2 569.89 cM,标记密度为1.02 bin/cM,标记间平均距离为0.98 cM。bin标记在每条染色体上的分布疏密不均,且密集区域不同,其中第1染色体的遗传距离最长(417.96 cM),且标记密度也最大,为1.14 bin/cM;第10染色体的遗传距离最短,为158.69 cM;第3染色体的标记密度最小,为0.89 bin/cM。依据bin在各染色体上的信息,计算bin两两之间的重组率(图1-B),表明邻近bin之间连锁程度较高,距离较远的bin之间连锁程度低,bin map质量较好。
2.2 重组自交系群体以及亲本的表型分析
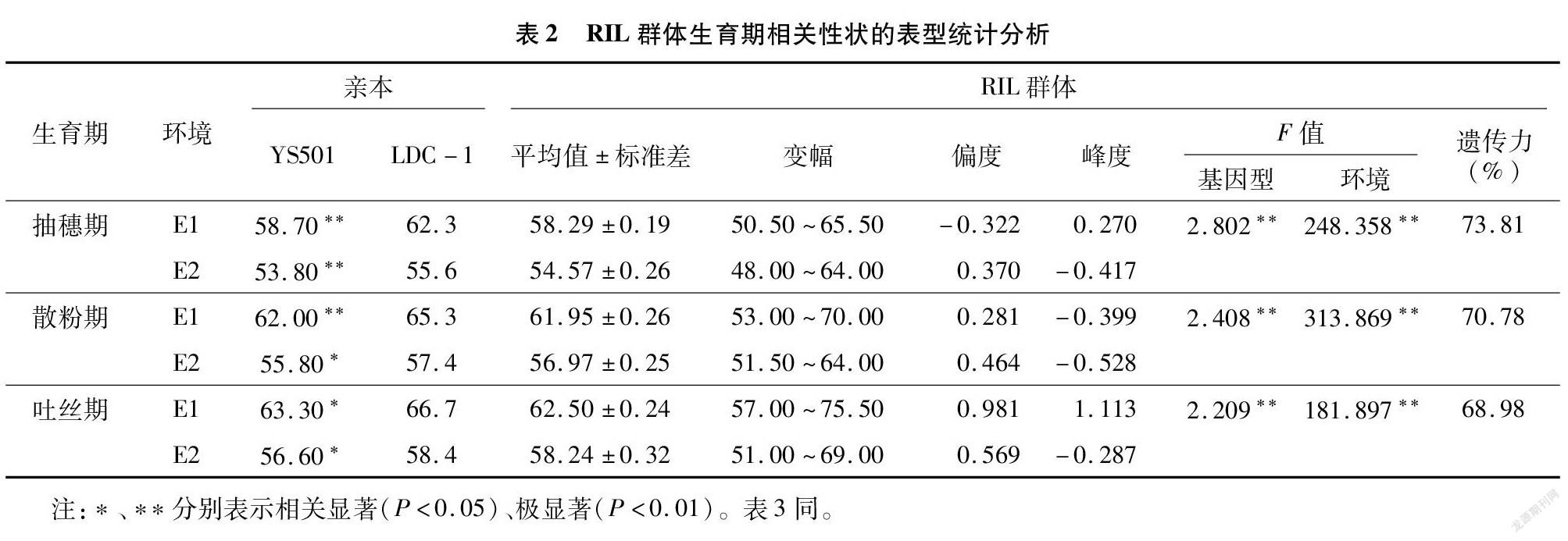
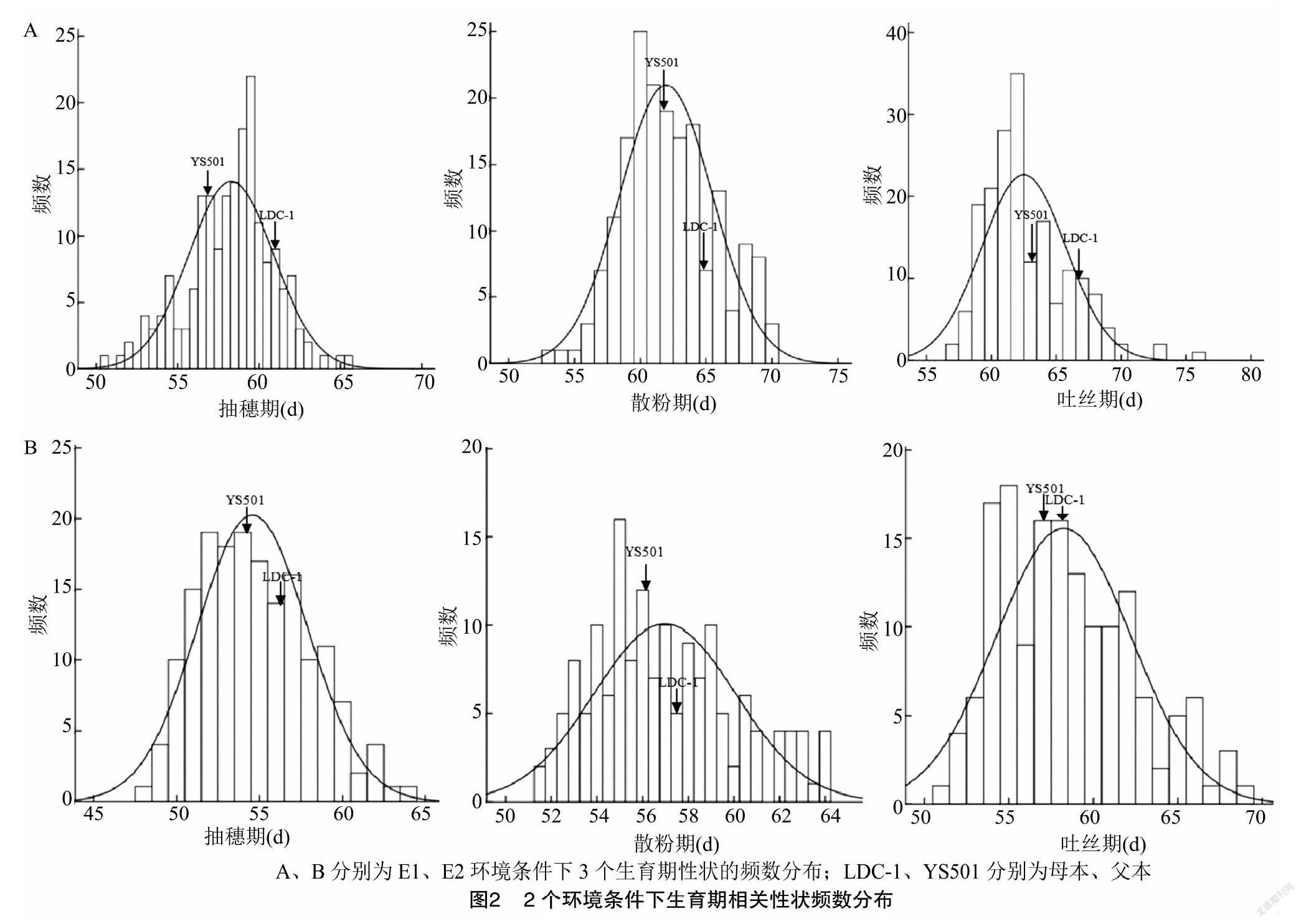
对RIL群体和亲本的生育期相关性状进行描述性统计分析(表2),发现2个环境中父母本生育期相关性状均存在显著差异,其中抽穗期的差异最大,LDC-1比YS501平均延迟了2.7 d。RIL群体中3个生育期的数据在不同环境的偏度和峰度绝对值均小于1(吐丝期峰度除外),大都近似正态分布(图2)。3个性状的遗传差异和环境差异都达到极显著水平,各个性状的广义遗传力为68.98%~73.81%(表2)。
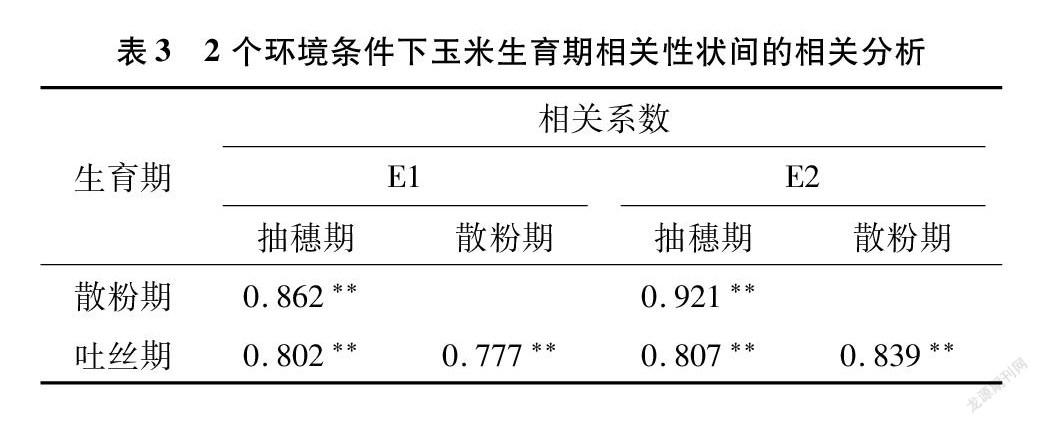
对2个不同环境条件下玉米RIL群体的抽穗期、散粉期和吐丝期进行相关性分析(表3),结果表明,2个环境条件下玉米RIL群体抽穗期、散粉期、 吐丝期两两之间均呈极显著正相关,相关系数均大于0.7。
2.3 生育期相关性状的QTL定位
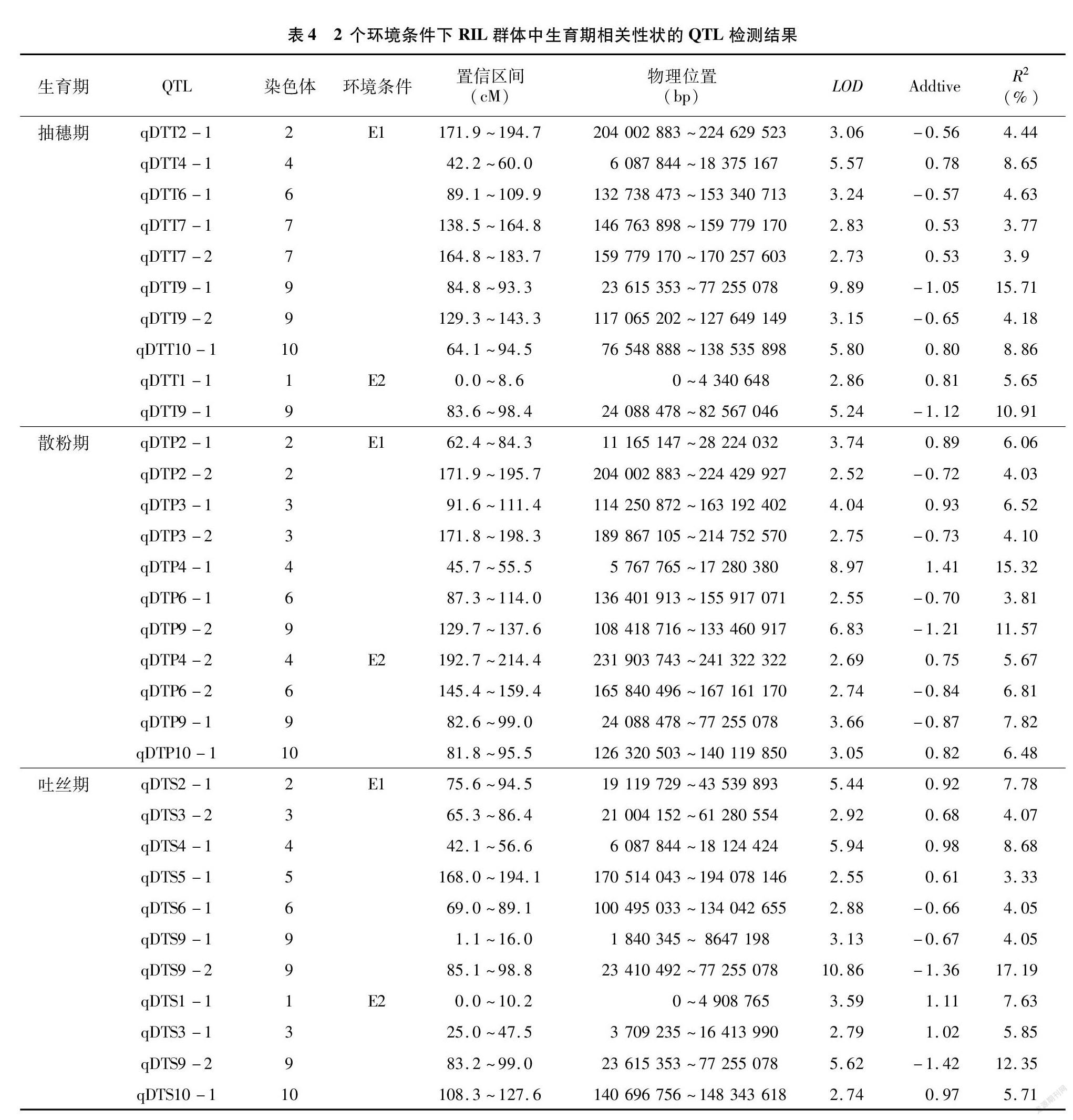
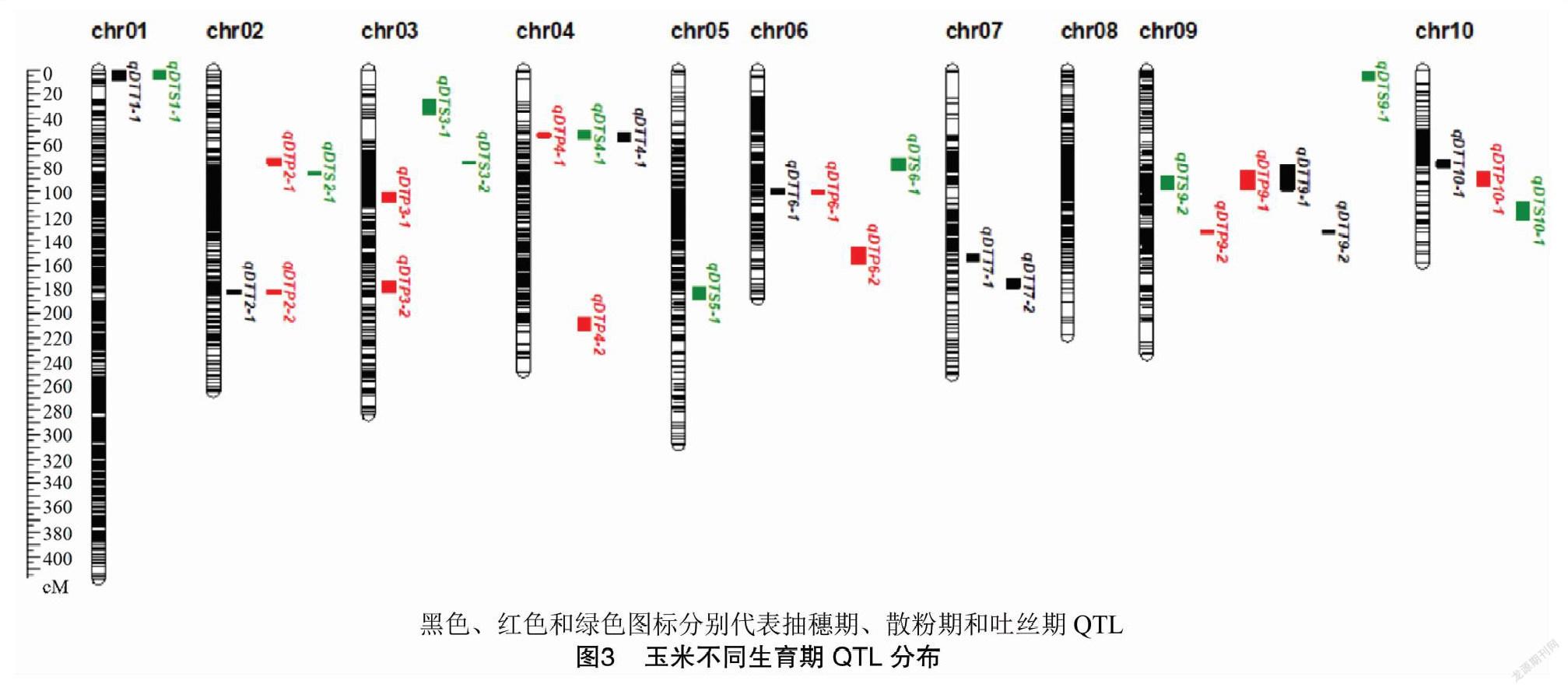
采用复合区间作图法(CIM)对抽穗期、散粉期和吐丝期3个性状进行QTL检测,于2020年扬州夏播和2021年镇江夏播2个环境中共检测到32个调控目标性状的QTLs(表4、图3),LOD值为2.52~10.86,表型变异贡献率为3.33%~17.19%。
由2个环境的定位结果可知,共检测到10个控制抽穗期的QTLs,分布在第1、2、4、6、7、9、10染色体上,解释了3.77%~15.71%的表型变异,LOD值为2.73~9.89。进一步研究发现,在第9染色体上的QTL qDTT9-1于2个环境中重复检测到,且该QTL表型贡献率最大,在E1、E2环境分别为15.71%、10.91%,加性效应均为负值,推测可能是同一QTL,来自母本LDC-1的等位基因对抽穗期起到了提前的作用。
在2个不同的环境中共检测到11个控制散粉期的QTL,分布在第2、3、4、6、9、10染色体上,LOD值为2.52~8.97,解释了3.81%~15.32%的表型变异。通过分析发现,于E1环境定位到的qDTP4-1对表型的贡献率最大,该QTL加性效应为1.41,解释了15.32%的表型变异,增效基因来自母本LDC-1。
对于检测到的吐丝期相关QTL,11个QTL分布于除第7、8染色体外的其余8条染色体上,解释的表型贡献率为3.33%~17.19%,LOD值为2.55~10.86。进一步分析发现, qDTS9-2在2个环境中均被检测到,且2个环境条件下的表型贡献率均高于10%,在E1、E2环境分别为17.19%、12.35%,LOD值最高为10.86,为吐丝期主效QTL,加性效应为负,该增效基因来自父本YS501。
3 讨论与结论
本研究于2个环境中共检测到32个控制生育期相关性状的QTLs,分布在除了第8染色体以外的9条染色体上。其中检测到的部分QTL与前人具有一致性,本研究检测到的qDTP3-2与曹浩飞等基于单片段代换系群体在3号染色体上umc1844-umc1320-bnlg1182标记区间内检测到的散粉期QTL qDTA3-2的定位区间重叠[18];7号染色体上的qDTT7-2与高世斌等利用N87-1×9526在7号染色体标记区间bnlg1805内定位到的开花期相关性状QTL区间重叠[19];9号染色体上的qDTT9-1、qDTS9-2与魏海忠等利用玉米自交系80007和80044所构建的重组自交系群体在第9染色体上于标记bnlg127-umc2337内定位到的生育期相关性状主效QTL区间重叠[20],这些与前人相似的结果说明本研究QTL检测结果的真实性与可靠性。进一步分析发现,其中qDTT9-1、qDTS9-2于E1、E2 2个环境中均能检测到,且表型贡献率较大(大于10%),上述QTL可能具有较高的育种利用价值,为后续开展精细定位与基因克隆等方面的研究奠定了基础。另外本研究还存在一些前人未发现的新QTL,但只在单环境中被检测到,如qDTT4-1、qDTP4-1等,这些QTL仍需进一步利用不同群体验证其真实性。
生育期作为典型的数量性状,对环境的变化较为敏感,因此在不同环境下控制生育期的基因表达可能不尽相同,这就会导致QTL检测结果有所差异。在本研究中,通过对2个环境的QTL检测结果分析发现,不同环境下的定位结果差异较大,只有2个QTL在2个环境中均被检测到,大多数QTL只在单环境中被检测到,QTL在多个环境下的重复性不高,这可能是由于试验的环境差异所致。相关研究也表明,环境的差异对生育期相关性状的影响较大,其中气温和光照是最主要的影响因素[21]。本试验中2个环境条件下的播种时间以及地理位置等因素导致试验环境间的气温和光照差异较为明显,这可能是导致控制抽穗期等性状的基因定位结果存在差异的原因。
此外,众多研究者提出不同性状的QTL位置相同应该与表型数据的相关性有关[22-23]。在本研究中生育期相关性状的表型数据在同一环境下高度正相关,而检测结果也出现了QTL成簇分布的现象,如于第4染色体相同区间检测到控制多个生育期相关性状的QTL簇,在第9染色体相同位置78.1~99.8 cM内检测到到多个QTL成簇分布(图3)。
参考文献:
[1]兰进好. 玉米开花期相关性状的QTL分析[J]. 西北植物学报, 2010,30(3):471-480.
[2]刘志斋,郭荣华,石云素,等. 中国玉米地方品种核心种质花期相关性状的表型多样性研究[J]. 中国农业科学,2008,41(6):1591-1602.
[3]胡彦民,吴 欣,李翠香,等. 玉米制种花期相关性状的QTL分析[J]. 南京农业大学学报,2008,31(1):11-16.
[4]孙婷婷,姜 昱,沈 鑫,等. 利用F2 ∶3群体对玉米花期相关性状的QTL分析[J]. 玉米科学,2013,21(2):42-46.
[5]Veldboom L R,Lee M,Woodman W L.Molecular marker-facilitated studies in an elite maize population:I.Linkage analysis and determination of QTL for morphological traits[J]. Theoretical and Applied Genetics,1994,88(1):7-16.
[6]Austin D F,Lee M.Genetic resolution and verification of quantitative trait loci for flowering and plant height with recombinant inbred lines of maize[J]. Genome,1996,39(5):957-968.
[7]公 强,王天宇,谭学林,等. 玉米优异早熟种质单330开花相关性状的QTL分析[J]. 植物遗传资源学报,2006,7(4):437-441.
[8]李玉玲,李学慧,董永彬,等. 利用相同来源F2 ∶3和BC2S1群体定位玉米生育期QTL[J]. 华北农学报,2007,22(6):38-43.
[9]Buckler E S,Holland J B,Bradbury P J,et al. The genetic architecture of maize flowering time[J]. Science,2009,325(5941):714-718.
[10]Coles N D,McMullen M D,Balint-Kurti P J,et al. Genetic control of photoperiod sensitivity in maize revealed by joint multiple population analysis[J]. Genetics,2010,184(3):799-812.
[11]Thornsberry J M,Goodman M M,Doebley J,et al. Dwarf8 polymorphisms associate with variation in flowering time[J]. Nature Genetics,2001,28(3):286-289.
[12]Colasanti J,Tremblay R,Wong A Y M,et al. The maize INDETERMINATE1 flowering time regulator defines a highly conserved zinc finger protein family in higher plants[J]. BMC Genomics,2006,7:158.
[13]Colasanti J,Yuan Z,Sundaresan V. The indeterminate gene encodes a zinc finger protein and regulates a leaf-generated signal required for the transition to flowering in maize[J]. Cell,1998,93(4):593-603.
[14]Salvi S,Sponza G,Morgante M,et al. Conserved noncoding genomic sequences associated with a flowering-time quantitative trait locus in maize[J]. Proceedings of the National Academy of Sciences of the United States of America,2007,104(27):11376-11381.
[15]Muszynski M G,Dam T,Li B L,et al. Delayed flowering1 encodes a basic leucine zipper protein that mediates floral inductive signals at the shoot apex in maize[J]. Plant Physiology,2006,142(4):1523-1536.
[16]Wang D,Portis A R.Increased sensitivity of oxidized large isoform of ribulose-1,5-bisphosphate carboxylase/oxygenase (rubisco) activase to ADP inhibition is due to an interaction between its carboxyl extension and nucleotide-binding pocket[J]. The Journal of Biological Chemistry,2006,281(35):25241-25249.
[17]McCouch S R,Chen X,Panaud O,et al. Microsatellite marker development,mapping and applications in rice genetics and breeding[J]. Plant Molecular Biology,1997,35(1/2):89-99.
[18]曹浩飛,王 彬,毛克举,等. 基于单片段代换系群体的玉米开花期性状QTL分析[J]. 河南农业大学学报,2014,48(1):6-10.
[19]高世斌,赵茂俊,潘光堂,等. 干旱胁迫与正常环境下控制玉米开花期性状的QTL鉴定[J]. 西南农业学报,2005,18(5):593-597.
[20]魏海忠,商 伟,钟世宜,等. 利用重组自交系群体定位玉米生育期相关性状QTL[J]. 玉米科学,2014,22(1):49-55.
[21]刘月娥. 玉米对区域光、温、水资源变化的响应研究[D]. 北京:中国农业科学院,2013.
[22]Abler B S B,Edwards M D,Stuber C W.Isoenzymatic identification of quantitative trait loci in crosses of elite maize inbreds[J]. Crop Science,1991,31(2):267-274.
[23]Paterson A H,Damon S,Hewitt J D,et al. Mendelian factors underlying quantitative traits in tomato:comparison across species,generations,and environments[J]. Genetics,1991,127(1):181-197.
