光照对紫叶稠李花色素苷合成调控通路的影响
2022-05-06蔡建超张克中崔金腾
马 赫,蔡建超,张克中,崔金腾
(北京农学院园林学院/城乡生态环境北京实验室/北京市乡村景观规划设计工程技术研究中心,北京 102206)
紫叶稠李(Padus virginiana)是蔷薇科稠李属落叶乔木,株高10—14 m,是一种珍贵的彩叶树种,在我国北方,气温达到一定程度,紫叶稠李的初生叶由绿色逐渐转为紫红色,叶色靓丽极具观赏价值[1-2]。
植物叶色变化主要由色素含量的相对变化引起,花青素是植物体内的天然色素[3],可以遮挡部分强光,帮助植物抵御环境胁迫,降低光合作用对叶片的伤害[4],朱书香等[5]通过研究4种李属植物,发现花色素苷能够形成“光屏障”,吸收植物体内的过剩光,缓解强光对植物光合作用的影响。
花青素的合成以苯丙氨酸为底物,经酶促反应、糖基化、酰基化等过程最终达到稳定状态,参与合成的有PAL、CHS、CHI、F3H、F3’H、DFR、ANS、UFGT等结构基因及MYB、bHLH、WD40等转录因子[6]。MYB转录因子对植物积累花青素具有重要意义,可通过调控花色素苷水平调控苹果果皮的红色和绿色条纹[7]。在杨树中,MYB6正向调控花青素和单宁生物合成。MYB、bHLH和WD40互作可形成三元复合物,参与调节晚期类黄酮生物合成基因的表达[8]。
本研究基于花青素光保护理论对紫叶稠李进行遮光处理,结合转录组测序及生物信息学分析,对花青素合成通路上的关键结构基因和MYB家族转录因子MYB6和MYB10进行验证,以期为紫叶稠李花色素苷调控机制提供理论基础。
1 材料与方法
1.1 试验材料
北京农学院实践基地中陆地生长的2年生紫叶稠李(Padus virginiana)扦插苗。
1.2 遮光处理
选取长势良好的紫叶稠李60棵,30棵进行20%光照处理(采用80%遮阳网),另外30棵在自然条件全光照下生长。试验期间,进行常规管理,保持植株长势良好。遮光处理前1 d采集1次叶片,采集部位为顶端倒3叶,命名为Day0,处理30 d后,再分别采集1次叶片,20%光照处理组命名为ST_Day30,自然条件全光照处理组命名为Day30。试验时间为2019年4月至5月。
1.3 叶色参数测定
采用柯尼卡美能达(中国)投资公司的色彩色差计(CR-400/410)测定叶色参数,L*表示照度,相当于亮度,L*的值域由0到100,表示由黑色向白色转变。a*和b*的值域都是-127至+128,a*表示由绿色向红色转变;b*表示由蓝色向黄色转变,所有颜色以这三个值交互变化表示。
1.4 花色素苷含量测定
采用盐酸提取比色法测定花色素苷含量[5],称取植物样品0.50 g,置于离心管中,加0.1 mol/L盐酸溶液20 mL,使样品完全浸于溶液中,置于32℃恒温箱中浸提8 h,使用光径1 cm的比色皿,以0.1 mol/L盐酸为空白对照,测量波长530 nm下的吸光值。花色素苷含量(mg/L)=A(530)/0.1×样品质量(g)
1.5 RNA的提取和转录组测序
使用北京艾德莱生物科技有限公司EASYspin Plus RNA快速提取试剂盒,提取紫叶稠李叶片总RNA。通过1%琼脂糖凝胶电泳检测RNA完整性。使用Nanodrop2000检测RNA的浓度和纯度。采用Illumina Hiseq 2500进行转录组测序,由上海美吉生物医药科技有限公司完成。
1.6 基因功能注释、转录因子预测和差异基因分析
将拼接得到Unigene序列与Nr、Swiss-Prot、Pfam、KOG、GO和KEGG六大数据库比对获得功能注释,与PlantTFDB数据库比对得到转录因子信息,利用DEGseq进行组间差异表达分析,通过与GO和KEGG数据库比对,获得差异基因的功能注释及通路注释。
1.7 关键基因的qRT-PCR
使用北京全式金生物技术有限公司试剂盒TransScriptOne-Step gDNA Removal and cDNA Synthesis SupperMix将RNA反转录为cDNA,PCR程序设定为:55℃30 min,85℃5 s。使用北京全式金生物技术有限公司试剂盒TransScripTtop Green qPCR SupperMix配置荧光定量体系,qPCR程序设定为:94℃30 s,94℃5 s,60℃10 s,70℃10 s,45 cycles。所用引物见表1。根据富集结果筛选出花色素苷代谢通路PAL、CHS、CHI、F3H、F3’H、DFR、ANS、UFGT八个关键功能基因及转录因子MYB6和MYB10并进行实时荧光定量分析。

表1 引物序列Table 1 Primers and sequences
2 结果与分析
2.1 遮光对紫叶稠李叶色参数的影响
光照是影响彩叶植物叶色变化的重要环境因子之一,在长期的进化过程中,植物通过改变形态结构、生理生化等方式应对复杂的光环境[1]。由图1和图2可知,自然全光照的叶片由绿色变为紫红色,80%遮光处理30 d后的紫叶稠李叶片并未完全变色,与全光照30 d的叶片相比,a*值减小,b*值增加,叶片亮度L*值增强,叶面与叶背颜色不同,叶面偏绿色,叶背偏紫红色。

图1 光照强度对紫叶稠李叶片颜色变化的影响Fig.1 Effects of light intensity on leaf color change of Padus virginiana
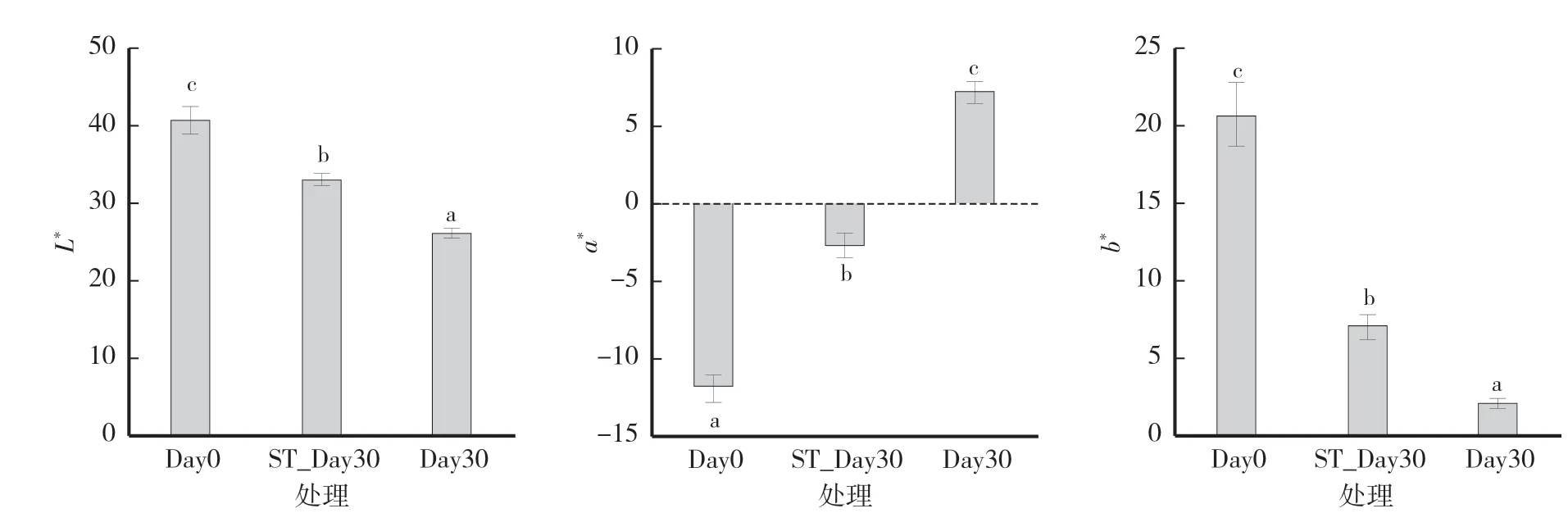
图2 遮光对紫叶稠李叶色参数的影响Fig.2 Effects of shading on leaf color parameters of Padus virginiana
2.2 遮光对紫叶稠李叶片中花色素苷含量的影响
光照可通过促进可溶性糖的积累来激活花色素苷合成的某些酶类,从而促进花色素苷的合成。全光照30 d后叶片花色素苷含量显著提高,而遮光处理30 d的叶片,花色素苷虽然也有积累,但伴随着光照强度的减弱,积累量明显小于全光照组(图3)。
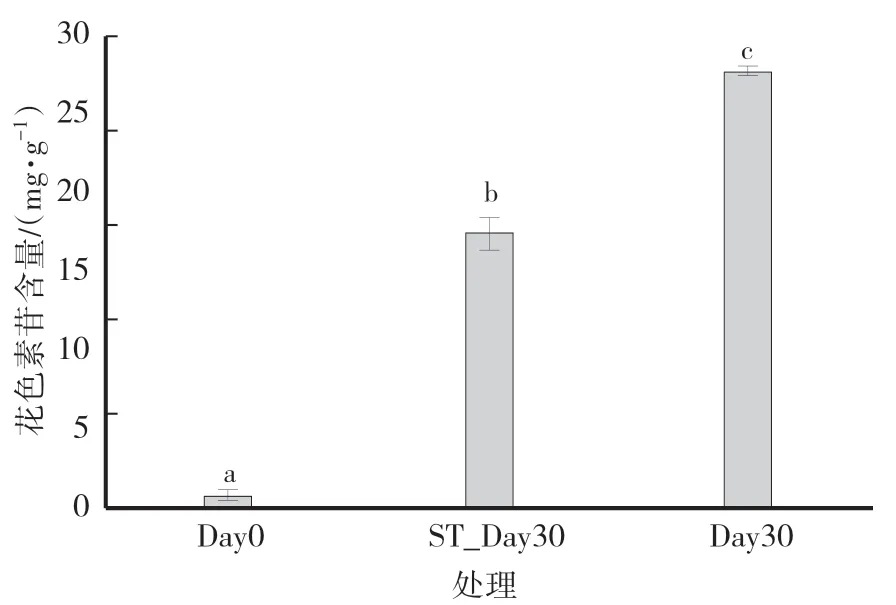
图3 遮光对紫叶稠李叶片花色素苷含量的影响Fig.3 Effects of shading on the anthocyanin content of Padus virginiana
2.3 紫叶稠李转录组测序质量统计
对Day0、Day30、ST_Day30三个试验组进行转录组测序,设置两次重复,总共获得145.87GB的Clean Data,具体结果见表2。Day0、Day30、ST_Day30分别获得Clean reads 102 560 220、118 348 698、112 063 772,单次重复获得的Clean reads大于46 931 024,N50值1 266 bp,Q20值超过98%,Q30值超过94%,GC含量处于46%—47.1%,无AT和GC分离,测序数据质量较高。图4显示,Unigene的长度分布于1—400区间的最多,达到了40 000以上,随着分布区间的变长,数量逐渐减少。
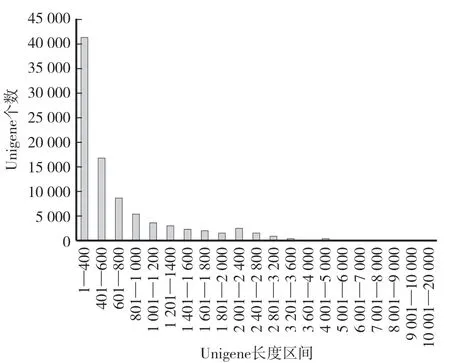
图4 紫叶稠李Unigene长度分布Fig.4 Length distribution of Unigenes of Padus virginiana

表2 质控数据统计Table 2 Quality control data statistics
2.4 紫叶稠李转录组Unigene的功能注释
本试验中紫叶稠李为无参转录组测序,将该次转录组测序获得的所有转录本与六大数据库(NR,Swiss-Prot,Pfam,COG,GO和KEGG数据库)进行比对,E-value值设为1e-3,Unigene所占比例分别为52.3 %、42.83%、34.8%、10.17%、20.62%、27.41%、58.62%,Unigene在NR和KEGG数据库中比对占比最高(表3)。
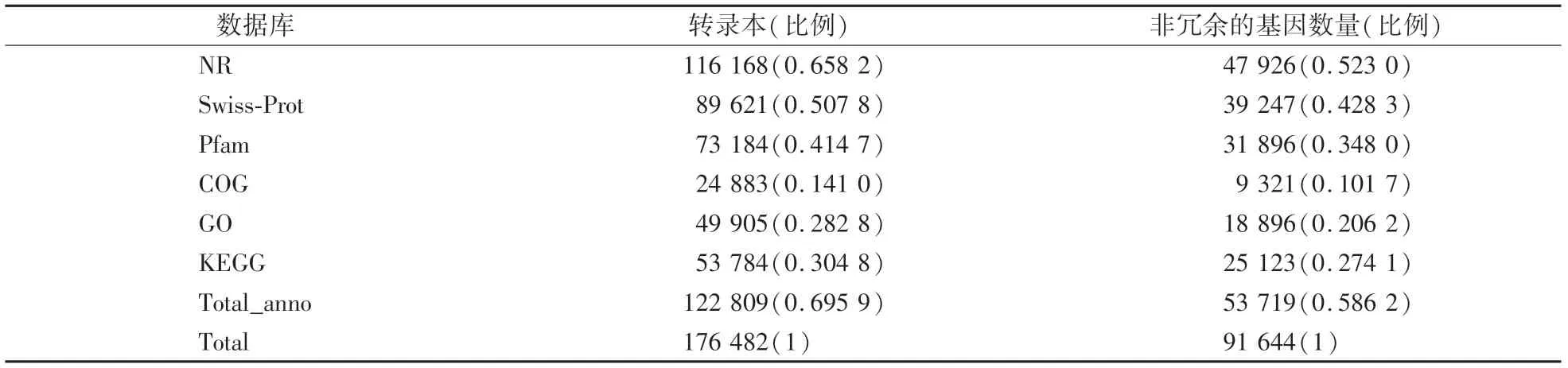
表3 Unigene与数据库比对结果Table 3 Comparison of Unigene and six databases
NCBI_NR为综合数据库,其中包含Swiss-Prot、PIR、PRF、PDB等蛋白质数据库中非冗余的数据以及从GenBank和RefSeq的CDS数据库中翻译所得的蛋白质数据。通过与NR数据库的比对,可以查看紫叶稠李转录本序列与相近物种的相似情况,以及同源序列的功能信息。NR注释显示紫叶稠李与欧李(Prunus avium,31%)、桃(Prunus persica,24.17%)、梅(Prunus mume,14.52%)3个物种同源关系最近(图5)。

图5 紫叶稠李NR库比对物种分布占比Fig.5 Distribution of species distribution in Padus virginiana NR library
2.5 遮光对紫叶稠李基因表达的影响
设置Day30_vs_ST_Day30、Day0_vs_Day30、Day0_vs_ST_Day30三个组件对比分析。对不同样品间的raw counts进行基于TMM方法的标准化,默认利用DEGseq软件进行组间差异表达分析,默认阈值:padjust<0.05&|log2FC|>=1。以基因在两个样本间表达差异的倍数变化值为横坐标,P值为纵坐标,绘制差异基因表达火山图(图6),从图中可看出,Day30_vs_ST_Day30差异表达基因共2 432条,其中上调1 396条,下调1 036条;Day0_vs_Day30差异表达基因共2 811条,其中上调806条,下调2 005条;Day0_vs_ST_Day30差异表达基因共3 368条,上调1 376条,下调1 992条。
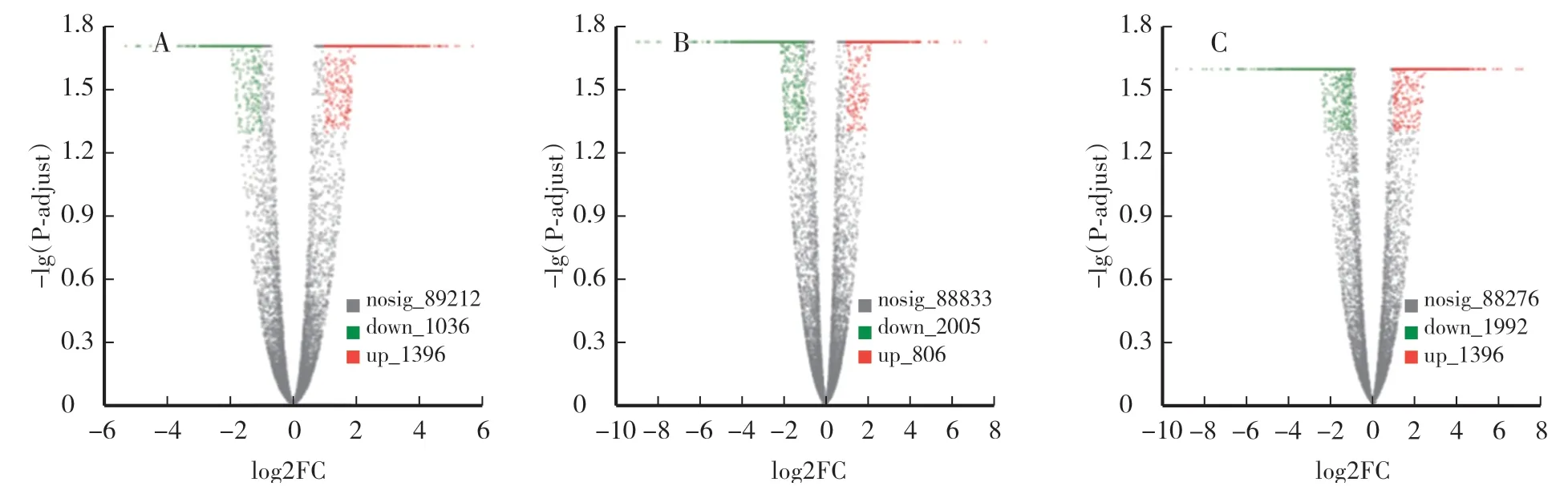
图6 遮光处理下紫叶稠李差异基因表达分析Fig.6 The analysis of differential gene expression of Padus virginiana in shading
通过与GO数据库比对,对紫叶稠李的差异基因按照其细胞组分(Cellular Component)、分子功能(Molecular Function)和参与的生物过程(Biological Process)进行分类注释,筛选显著富集在前20位的Go Term绘制折线图(图7),设置筛选参数P值(FDR)<0.05,由图7可知,Day30 vs ST_Day30有1 125条基因富集到177个GO term中,其中,主要富集在细胞大分子代谢过程(Cellular macromolecule metabolic process)、细胞过程(Cellular process)、细胞内部分(Intracellular part)、氧化还原过程(Oxidation-reduction process)、氧化还原酶活性(Oxidoreductase activity)、氮化合物代谢过程(Nitrogen compound metabolic process);Day0 vs Day30有1 396条基因富集到211个GO term中,其中,主要富集在碳水化合物代谢过程(Carbohydrate metabolic process)、细胞碳水化合物代谢过程(Cellular carbohydrate metabolic process)、多糖代谢过程(Polysaccharide metabolic process)、细胞壁组织(Cell wall organization)、细胞多糖代谢过程(Cellular polysaccharide metabolic process)、细胞外区域(Extracellular region)、蛋白质复合物(Protein complex)、外部封装结构(External encapsulating structure);Day0 vs ST_Day30有1 069条基因富集到215个GO term中,其中,主要富集在催化活性(Catalytic activity)、细胞内部分(Intracellular part)、氧化还原过程(Oxidation-reduction process)。

图7 遮光处理下紫叶稠李差异基因GO富集分析Fig.7 The analysis of differentially expressed genes in GO enrichment of Padus virginiana in shading
通过与KEGG数据库比对,获得紫叶稠李的基因或转录本对应的KO编号,根据KO编号获得Unigene可能参与的具体生物学通路情况。紫叶稠李KEGG注释到遗传信息处理(Genetic Information Processing),代谢(Metabolism),环境信息处理(Environmental Information Processing),细胞过程(Cellular Processes),生物体系统(Organismal Systems)等通路,筛选显著富集在前10位的差异基因绘制KEGG代谢通路气泡图(图8),设置筛选参数P值(FDR)<0.05。在Day30 vs ST_Day30显著富集在前10位的代谢通路中,苯丙氨酸代谢(Phenylalanine metabolism)、淀粉和蔗糖代谢(Starch and sucrose metabolism)、氰氨基酸代谢(Cyanoamino acid metabolism)、植物激素信号转导(Plant hormone signal transduction)、脂肪酸生物合成(Fatty acid biosynthesis)5条代谢途径富集的差异基因较多,分别为50、38、27、38和15个差异基因;Day0 vs Day30显著富集在前10位的代谢通路中,苯丙烷类生物合成(Phenylpropanoid biosynthesis)、氨基糖和核苷酸糖代谢(Amino sugar and nucleotide sugar metabolism)、植物激素信号转导(Plant hormone signal transduction)、脂肪酸生物合成(Fatty acid biosynthesis)和氰氨基酸代谢(Cyanoamino acid metabolism)5条代谢途径富集的差异基因较多,分别为40、32、32、24和24个差异基因;Day0 vs ST_Day30显著性富集在前10位的代谢通路中,苯丙烷类生物合成(Phenylpropanoid biosynthesis)、脂肪酸生物合成(Fatty acid biosynthesis)、淀粉和蔗糖代谢(Starch and sucrose metabolism)、氨基糖和核苷酸糖代谢(Amino sugar and nucleotide sugar metabolism)和类黄酮生物合成(Flavonoid biosynthesis)5条代谢途径富集的差异基因较多,分别为36、27、46、36和18个差异基因。

图8 遮光处理下紫叶稠李差异基因KEGG富集分析Fig.8 The analysis of differential expression gene KEGG enrichment of Padus virginiana in shading
Day0 vs ST_Day30有279条差异基因显著富集到14条代谢通路上,为苯丙烷类生物合成(Phenylpropanoid biosynthesis)、脂肪酸生物合成(Fatty acid biosynthesis)、淀粉和蔗糖代谢(Starch and sucrose metabolism)、氨基糖和核苷酸糖代谢(Amino sugar and nucleotide sugar metabolism)、类黄酮生物合成(Flavonoid biosynthesis)代谢途径。
2.6 紫叶稠李转录因子统计
转录因子是一类能与基因5’端上游特定序列专一性结合,调控目的基因以特定强度在特定时空表达的蛋白质分子。通过与PlantTFDB 4.0数据库比对,得到紫叶稠李转录因子1 261条,其中MYB超级家族最多,共有213条,占总数的17%,数量是其他转录因子的两倍以上,AP2/ERF家族96条,bHLH家族87条,NAC家族68条,bZIP家族59条(图9)。
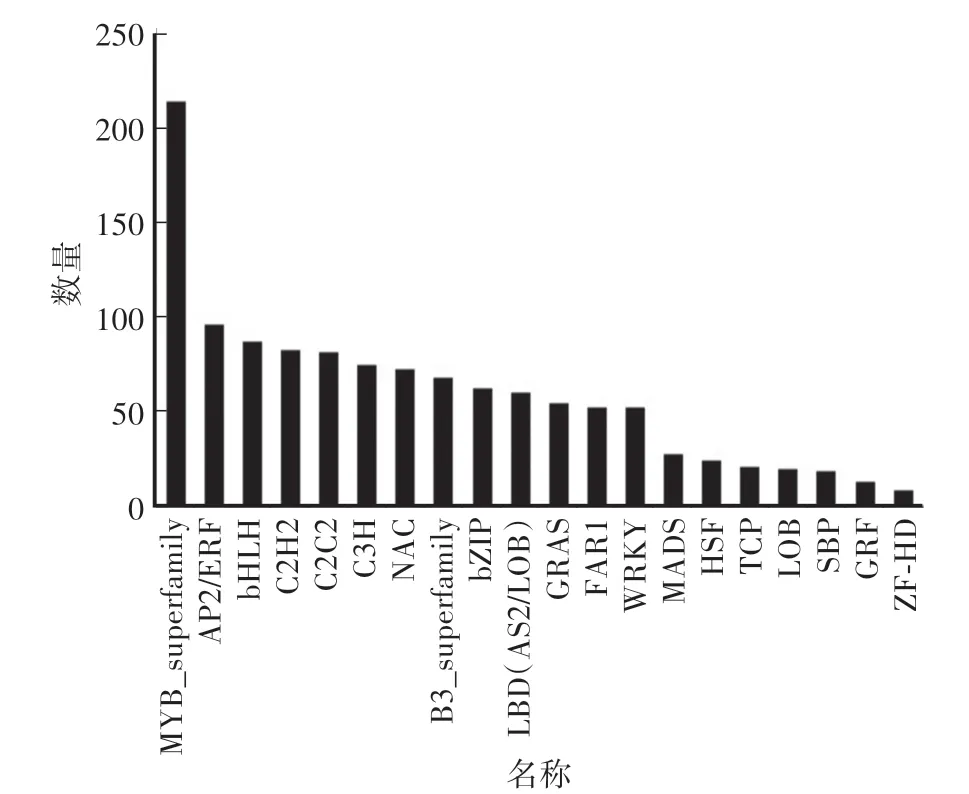
图9 遮光处理下紫叶稠李转录因子统计Fig.9 Transcription factor statistics of Padus virginiana in shading
2.7 紫叶稠李花青素合成途径关键基因的qRT-PCR验证
对比Day30自然全光照条件,紫叶稠李在经过30 d的遮光处理后,花青素合成通路上的关键酶PAL、CHS、CHI、F3H、F3’H、DFR、ANS、UFGT均呈现下调趋势,花青素合成在遮光下受到了抑制,MYB6与MYB10呈表达上调趋势(图10),说明其对花色素苷合成途径相关基因起到负调控的作用。
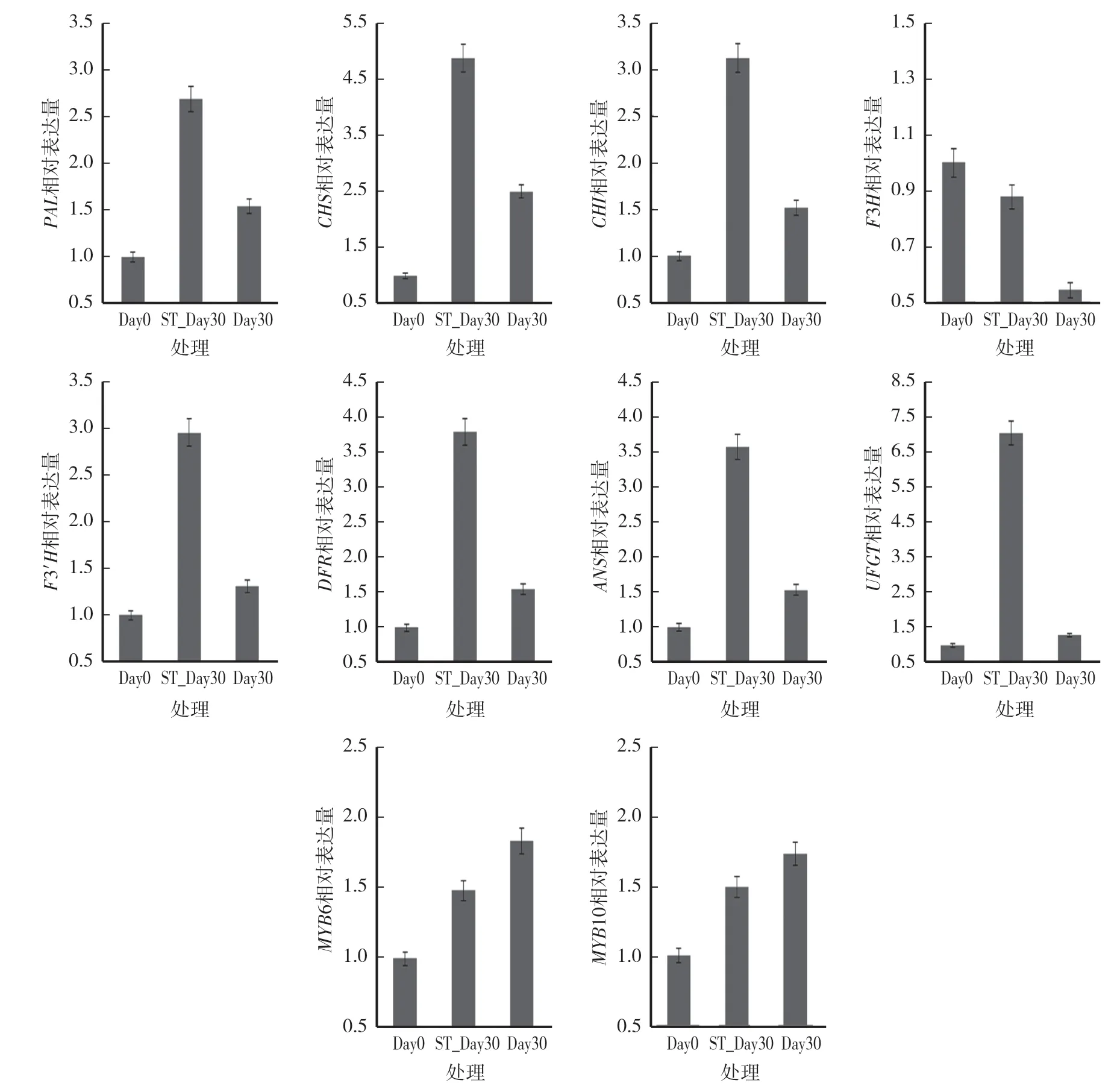
图10 紫叶稠李花青素合成途径关键基因的荧光定量分析Fig.10 The qRT-PCR verification of key genes in the anthocyanin synthesis pathway of Padus virginiana
3 结论与讨论
孟祥春等[9]研究发现,非洲菊遮光后花色素的积聚减少,无法进行正常上色。PAL苯丙氨酸解氨酶是连接初生代谢与次生代谢途经的纽带,是多酚类代谢的起始酶[10-11]。顾辰辰等[12]对茶树短期遮光的影响研究表明,短期遮光处理对茶树花色素苷代谢通路关键酶基因的表达影响很大,PAL、F3’H、DFR、UFGT出现下调现象。周波等[13]研究发现,草莓果实花色素苷的合成量受到光敏感度的影响,光不敏感草莓果实的花色素苷合成相关基因CHS、DFR、ANS遮光与不遮光表达没有明显差异,而光敏感草莓果实表达量受光部分要高于遮光部分。F3’H基因的上调会导致Penstemon barbatus花从蓝色到红色的变化[14],说明紫叶稠李叶片的花色素苷含量与其有密切关系,UFGT是花色素苷合成过程中最后一个酶,主要是在花色素苷合成后,对碳骨架起到糖基化修饰的作用[15],它能将不稳定的花色素苷进行糖基化,使其转变为稳定的花色素苷。UFGT基因的表达与花色素苷含量变化趋势相同,这一结果与苹果、梨、葡萄中的报道一致[16-17]。
调节植物花色素苷代谢途径的转录因子主要有MYB、bHLH和WD40,能与结构基因的启动子结合,并影响花色素苷生物合成途径中一个或多个结构基因的表达,从而改变花色素苷的代谢[18-19]。Yao等[20]研究发现,转录因子PyMYBll4和PyMYB10存在相互作用及功能叠加的效应,使花色素苷的生物合成得到加强。
本研究发现,遮光对紫叶稠李叶片淀粉和蔗糖代谢、苯丙烷类生物合成、植物激素信号转导途径影响不大,可能是植物适应环境并进行正常生长发育的必要生命代谢活动,氰氨基酸代谢途径的富集说明植物启动了自身的防御机制以抵抗病虫害,另外,遮光也较显著影响了类胡萝卜素生物合成过程、α-亚麻酸的代谢过程,半萜类三萜类生物合成过程及玉米素合成过程。遮光对植物体内类黄酮生物合成有显著影响,类黄酮是植物重要的一类次生代谢产物,花色素苷是类黄酮合成途径的一个分支。本研究发现,遮光对紫叶稠李叶片的着色有显著影响,并减少了叶片中的花色素苷合成,通过转录组测序分析发现,遮光与全日照条件下的差异基因主要富集在类黄酮生物合成代谢通路,进一步说明光照是紫叶稠李叶片着色和花色素苷代谢的关键因素之一,荧光定量分析显示,遮光使花色素苷合成过程中的关键酶基因PAL、CHS、CHI、F3H、F3’H、DFR、ANS、UFGT表达下调,关键转录因子MYB6、MYB10表达上调。
