葡萄膜黑色素瘤生存预后相关lncRNA的初步筛选及相关性分析
2022-04-29尚会平林祥祥
尚会平 林祥祥
作者单位:1 温州医科大学附属眼视光医院 眼视光学和视觉科学国家重点实验室,温州 325027;2 温州医科大学附属第一医院 浙江省神经老化与疾病研究重点实验室,温州 325015
葡萄膜黑色素瘤(Uveal melanoma,UM)是一种恶性肿瘤,起源于眼睛中的黑色素细胞。在加拿大,UM的年发病率为每年每百万人3.75例。在美国,UM的总发病率为每百万人4.637 例。UM转移具有倾向性,大约一半的UM患者会发生转移,尤其是在肝脏。大约50%的UM患者在诊断后10年内会发生转移。多年来,随着治疗策略的不断进步,肿瘤的控制率有所提高,但生存率尚无明显提高。
在以往的研究中,发现了一系列与UM 预后相关的基因。其中黑色素瘤特异性抗原基因(Preferably presented as an antigen of melanoma
,PRAME)
与肿瘤的直径、转移与存活率均呈正相关关系。在83%的UM中发现鸟嘌呤核苷酸结合蛋白α亚基q基因(G protein subunit alpha Q
,GNAQ)
和鸟嘌呤核苷酸结合蛋白α亚基11基因(G protein subunit alpha 11
,GNA11)
的点突变与肿瘤的发生发展相关。但是,随后的研究发现,GNAQ
和GNA11
与UM 的转移及生存率没有良好的相关性。在对转移性和非转移性UM的预后分析中发现,p52与各种类型的UM预后相关,而RelB的蛋白表达水平与非转移性UM的生存率相关。通过共表达网络分析(The weighted gene co-expression network analysis,WGCNA),研究人员鉴定出了与UM预后相关的一系列关键基因,如:slic17A7
、ntrkrk2
、Abtb1
和AdpRHL1
等。但是,到目前为止,与UM预后相关的基因鉴定工作鲜见报道。本研究从长链非编码RNA(Long non-coding RNA,lncRNA)角度出发,寻找与UM预后生存相关的lncRNA,并通过内源竞争RNA(Competing endogenous RNAs,ceRNA)机制的探讨,分析候选lncRNA 参与UM发生发展的可能分子机制。1 资料与方法
1.1 材料
2019年7月13日从肿瘤基因组图谱数据库(The Cancer Genome Atlas,TCGA,https://cancergenome.nih.gov/)下载最新更新的全部80 例葡萄膜黑色素瘤患者(Project ID:TCGA-UM)的原发灶RNA-seq测序数据(HTSeq-FPKM)、miRNA-seq测序数据及其临床信息,并收集相关的临床数据,包括年龄、性别、肿瘤TNM分期和生存时间等信息。纳入的TCGA-UVM均为原发灶样本,且均已经过患者病理信息、肿瘤样本纯度、测序数据质量等步骤的质量控制。
1.2 分析方法
1.2.1 UM预后相关lncRNA筛选
使用转录组相关软件Cuffdiff(2.2.1 版本)将FPKM 值转换为每百万次阅读的转录本数据(Transcripts Per Million Reads,TPM)。使用统计计算软件R survival包(4.0.5版本)和生存分析曲线绘制软件survminer包(0.4.9版本)分析TCGA数据库中的UM肿瘤信息(TCGA-UM)中lncRNAs表达与生存预后的关系。通过检索RNALocate数据库(http://www.rna-society.org/rnalocate/)筛选可能定位于细胞质的候选lncRNAs。
1.2.2 LncRNA的靶miRNA预测及与UM生存预后相关的靶miRNA筛选
通过mircode数据库(http://www.mircode.org/)检索lncRNA可能可以结合的miRNA家族,再通过Targetscan数据库(http://www/targetscan.org/vert_72/)检索miRNA家族对应的miRNA。使用R survival包和survminer包分析TCGA数据库中miRNAs表达对UM生存预后的影响,筛选与UM生存预后相关的靶miRNA。
1.2.3 miRNA-靶基因关系的多数据库验证及靶基因KEGG通路富集分析
通过targetscan数据库(http://www.targetscan.org/),miRTarBas数据库(http://mirtarbase.mbc.nctu.edu.tw/)和miRDB数据库(http://mirdb.org/)检索候选miRNA对应的靶基因。采用KEGG功能富集及其可视化软件clusterProfiler(4.1版本)对基因集进行KEGG通路富集分析。
1.2.4 LncRNA-miRNA-mRNA分析
使用生物信息学可视化软件cytoscape(3.9.0版本)对lncRNA-miRNA作用关系及miRNA调控靶基因进行可视化分析。
1.3 统计学方法
采用R软件(4.0.0 版本)进行统计、生物信息学分析并作图。使用STATS包的Loess函数,对TCGA-UM RNA-seq中的所有基因的TPM值和生存时间进行局部加权回归(Loess回归)分析,将回归模型预测时间与实际生存时间值进行Spearman相关性分析,P
<0.05 且r
>0.8,则认为该基因与生存预后明显相关。预后相关miRNAs的筛选方法同上。使用 clusterProfiler软件(3.18.0版本)对所有候选功能基因进KEGG通路功能分析,使用 Benjamini-Hochberg法对多重检验的P
值进行校正,以校正后P
<0.05为富集有统计学意义。2 结果
2.1 与生存预后相关的lncRNAs
本研究筛选到579个表达水平与生存预后明显相关的lncRNAs(P
<0.05),见表1。作为ceRNA中发挥对下游靶基因调控作用的lncRNA,一般位于细胞质中。因此,通过 RNALocate数据库,从候选lncRNAs中进一步筛选到17 个定位于细胞质的lncRNAs,见表2。2.2 lncRNA的靶miRNA鉴定结果
lncRNA可以作为竞争性结合位点结合miRNA,调节下游基因表达。因此,我们利用Mircode数据库检索17个候选lncRNA可能结合的miRNA家族,并通过targetscan数据库检索对应的miRNA。最终,17个lncRNAs共找到70个miRNA家族以及对应的154个靶miRNA分子,见表3。
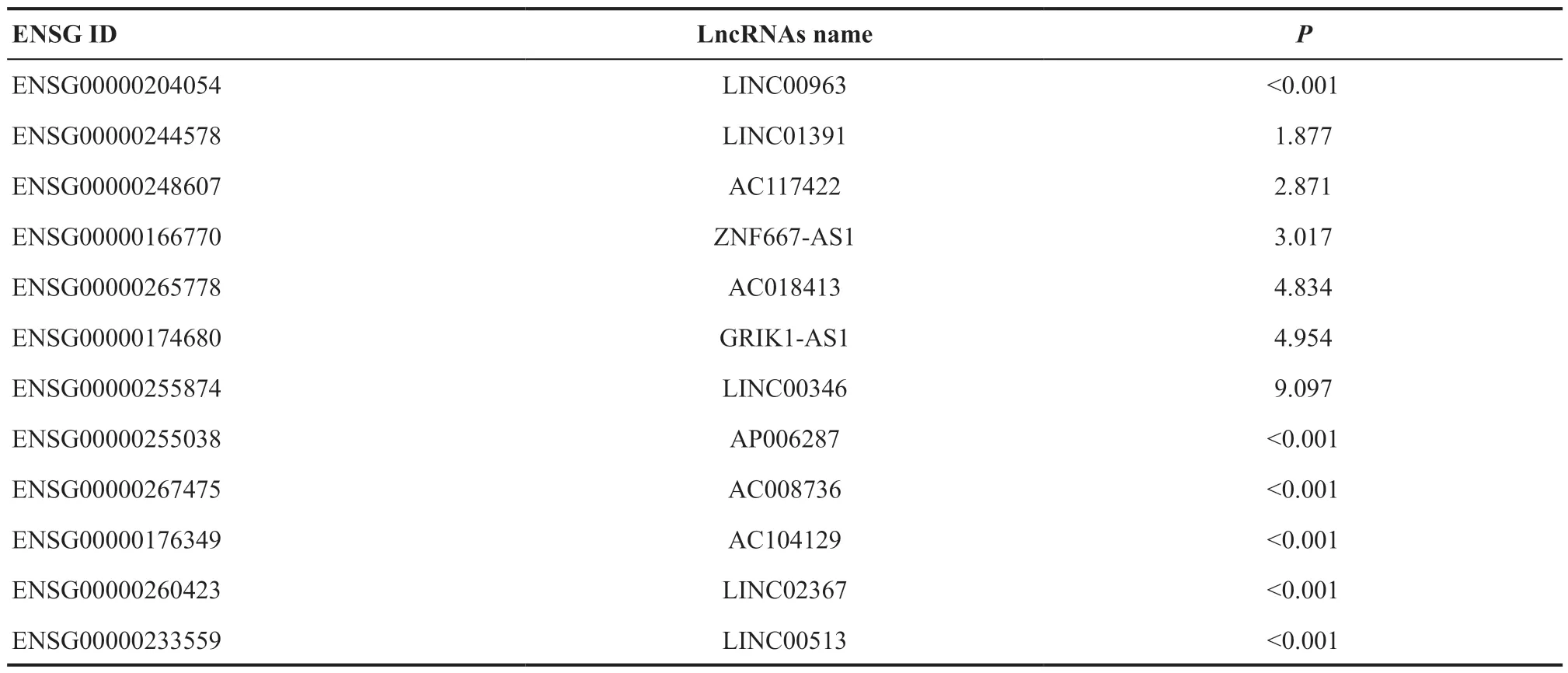
表1.表达与生存预后明显相关的lncRNAsTable 1.Expression of lncRNAs significantly correlated with survival prognosis
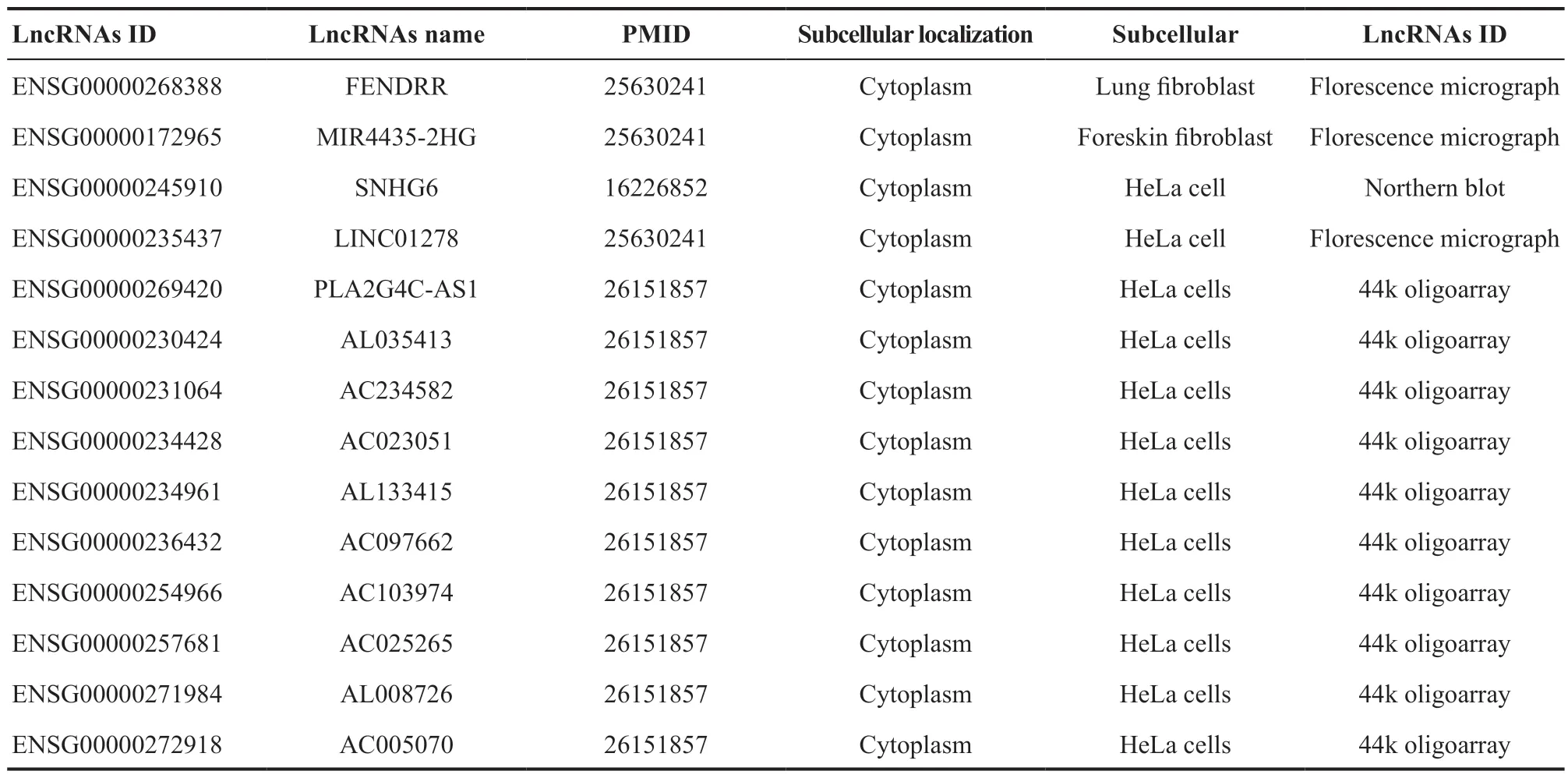
表2.定位于细胞质的候选lncRNAsTable 2.Candidate lncRNAs localized in cytoplasm

表3.lncRNAs的下游靶miRNAs筛选Table 3.Downstream target miRNAs of lncRNAs
2.3 候选lncRNA下游与生存相关miRNA-mRNA的鉴定
在鉴定出的154个miRNA基础上,为进一步明确这些miRNA是否与UM生存预后相关。通过Loss回归分析,我们发现120个miRNA与UM的生存预后明显相关(P
<0.05)。然后,在154个靶miRNA和与UM预后相关的120个miRNA中,通过Venn分析筛选出共同的miRNA,最终筛选到与UM预后相关且被lncRNA可能吸附的15 个前体miRNA。见图1A。MicroRNA(miRNA)通过与下游功能基因的靶向结合实现对靶基因的转录表达调控。因此,通过targetscan数据库(http://www.targetscan.org/)、miRTarBase数据库(http://mirtarbase.mbc.nctu.edu.tw/)和miRDB数据库(http://mirdb.org/)检索15个前体miRNA对应的靶基因,并通过venn方法分析,筛选3个数据库中同时存在的miRNA-mRNA。结果发现在miRTarBase数据库中检索到8 053 个miRNA-mRNA,miRDB数据库中检索到23 646个miRNA-mRNA,targetscan数据库中检索到46 377个miRNA-mRNA。多数据库验证,共筛选到630对共有的miRNA-mRNA,其中29个成熟miRNA和578个基因。见图1B。

图1.UM预后相关miRNA-mRNAA:候选lncRNAs下游靶miRNAs和UM预后相关miRNA的Venn分析;B:Targetscan、miRTarBas、miRDB数据库中共有的候选miRNA-mRNAFigure 1.MiRNA-mRNA associated with prognosis of uveal melanoma.A:Venn analysis of downstream target miRNAs of candidate lncRNAs and prognostic related miRNAs in uveal melanoma.B:Candidate miRNA-mRNAs common to TargetScan,miRTARbas,and miRDB databases.
2.4 预后相关lncRNA-miRNA-mRNA的ceRNA调控网络
ceRNA调控网络中,lncRNA结合miRNA,调控基因表达。我们从17个候选lncRNA中鉴定出了9个可能发挥ceRNA功能的lncRNA(见表4),这些lncRNA调控578 个功能基因的表达。然后,对这些候选功能基因进行KEGG功能分析,结果发现这些基因集中表达在前列腺癌和胰腺癌中。此外,这些功能基因也集中表达在PI3K-Akt、MAPK以及Hippo等肿瘤相关的关键信号通路,说明鉴定的578个基因中确实存在参与肿瘤发生发展过程的关键分子,见图2A。最后,使用cytoscape软件绘制ceRNA调控网络。同时我们对富集到的黑色素瘤KEGG通路进行了可视化。图2B展示了SNHG6、LINC01278等lncRNA通过调控miRNA-mRNA参与UM发生发展过程。
3 讨论
UM恶性程度高、致死率强。尽管在放化疗的作用下,眼部肿瘤的控制得到了明显改善。一旦发生转移,患者的生存时间将明显缩短,其死亡率目前尚无明显改变。因此,寻找UM生存预后相关的诊断指标对改善患者的预后具有重要的临床价值。如类似EZH2抑制剂3-deazaneplanocin A(DZNep)可抑制人UM细胞增殖,具有治疗UM的应用前景。人的3 号染色体缺失及某些基因表达水平差异可能是用于评估UM预后的可靠指标,UM通常含有激活G蛋白α-q信号级联的突变,影响PLCB4 或CYSLTR2 的表达。其中,UM细胞中的BAP1
基因的突变与肿瘤大小和睫状体损伤相关,而在家族性UM和家族性癌症综合征(皮肤黑色素瘤、间皮瘤和其他肿瘤)中,BAP1
的突变似乎也特别常见。除此之外,PRAME
、EIF1AX
、FOXO3
、ITPR2
、GNAQ
和GNA11
等基因的突变,也被认为参与了UM的病理进程。lncRNAs的大小超过200 bp,可以通过调控基因表达来调控细胞增殖、分化和凋亡等一系列重要的细胞过程。lncRNAs可以通过与细胞内遗传物质,包括DNA、RNA和蛋白质的相互作用,发挥各种生物学功能。因此,lncRNAs在转录抑制、转录激活、染色体修饰等方面均发挥重要作用。越来越多的研究证明,lncRNAs参与了肿瘤细胞的恶性过程,包括增殖、侵袭和干细胞特性。到目前为止,大多数lncRNAs的生物学功能仍处于未知状态。已有的相关报道中,lncRNAs参与基因表达调控的主要分子机制之一是通过竞争性结合靶标miRNA,从而解除miRNA对下游功能基因的转录抑制。如在胰腺癌中,LINC00673能够增强PTPN11 与E3 泛素连接酶PRPF19 的相互作用,从而增强S TAT 1 依赖的抗肿瘤效应,LINC00673 第4 外显子的点突变(G>A)使得LINC00673能特异性结合miR-1231,更容易表现出肿瘤易感性。在与UM生存预后相关的lncRNA
中,linc00963(MetaLnc9)已被证明在非小细胞肺癌组织中高表达,且与不良预后呈正相关,Linc00963的高表达促进了非小细胞肺癌细胞的迁移、入侵以及增强转移能力。在肝癌中,LINC01391可诱导β-catenin与ICAT相互作用增强,通过抑制β-catenin与TCF/LEF相互作用来调控肿瘤的生长。这说明筛选出的lncRNA分子确实可能在肿瘤的发生发展及转移中发挥重要功能。

表4.lncRNA靶向miRNA及mRNATable 4.lncRNA targeting miRNA and mRNA

图2.UM预后相关lncRNA-miRNA-mRNA的调控机制A:UM预后相关lncRNA下游靶基因的KEGG功能富集图;B:UM预后相关lncRNA-miRNA-mRNA通路调控黑色素瘤发生发展进程Figure 2.Regulation mechanism of lncRNA-miRNA-mRNA related to prognosis of uveal melanoma.A:KEGG function enrichment diagram of downstream target gene of lncRNA related to prognosis of UM.B:Prognostic related lncRNA-miRNA-mRNA pathway of UM regulates the occurrence and development of melanoma.UM,uveal melanoma.
在对lncRNA的下游靶基因miRNA及功能基因的预测中,我们鉴定出了630 对候选miRNAmRNA。在对功能基因的KEGG分析中,发现鉴定出的lncRNA的下游调控基因主要富集在参与肿瘤病理进程的信号通路中。以SNHG6 为例,我们发现SHNG6 可能通过调控has-miR-214-5p、hasmiR-214-3p、has-miR-let-7b-5p、has-miR-let-7c-3p等microRNA,调节下游的功能基因BAX
、IGF1R
、KRAS
、PIK3R1
、PDGFD
等。之前已有学者发现SNHG6 的表达升高与结直肠癌进展相关,并证明SNHG6可通过与miR-26a、miR-26b、miR-214相互作用来调控EZH2,促进细胞的生长、迁移和侵袭。在胶质瘤中,SNHG6可以竞争性地吸收miR-543,从而调节胶质瘤细胞中LMO3的表达及肿瘤的生长。在肝癌中,SNHG6作为ceRNA调控网络中竞争性结合的位点,使has-let-7c-5p、c-Myc表达上调,促进了肝癌细胞的增殖。但是,在UM中,关键lncRNA的ceRNA机制研究鲜见报道。这意味着本研究鉴定出的SHNG6 信号通路可能在UM的发生发展、迁移及侵袭等方面扮演着重要角色。因此,这些候选的与UM预后相关的lncRNA及其信号通路有待后续进一步的功能验证。综上,本研究通过构建ceRNA网络,探索与UM相关的lncRNA及其下游调控关系。筛选出的9 个可能发挥ceRNA功能的lncRNA可为研究UM的进展机制提供依据,SHNG6、LINC01278 等lncRNA可能在UM发生、迁移及侵袭等方面起重要作用。其对应的网络调控关系为后续更深入研究lncRNA-miRNA-mRNA调控机制及探索UM治疗靶点提供了参考和方向。
利益冲突申明
无任何利益冲突作者贡献声明
尚会平:参与选题、设计、资料的分析和解释和修改论文的结果、结论。林祥祥:收集数据;参与选题、设计及资料的分析和解释;撰写论文;根据编辑部的修改意见进行修改