加权基因共表达网络在肌少症基因筛选中的应用研究
2022-04-24黄玲莉
黄玲莉
(上海理工大学健康科学与工程学院,上海 200093)
0 引言
肌少症是一种随着年龄增长而导致进行性肌肉量减少、肌肉力量下降或躯体功能减退的老年综合征。患有肌少症的个体会因骨骼肌衰退、骨—肌单位萎缩,容易发生跌倒造成骨折,并且该症状与认知障碍、呼吸系统疾病和心血管疾病相关,会导致患者生活质量下降。研究表明,从40岁至80岁,人体的骨骼肌总量将下降30%~50%;60岁后,肌肉功能每年将下降3%;80岁后,患肌少症的概率高达67.1%。
随着中国进入老龄化社会,肌少症的预防和治疗已受到了人们的广泛关注,但目前人们对肌少症的认识仍然较少,发病机理尚不明晰。因此,迫切需要挖掘肌少症的相关基因用于研究其发病机理,并为预防、诊断和治疗该疾病提供参考。
1 研究现状
随着步入大数据时代,高通量测序技术、网络方法和计算机技术快速发展,从计算角度挖掘疾病的生物标志物或治疗靶点已成为目前的研究热点。王莉华等通过差异基因分析法对肌少症进行分析,但该方法忽略了基因间的内部相关性,即具有相同或相似表达的基因可能具有相似功能。Shin等利用TargetScan算法从Dlk1-Dio3 miRNA中挖掘有关肌少症的靶点基因,但该算法的处理对象为单个miRNA,无法同时考虑全基因组信息。为此,Zillikens等提出了全基因组关联分析方法(Genome-wide Association Study,GWAS)以解决TargetScan算法中存在的问题,该方法从基因HSD17B11、VCAN、ADAMTSL3、IRS1、FTO中发现了有关肌少症的5个新位点,但GWAS会随着测序数据量的增加,影响计算速度。同时,GWAS存在多重检验问题,会导致筛选的基因不完全是致病基因。
然而,加权基因共表达网络分析(Weighted Gene Coexpression Network Analysis,WGCNA)的提出为解决以往研究存在的问题提供了新的思路。该方法是一种基于拓扑矩阵所构建的共表达网络,使用无监督聚类法确定簇的数据挖掘方法。相较于上述方法,WGCNA可从整体基因组进行研究,通过确定基因表达水平间的关联及生物功能的相似性,将基因集分为一个个模块以挖掘关键基因,这样既能缩小筛选范围,减少计算量,又能较好地控制多重检验的假阳性率,提高识别准确率。
目前,WGCNA已被广泛应用于肿瘤、帕金森、糖尿病等疾病的研究当中,其稳定性和有效性已在多项研究中得以证实。例如,Su等使用WGCNA筛选卵巢癌的关键基因。Haase等将WGCNA应用于研究Rett综合症。而本文首次运用WGCNA分析肌少症,以期为肌少症的致病机制及治疗研究提供新的生物标志物或候选靶点分子。
2 基于WGCNA的基因识别
本文利用WGCNA筛选肌少症基因,具体实验过程包含以下6个部分:①对原始数据进行预处理;②依据基因间的关联性构建共表达网络;③利用共表达网络对基因集进行模块识别;④将划分后的模块与外部性状进行关联分析,以确定关键模块;⑤在关键模块中筛选肌少症的枢纽基因;⑥对枢纽基因进行功能验证。
2.1 数据预处理
本文使用的肌少症相关基因表达数据和临床信息(GSE111016)来源于美国国立生物信息中心的GEO(Gene Expression Omnibus)数据库(https://www.ncbi.nlm.nih.gov/gds/),该数据库是目前国际上最全面的高通量基因表达公共数据库,其中包含了20例肌少症患者和20例健康对照者,然而发现数据库中的样本3存在数据缺失问题,需要将其剔除。
图1为通过类平均法计算样本间的距离所得到的样本聚类树。由图1可见,将聚类高度设为77时,样本11、样本13和样本18为离群样本,需要先将其剔除。然后,对剩余的基因表达数据进行log2转换,并进行TMM归一化处理,最终得到36例临床信息的16 879个基因表达数据用于后续研究分析。
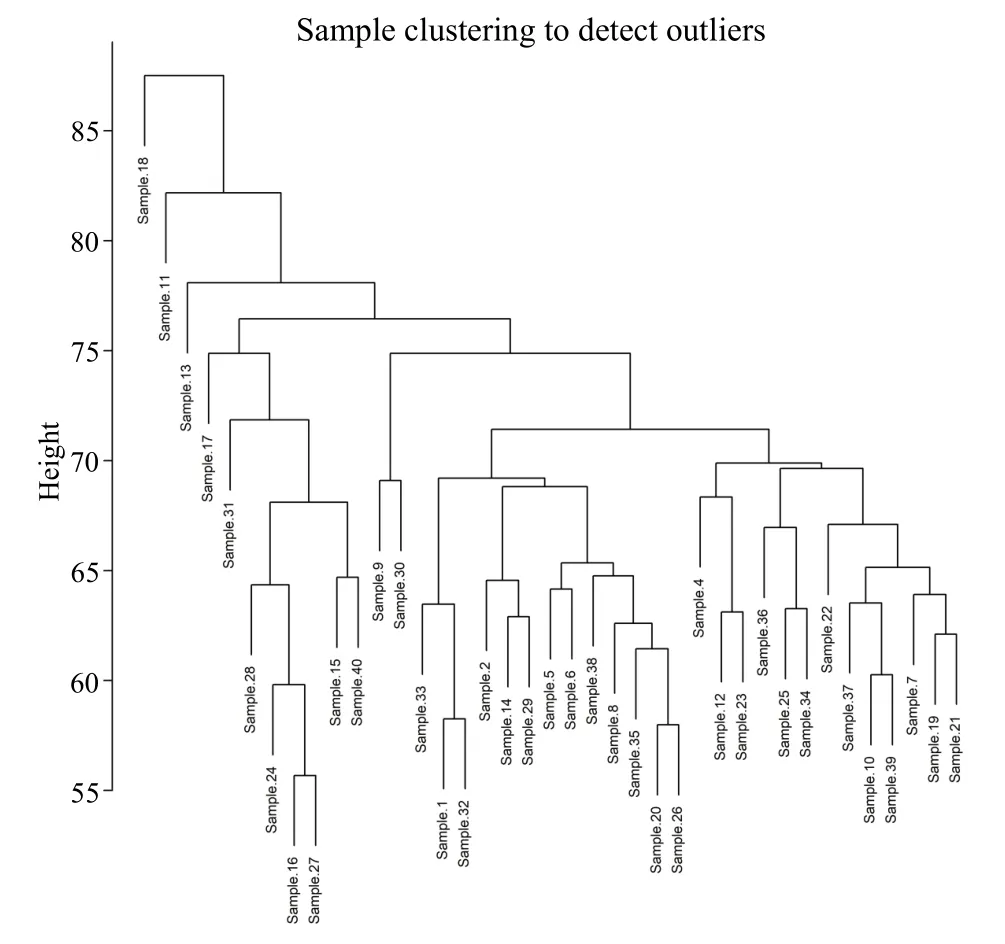
Fig.1 Sample hierarchical clustering tree图1 样本分层聚类树
2.2 共表达网络构建
基因共表达网络关系的构建通常由邻接矩阵定义,该矩阵的元素大小表示两个节点间的连接强度。本研究在构建邻接矩阵前,先按式(1)、式(2)定义一个中间变量,即相似度矩阵S。
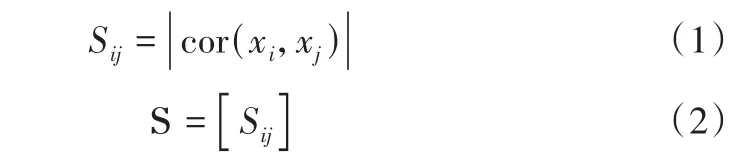
x
,x
表示任意两个基因。然后,利用阈值化方法将相似度矩阵S转化为邻接矩阵A,传统邻接矩阵计算公式为:
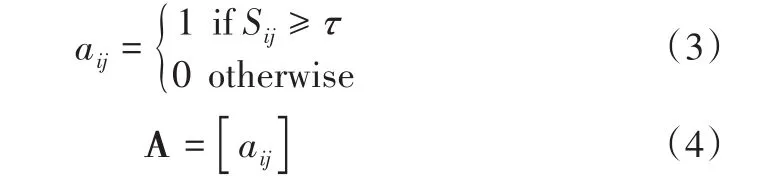
其中,τ为所取阈值。
但由于该阈值法为硬阈值,无法反映共表达信息的连续性,易丢失部分信息,并且该方法只能表示基因间是否存在联系,无法表示关系的强弱。为此,本文通过引入软阈值β
对相似度进行幂运算,计算公式如式(5):
β
≥1。接下来,使用R软件的pick Soft Threshold()函数对软阈值进行筛选,并对筛选出的软阈值进行无尺度拓扑检查。该操作决定了所构建网络是否符合无尺度网络,幂运算使得之前关系紧密的基因不受影响或者影响较小,而相关性较弱的基因相关性则会明显下降,进而表示关系的强弱。
考虑到两个基因间除直接影响外,还会受到其它基因的间接调控,本文根据拓扑重叠公式将邻接矩阵A转化为拓扑重叠矩阵Ω。该矩阵既能在网络拓扑水平上体现基因间的强度大小,还能减少噪音和假阳性,使结果更稳健。计算公式如式(6)-式(8)所示:

I
表示引入第3个基因u
之后,第i
个基因和第j
个基因新的连通性大小。2.3 基因模块识别
本文使用无监督聚类法确定基因模块,具体通过层次聚类算法实现,如公式(9)所示:

同时,利用动态剪切树法将层次聚类树切分为若干模块,每个模块表示共表达程度较高的一类基因。
2.4 关键模块确定
首先对每个模块矩阵进行主成分分析,得到模块特征值。然后,提取聚类后的模块特征值与外部性状向量,以计算Pearson的相关系数。最后,选择与肌少症具有显著关系(p
<0.05)的关键模块进行后续枢纽基因的筛选。同时,提取关键模块内每个基因的基因显著性值(Gene Significance,GS)和模块身份值(Module Membership,MM)以进一步验证所选模块的可靠性。通过分别计算GS和MM的相关系数,若其结果越显著(p
<0.05),则表明所选模块越稳健。2.5 枢纽基因筛选
计算关键模块内各基因的|MM|、|GS|值,可有效提高筛选枢纽基因的准确性。因此,本文通过分析大量数据后,选择|MM|>0.8、|GS|>0.2、P
<0.05为筛选条件。2.6 基因功能验证
GO(Gene Ontology)是研究基因功能的重要数据库,现已被广泛用于分析、验证高通量基因数据。基于GO对筛选出的基因进行富集分析,当P
<0.05时表示基因组与对应的功能具有统计学意义,证明所选基因是合理、有效的。3 实验结果与分析
3.1 WGCNA结果与分析
通过pick Soft Threshold()函数得出理想软阈值为5,其无尺度拓扑分布检验结果如图2所示。其中,k
为连接度,横坐标log10(k
)表示某基因连接数的对数,纵坐标表示该基因出现概率的对数,拟合系数R=0.84>0.8,斜率为-2.29,呈线性关系,表明所选软阈值符合无尺度拓扑分布,所构建的网络具有较强的鲁棒性。如表1所示,WGCNA将过滤后的肌少症公共数据集聚类为29个基因模块。其中,不同颜色代表不同基因模块,结果跟理论预期相一致,每个模块都包含具有相似功能的多个基因。

Fig.2 Scale free topology check with soft threshold of 5图2 软阈值为5的无尺度拓扑检查
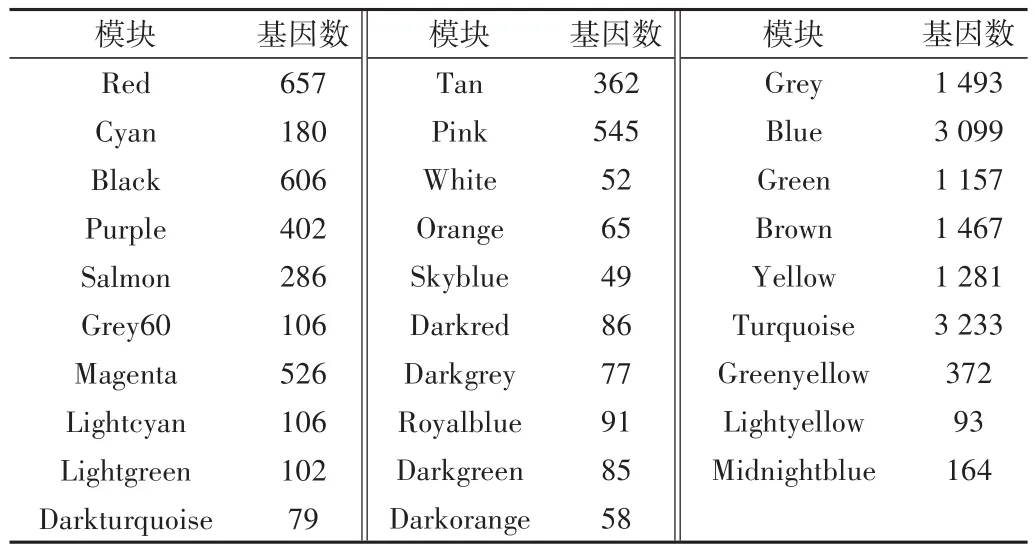
Table 1 Number of module genes表1 模块基因数
图3为识别出的29个基因模块与样本表型(肌少症和健康受试者对照性状)关系的可视化图(彩图扫OSID码可见)。其中,红色表示正相关,蓝色表示负相关,颜色越深则相关性越强,可见Skyblue模块(r
=0.53,p
=9E-04)、Turquoise模块(r
=0.38,p
=0.02)和Black模块(r
=0.37,p
=0.02)与肌少症具有显著正相关性,而与对照组呈负相关。因此,可确定这3个模块为肌少症的关键模块。此外,所选关键模块的GS和MM值也表明了筛选结果的可靠性。实验从3个关键模块中共识别出了86个枢纽基因,并通过GO验证了69个,该结果与当前文献记载肌少症是多基因参与的复杂疾病的结论相一致。实验结果表明,WGCNA在识别枢纽基因时,准确率达到了80.2%。表2为关键模块中的枢纽基因。其中,Skyblue模块存在17个枢纽基因,验证成功15个;Turquoise模块存在68个枢纽基因,验证成功53个;Black模块只有1个枢纽基因并已成功验证。
表3为验证成功的基因,可发现大多数基因是由多个路径共同作用,并且根据GO_ID从GO库查找对应的功能发现,肌少症的枢纽基因主要通过调节因子活性转录、肽基赖氨酸修饰、组蛋白修饰等生物学过程影响肌少症。
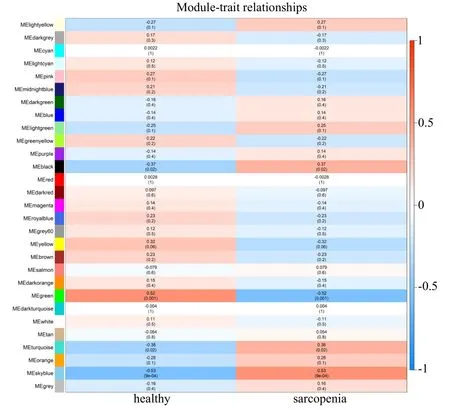
Fig.3 Correlation analysis between key modules and sarcopenia图3 关键模块与肌少症的关联分析

Table2 Hub genes in key modules and their verification information表2 关键模块中的枢纽基因及其验证信息
3.2 实验对比
为验证算法的有效性,本文选用目前基因筛选常用的差异基因分析法进行对比。实验基于R软件的limma包,通过差异基因分析法对实验数据筛选肌少症基因,并利用GO对其进行验证。通过实验分析表明,差异基因分析法可从16 879个基因中识别出5个肌少症基因(SCTR-AS1、ANKRD18B、ITLN1、RPS16P1、OVOS2),并成功验证了ITLN1(GO:0070206,p
=0.002)、OVOS2(GO:0010951,p
=0.03),准确率为40%。WGCNA识别出了86个肌少症相关基因,准确率达到了80.2%,相较于差异基因分析法,该方法更高效,准确率约提高了40%。
Table3 Results of genefun ction validation analysis表3 基因功能验证分析结果
4 结论
由于肌少症是多基因参与的复杂疾病,对发病机制的探究造成了很大的困难,利用现代计算机技术发掘其致病基因是一种行之有效的方法。WGCNA分析算法是目前基因共表达网络研究中最为常用的生物信息学分析方法,本文首次将该算法应用于分析肌少症,成功筛选出86个与肌少症相关的枢纽基因,并验证了69个具有真实生物信息基因,相较于传统方法,该方法考虑信息更全面,准确率约提高了40%。
验证成功的基因为探索肌少症的发病机制提供了新的思路,也为临床诊断、治疗靶点提供了参考。然而,实验所用样本数较少,后期将会寻找更合适的数据以进一步提高识别准确率。
