瓜螺线粒体全基因组密码子偏好性分析
2022-04-21何积翠喻达辉白丽蓉
何积翠 喻达辉 白丽蓉


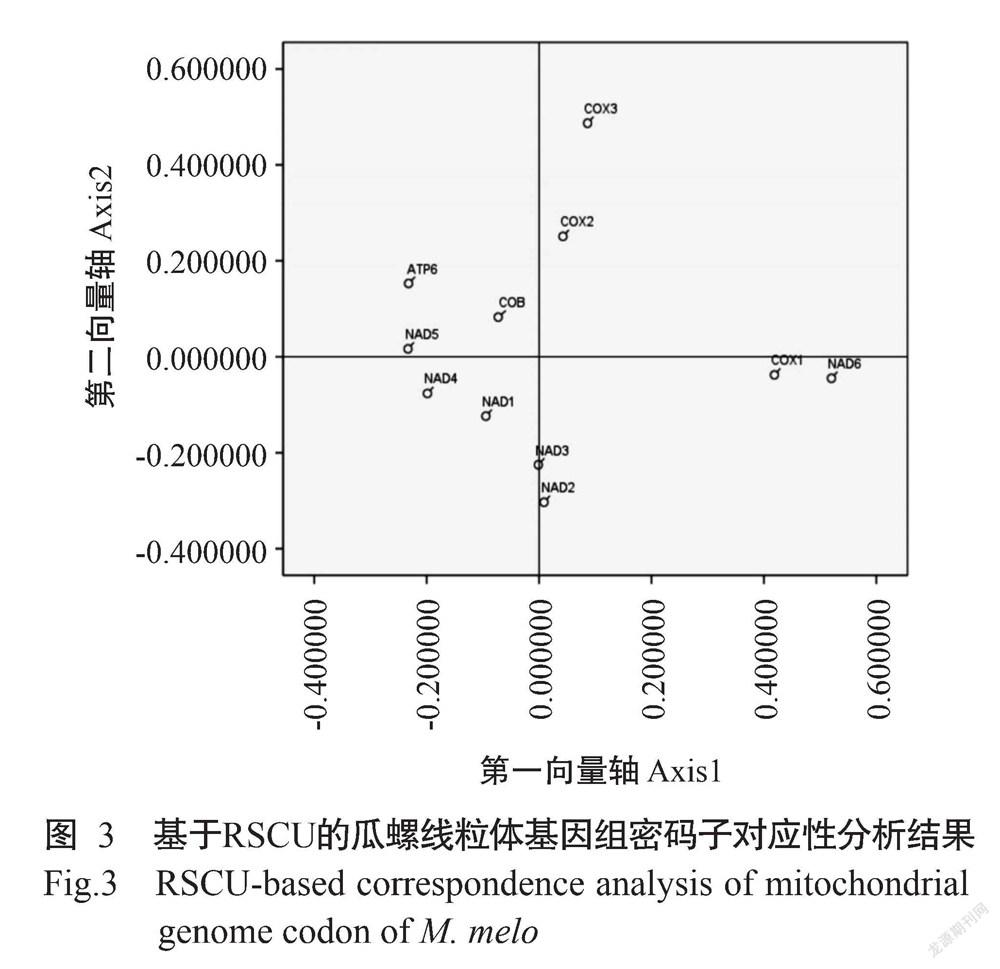
摘要:【目的】探究瓜螺(Melo melo)線粒体全基因组密码子使用偏好性,明确影响密码子偏好性的因素,为提高基因表达效率及了解瓜螺线粒体基因组的特性和进化方向提供科学依据。【方法】以瓜螺线粒体全基因组序列为研究对象,从中筛选出11条长度大于300 bp且以ATG为起始密码子的非重复基因序列,利用CodonW 1.4.2、Excel 2017及SPSS 22.0等软件进行瓜螺线粒体全基因组密码子偏好性分析。【结果】不同基因序列密码子位置所对应的G+C含量不同,其中,GC1(密码子第1位所代表的G+C含量)、GC2(密码子第2位所代表的G+C含量)、GC3(密码子第3位所代表的G+C含量)的变化范围分别为35.20%~51.90%、16.40%~40.60%和13.10%~34.90%,对应平均值为41.15%、32.29%和23.71%,GC1和GC2明显高于GC3;密码子适应指数(CAI)在0.110~0.180,平均为0.137;密码子偏好性指数(CBI)在-0.272~-0.090,平均为-0.175;最优密码子使用频率(FOP)在0.216~0.339,平均为0.277;有效密码子数(ENC)在36.84~53.75,平均为45.59;总平均亲水性(GRAVY)在0.480~1.394,平均为0.968。有30个同义密码子的相对使用度(RSCU)大于1.00,且主要以A/U结尾。中性绘图分析发现,GC1和GC2平均值(GC12)与GC3的相关系数为-0.438,相关性不显著(P>0.05,下同);ENC-plot绘图分析结果显示,各散点(基因)均位于标准曲线附近,其中,NAD4、COX2和NAD3基因位于标准曲线上方,COX1、ATP6、NAD1、NAD6、COB、NAD5、COX3及NAD2等8个基因位于标准曲线下方;在对应性分析中,第一、第二、第三、第四向量轴的贡献率分别23.12%、15.19%、13.17%和9.63%,前4轴累计差异为61.12%。综合高频密码子与高表达密码子,最终筛选出4个最优密码子(UUU、GUU、ACU和CAU),且均以U结尾。【结论】瓜螺线粒体全基因组密码子偏好以A/U结尾的形式,筛选出的4个最优密码子(UUU、GUU、ACU和CAU)均以U结尾。除了自然选择在瓜螺线粒体全基因组密码子偏好性形成中占主导地位外,突变压力和碱基组成也影响其密码子偏好。因此,可通过瓜螺目的基因密码子优化,有效提高外源基因表达且优化性状决定基因,从而加速瓜螺优良亲本培育的进程。
关键词: 瓜螺;线粒体;基因组;密码子偏好性;自然选择
中图分类号: S917 文献标志码: A 文章编号:2095-1191(2022)01-0191-09
Codon usage bias analysis in mitochondrial genome of Melo melo
HE Ji-cui, YU Da-hui, BAI Li-rong*
(Beibu Gulf University/Guangxi Key Laboratory of Beibu Gulf Marine Biodiversity Conservation,
Qinzhou, Guangxi 535011, China)
Abstract:【Objective】The use preference of mitochondrial whole genome codon of Melo melo was investigated, and the factors affecting codon preference were analyzed, to improve gene expression efficiency,study the characteristics, ecological adaptation and evolutionary direction of mitochondrial genome of M. melo. 【Method】Based on the mitochondrial genome sequence of M. melo, eleven non-repetitive sequences with length more than 300 bp and ATG as the starting codon were selected as the research objects, and the codon bias was analyzed by using the softwares of CodonW 1.4.2, Excel 2017, SPSS 22.0. 【Result】The G+C content corresponding to codon positions of different gene sequence varies, including GC1(G+C content represented by codon position 1), GC2(G+C content represented by codon position 2), GC3(G+C content represented by codon position 3), was 35.20%-51.90%, 16.40%-40.60%, 13.10%-34.90%, corresponding average of 41.15%, 32.29%, and 23.71%, respectively. The content of GC1 and GC2 was higher than that of GC3. The codon adaptation index (CAI )value ranged from 0.110 to 0.180, with an average of 0.137. The codonbiasindex (CBI) was at -0.272 to -0.090, with an average of -0.175. The optimal codon usage frequency (FOP) was from 0.216-0.339, with an average of 0.277. The effective codon number (ENC) ranged from 36.84 to 53.75, with an average of 45.59; grand average of hydropathy (GRAVY) ranged from 0.480 to 1.394, with an average of 0.968. There were 30 codons with relative synonymous codon usage (RSCU)>1.00, mainly ending with A/U. Neutral mapping analysis showed that the correlation coefficient between the mean of GC1 and GC2(GC12) and GC3 was -0.438, the correlation was not significant(P>0.05, the same below). ENC-plot analysis showed that all genes were located near the standard curve, among them, NAD4, COX2 and NAD3 genes were located above the standard curve, and eight genes including COX1, ATP6, NAD1, NAD6, COB, NAD5, COX3 and NAD2 were located below the standard curve. Correspondence analysis showed that the first, second, third and fourth axis contributed 23.12%, 15.19%, 13.17% and 9.63% of the difference, respectively. The total contribution rate of the first four vectors was 61.12%. Combining high frequency codons and high expression codons, four optimal codons (UUU, GUU, ACU and CAU) were selected, all ending with U. 【Conclusion】The whole mitochondrial genome codon of M. melo mainly prefers to use the codon ending in A/U, and the four selected optimal codons (UUU, GUU, ACU, and CAU) end with U. In addition to dominating natural selection in genome-wide codon preference formation in M. melo mitochondria, mutational pressure and base composition also influence their codon preference. Codon optimization of the target gene can effectively improve the foreign gene expression and optimize the trait determination gene, so as to accelerate the process of excellent parental cultivation.
Key words: Melo melo; mitochondria; genome; codon bias; natural selection
Foundation items: National Key Research and Development Program of China(2018YFD0901406); Guangxi Key Research and Development Project(2018AB52002); Guangxi Natural Science Foundation (2021GXNSFAA075008)
0 引言
【研究意义】自然界中DNA序列以三联密码子法则翻译成氨基酸,经组装修饰后形成具有特定空间结构和功能的蛋白(Zhang et al.,2011)。遗传信息主要是按照“中心法则”进行传递,即从DNA转录为mRNA,最后翻译成氨基酸的过程(段晓克,2015),而准确识别编码不同氨基酸密码子是保证遗传信息正确表达的关键(杨国锋等,2015)。在生物体内的蛋白翻译过程中,编码同一种氨基酸的不同密码子称之为同义密码子(Synonymous codon)(吴宪明等,2007)。在密码子使用过程中,会出现某些密码子使用频率高于其他同义密码子使用频率的现象,称为密码子使用偏好性(Codon usage bias,CUB)(武志娟和钟金城,2012)。密码子偏好性是导致外源基因在宿主中表达量降低的原因之一,且受自然选择和突变压力等因素的共同影响。因此,研究密码子偏好性对揭示物种进化关系及提高外源基因的表达具有重要作用,同时对进一步了解生物基因或基因组特征和生态适应等具有重要意义。【前人研究进展】至今,线粒体基因组密码子使用模式已在小麦(Triticum aestivum)、拟南芥(Arabidopsis thaliana)和烟草(Nicotiana tabacum)等农作物(Zhou and Li,2009),以及壮体长春鳊(Parabramis pekinensis strenosom)、团头鲂(Megalobrama amblycephala)(段晓克,2015)、真白鲑(Coregonus lavaretus)(朱秀芳等,2015)、小鲵科(姜艳,2016)、斑石鲷(Oplegnathus punctatus)和条石鲷(O. fasciatus)(孟乾等,2020)等水生生物上得到广泛研究及应用,有效解释了影响其密码子偏性形成的原因,也为了解系统发育、分子进化过程及遗传育种等提供了理论依据(徐苏丽等,2013;蒋玮等,2014;Barbhuiya et al.,2020)。此外,张志东等(2019)利用生物信息学方法对文蛤属(Meretrix)贝类进行密码子偏好性分析,明确其密码子偏好性及特征,同时比较文蛤与近缘种密码子使用偏好性的差异;孟乾等(2020)利用中性绘图分析及聚类分析等生物信息学方法,从mtDNA水平上分析斑石鲷和条石鲷编码蛋白序列的密码子使用偏好性,并与其他鱼类的密码子偏好性进行对比;慎佩晶等(2020)通过对罗氏沼虾(Macrobrachium rosenbergii)相关的同義密码子进行密码子偏好性分析,发现其偏向于以T或A结尾的密码子,与黑腹果蝇(Drosophila melanogaster)、中华绒螯蟹(Eriocheir sinensis)及酵母(Saccharomyces cervisiae)等相比,罗氏沼虾具有明显不同的密码子偏好性。【本研究切入点】瓜螺(Melo melo)又称油螺或红螺,隶属于软体动物门(Mollusca)腹足纲(Gastropoda)新腹足目(New Gastropoda)涡螺科(Oncomelania)(郑小东和曲学存,2013;Zhong et al.,2019)。瓜螺腹足部肉质肥厚、营养丰富、味道鲜美,且具有清火明目之功效,但近年来因采捕野生数量不断减少,导致其种质资源日渐枯竭。培育具有优良性状的亲本繁殖后代对保护瓜螺种质资源具有重要意义,而明确密码子偏好性能有效提高外源基因的表达并优化性状决定基因,进而加速优良亲本的培育进程。【拟解决的关键问题】以瓜螺线粒体全基因组序列为研究对象,利用CodonW 1.4.2、Excel 2020及SPSS 20.0等软件探究瓜螺线粒体全基因组密码子的使用偏好性,明确影响密码子偏好性的因素,为提高基因表达效率及了解瓜螺线粒体基因组的特性和进化方向提供科学依据。
1 材料与方法
1. 1 序列来源与分析
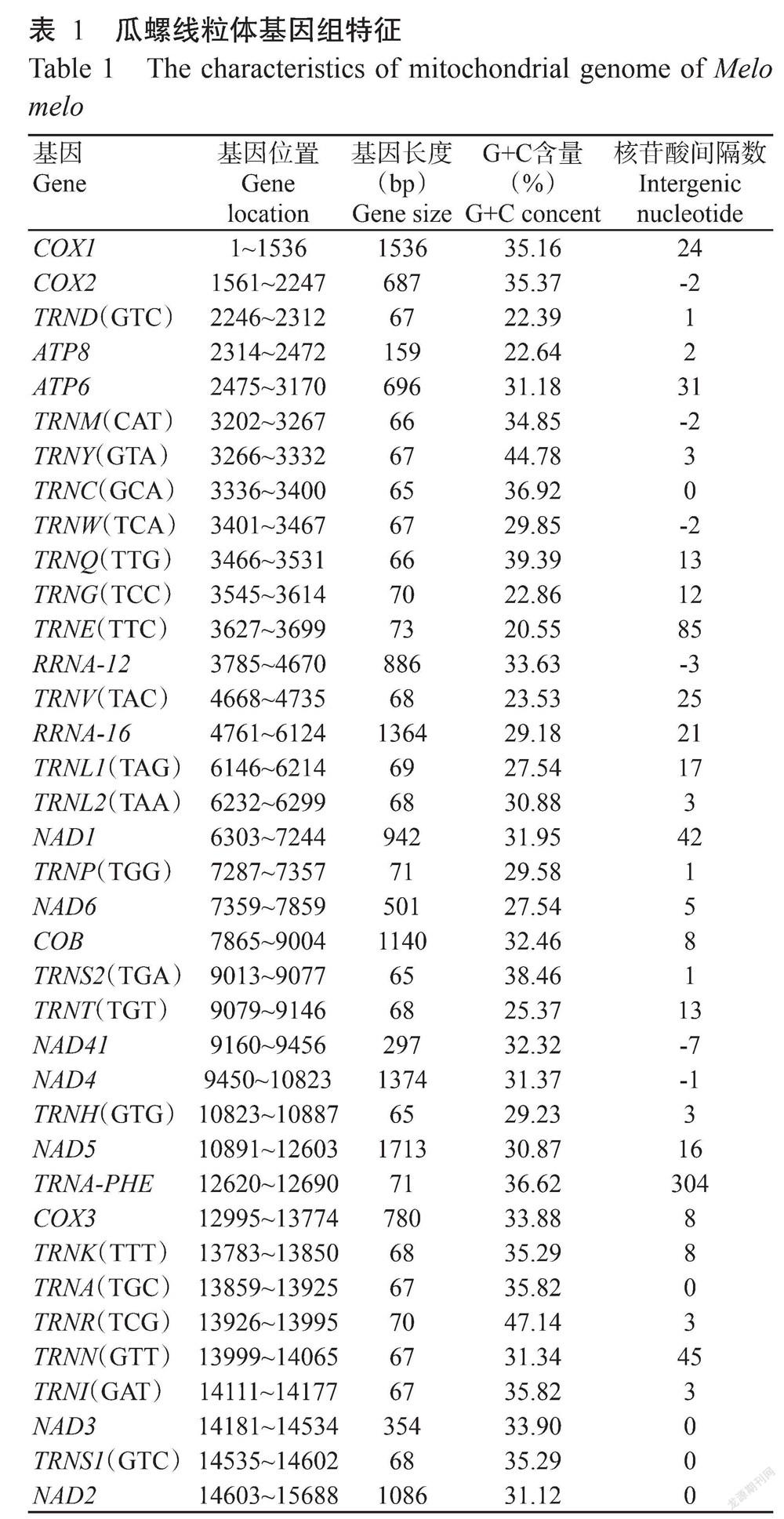
从NCBI细胞器基因组数据库中搜索瓜螺拉丁名:Melo melo,检索并下载瓜螺线粒体全基因组序列及其编码区(Coding DNA sequence,CDS),瓜螺线粒体全基因组序列全长15721 bp,包含37个基因。为确保试验数据的准确性,本研究选取11条长度大于300 bp且以ATG为起始密码子的非重复基因序列进行密码子偏好性分析(柳燕杰等,2020;王宇等,2020),并利用CodonW 1.4.2、Excel 2017及SPSS 22.0等软件进行统计分析。
1. 2 密码子偏好性参数计算
以选取的CDS序列为研究对象,采用CodonW 1.4.2分析密码子偏好性参数,包括密码子G+C总含量及GC1(密码子第1位所代表的G+C含量)、GC2(密码子第2位所代表的G+C含量)、GC3(密码子第3位所代表的G+C含量)、GC3S(同义密码子第3位的G+C含量)、密码子适应指数(Codon adaption index,CAI)、密码子偏好性指数(Codon bias index,CBI)、最优密码子使用频率(Frequency of optimal codon,FOP)、有效密码子数(Effective number of codon,ENC)、同义密码子相对使用度(Relative synonymous codon usage,RSCU)及蛋白疏水性均值(Grand average of hydropathy,GRAVY)(王鹏良等,2019;赵森等,2019)。其中,ENC的范围在20~61,ENC越小说明偏好性越强,通常以35为标准,当ENC小于35时偏好性较强,反之则越弱(王媛媛和杨美青,2021)。RSCU为某一特定密码子使用频率/其在无偏好性时的使用频率,以1.00为标准,当RSCU小于1.00时说明该密码子使用频率较低,RSCU等于1.00时说明该密码子无偏好性,而RSCU大于1.00时表明该密码子使用频率较高(吴宪明等,2007)。CAI的变化范围在0~1,CAI越小说明密码子偏好性越弱(吴宪明等,2007;周丹等,2013)。
1. 3 中性绘图分析
中性绘图是初步判断影响密码子使用偏好因素的方法之一(原晓龙等,2020b),是以GC3为横坐标、GC12为纵坐标,绘制散点图。GC12是GC1和GC2的平均值。中性绘图可根据密码子的前2位碱基和第3位碱基的关系,而判断密码子使用偏好是由突变压力还是自然选择所造成。如果散点均在对角线上,说明密码子偏好性受突变压力的影响,否则就是受自然选择的影响(梁晓静等,2020;原晓龙等,2020a)。
1. 4 ENC-plot绘图分析
以GC3S为横坐标、ENC为纵坐标,绘制散点图,且在坐标系中绘制ENC的标准曲线。如果散点均在標准曲线附近,说明密码子偏好性只受突变压力的影响;若散点均位于标准曲线下方,说明密码子偏好更多是受自然选择的影响(吴宪明等,2007;王鹏良等,2018)。标准曲线公式如下:
ENC=2+GC3S+[29GV3S2+(1-GC3S2)] (1)
为了更准确地体现ENC期望值(ENCexp)与ENC实际值(ENCobs)的差异,而统计ENC比值(ENCRatio)频数分布情况,具体公式如下:
ENCRatio=[ENCexp-ENCobsENCexp] (2)
当ENCexp与ENCobs接近时,表明基因密码子受突变压力的影响;而ENCexp与ENCobs差别较大时,说明基因密码子受自然选择的影响(赵洋等,2016;赵婉清等,2020)。
1. 5 对应性分析
参照王鹏良等(2018)的方法进行对应性分析,根据对应性分析得到影响瓜螺线粒体全基因密码子偏好性的因素。
1. 6 最优密码子确定
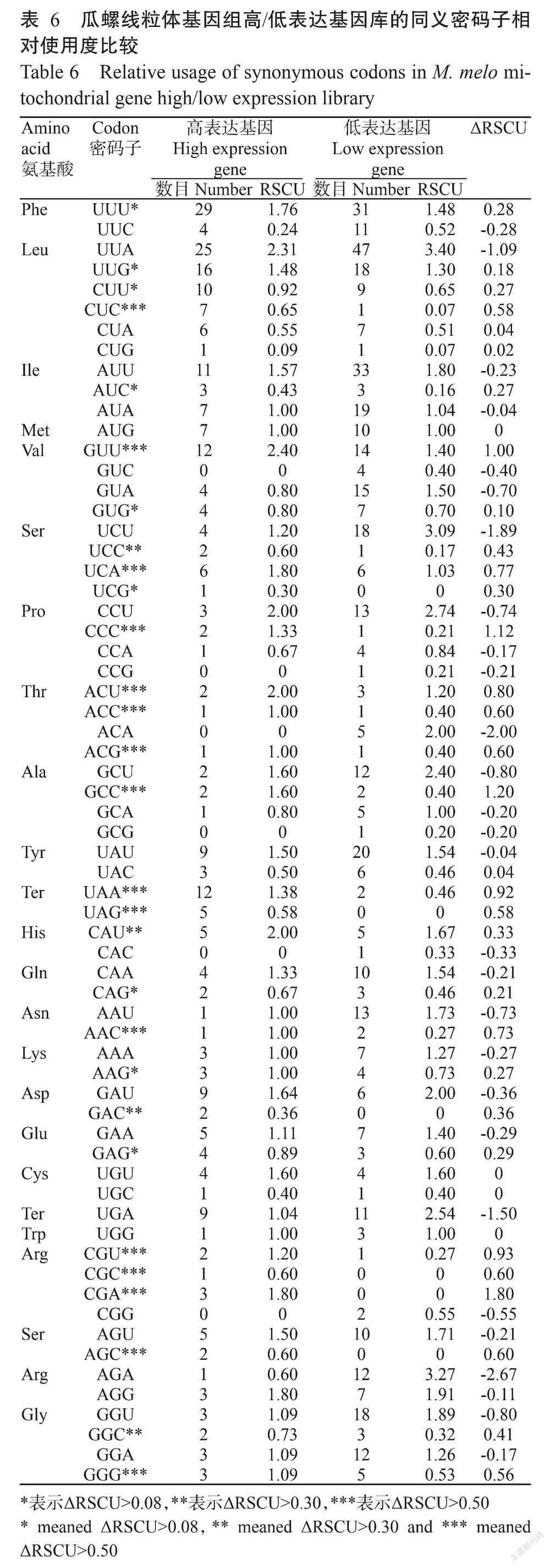
以ENC为标准,对所有参试基因按从大到小的顺序进行排列,分别从ENC的两端选取20%的参试基因(各选2个),建立高表达基因库和低表达基因库(姜艳等,2016)。
ΔRSCU=RSCUH-RSCUL
将高表达基因库(RSCUH)与低表达基因库(RSCUL)的同义密码子相对使用度差值(ΔRSCU)>0.08的判为高表达密码子,同义密码子相对使用度(RSCU)>1.00的密码子为高频密码子。既是高表达又是高频的密码子即为最优密码子(Optimal codon)(段晓克,2015;姜艳等,2016)。
2 结果与分析
2. 1 密码子组成分析结果
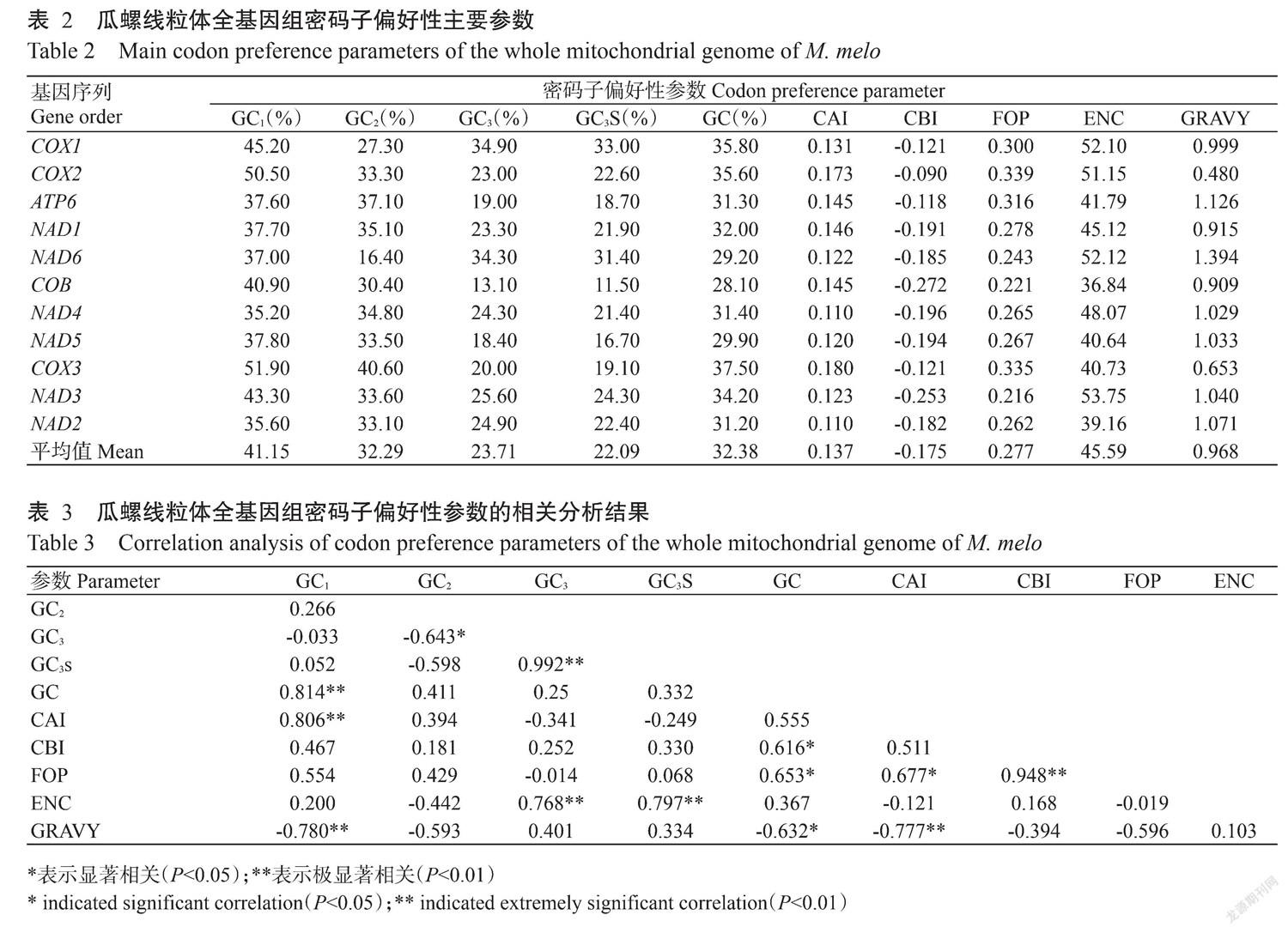
瓜螺线粒体基因组是一个总长为15721 bp的环状DNA分子,包含有37个基因,G+C含量为31.66%,A+T含量为68.34%,说明瓜螺表现出对A和T碱基的偏好性。瓜螺线粒体基因组特征如表1所示。根据密码子使用偏好分析要求,筛选得到11条非重复且长度大于300 bp的基因序列,采用CodonW 1.4.2对筛选获得的11条基因序列进行分析,结果(表2)显示,不同基因序列密码子位置所对应的G+C含量也不同,其中,GC1的范围在35.20%~51.90%,GC2的范围在16.40%~40.60%,GC3的范围在13.10%~34.90%,GC1、GC2、GC3的平均值分别为41.15%、32.29%和23.71%,说明瓜螺线粒体全基因组密码子偏好以碱基U或A结尾的密码子为主。CAI的范围在0.110~0.180,平均为0.137,说明瓜螺密码子偏好性较弱;CBI的范围在-0.272~-0.090,平均为-0.175;FOP的范围在0.216~0.339,平均为0.277;ENC的范围在36.84~53.75,平均为45.59,且ENC均大于35,也说明瓜螺密码子偏好性较弱;GRAVY的范围在0.480~1.394,平均为0.968。
密码子偏好性参数相关分析结果(表3)表明:GC1与GC2和GC3的相关性不显著(P>0.05,下同);GC1与GC和CAI呈极显著正相关(P<0.01,下同),与GRAVY呈极显著负相关,其相关系数分别为0.814、0.806和-0.780;GC2与GC3呈显著负相关(P<0.05,下同),其相关系数为-0.643,与其他参数的相关性均未达显著水平;GC3与GC3S和ENC呈极显著负相关,其相关系数分别为0.992和0.768;GC3S与ENC呈极显著正相关,其相关系数为0.797。可见,GC3和GC3S对瓜螺线粒体全基因组密码子偏好性有较大的影响作用,但二者与GC、CAI、CBI、FOP和GRAVY的相关性均不显著。GC与CBI和FOP呈显著正相关,其相关系数分别为0.616和0.653;CAI与FOP呈显著相关,与GRAVY呈极显著负正相关,相关系数分别为0.677和-0.777;CBI与FOP呈极显著正相关,其相关系数为0.948。
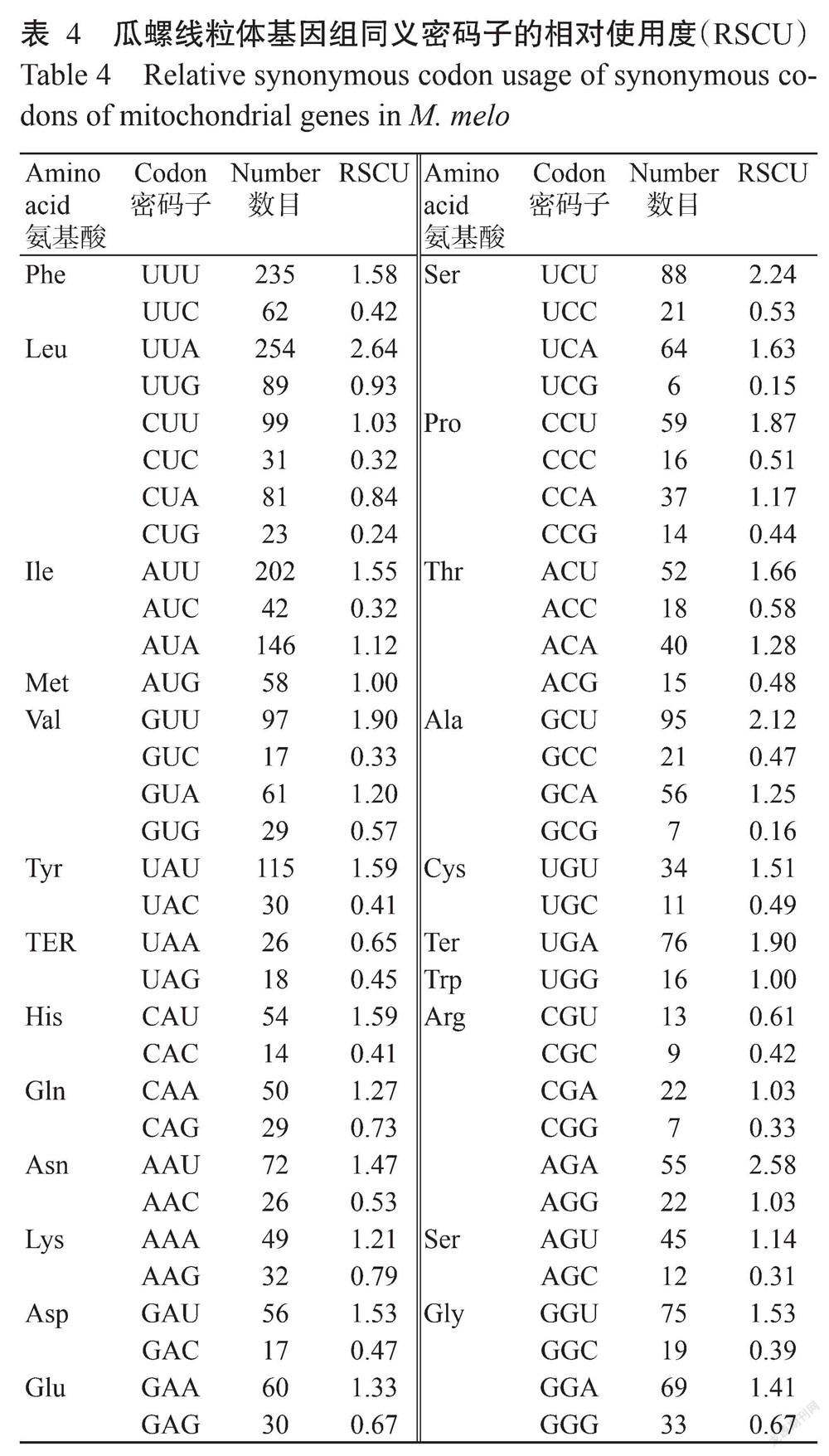
RSCU分析结果(表4)显示,有30个同义密码子的RSCU大于1.00。在这30个同义密码子中,以碱基U结尾的同义密码子有15个,以碱基A结尾的有14个,以碱基G结尾的仅有1个,即以碱基U和A结尾的同义密码子数目占总同义密码子数目的96.67%,说明瓜螺线粒体全基因组密码子偏好以碱基U或A结尾。
2. 2 中性绘图分析结果
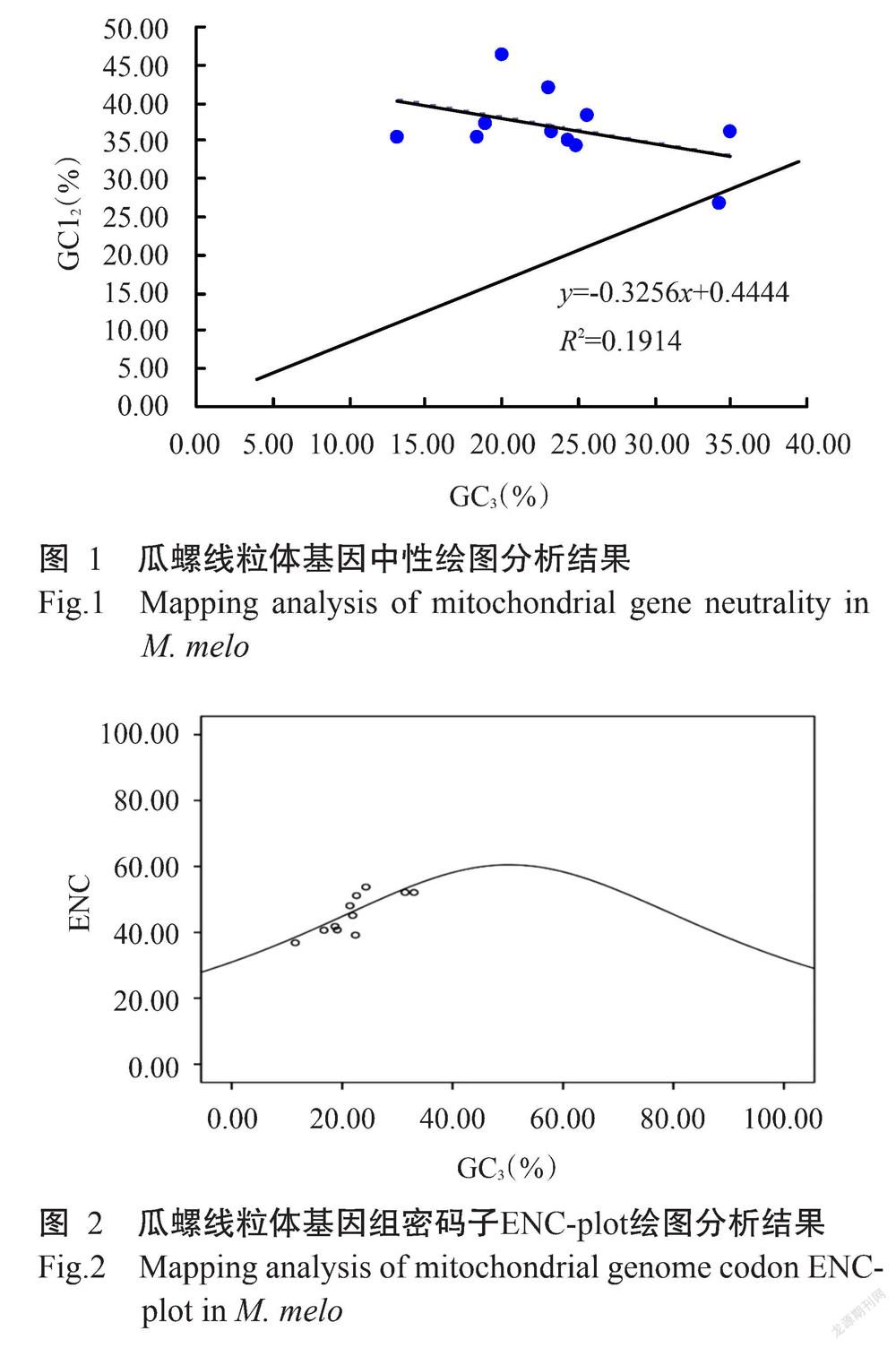
瓜螺线粒体基因中性绘图分析结果(图1)表明,GC3的范围在13.10%~34.90%,GC12的范围在26.70%~46.25%。GC12与GC3的相关系数为-0.438,相关性不显著;回归方程为y=-0.3256x+0.4444(R2=0.1914),说明GC12与GC3的相关性不强。在图1中,几乎所有的散点(基因)都在对角线上方,仅有1个基因在对角线下方,且GC12均高于GC3,说明瓜螺线粒体基因组密码子偏好虽然受突变压力的影响,但受自然选择的影响作用更大。
2. 3 ENC-plot绘图分析结果
采用SPSS 22.0进行ENC-plot绘图分析,以GC3S为横坐标、ENC为纵坐标建立直角坐标系图,将各参数导入该坐标系,并根据公式(1)添加标准曲线。瓜螺线粒体基因组密码子ENC-plot绘图分析结果(图2)表明,各散点(基因)均位于标准曲线附近,其中,NAD4、COX2和NAD3基因位于标准曲线上方,COX1、ATP6、NAD1、NAD6、COB、NAD5、COX3及NAD2等8个基因位于标准曲线下方。为了更准确地体现ENCexp与ENCobs的差异,通过公式(2)计算ENCRatio,并统计其频数分布情况。由表5可知,有63.64%基因的ENCRatio范圍在0~0.1,18.18%基因的ENCRatio范围在-0.1~0,ENCRatio范围在-0.2~-0.1和0.1~0.2的基因各占9.09%,说明突变压力和自然选择均对瓜螺线粒体全基因组密码子偏好性的有影响,但以自然选择占主导地位。
2. 4 对应性分析结果
对应性分析结果表明:第一、第二、第三和第四向量轴的贡献率分别为23.12%、15.19%、13.17%和9.63%,前4轴的累计差异为61.12%。第一和第二向量轴的贡献率均大于15.00%,说明第一、第二向量轴是瓜螺线粒体全基因组密码子偏好性的主要影响因素。第一向量轴与GC3和GC3S呈极显著正相关,其相关系数分别为0.803和0.804,与GC2呈极显著负相关,相关系数为-0.750,说明碱基差异对瓜螺线粒体全基因密码子偏好性有一定影响;第一向量轴与GC1、GC、CAI、CBI、FOP、ENC的相关系数分别为0.272、0.231、0.013、0.208、-0.007和0.539,相关性均不显著。为更好地观察瓜螺线粒体全基因密码子偏好性,以第一向量轴为横坐标、第二向量轴为纵坐标绘制对应性分析图,结果(图3)发现ATP6、COB、NAD5、NAD4、NAD1、COX2、NAD3及NAD2等8个基因相对较集中,说明这8个基因密码子使用偏好较接近,而另外3个基因相对较分散,可能是密码子使用偏好存在明显差异。
2. 5 最优密码子分析结果
高、低表达基因库的ΔRSCU如表6所示。按ΔRSCU>0.08的标准,共确定获得29个瓜螺线粒体全基因组高表达密码子,其中,0.30>ΔRSCU>0.08的有9个密码子(表6中以*标注),0.50>ΔRSCU>0.30的有4个密码子(表6中以**标注),ΔRSCU>0.50的有16个密码子(表6中以***标注)。在这些高表达密码子中,以C结尾的密码子有11个,以G结尾的有9个,以U结尾的有6个,以A结尾的有3个。将表4中的30个高频密码子与表6中的29个高表达密码子进行综合分析,确定既是高表达密码子又是高频密码子的密码子为最优密码子,最终确定出4个最优密码子,分别为UUU、GUU、ACU和CAU,对应编码苯丙氨酸(Phe)、缬氨酸(Val)、组氨酸(His)和苏氨酸(Thr)4个氨基酸残基,且这4个最优密码子均以U结尾,说明最优密码子偏好以U结尾的形式。
3 讨论
同义密码子的主要区别在其第3位碱基上。本研究的密码子偏好性参数相关分析结果表明,GC1与GC2和GC3相关不显著,GC2与GC3呈极显著负相关,且GC1和GC2明显高于GC3,说明瓜螺线粒体全基因组偏好使用以A/U结尾的密码子,RSCU分析结果(RSCU>1.00的密码子有30个,以A/U结尾的有29个)也支持这一观点,与张志东等(2019)分析5种文蛤属贝类线粒体基因密码子使用偏好性、孟乾等(2020)分析斑石鲷和条石鲷线粒体基因组密码子使用偏好性、慎佩晶等(2020)分析罗氏沼虾转录组线粒体密码子使用偏好性得出的结论相似;不同的是斑石鲷和条石鲷线粒体基因组密码子偏好以A/C结尾的形式(孟乾等,2020)、罗氏沼虾转录组线粒体密码子偏好以A/T/U结尾的形式(慎佩晶等,2020),李喜莲等(2014)分析螯虾次目功能基因密码子偏好时则发现其偏好于C/G结尾的密码子,说明不同生物的线粒体基因密码子使用规律具有相似性,但也存在一定差异。瓜螺线粒体基因组ENC平均值为45.59,参照王媛媛和杨美青等(2021)的研究结论(ENC>35表明密码子偏好性较弱,ENC<35则密码子偏好性较强)可判定,瓜螺线粒体基因组密码子为弱偏好性,其CAI参数数据也支持这一观点。
影响密码子偏好性的因素诸多,包括突变压力、自然选择、碱基组成及tRNA丰度等(吴宪明等,2007;梁菲菲,2010;李若愚等,2021)。一般认为影响密码子偏好的两大主要因素是突变压力和自然选择(Hershberg and Petrov,2008;吴炜倩等,2020)。本研究结果表明,GC3和GC3S与ENC均呈极显著相关,在对应性分析中第一向量轴与GC3和GC3S呈极显著正相关,其相关系数分别为0.803和0.804,说明碱基组成也是影响瓜螺线粒体全基因组密码子偏好性的因素之一;GRAVY与CBI和FOP的相关性不显著,说明瓜螺线粒体全基因组密码子偏好性对蛋白的亲/疏水性几乎没有影响。此外,由中性绘图分析和ENC-plot绘图分析可知,瓜螺线粒体全基因组密码子偏好性虽然受突变压力的作用,但更多的是受自然选择所影响。
4 结论
瓜螺线粒体全基因组密码子偏好以A/U结尾的形式,筛选出的4个最优密码子(UUU、GUU、ACU和CAU)均以U结尾。除了自然选择在瓜螺线粒体全基因组密码子偏好性形成中占主导地位外,突变压力和碱基组成也影响瓜螺线粒体基因组密码子偏好。因此,可通过瓜螺目的基因密码子优化,有效提高外源基因表达且优化性状决定基因,从而加速瓜螺优良亲本培育的进程。
参考文献:
段晓克. 2015. 壮体长春鳊线粒体基因组全长测定及团头鲂密码子偏好模型分析[D]. 武汉:华中农业大学. [Duan X K. 2015. Studies on complete mitochondrial genome of Parabramis pekinensis strenosoma and codon usage patterns of Megalobrma amblycephala[D]. Wuhan:Huazhong Agricultural University.] doi:10.7666/d.Y2803757.
姜艳,张全星,管德龙,许升全. 2016. 小鲵科动物Cytb基因密码子偏好性分析[J]. 陕西师范大学学报(自然科学版),44(4):77-82. [Jiang Y,Zhang Q X,Guan D L,Xu S Q. 2016. Codon bias of Cytb genes in hynobiidae[J]. Journal of Shaanxi Normal University(Natural Science Edition),44(4):77-82.] doi:10.15983/j.cnki.jsnu.2016.04.345.
姜艳. 2016. 小鲵科线粒体基因组密码子偏好性及进化关系的分析[D]. 西安:陕西师范大学. [Jiang Y. 2016. Analysis of codon bias and evolutionary relationship in mitochondrial genome of Hynobidae[D]. Xi’an:Shaanxi Normal University.]
蒋玮,吕贝贝,何建华,王金斌,吴潇,武国干,鮑大鹏,陈明杰,张劲松,谭琦,唐雪明. 2014. 草菇密码子偏好性分析[J]. 生物工程学报,30(9):1424-1435. [Jiang W,Lü B B,He J H,Wang J B,Wu X,Wu G G,Bao D P,Chen M J,Zhang J S,Tan Q,Tang X M. 2014. Codon usage bias in the straw mushroom Volvariella volvacea[J]. Chinese Journal of Biotechnology,30(9):1424-1435.] doi:10. 13345/j.cjb.130623.
李若愚,张小丹,马昕怡,郭瑞,颜少宾,金光,周平. 2021. 桃基因密码子使用模式及其偏好性分析[J]. 分子植物育种,19(3):799-807. [Li R Y,Zhang X D,Ma X Y,Guo R,Yan S B,Jin G,Zhou P. 2021. Analysis of codon usage patterns and codon usage bias in peach (Prunus persica)[J] Molecular Plant Breeding,19(3):799-807.] doi:10. 13271/j.mpb.019.000799.
李喜莲,杨元杰,李倩,刘金殿,顾志敏. 2014. 螯虾次目功能基因密码子偏好性研究[J]. 浙江农业学报,26(4):862-867. [Li X L,Yang Y J,Li Q,Liu J D,Gu Z M. 2014. Analysis on codon bias of functional gene of Astacidea[J]. Acta Agriculturae Zhejiangensis,26(4):862-867.] doi:10.3969/j.issn.1004-1524.2014.04.04.
梁菲菲. 2010. 密码子偏性的影响因素及研究意义[J]. 畜牧与饲料科学,31(1):118-119. [Liang F F. 2010. Influen-cing factors of codon bias and its research significance[J]. Animal Husbandry and Feed Science,31(1):118-119.] doi:10.16003/j.cnki.issn1672-5190.2010.01.077.
梁晓静,朱昌叁,李开祥,安家成,王鹏良. 2020. 香樟转录组基因密码子偏好性分析[J]. 广西植物,41(12):2077-2083. [Liang X J,Zhu C S,Li K X,An J C,Wang P L. 2020. Genes codon bias of transcriptome in Cinnamomum camphora[J]. Guihaia,41(12):2077-2083.] doi:10. 11931/guihaia.gxzw202003074.
柳燕杰,田旭平,李倩. 2020. 美国红梣叶绿体基因组密码子偏好性分析[J]. 江苏农业科学,48(15):83-88. [Liu Y J,Tian X P,Li Q. 2020. Analysis of codon bias in chloroplast genome of Fraxinus pennsylvanica[J]. Jiangsu Agricultural Science,48(15):83-88.] doi:10.15889/j.issn.1002- 1302.2020.15.014.
孟乾,张志勇,张志伟,张曹进,陈淑吟,祝斐,贾超峰,曹广勇. 2020. 斑石鲷和条石鲷线粒体基因组密码子使用分析[J]. 水产科学,39(5):702-709. [Meng Q,Zhang Z Y,Zhang Z W,Zhang C J,Chen S Y,Zhu F,Jia C F,Cao G Y. 2020. Codon bias and cluster analysis on coding regions of mtDNA in rock porgy Oplegnathus punctatus and rock bream O. fasciatus[J]. Fisheries Science,39(5):702-709.] doi:10.16378/j.cnki.1003-1111.2020.05.008.
慎佩晶,张宇飞,李喜莲,高强,徐洋,黄振远,蒋文枰,陈雪峰. 2020. 罗氏沼虾转录组密码子使用偏好性分析[J]. 现代农业科技,(16):175-178. [Shen P J,Zhang Y F,Li X L,Gao Q,Xu Y,Huang Z Y,Jiang W P,Chen X F. 2020. Analysis of codon usage bias based on Macrobrachium rosenbergii transcriptome[J]. Modern Agricultural Science and Technology,(16):175-178.] doi:10.3969/j.issn.1007-5739.2020.16.103.
王鹏良,吴双成,杨利平,王华宇,陈乃明,张照远. 2019. 巨桉叶绿体基因组密码子偏好性分析[J]. 广西植物,39(12):1583-1592. [Wang P L,Wu S C,Yang L P,Wang H Y,Chen N M,Zhang Z Y. 2019. Analysis of codon bias of chloroplast genome in Eucalyptus grandis[J]. Guihaia,39(12):1583-1592.] doi:10.11931/guihaia.gxzw201810027.
王鹏良,杨利平,吴红英,农有良,吴双成,肖玉菲,覃子海,王华宇,刘海龙. 2018. 普通油茶叶绿体基因组密码子偏好性分析[J]. 广西植物,38(2):135-144. [Wang P L,Yang L P,Wu H Y,Nong Y L,Wu S C,Xiao Y F,Qin Z H,Wang H Y,Liu H L. 2018. Condon preference of chloroplast genome in Camellia oleifera[J]. Guihaia,38(2):135-144.] doi:10.11931/guihaia.gxzw201708001.
王宇,周俊良,唐冬梅,仲偉敏,马玉华,张敏. 2020. 阔叶猕猴桃叶绿体基因组特征及密码子偏好性分析[J]. 种子,39(5):13-19. [Wang Y,Zhou J L,Tang D M,Zhong W M,Ma Y H,Zhang M. 2020. Analysis of chloroplast genome characteristics and codon preference in broad-leaf kiwifruit[J]. Seed,39(5):13-19.] doi:10.16590/j.cnki. 1001-4705.2020.05.013.
王媛媛,杨美青. 2021. 蒙古韭叶绿体基因组密码子使用偏好性分析[J]. 分子植物育种,19(4):1084-1092. [Wang Y Y,Yang M Q. 2021. Analysis of the codon usage bias in the chloroplast genome of Allium mongolicum Regel[J]. Molecular Plant Breeding,19(4):1084-1092.] doi:10.13271/ j.mpb.019.001084.
吴炜倩,胡源,胡接力,黄爱龙,涂增. 2020. 乙型肝炎病毒密码子偏好性及影响因素分析[J]. 中国病原生物学杂志,15(12):1404-1410. [Wu W Q,Hu Y,Hu J L,Huang A L,Tu Z. 2020. Codon usage bias of the hepatitis B virus and factors influencing it[J]. Journal of Parasitic Biology,15(12):1404-1410.] doi:10.13350/j.cjpb.201208.
吴宪明,吴松锋,任大明,朱云平,贺福初. 2007. 密码子偏性的分析方法及相关研究进展[J]. 遗传,29(4):420-426. [Wu X M,Wu S F,Ren D M,Zhu Y P,He F C. 2007. The analysis method and progress in the study of codon bias[J]. Hereditas(Beijing),29(4):420-426.] doi:10. 3321/j.issn:0253-9772.2007.04.006.
武志娟,钟金城. 2012. 密码子偏性及其应用[J]. 生物学通报,47(4):9-11. [Wu Z J,Zhong J C. 2012. Codon usage bias and its application[J]. Bulletin of Biology,47(4):9-11.] doi:10.3969/j.issn.0006-3193.2012.04.004.
徐苏丽,王俊峰,陈士华. 2013. 稻瘟病病菌线粒体基因密码子使用偏好性分析[J]. 河南农业大学学报,47(6):722-726. [Xu S L,Wang J F,Chen S H. 2013. Analysis on codon usage preference of mitochondria genome of Magaporthe orisea[J]. Journal of Henan Agricultural University,47(6):722-726.] doi:10.16445/j.cnki.1000-2340.2013. 06.011.
楊国锋,苏昆龙,赵怡然,宋智斌,孙娟. 2015. 蒺藜苜蓿叶绿体密码子偏好性分析[J]. 草业学报,24(12):171-179. [Yang G F,Su K L,Zhao Y R,Song Z B,Sun J. 2015. Analysis of codon usage in the chloroplast genome of Medicago truncatula[J]. Acta Prataculturae Sinica,24(12):171-179.] doi:10.11686/cyxb2015016.
原晓龙,李云琴,王毅,张劲峰. 2020a. 西南桦叶绿体基因组密码子偏好性分析[J]. 基因组学与应用生物学,39(12):5758-5764. [Yuan X L,Li Y Q,Wang Y,Zhang J F. 2020a. Analysis of codon usage in the chloroplast genome of Betula alnoides[J]. Genomics and Applied Bio-logy,39(12):5758-5764.] doi:10.13417/j.gab.039.005758.
原晓龙,李云琴,张劲峰,王毅. 2020b. 乳油木叶绿体基因组密码子偏好性分析[J]. 分子植物育种,18(17):5658-5664. [Yuan X L,Li Y Q,Zhang J F,Wang Y. 2020b. Codon usage bias analysis of chloroplast genome in Vitellaria paradoxa[J]. Molecular Plant Breeding,18(17):5658-5664.] doi:10.13271/j.mpb.018.005658.
张志东,陈爱华,吴杨平,张雨,陈素华,曹奕,曹广勇,田镇. 2019. 5种文蛤属贝类线粒体基因密码子偏好性分析[J]. 海洋渔业,41(5):589-595. [Zhang Z D,Chen A H,Wu Y P,Zhang Y,Chen S H,Cao Y,Cao G Y,Tian Z. 2019. Analysis of codon usage bias in five species of Meretrix[J]. Marine Fisheries,41(5):589-595.] doi:10.13233/j.cnki.mar.fish. 2019.05.006.
赵森,邓力华,陈芬. 2020. 秋茄叶绿体基因组密码子使用偏好性分析[J]. 森林与环境学报,40(5):534-541. [Zhao S,Deng L H,Chen F. 2020. Codon usage bias of chloroplast genome in Kandelia obovata[J]. Journal of Forest and Environment,40(5):534-541.] doi:10.13324/j.cnki.jfcf.2020.05.011.
赵婉清,张敏,柳大军,雷慧,伊文博,刘佳,高志忠,张虎芳. 2020. 菜蝽线粒体基因组密码子偏好性分析[J]. 山西农业科学,48(7):1005-1009. [Zhao W Q,Zhang M,Liu D J,Lei H,Yi W B,Liu J,Gao Z Z,Zhang H F. 2020. Ana-lysis of codon usage bias in mitochondrial genome of Eurydema dominulus[J]. Journal of Shanxi Agricultural Scien-ces,48(7):1005-1009.] doi:10.3969/j.issn.1002-2481. 2020.07. 01.
赵洋,刘振,杨培迪,成杨,杨阳. 2016. 密码子偏性分析方法及茶树中密码子偏性研究进展[J]. 茶叶通讯,43(2):3-7. [Zhao Y,Liu Z,Yang P D,Cheng Y,Yang Y. 2016. Codon bias analysis method and research progress on codon bias in Camellia sinensis[J]. Tea Communication,43(2):3-7.] doi:10.3969/j.issn.1009-525X.2016.02.001.
郑小东,曲学存. 2013. 中国水生贝类图谱[M]. 青岛:青岛出版社. [Zheng X D,Qu X C. 2013. Atlas of aquatic molluscs in China[M]. Qingdao:Qingdao Press House.]
周丹,薛仁余,张晓峰,杜科,马吉敏,顾颖,孙效文. 2013. 鲤和斑马鱼HOX基因家族同义密码子使用偏性的分析[J]. 水产学杂志,26(2):19-25. [Zhou D,Xue R Y,Zhang X F,Du K,Ma J M,Gu Y,Sun X W. 2013. Sysnonymous codon bias of HOX gene family in common carp(Cyprinus carpio) and zebrafish(Danio rerio)[J]. Chinese Journal of Fisheries,26(2):19-25.] doi:10.3969/j.issn.1005-3832.2013.02.004.
朱秀芳,郭宇超,米琦,刘顿. 2015. 真白鲑线粒体蛋白编码基因的密码子偏性研究[J]. 凯里学院学报,33(6):60-63. [Zhu X F,Guo Y C,Mi Q,Liu D. 2015. Analysis on codon usage of mitochondrial protein-coding genes of Coregonus lavaretus[J]. Journal of Kaili University,33(6):60-63.] doi:10.3969/j.issn.1673-9329.2015.06.18.
Barbhuiya P A,Uddin A,Chakraborty S. 2020. Analysis of compositional properties and codon usage bias of mitochondrial CYB gene in anura,urodela and gymnophiona[J]. Gene,751:144762. doi:10.1016/j.gene.2020.144762.
Hershberg R,Petrov D A. 2008. Selection on codon bias[J]. Annual Review of Genetics,42:287-299. doi:10.1146/annurev.genet.42.110807.091442.
Zhang L,Guo Y,Luo L,Wang Y P,Dong Z M,Sun S H,Qiu L J. 2011. Analysis of nuclear gene codon bias on soybean genome and transcriptome[J]. Acta Agronomica Sinica,37(6):965-974. doi:10.1016/S1875-2780(11)60028-X.
Zhong S P,Huang G Q,Liu Y H,Huang L H. 2019. The complete mitochondrial genome of marine gastropod Melo melo(Neogastropoda:Volutoidea)[J]. Mitochondrial DNA Part B,4(2):4161-4162. doi:10.1080/23802359.2019. 1693293.
Zhou M,Li X. 2009. Analysis of synonymous codon usage patterns in different plant mitochondrial genomes[J]. Molecular Biology Reports,36(8):2039-2046. doi:10.1007/s11033-008-9414-1.
(責任编辑 陈德元)
