基于叶绿体DNA序列atpI-rsp2和psbC-trnS 的山药种质资源遗传多样性分析
2022-04-21张静珍张文英雷剑柴沙沙靳晓杰王崇杨园园程贤亮杨新笋王连军
张静珍 张文英 雷剑 柴沙沙 靳晓杰 王崇 杨园园 程贤亮 杨新笋 王连军



摘要:【目的】基于叶绿体DNA(cpDNA)序列atpI-rsp2和psbC-trnS分析山药种质资源的遗传多样性,为山药种质资源鉴定、创新利用及新品种选育提供理论依据。【方法】以来自14个省(区)的64份山药种质为材料,对其atpI-rsp2和psbC-trnS序列进行多态性扩增并测序,经拼接、比对后,利用MEGA 7.0计算种质间的遗传距离并构建系统发育进化树,并利用DNAsp 5.0分析核苷酸多态性,采用NET Framework 4.6.1绘制单倍型间的中介邻接网络结构。【结果】atpI-rsp2和psbC-trnS序列合并序列长度为1938 bp,共有117个插入/缺失位点(IS)和14个变异位点(Vs),转换率(Si)为49.1%,总体转换/颠换偏倚率(R)为0.928,共产生30种单倍型,其中有21种为独享单倍型,9种为共享单倍型,单倍型多样性(Hd)和核苷酸多样性(p)分别为0.9206和0.00139。atpI-rsp2序列和psbC-trnS序列及合并序列的Tajima’s D、Fu and Li’s D*和F*均为负值,其差异均未达显著水平(P>0.10),說明这2个序列在进化上符合中性进化模式。64份山药种质的遗传距离为0~0.003865,平均遗传距离均为0.001400。中介邻接网络结构分析结果显示,30种单倍型可分为3类,其中,H5为较原始单倍型。【结论】64份山药种质资源的遗传距离较近,遗传背景相似,可能是由于长期地区间引种导致,与地理距离不完全相关。atpI-rsp2和psbC-trnS序列可用于山药物种鉴定和系统进化分析等研究领域。
关键词: 山药;叶绿体DNA(cpDNA);atpI-rsp2;psbC-trnS;遗传多样性;遗传分化
中图分类号: S632.102.4 文献标志码: A 文章编号:2095-1191(2022)01-0125-09
Genetic diversity of yam germplasm revealed by chloroplast DNA sequences atpI-rsp2 and psbC-trnS
ZHANG Jing-zhen1,2, ZHANG Wen-ying2,LEI Jian1, CHAI Sha-sha1, JIN Xiao-jie1,
WANG Chong1,2, YANG Yuan-yuan1, CHENG Xian-liang1,
YANG Xin-sun1*, WANG Lian-jun1*
(1Hubei Sweet Potato Engineering and Technology Research Centre, Institute of Food Corps, Hubei Academy of Agricultural Sciences/Hubei Key Laboratory of Food Crops Germplasm and Genetic Improvement,
Wuhan 430064, China; 2College of Agriculture,Yangtze University, Jingzhou 434025, China)
Abstract:【Objective】The genetic diversity of 64 yam germplasm was analyzed based on the chloroplast DNA(cp-DNA) sequences atpI-rsp2 and psbC-trnS to provide a theoretical basis for the selection, innovative utilization and bree-ding of yam germplasm resources. 【Method】The atpI-rsp2 and psbC-trnS sequences of 64 yam germplasm collected from 14 provinces(regions) were amplified and sequenced for polymorphism. After mosaic and comparison, the genetic distance between germplasms was calculated by MEGA 7.0 and a phylogenetic tree was constructed. The nucleotide polymorphisms were analyzed by DNAsp 5.0 and NET Framework 4.6.1 was used to draw the intermediate adjacency network structure between haplotypes. 【Result】The combined sequence length of atpI-rsp2 and psbC-trnS was 1938 bp, with contained 117 insert/missing sites(IS) and 14 variable sites(Vs). The conversion frequency(Si) was 49.1%, and the overall transition/transversion bias(R) value was 0.928, resulting in 30 haplotypes. Of these, 21 haplotypes were exclusive and 9 haplotypes were shared, corresponding to a haplotype diversity index value(Hd) of 0.92063 and nucleotide diversity value(p) of 0.00139. The Tajima’s D values, Fu and Li’s D * and F * of the atpI-rsp2 sequence and the psbC-trnS sequence and the merged sequences were all negative, and the difference was not significant(P>0.10), suggesting the sequences in yams fitted the neutral evolutionary model. The genetic distance of 64 yam germplasm ranged from 0 to 0.003865 with an average of 0.001400. The results of the structure analysis of the intermediate adjacency network showed that the 30 haplotypes could be divided into three types, H5 was the primitive haplotype. 【Conclusion】The 64 yam germplasm resources have close genetic distance and similar genetic background, which may be caused by long-term interregional introduction. It is not entirely correlated with geographical distance. atpI-rsp2 and psbC-trnS sequences can be used for species identification and the phylogenetic analysis of yam.
Key words: yam; chloroplast DNA(cpDNA); atpI-rsp2; psbC-trnS; genetic diversity; genetic differentiation
Foundation items: Hubei Agricultural Science and Technology Innovation Center Funding Project(2020-620-000-001-007); Characteristic Discipline Project of Hubei Academy of Agricultural Sciences(2021)
0 引言
【研究意义】山药(Dioscorea spp.)是百合目(Li-liales)薯蓣科(Dioscoreaceace)薯蕷属(Dioscorea)一年生或多年生缠绕性藤本植物,可药食两用(雷伏贵,2011),主要分布于我国亚热带地区,具有丰富的山药种质资源。近年来,由于人类长期引种驯化、历史变迁等因素,使山药种质资源遗传背景复杂,难以从形态学角度进行准确鉴定和分类(雷伏贵,2011)。DNA序列分析是植物鉴定和分类的重要方法,可在较短时间内为传统园艺学分类及物种鉴定提供依据。植物叶绿体DNA(Chloroplast DNA,cpDNA)为双链共价闭合环状分子,其分子量小,约120~210 kb,母系遗传,结构简单,其非编码区序列进化速率比编码区(CDS)快,适用于低分类类群和种内的系统学研究(高丽杨等,2017)。常用的cpDNA序列有rbcL、matK、trnL-trnF和trnH-psbA等。基于cpDNA分子水平对山药种质资源进行遗传多样性分析,对于山药种质资源开发、保护和鉴定具有重要研究意义。【前人研究进展】目前,叶绿体DNA非编码区域已广泛应用于木材树种(张蓉等,2014)、黑藻(付春霖等,2017)、梨(齐丹等,2018)、何首乌(张宏意等,2018)、楸子(高源等,2020)等植物的遗传多样性研究。张蓉等(2014)基于木材树种的cpDNA序列trnL、trnL-trnF、trnH-trnK和psbC-trnS构建其系统发育进化树,结果发现这些序列对木材树种鉴定成功率较高。马双姣等(2017)利用高通量测序技术对薯蓣(D. opposita)和叉蕊薯蓣(D. collettii)cpDNA序列进行测序分析,经过多序列比对发现薯蓣属叶绿体基因组中非编码区序列变异高于保守的编码区,从中筛选出5个蛋白编码基因和5个基因间隔区作为鉴定薯蓣属植物的特异性DNA条形码序列,但这些DNA条形码在薯蓣属物种中的鉴定效率需大量种质资源进行验证分析,且对叶绿体全基因组进行比对能更加全面地反映薯蓣属物种的序列变异情况,可为叶绿体基因组序列中筛选特异性DNA条形码鉴定序列提供参考。付春霖等(2017)对黑藻cpDNA分子标记进行筛选并研究其遗传多样性,结果发现黑藻居群遗传差异的主要影响因子为生境差异和地理隔离。齐丹等(2018)利用5个叶绿体非编码区序列trnL-trnF-1、trnL-trnF-2、trnS-psbC、accD-psaI、rps16-trnQ和1个基因区域rbcL对我国秦岭淮河以南地区的188份梨种质资源进行遗传多样化及演化分析,根据cpDNA序列变异信息可推测秦岭淮河以南地区的砂梨和白梨亲缘关系较近,同时来自湖南地区的砂梨种质资源具有丰富的遗传多样性。张宏意等(2018)利用psbA-trnH序列对7个省(区)15个居群共116份何首乌种质DNA进行PCR扩增及测序分析,并分析cpDNA序列信息,结果显示何首乌的遗传变异丰富,聚类结果和种质产地分布较一致,且不同产地的何首乌psbA-trnH序列遗传变异较显著,为何首乌种源分子鉴定提供理论依据。高源等(2020)利用cpDNA序列trnH-psbA、trnS-trnG spacer+intron、trnT-5´trnL和5′trnL-trnF对楸子种质遗传多样性进行研究,结果表明楸子的遗传变异主要存在于群体内部,与地理距离不完全相关。【本研究切入点】我国具有丰富的山药种质资源,但对其分类鉴定尚缺乏可靠的分子依据。目前鲜见基于cpDNA序列对我国山药种质资源进行遗传多样性的研究报道。【拟解决的关键问题】以64份山药种质资源为研究对象,基于前人(马双姣等,2017;沈奇等,2019)研究结果筛选适宜的cpDNA基因间隔区序列atpI-rsp2和psbC-trnS,对其进行扩增及测序,并分析山药种质的亲缘关系及遗传多样性,为我国山药种质资源系统进化、种质资源保护及育种亲本选择提供理论参考。
1 材料与方法
1. 1 试验材料
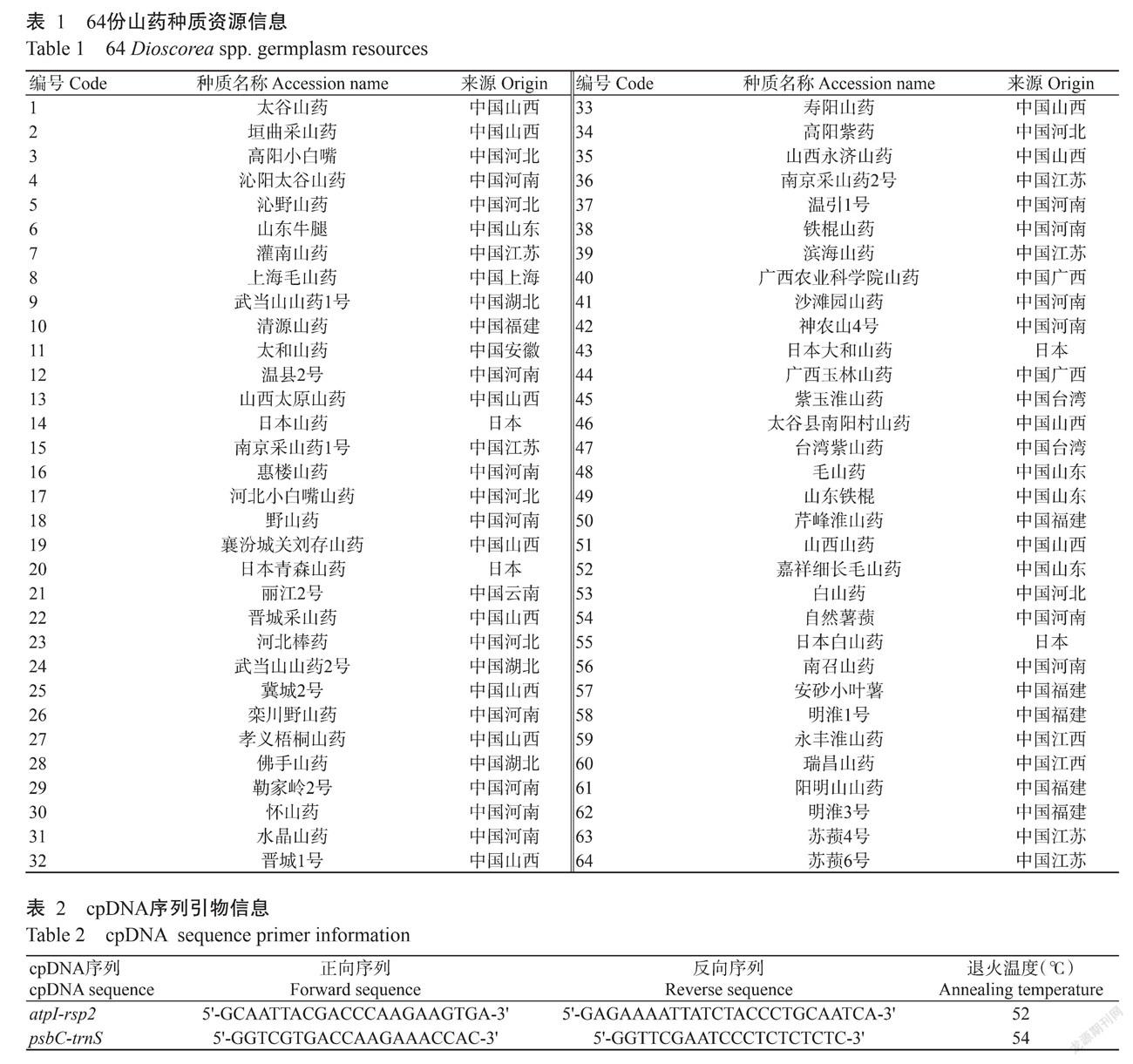
供试的64份山药种质资源为河南省焦作市温县山药种质资源圃(表1)。Easy Taq DNA聚合酶、10×Easy Taq Buffer for PAGE、dNTPs等均购自武汉全式金生物技术有限公司。主要仪器设备:离心机(Eppendorf,德国)、凝胶成像仪(Bio-Rad,美国)、NanoDrop2000分光光度计(Thermo,美国)和PCR仪(Bio-Rad,美国)。
1. 2 试验方法
1. 2. 1 DNA提取 采用改良的CTAB法提取山药幼嫩叶片的DNA(张安世等,2009)。使用NanoDrop2000分光光度计测量DNA浓度,将山药种质DNA溶液稀释至50~60 ng/μL,取2 μL稀释后的DNA用1.0%琼脂糖凝胶电泳(恒定电压120 V,20 min)进行检测。
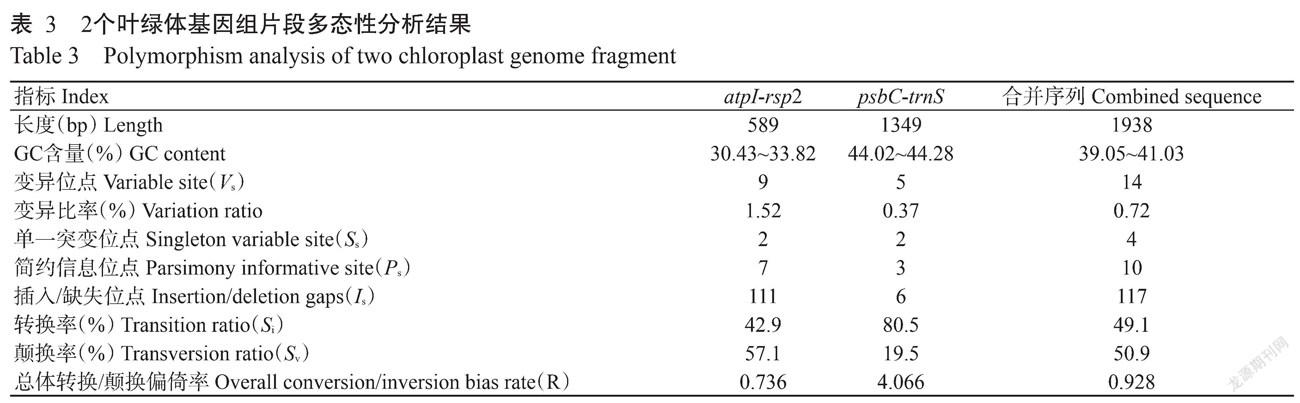
1. 2. 2 PCR扩增 参照马双姣等(2017)设计的特异引物,用于山药cpDNA的PCR扩增和测序。根据扩增条带的清晰度、稳定性及多态性共筛选出2对cpDNA非编码区序列(atpI-rsp2和psbC-trnS)引物,如表2所示。反应体系25.0 μL:2.5 μL 10×Buffer,10 mmol/L dNTPs 2.0 μL,10 μmol/L上、下游引物各1.0 μL,5 U/mL Taq DNA聚合酶0.5 μL,50 ng/μL DNA模板1.0 μL,ddH2O补足至25.0 μL。扩增程序:94 ℃ 5 min;94 ℃ 30 s,52 ℃(或54 ℃) 30 s,72 ℃ 1 min,进行35个循环;72 ℃延伸5 min;4 ℃保存。取2.0 μL的PCR扩增产物通过1.0%琼脂糖凝胶电泳(恒定电压120 V,20 min)进行检测,将PCR产物送至由天一辉远(武汉)生物科技有限公司进行正、反双向测序。
1. 2. 3 序列特征分析 利用DNAMAN 7.0对双向测序成功的序列进行比对,并去除两端的不可靠的侧翼序列,再生成用于MEGA 7.0和DNAsp 5.0分析的FASTA数据格式。利用DNAsp 5.0计算插入/缺失位点[Insertion/deletion(Indel),Is)]和突变位点数目,包括单一突变位点(Singleton variable site,Ss),简约信息位点(Parsimony informative site,Ps)。利用MEGA 7.0对对位排列后的序列进行序列长度、变异位点(Variable site,Vs)、颠换率(Transversion ratio,Sv)、转换率(Transition ratio,Si)、总体转换/颠换偏倚率(R)(Si/Sv)和GC含量等序列特分析。
1. 2. 4 倍性多态性分析 利用DNAsp 5.0计算单倍型数目(Number of haplotype,h)、单倍型多态性(Haplotype diversity,Hd),核苷酸多样性(Nucleotide diversity,p)和平均核苷酸差异(Average number of nucleotide difference,k),并计算Tajima’s D、Fu and Li’s D*和Fu and Li’s F*进行中性检验(Neutrality tests)。
1. 2. 5 系统发育进化树及单倍型间的进化网络图的构建 根据比对结果,利用MEGA 7.0中的Kimura-2-parameter(K2P)模型计算遗传距离,以最大似然法(ML)构建64份山药种质的系统发育进化树,以邻接法(NJ)构建单倍型系统发育进化树,对分支的可靠性使用Bootstrap(1000次重复)进行评价分析(Kumar et al.,2016)。自展数值>75%表示支持率高,50%~74%表示弱支持率,<50%表示不支持(朱元娣等,2014)。采用NET Framework 4.6.2以最大简约法绘制单倍型间的中介邻接网络结构(Polzin and Daneshmand,2003)。
2 结果分析
2. 1 序列特征分析
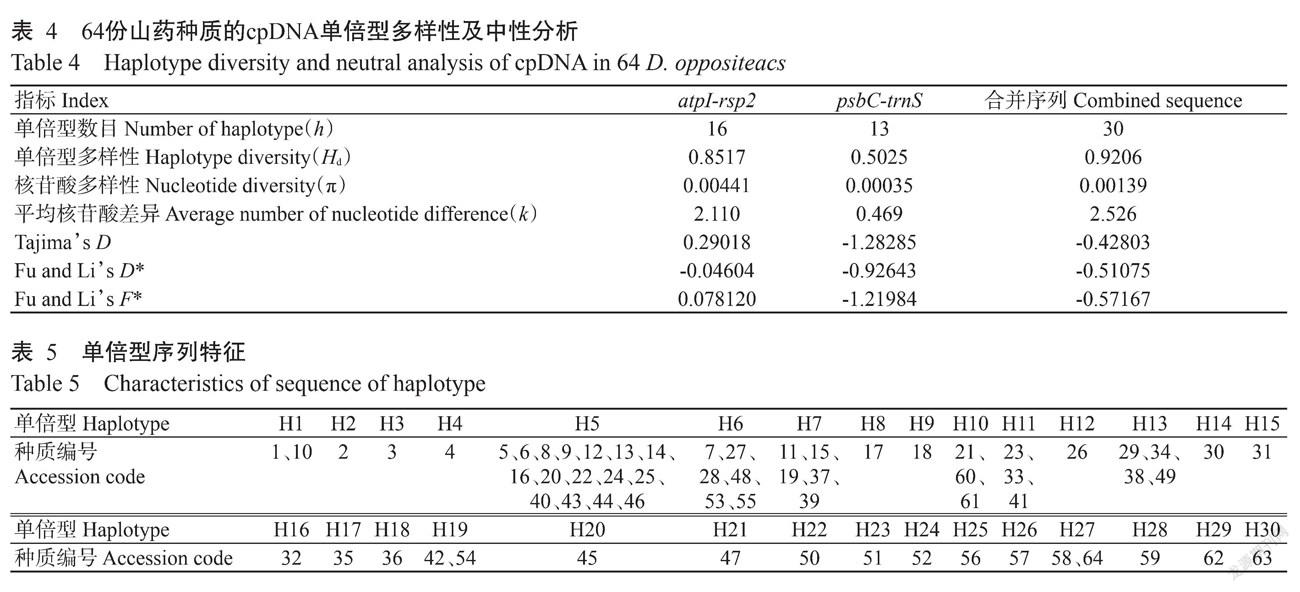
利用cpDNA序列atpI-rsp2和psbC-trnS的扩增引物进行PCR扩增及其产物电泳检测,结果发现2条清晰的目的条带,大小分别约600和1300 bp(图1)。根据这2个片段的双向测序结果,去除两端的不可靠的侧翼序列后,分别获得了589和1349 bp的片段序列(表3)。atpI-rsp2序列的GC含量为30.44%~31.15%,AT含量为67.85%~69.67%;psbC-trnS序列的GC含量为44.02%~44.28%;AT含量为55.72%~55.98%。
atpI-rsp2序列的保守位点为469个,变异位点(Vs)共9个,约占扩增位点总数的1.52%,其中包括7个简约信息位点(Ps)和2个单一突变位点(Ss),插入/缺失位点(Is)111个,转换率(Si)为42.9%,颠换率(Sv)为57.1%,总体转换/颠换偏倚率(R)为0.736;psbC-trnS序列的多态性较低,有2个单一突变位点(Ss),3个简约信息位点(Ps),6个插入/缺失位点(Is),转换率(Si)80.5%,而颠换率(Sv)仅占19.5%,均为C与T之间的转换,总体转换/颠换偏倚率(R)为4.066。
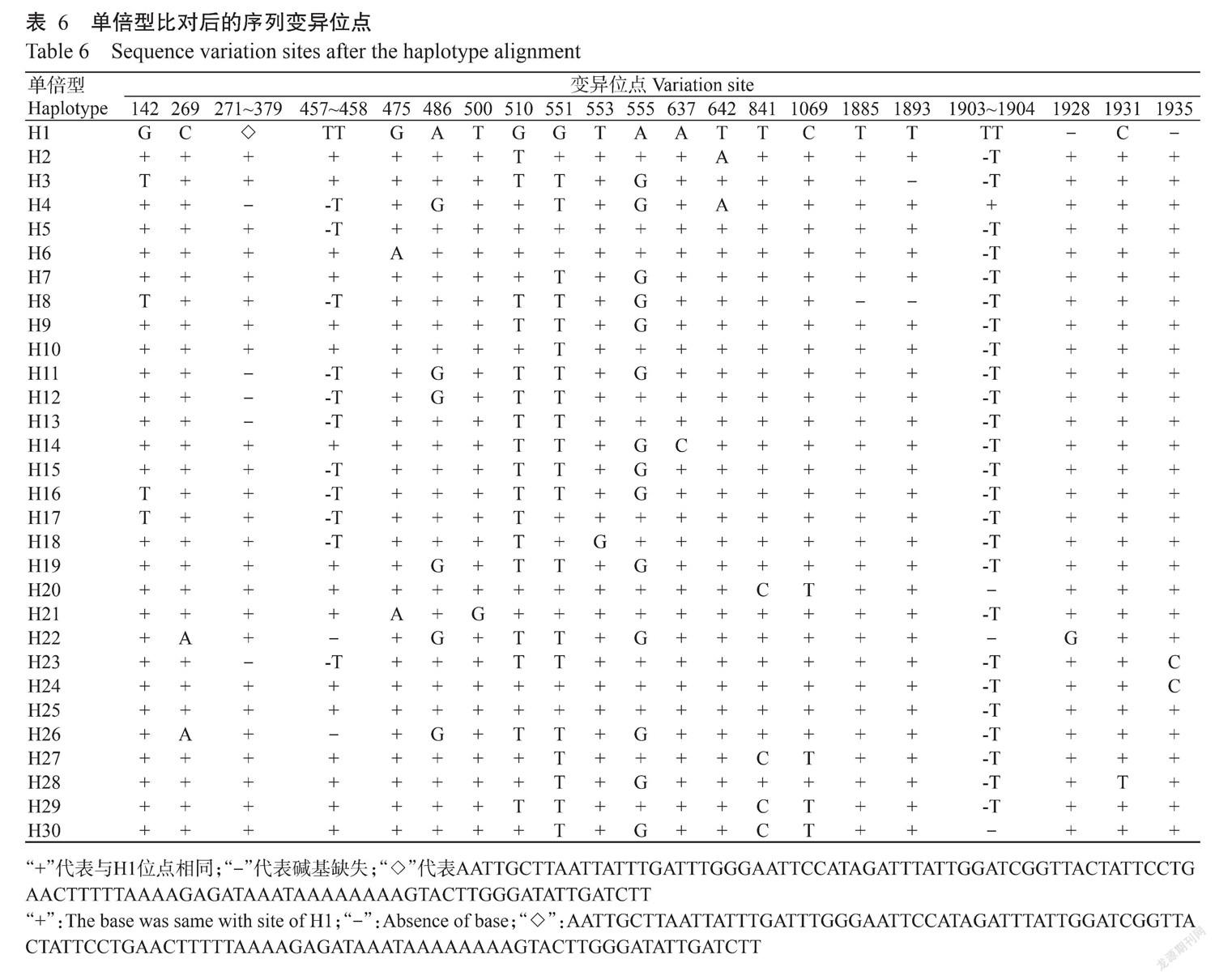
atpI-rsp2和psbC-trnS合并后对位排列长度为1938 bp,GC含量为39.05%~41.03%,共有131个突变位点,其中包括14个变异位点(Vs)和117个插入/缺失位点(Is)(表3)。合并序列的10个简约信息位点(Ps)分别出现在142、269、475、486、510、551、555、642、841和1069位点;4个单一突变位点(Ss)分别出现在500、553、637和1931位点,7个插入/缺失片段分别在271~397、457~458、1885、1893、1903~1904、1928和1935,共117个位点;转换率(Si)为49.1%,总体转换/颠换偏倚率(R)为0.928。
2. 2 倍性多态性分析及中性检验
分别对atpI-rsp2序列和psbC-trnS序列进行单倍型多样性分析,结果(表4)表明,在64份山药种质中,atpI-rsp2和psbC-trnS序列的单倍型数(h)分别为16和13个,atpI-rsp2序列的單倍型多样性(Hd)和核苷酸多样性(p)较高,分别为0.8517和0.00441;合并序列的单倍型(h)为30种,即H1~H30(表5),单倍型多样性(Hd)和核苷酸多样性(p)分别为0.9206和0.00139。atpI-rsp2和psbC-trnS序列及二者合并序列的平均核苷酸差异(k)分别为2.110、0.469和2.526。
为检验山药居群是否发生扩张,对atpI-rsp2和psbC-trnS序列进行中性检验,结果(表4)显示,atpI-rsp2序列和psbC-trnS序列及二者合并序列的Tajima’s D、Fu and Li’s D*和F*均为负值,且差异均未达显著水平(P>0.10),符合中性进化模式,表明从物种水平上山药atpI-rsp2序列和psbC-trnS序列及合并序列符合中性进化假设。
2. 3 聚类分析结果
采用MEGA 7.0中的K2P模型计算山药不同种质间cpDNA序列的遗传距离,结果显示,山药种质间遗传距离为0~0.003865,平均遗传距离为0.001400,其中,沁阳太谷山药、芹峰淮山药和安砂小叶薯3个种质与紫玉淮山药遗传距离最大,均为0.003865,与台湾紫山药遗传距离次之,均为0.003864。
为了解64份供试山药种质的群体遗传结构,利用ML构建系统发育进化树,结果如图2所示。64份山药种质可分为八大类,其中,河北的白山药和日本的白山药聚在第Ⅱ类中,山西的太谷山药和太谷县南阳村山药聚类在第Ⅴ类中,推测上述种质存在同物异名的现象,或存在地区间引种现象;第VII类中的种质大部分来自江苏、福建和江西等地,其中,苏蓣4号和苏蓣6号紫山药由江苏徐淮地区徐州农业科学研究所选育,福建省三明市农业科学院选育的明淮1号、明淮3号、阳明山山药及台湾的紫玉淮山药均为紫山药,上述6个种质聚在一个小分支,表明其亲缘关系较近;第Ⅷ类中的种质主要来自河南、山西和山东,其中,山东铁棍和河南铁棍山药聚在一个小分支,推测山东铁棍为河南铁棍山药的引种,为同一种质。
从已获得的64条序列中定义了30种单倍型(H1~H30),单倍型比对后变异位点如表6所示。其中,H5为共享单倍型,在所有种质中出现了16次,占25.0%,单倍型H6出现了6次(9.3%),H7出现了5次(7.8%)。在30种单倍型中,有21种单倍型为独享单倍型。由单倍型系统发育进化树(图3)可看出,分支上显示的置信度较低,H14和H15单倍型的置信度仅为9,说明单倍型差异较小,不能形成可靠分支。
2. 4 山药cpDNA单倍型中介网络关系
根据中介邻介网络(MJ)算法和最大简约算法(MP)优化计算结果并分析2个cpDNA序列组合的单倍型关联图(图4)。单倍型的中介矢量位点分别用H和mv表示,其中mv代表可能出现的原始单倍型。分析结果显示,单倍型H5和H7为古老单倍型,H4和H21为较进化单倍型;单倍型H5、H7、H10、H15、H19、H25、H20和H27与mv1~mv6位于躯干位置上,分化时间早,其中单倍型H5和H7在序列上表现为3个碱基差异。山药单倍型可分成3种类型:Type A(在atpI-rsp2中存在长度为109 bp的缺失)、Type B(在psbC-trnS片段缺失)和Type C(在atpI-rsp2片段缺失的)。其中,Type A包括5个单倍型H4、H11、H12、H13和H23,由mv4延伸出来。Type B包括18个单倍型,可分为3个组(B1~B3),B1组包括单倍型H1、H2、H6、H21、H24和H25,均由H5单倍型派生出来;B2组包括单倍型H20、H27、H29和H30,由mv6延伸出来;B3组包括单倍型H7、H9、H10、H14、H19和H28,由mv5延伸出来;Type C包括3组(C1~C3),C1组包括单倍型H17和H18,由mv1延伸出来,C2组包括单倍型H3、H8、H15和H16,由mv2和mv4延伸出来;C3组包括H22和H26,由mv2延伸出来,为独有单倍型。
3 讨论
目前对山药种质资源进行鉴定及保护是山药产业发展的重要环节。随着地区间互相引种,导致同物异名等现象日益突出,通过形态特征鉴定种质的差异难度增大。程月琴等(2019)利用cpDNA序列trnQ-rps16和psbM-trnD对20个怀山药品种及其他20个山药品种进行鉴定,结果表明,怀山药在trnQ-rps16序列出现了长度为210 bp的缺失,说明该序列可有效区分怀山药和其他山药品种。本研究也发现,怀山药为独有单倍型(H14),在序列上存在3个碱基差异,可有效区分于其他种质。本研究通过atpI-rsp2和psbC-trnS序列分析发现,64份山药种质的遗传距离为0~0.003865,表明种质间遗传距离较近,遗传背景相似,与地理距离不完全相关,可能是由于长期地区间引种所导致。太谷山药原产于山西,铁棍山药原产于河南温县,形态相似,两者存在109 bp的碱基缺失(270~379 bp)和3个碱基差异,遗传距离为0.0011,说明太谷山药和铁棍山药间存在明显的遗传变异,亲缘关系较远,为鉴别山药种质提供依据,与吴志刚(2012)利用cpDNA序列rbcL序列对14份山药种质的分析结果相似,14份山药种质的平均遗传距离为0.0044,太谷山药和铁棍山药存在11处碱基差异,说明atpI-rsp2和psbC-trnS序列可有效鉴定山药品种。
本研究利用筛选出cpDNA非编码区序列atpI-rsp2和psbC-trnS对来自不同地区64份山药种质进行遗传多样性分析,结果显示,在64份山药种质中共发现131个突变位点,在整个区段均有分布,共产生了30种单倍型,具有较高的单倍型多样性,其单倍型数量高于李亚属物种(章秋平等,2017)和茶树(刘振等,2020)的单倍型数量,可能是由于山药种质具有更强的环境适应能力。由于H5单倍型分布最广泛,推测这种单倍型是山药植株适应环境变化并在居群中稳定存在的优势单倍型。在30种单倍型中,有9种为共享单倍型,有21种单倍型是独享单倍型,其中,来自河南的山药种质的单倍型数量最多,共14种单倍型,表明该地区山药种质资源较丰富,对不同环境具有较强的适应能力,这可能与物种特点、生态环境变化和栽培习性有关。
64份山药种质的30种单倍型中,有8种单倍型H5、H7、H10、H15、H19、H25、H20和H27与mv1~mv6位于躯干位置上,其中,单倍型H5包含了来自9个地区的16个种质,是所有单倍型中种质数量最多,来源最广泛的类型。若单倍型在种群或居群中覆盖范围最广、出现频率最高,并处于单倍型网络的中心位置,故推测这种单倍型是所有单倍型中最古老的类型(Posada and Crandall,2001),以H5为中心呈星状辐射,说明其发生种群扩张。可见,本研究基于cp-DNA非编码区序列atpI-rsp2和psbC-trnS对山药种质遗传多样性进行分析,结果发现了大量的变异位点,获得了较全面的遗传信息。由于不同来源的山药群体之間的遗传关系和遗传分化不完全与地理距离相关的情况,可能与群体数量有关。因此,在今后研究中,应加大种质资源收集力度和增加采集地点,目标是以较少的种质代表该地区的遗传变异,需要结合其他分子标记,比如核基因或线粒体基因组的分子标记,或进一步加深对山药居群的遗传机制研究,为山药系统发育进化分析提供新思路。
4 結论
64份山药种质资源的遗传距离较近,遗传背景相似,可能是由于长期地区间引种导致,与地理距离不完全相关。atpI-rsp2和psbC-trnS序列可用于山药物种鉴定和系统进化分析等研究领域。
参考文献:
程月琴,李萍萍,王梦云,王昱程,王红卫. 2019. 基于叶绿体DNA变异的怀山药鉴定体系构建[J]. 河南农业大学学报,53(5):759-765. [Cheng Y Q,Li P P,Wang M Y,Wang Y C,Wang H W. 2019. Construction of the system for the identification of Huai-yam(Dioscorea Opposita) based on chloroplast DNA variation[J]. Journal of Henan Agricultural University,53(5):759-765.] doi:10.16445/j.cnki.1000-2340.2019 0919.001.
付春霖,戴小康,李小燕,刘星. 2017. 黑藻cpDNA分子标记的筛选及其遗传多样性的初步研究[J]. 植物科学学报,35(1):87-92. [Fu C L,Dai X K,Li X Y,Liu X. 2017. Scree-ning cpDNA markers and preliminary study on the gene-tic diversity of Hydrilla verticillata from China[J]. Plant Science Journal,35(1):87-92.] doi:10.11913/PSJ.2095-0837.2017.10087.
高丽杨,夏惠,谢玥,沈妍秋,王秀,梁东. 2017. 基于cpDNA序列的猕猴桃种质资源多态性分析[J]. 分子植物育种,15(9):3751-3758. [Gao L Y,Xia H,Xie Y,Shen Y Q,Wang X,Liang D. 2017. Molecular polymorphic analyses for the germplasms of Actinidia based on cpDNA[J]. Molecular Plant Breeding,15(9):3751-3758.] doi:10. 13271/j.mpb.015.003751.
高源,王大江,王昆,丛佩华,张彩霞,李连文,朴继成. 2020. 基于叶绿体DNA分析的楸子种质遗传多样性研究[J]. 园艺学报,47(5):853-863. [Gao Y,Wang D J,Wang K,Cong P H,Zhang C X,Li L W,Piao J C. 2020. Genetic diversity of Malus prunifolia germplasms based on chloroplast DNA analysis[J]. Acta Horticulturae Sinica,47(5):853-863.] doi:10.16420/j.issn.0513-353x.2019-0609
雷伏贵. 2011. 山药种质资源遗传多样性的ISSR分析[D]. 福州:福建农林大学. [Lei F G. 2011. Analysis of genetic diversity of yam of germplasm rescources[D]. Fuzhou:Fujian Agriculture and Forestry University.]
刘振,成杨,杨培迪,赵洋,宁静,杨阳. 2020. 基于nSSR和cpDNA序列的城步峒茶群体遗传多样性和结构研究[J]. 茶叶科学,40(2):250-258. [Liu Z,Cheng Y,Yang P D,Zhao Y,Ning J,Yang Y. 2020. Genetic diversity and structure of Chengbudong tea population revealed by nSSR and cpDNA markers[J]. Journal of Tea Science,40(2):250-258.] doi:10.13305/j.cnki.jts. 2020.02.011.
马双姣,周建国,李滢,陈新连,吴明丽,孙伟,李永华,宋经元,姚辉. 2018. 薯蓣和叉蕊薯蓣叶绿体基因组及特异性DNA条形码鉴定序列筛选研究[J]. 中国科学:生命科学,48:571-582. [Ma S J,Zhou J G,Li Y,Chen X L,Wu M L,Sun W,Li Y H,Song J Y,Yao H. 2018. Complete chloroplast genomes of Dioscorea opposite and D. collettii and screening specific DNA barcodes[J]. Scientia Sinica Vitae,48:571-582.] doi:10.1360/N052017-00160.
齐丹,常耀军,曹玉芬,胡红菊,田路明,董星光,张莹,霍宏亮,徐家玉,张小双,刘超. 2018. 基于叶绿体DNA信息的南方梨属种质的遗传多样性和演化分析[J]. 园艺学报,45(12):2308-2320. [Qi D,Chang Y J,Cao Y F,Hu H J,Tian L M,Dong X G,Zhang Y,Huo H L,Xu J Y,Zhang X S,Liu C. 2018. Genetic diversity and phylogenetics of pear(Pyrus L.) germplasm resources from South China revealed by chloroplast DNA[J]. Acta Horticulturae Sinica,45(12):2308-2320.] doi:10.16420/j.issn.0513-353x. 2018-0176.
沈奇,潘月云,臧春鑫,赵志平,关潇,张银东. 2019. 基于叶绿体 DNA 单倍型的蒙古沙冬青遗传多样性格局探究[J]. 分子植物育种,17(4):1378-1384. [Shen Q,Pan Y Y,Zang C X,Zhao Z P,Guan X,Zhang Y D. 2019. Study on distribution pattern of genetic diversity of Ammopiptanthus mongolicus based on the haplotype of chloroplast DNA[J]. Molecular Plant Breeding,17(4):1378-1384.] doi:10.13271/j.mpb.017.001378.
吴志刚. 2012. 山药种质资源遗传亲缘关系与药材质量研究[D]. 广州:广州中医药大学. [Wu Z G. 2012. Study on genetic relationship and crude drugs quality of yam germplasm resources[D]. Guangzhou:Guangzhou University of Chinese Medicine.]
张安世,邢智峰,刘永英,张为民. 2009. 苔藓植物DNA不同提取方法的比较分析[J]. 河南科学,27(5):559-562. [Zhang A S,Xing Z F,Liu Y Y,Zhang W M. 2009. Comparison and analysis on the different methods of DNA extraction methods from Bryophytes[J]. Henan Science,27(5):559-562.] doi:10.13537/j.issn.1004-3918.2009. 05.008.
张宏意,袁林林,饶秋红,李威,严寒静. 2018. 不同产地何首乌叶绿体psbA-trnH基因序列分析[J]. 中草药,49(5):1146-1149. [Zhang H Y,Yuan L L,Rao Q H,Li W,Yan H J. 2018. Sequence analysis of psbA-trnH gene in chloroplast of Polygonum multiflorum from different produ-cing areas[J]. Chinese Traditional and Herbal Drugs,49(5):1146-1149.] doi:10.7501/j.issn.0253-2670.2018. 05.024.
张蓉,殷亚方,徐魁梧,叶克林. 2014. 基于DNA的木材树种识别研究进展[J]. 南京林业大学学报(自然科学版),38(1):151-156. [Zhang R,Yin Y F,Xu K W,Ye K L. 2014. Progress of wood identification based on DNA methods[J]. Journal of Nanjing Forestry University(Natural Sciences Edition),38(1):151-156.] doi:10.3969/j.issn.1000-2006.2014.01.027.
章秋平,魏潇,刘威生,董文轩,刘宁,张玉萍,徐铭,刘硕,张玉君,马小雪. 2017. 基于叶绿体DNA序列trnL-F分析李亚属植物的系统发育关系[J]. 果树学报,34(10):1249-1257. [Zhang Q P,Wei X,Liu W S,Dong W X,Liu N,Zhang Y P,Xu M,Liu S,Zhang Y J,Ma X X. 2017. Phylogenetic relationship in the plants of subgenus Prunophora(Rosaceae) inferred from the chloroplast DNA region,trnL-F[J]. Journal of Fruit Science,34(10):1249-1257.] doi:10.13925/j.cnki.gsxb.20170098.
朱元娣,曹敏格,許正,王昆,张文. 2014. 基于ITS和matK序列探讨新疆野苹果与中国苹果的系统演化关系[J]. 园艺学报,41(2):227-239. [Zhu Y D,Cao M G,Xu Z,Wang K,Zhang W. 2014. Phylogenetic relationship between Xinjiang wild apple(Malus sieversii Roem.) and Chinese apple(Malus×domestica subsp. chinesnsis)based on ITS and matK sequences[J]. Acta Horticulturae Sinica,41(2):227-239.] doi:10.16420/j.issn.0513-353x.2014. 02.027.
Kumar S,Stecher G,Tamura K. 2016. MEGA7:Molecular evolutionary genetics analysis version 7.0 for Bigger Datasets[J]. Molecular Biology and Evolution,33(7):1870-1874. doi:10.1093/molbev/msw054.
Polzin T,Daneshmand S V. 2003. On Steiner trees and minimum spanning trees in hypergraphs[J]. Operations Research Letters,31(1):12-20. doi:10.1016/S0167-6377(02)00185-2.
Posada D,Crandall K A. 2001. Intraspecific gene genealogies:trees grafting into networks[J]. Trends in Ecology and Evolution,16:37-45. doi:10.1016/S0169-5347(00)02026-7.
(责任编辑 陈 燕)
