基于简化基因组测序的不同干形云南松遗传分析*
2022-04-07吴治洋甘沛华刘子腾朱梅彩何承忠
吴治洋,纵 丹,3,甘沛华,刘子腾,朱梅彩,何承忠,3
(1.西南林业大学,云南省高校林木遗传改良与繁育重点实验室,云南 昆明 650224;2.西南林业大学,西南地区生物多样性保育国家林业和草原局重点实验室,云南 昆明 650224;3.西南林业大学,西南山地森林资源保育与利用教育部重点实验室,云南 昆明 650224)
云南松(Pinus yunnanensis)是中国荒山荒地造林的主要树种,也是西南地区重要的用材树种,具有生长快、材质较好、耐干旱和木材用途广泛等优良特点[1-3]。云南松以云贵高原为起源中心和分布中心,分布于N23°~30°、E96°~108°,其辐射分布范围包括云南大部分地区、西藏东南部、四川西南部、广西西部和贵州西部[4-6]。云南省的云南松林地面积达344.84 hm2,占林分总面积的52%,是主要的森林植被覆盖类型之一[4,7],在经济、社会和环境的可持续发展中具有其他树种不可替代的重要作用[8]。随着气候变迁、人类干扰活动加剧及自然灾害频发等因素,云南松的生存状况不容乐观。当前云南松林分中80%以上是纯林,林分组成极其简单[9]。绝大多数云南松林分由低矮、弯曲和扭曲等个体构成,呈现为低质低效林,林分表现出严重的衰退现象,优良遗传资源正在急剧减少[1,4,9-10]。云南松干形扭曲形成的机制还存在较大争议,属于遗传因素决定,还是环境因素引起尚不明确。
简化基因组测序(reduced-representation genome sequencing,RRGS)技术基于二代高通量测序技术,通过限制性内切酶对基因组进行打断,经过添加接头和过滤等筛选基因组特异性长度区域进行测序,进而开发大量遗传标记[11-12]。该技术大大降低了基因组的复杂性,同时具有不依赖参考基因组而获得大量遗传标记、操作简单和成本较低等优点。目前,RRGS 技术主要被用于分子标记开发、群体遗传及系统发生学、高密度遗传图谱构建和QTL 定位等[13-14]。在裸子植物中,胚乳来源于雌配子体,存在于受精作用前,由大孢子叶直接发育而来,为单倍体,具有染色体来源于母本的特点。基于此,本研究利用RRGS 技术中基于测序的基因分型 (genotyping by sequencing,GBS)双酶切(ddGBS)对不同干形云南松胚乳和针叶进行测序分析,根据单倍体胚乳和二倍体针叶的组内及组间序列比对,在全基因组范围中挖掘不同干形云南松SNP 标记并分析其特征,揭示不同干形云南松遗传变异特点,为进一步探究云南松干形变异机制提供科学依据。
1 材料与方法
1.1 试验材料
从云南省楚雄彝族自治州永仁县方山云南松纯林居群中,选取树龄相近的直干形云南松5 株和扭纹角>30o的扭干形云南松6 株,分别采集针叶和球果,同类干形的样株相隔50 m 以上。针叶样品装入冻存管后立即放入液氮中冻存;球果带回实验室,人工脱粒后去除外种皮和合子胚,仅保留胚乳,按单株将人工剥取的胚乳装入冻存管,置于液氮中冻存。样本分为4 组,即直干形云南松胚乳组(ES01、ES02、ES03、ES04 和ES05)、直干形云南松针叶组(NS01、NS02、NS03、NS04和NS05)、扭干形云南松胚乳组(ET01、ET02、ET03、ET04、ET05 和ET06)以及扭干形云南松针叶组(NT01、NT02、NT03、NT04 和NT05、NT06)。
1.2 DNA 的提取及质量检测
采用HiPureSFPlant DNA Kit 试剂盒分别提取针叶和胚乳的总DNA,DNA 的完整性、纯度和质量浓度采用1%的琼脂糖凝胶电泳和核酸蛋白检测仪共同进行检测。合格的DNA 样品稀释至100 ng/μL,并置于-20 ℃冰箱保存,备用。
1.3 文库构建
ddGBS 文库构建及测序委托广州基迪奥生物科技有限公司承担完成。具体操作步骤:采用特定的2 种限制性内切酶 (EcoR I+NlaIII)对质检合格的基因组DNA 进行消化;再进行末端修复,添加A-尾和Illumina 测序接头;对300~400 bp的DNA 片段进行PCR 扩增富集;使用AMPure XP系统对PCR 产物进行纯化,使用Agilent 2100 生物分析仪(Agilent,USA)对测序文库进行检测,并使用实时定量PCR 进行文库定量,合格后采用Novaseq 6000 测序仪进行测序,测序策略为Illumina PE150。
1.4 高通量测序分析
Novaseq 6000 测序仪测序获得的原始图像数据经Base Calling 转化为序列数据,得到原始测序数据文件,结果以FASTQ 文件格式存储,包含reads 的序列以及碱基的测序质量。在测序数据下机之后,应用Fastp 软件对下机数据进行质量控制,过滤去除含adapter 的reads、含N 比例大于10%的reads 以及质量值(Q)≤10且碱基数占整条read 的50%以上的低质量reads,得到高质量的数据(high quality clean reads)用于后续信息分析。
1.5 基因组SNP 检测与开发
使用VAN DER AUWERA 等[15]的UnifiedGenotyper 模块将处理好的比对文件进行多个样本的变异检测,以期获得较高质量的SNP 位点,对检测所得的变异进行以下参数过滤:每15 bp 中存在超过3个SNP 位点、单位深度变异值(QD)<4、比对质量值离散程度(MQ)<40、Fisher’s 检测P值(FS)>60 和基因型质量值(GQ)<20 的异常序列,过滤后再分别进行个体聚类以及群体聚类分析。
1.6 群体分析
根据所得的SNP 信息计算样本之间的遗传距离,对样本进行聚类分析,从而推断出样本之间亲缘关系远近。利用treebest[16]邻近法构建进化树。使用PLINK[17]和GCTA[18]进行主成分分析。采用admixture 软件[19]进行群体结构分析。使用perl 编程对云南松直、扭干形之间的期望杂合度(He)、观测杂合度(Ho)、多态性信息含量(PIC)、观测等位基因数(Na)、有效等位基因数(Ne)、基因多样性指数(Nei)、Shannon 信息指数(I)、遗传分化指数(Fst)和基因流(Nm)等指标进行计算。
2 结果与分析
2.1 测序数据质控和过滤结果
由于简化基因组测序reads 为基因组DNA的酶切片段,其碱基组成分布检查主要是检测有无AT、GC 的分离现象。基于酶切片段的随机性和碱基互补配对原则,理论上每个测序循环的AT 和GC 含量相等,并在测序全过程中基本保持不变。本研究样品ES01 的测序碱基组成分布如图1 所示:AT 含量和GC 含量基本保持稳定,模糊碱基N 所占比例较低,表明未知碱基数较少,且测序样本受系统AT 偏好影响较小。结果表明:该样品的文库构建质量与测序质量均可满足后续相关分析。

图1 样品ES01 的碱基组成分布图Fig.1 Distribution of base composition of sample ES01
对质控后数据进行统计,共获得高质量序列数490 万个,4 组分析样本过滤后得到的高质量测序数据在14.30~17.15 G 之间,GC 含量在44.61%~50.60%之间,碱基质量Q30 比例在93.65%~95.19%之间(表1)。由此可知:所测序列的Q30 数据较高,表明碱基测序出错率很低,GC 分布合理,数据量达到分析要求,建库测序成功。
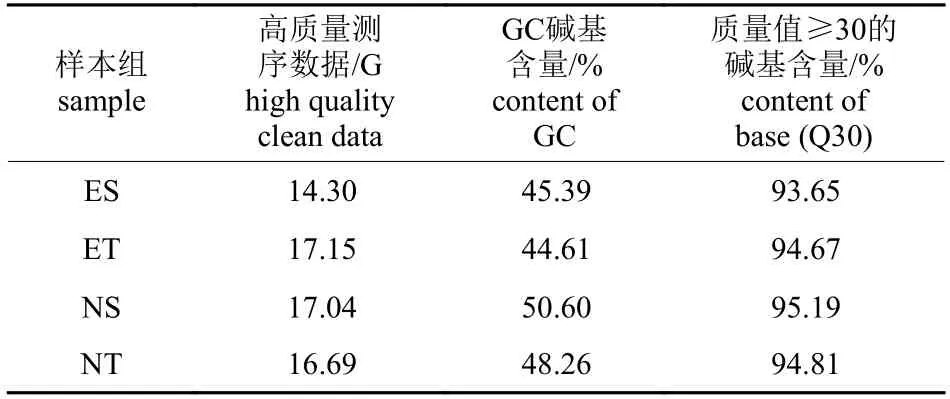
表1 云南松针叶和胚乳简化基因组DNA 测序数据Tab.1 ddGBS sequencing of endosperm and needle of Pinus yunnanensis
2.2 SNP 和InDel 位点检测
变异位点检测结果显示:共检测出772 240个变异,其中SNP 变异有762 699个,InDel 变异9 541个。在SNP 变异中,颠换(38.28%)小于转换(61.72%);其中,颠换类型中,置换比例从小到大依次为T→A (2.40%)=A→T (2.40%)< T→G (3.80%)< A→C (3.88%)< G→T (4.16%)< C→A(4.19%)< C→G (8.61%)< G→C (8.83%),而转换类型中,置换比例从小到大依次为A→G (14.72%)<T→C (14.83%)< G→A (15.96%)< C→T (16.21%)。在9 541个InDels 中,插入型变异共4 597个(48.18%),缺失型变异为4 944个(51.82%)。In-Del 的长度从1~53 bp 均有分布,主要以小片段(1~10 bp)分布为主。其中,单碱基InDels 的数量最多,达到5 207个;2 碱基InDels 次之,有1 211个;3 碱基InDels 变异为480个(图2)。
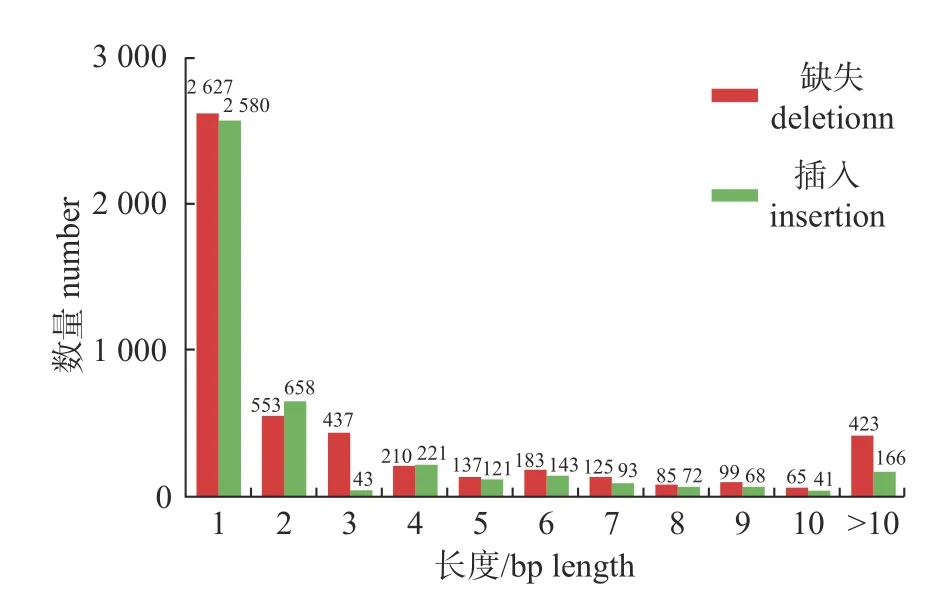
图2 InDel 变异类型Fig.2 Variation types of InDel
2.3 遗传变异分析
基于得到的762 699个SNP 标记,对云南松4个样本组分别进行遗传变异分析。结果(表2)显示:云南松直干型胚乳组(ES)、直干型针叶组(NS)、扭干型胚乳组(ET)和扭干型针叶组(NT)的期望杂合度(He)为0.203 9~0.229 9、观测杂合度(Ho)为0.100 2~0.145 7、多态性信息含量(PIC)为0.163 3~0.183 7、观测等位基因数(Na)为0.340 5~0.442 3、有效等位基因数(Ne)为1.347 2~1.388 4,基因多样性指数(Nei)为0.234 5~0.262 5、Shannon’s 信息指数(I)为0.304 3~0.344 8。由表2还可知:无论采用针叶材料(双亲型,二倍体)还是种子胚乳材料(单亲型,单倍体),云南松遗传变异参数较为接近,表明云南松个体具有较高的杂合性。
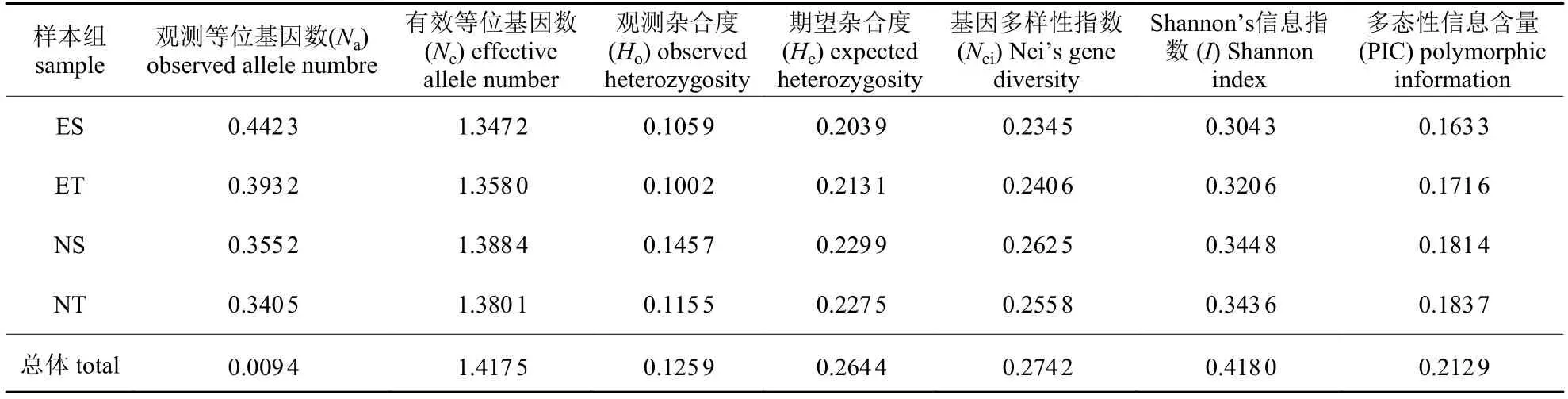
表2 不同组云南松遗传变异指标Tab.2 Genetic variation indexes of different groups of P.yunnanensis
4个样本组之间的遗传分化系数和基因流分析结果(表3)显示:组间的Fst值在0.023 2~0.143 7 之间,而不同干形针叶比较组(NS 与NT)和不同干形胚乳比较组(ES 与ET)的Fst值分别为0.107 1 和0.143 7,表明直、扭干形为中等程度遗传分化(0.05<Fst<0.15)。然而,不同组间基因交流丰富,Nm值均大于1,介于1.489 3~ 10.519 0之间。其中,直干形之间(Nm=6.363 4)及扭干形之间(Nm=10.519 0)的基因流值远高于直干形和扭干形云南松之间的基因流值(Nm针叶=2.083 9,Nm胚乳=1.489 3)。
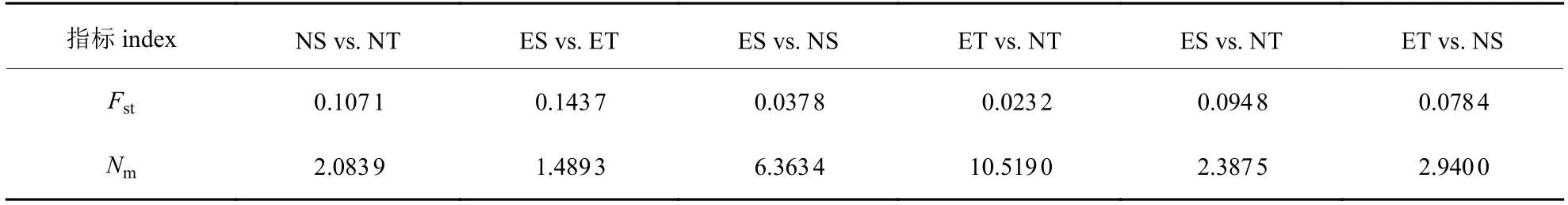
表3 云南松4个样本组间遗传分化系数和基因流Tab.3 Fst and Nm between four sample groups of P.yunnanensis
2.4 系统发育分析
采用treebest 对得到的762 699个SNP 位点构建进化树,把具有最低交叉验证错误率的分群数(K值)定义为最优分群数。当K值取值范围为1~9 时,随着K值增大,交叉验证错误率呈现下降趋势(图3a)。当K=2 时,群体分为2个类群,但直干形和扭干形样本区分不明显;当K=9 时,具有最低交叉验证错误率,样本分为9个类群(图3b)。由此可见,本研究中采集的22个云南松样本间遗传结构比较复杂。

图3 基于SNP 分析群体结构Fig.3 Analysis of population structure by SNPs
系统进化树(图4)显示:22个样本聚为2 大类,即针叶型类群和胚乳型类群。进一步分析发现:在针叶型类群中,除NT01 被聚类到胚乳组中,其余不同干形样本没有明显的分群现象;而在胚乳类群中,除ET06 样本外,不同干形的样本具有较好的分群现象。可见,采用胚乳材料进行不同干形云南松的分型比较可行。

图4 基于SNP 变异位点构建的系统进化树Fig.4 Phylogenetic tree based on SNPs
进一步以直干形样本SNP 位点的相同碱基为参照,筛选扭干形云南松样本的SNP 变异位点,共挑选出27 条序列中的39个SNP 位点。采用邻近法对39个SNP 位点绘制系统进化树,结果(图5)显示:不同干形的样本能被明确地区分,说明这些位点与云南松干形变异的发育调控相关。对这27 条序列进行数据库比对注释,仅有5 条序列被NR 和GO 数据库注释(图6),注释结果依次为多向耐药性蛋白(pleiotropic drug resistance protein 3,PDR3)、乙烯响应转录因子(ethylene-responsive transcription factor,ERF017 和RAP2-9)及MYB 相关蛋白340 (MYB-related protein 340),表明这些因子可能参与了云南松不同干形发育的调控作用。

图5 基于筛选的39个SNP 位点构建的系统进化树Fig.5 Phylogenetic tree based on selected 39 SNPs
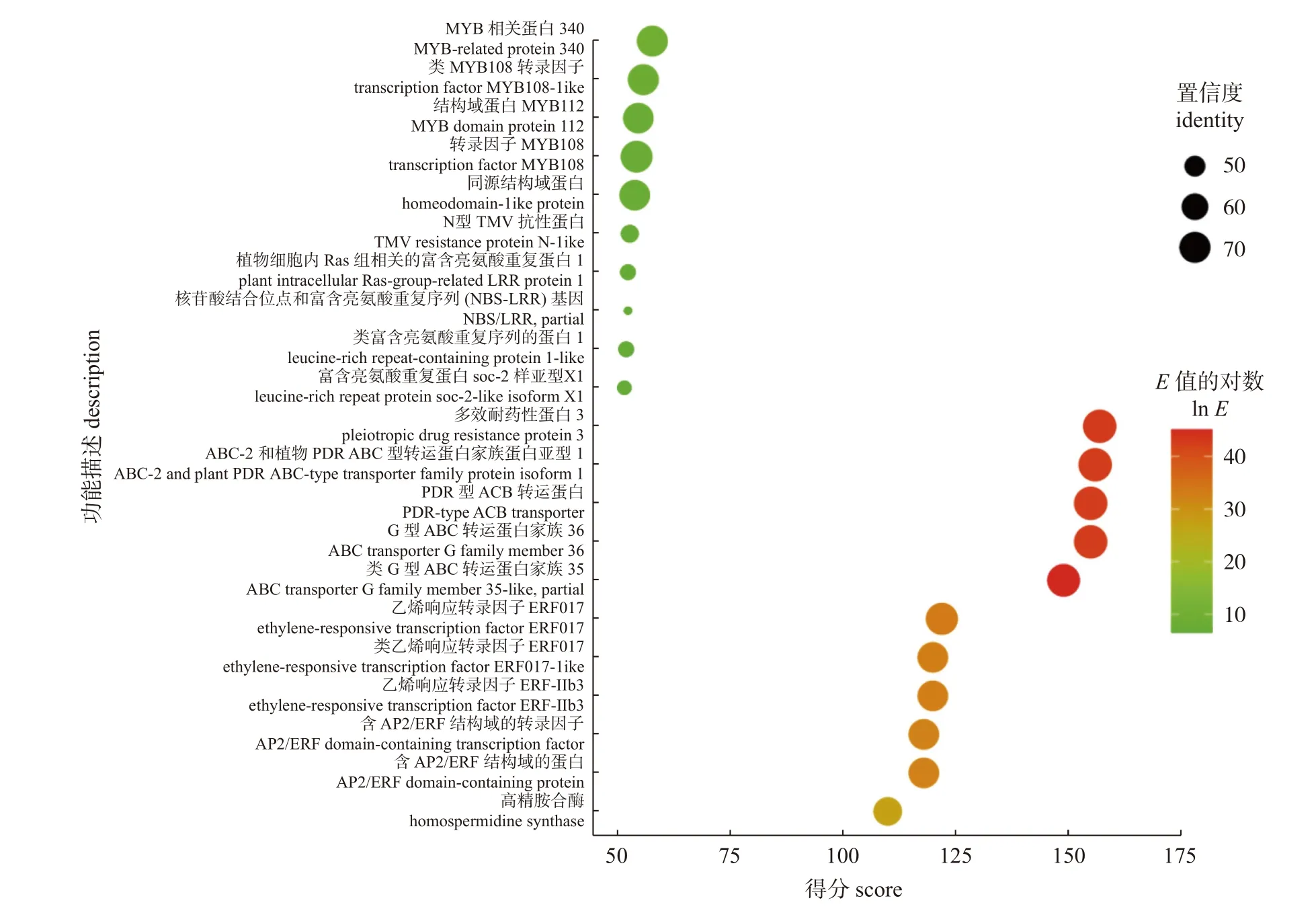
图6 含有SNP 位点的5个序列片段注释信息Fig.6 Annotation of five SNP location fragments
2.5 主成分分析
基于762 699个SNP 位点对云南松22个样本进行主成分分析,结果(图7)显示:不同干形云南松个体交错分布,没有产生明显的分群现象,表明云南松个体具有较高的杂合性,从而导致云南松干形变异的复杂性。
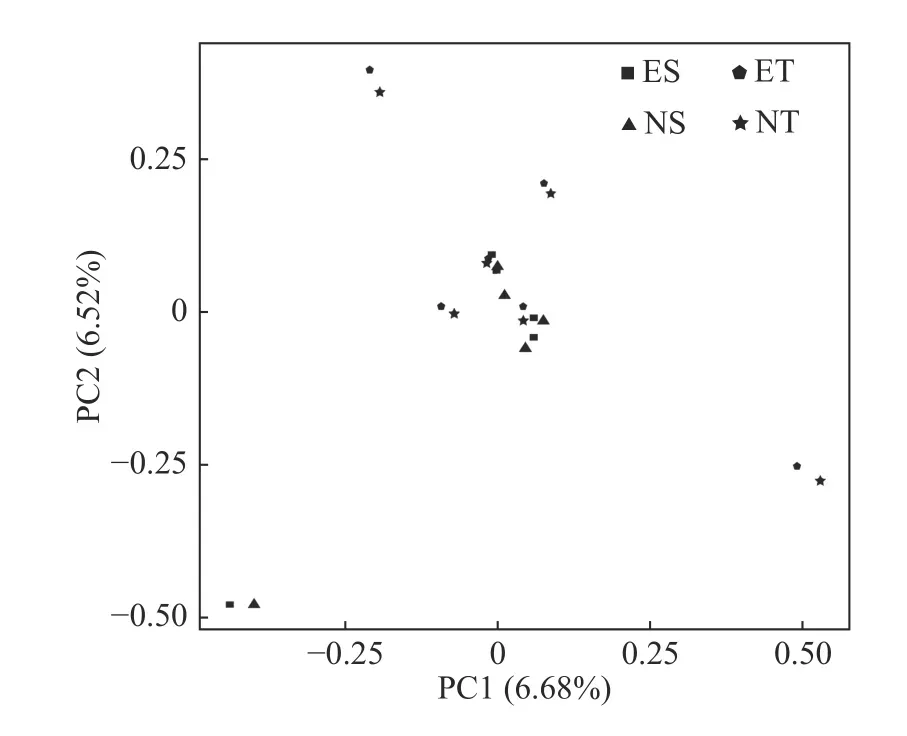
图7 22个云南松样本主成分分析Fig.7 Principal component analysis of 22 Pinus yunnanensis samples
3 讨论
简化基因组是基于二代高通量测序技术通过特定的限制性内切酶对基因组DNA 进行随机切割,降低基因组复杂性,进而展现基因组序列特征[20-22],其具有高通量、高准确性、高特异性和标记数量多等优点[23],尤其是适用于缺少相关参考基因组的物种,能有效简化基因组的复杂性。当前简化基因组技术广泛地应用于植物分子育种和遗传多样性研究[24]。段义忠等[25]基于SLAF-seq技术获得沙冬青56 295个多态性SLAF 标签,并对其遗传变异进行了分析;张序等[21]采用简化基因组技术对宽杯杜鹃进行单核苷酸多态性位点挖掘和遗传多样性分析,共开发出103 133个高质量SNP 位点,并对宽杯杜鹃的遗传多样性进行了分析。本研究分别以单倍型胚乳和二倍型针叶为材料,采用简化基因组技术开发出762 699个云南松高质量SNP 位点,表明云南松个体差异极丰富、遗传背景极复杂。
丰富的遗传多样性是一个物种应对环境变化的基础[26],观测杂合度、期望杂合度和多态性信息含量等参数被用来表征群体中遗传多样性的大小[27]。本研究共检测出SNP 位点762 699个和InDel 位点9 541个。云南松直干型胚乳组、云南松直干型针叶组、云南松扭干型胚乳组和云南松扭干型针叶组的期望杂合度介于0.203 9~0.229 9、观测杂合度介于0.100 2~0.145 7、多态性信息含量介于0.163 3~0.183 7,表明该研究中云南松样本的遗传多样性较低,这是由于本研究旨在初步揭示云南松基于简化基因组的SNP 位点分布特点,采用样本数较少。在云南松不同干形针叶组和胚乳组中,Fst值分别为0.107 1 和0.143 7 (0.05<Fst<0.15),表明直干形云南松和扭干形云南松之间存在中等程度遗传分化,该结果与遗传变异分析、系统发育分析及主成分分析结果基本一致。这一结果也与采用SRAP[28]、AFLP[29]和SSR[30-31]标记的结果基本吻合。同时,不同干形云南松之间的基因交流值均大于1,表明不同干形云南松之间不存在生殖隔离或基因交流障碍,扭干形云南松仅为云南松的一种类型。
当前云南松林中存在广泛且丰富的干形扭曲现象,其成因存在一定的争议。树木干形的生长发育与其分布的生境极其相关,干形的多样性极大地反映出林木对周边环境的适应性,同时也是进化的基础[1-2,7,32]。陈守常等[32]对四川省集中分布的云南松居群进行了长期观测调查,认为干形扭曲是云南松所具有的一种特性,林分条件(水分、土壤、年龄和疏密度)尤其疏密度是树木干形扭曲的决定性因素,而风在一定程度上也是促进和强化扭曲的外在因子。姜汉侨[33]结合云南松干形调查资料提出:树木干形扭曲是云南松的一种原有性状,具有遗传性,并认为在适宜的生境条件下,干形的扭曲度可以在一定程度上减小,而且干形的扭曲度变化是一种数量性状。SHELBORUNE 等[34]对火炬松进行了深入研究,认为树干通直度可遗传,直干型树木繁殖所得后代大部分也是树干通直,其遗传力为0.39。周蛟等[35]对云南松的遗传改良试验表明:优良林分速生林生长量遗传力为0.76,遗传增益为0.36,其子代种子可以改变云南松林分中树干弯曲及木材纹理扭曲的不良特性。由于云南松为雌雄同株植物,存在自花授粉和异花授粉,从而导致云南松林的个体之间存在较大差异[36]。同时,云南松花粉在风力作用下的水平传播距离可达数百公里,同时在垂直面上还有大量扩散[37]。当直干形和扭干形花粉或种子通过风落入同一林分时,就可能出现同一林分中直、扭干形共存现象。本研究材料采集于直干形和扭干形云南松共存的同一林分内,且部分不同干形云南松样本相邻分布。基于针叶材料开发获得的SNP 位点不能有效地区分直、扭干形云南松,但基于胚乳材料开发得到的SNP 位点能够较好地将2 种干形云南松分型。此外,对筛选出包含39个SNP 位点的27 条序列注释表明:虽然仅有5 条序列被注释,但注释预测的乙烯响应转录因子ERF017 和RAP2 均属于AP2/ERF植物转录因子家族,可通过响应乙烯、细胞分裂素、生长素及脱落酸和油菜素内酯等内源激素直接或间接调控植物生长发育及抗逆性[38-39]。基于此,认为云南松干形扭曲变异主要受遗传因素调控,但环境因素的协同作用增强了该特征的显著性。
4 结论
直、扭2 种干形云南松之间存在较为频繁的基因交流,但不同干形云南松间存在中度遗传分化,且个体差异极丰富、遗传背景极复杂。基于针叶材料开发得到的SNP 位点不能有效区分直、扭干形云南松,但基于胚乳材料开发得到的SNP 位点能够较好地将2 种干形云南松分型;对筛选出的包含39个SNP 位点的27 条序列注释分析表明:仅有5 条序列被注释到多向耐药性蛋白(PDR3)、乙烯响应转录因子 (ERF017,RAP2)及MYB 相关蛋白340 (MYB-related protein 340),可能在云南松直干形和扭干形的发育中发挥着调控作用,故认为云南松干形扭曲变异主要受遗传因素调控,但环境因素的协同作用增强了该特征的显著性。
