甜菜盐胁迫应答基因的鉴定与功能分析(Ⅰ)
2022-04-01李俊良
李俊良
(哈尔滨工业大学)
0 前言
植物为适应环境的变化需要不断地调整自己的生长及生命进程来应对这种变化。这种适应过程受到基因表达的动态调控,同时相关调控基因的表达水平通常也会随着环境的变化而实时改变。与生理水平的研究进展相比,作物在基因水平上对盐胁迫应答的分子机制研究可谓是突飞猛进[1]。表达谱测序是一种可以进行转录鉴定和表达定量的高通量测序方法,广泛用于研究基因在转录水平的表达差异。本研究使用表达谱测序技术并结合WGCNA方法从转录水平分析甜菜对盐胁迫应答的分子机理。
1 材料与方法
1.1 生物材料培育
实验选用了本课题组选育的适应性强/品质性状优良特别是耐盐性较强的栽培甜菜“O68”做为实验材料。将催芽后的甜菜种子定植于定植棉中,定植棉放置在育苗盘上,定期加入适量水,待子叶完全展开后将定植棉插入定植篮后移入培养槽,培养槽内装入以1/2浓度的改良的霍格兰营养液为培养基质,水培法培育甜菜幼苗至三对真叶期。培育条件设置为光照(16 h;24℃)/黑暗(8 h;18℃)。培养基质更换周期为3天。
1.2 盐胁迫处理与样品收集
选取长势一致的3对真叶期幼苗分为5组:(1)使用正常培养基质继续培育72 h后取样作为空白对照;(2)使用正常培养基质培育60 h后移入添加了300 mM NaCl的培养基质中继续培育12 h后取样作为盐处理12 h组;(3)使用正常培养基质培育48 h后移入添加了300 mM NaCl的培养基质中继续培育24 h后取样作为盐处理24 h组;(4)使用正常培养基质培育24 h后移入添加了300 mM NaCl的培养基质中继续培育48 h后取样作为盐处理48 h组;(5)直接使用添加了300 mM NaCl的培养基质中培育72 h后取样作为盐处理72 h组。表达谱测序使用1-5组的叶片和根共10组样品。全转录组测序和small RNA测序使用Ⅰ和Ⅱ的叶片和根作为样品。将1-5组的叶片和根样品混合后分别执行叶片和根的降解组测序实验。代谢组学使用1和2 的叶片样品。
叶片的取样部位为第3对真叶,根的取样部位为自根尖向上起约6 cm长的区域。生理指标测定所需样本经PBS漂洗后直接使用;高通量测序所需样本经PBS漂洗后使用铝箔包裹,置于液氮中冷冻30分钟后保存于-80℃待用。
1.3 表达谱测序挖掘甜菜盐胁迫应答基因
1.3.1 RNA的提取
称取0.5 g叶片或根样本,利用TRIZOL法(Invitrogen, CA, USA)提取每个样品的总RNA。使用Bioanalyzer 2100和RNA 6000 Nano LabChip检测RNA质量,以RIN值>7.0作为合格标准。为尽可能排除植株个体差异对结果的影响,制作高质量Total RNA预混池(分别提取6株相同处理叶片或根的RNA,质检合格后进行混合)用于高通量测序文库的构建。
1.3.2 表达谱测序文库构建与测序
对于RNA-seq,取1 μg总RNA使用Poly-Toligo磁珠(Invitrogen)分离Poly(A)mRNA。纯化后,使用二价阳离子(在高温下)将mRNA裂解成小片段。然后依照mRNA Seq sample preparation kit(Illumina, San Diego,USA)方案,将打碎后的RNA片段进行反转录构建最终的cDNA文库。文库的平均插入长度为300 bp(±50 bp)。按前述构建10组RNA-seq文库,每组设置3个生物学重复,总计30个cDNA文库。然后在联川生物的Illumina NovaseqTM6000平台上进行双端测序。
1.3.3 盐胁迫应答表达基因的鉴定与表达模式分析
首先使用Cutadapt[2]去除RNA-Seq数据原始数据中的的引物/适配器污染、低质量碱基和未确定碱基。然后利用FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)对序列质量进行验证。使用Bowtie 2[3]和Hierarchical Indexing for Spliced Alignment of Transcripts (HISAT)[4]将clean reads映射到甜菜参考基因组RefBeet-1.2上(http://bvseq.boku.ac.at/Genome/Download/RefBeet-1.2/)。使用StringTie[5]计算各样本的映射读数。在最终转录组生成后利用StringTie和edgeR[6]计算样本中各转录本的表达量。FPKM(fragments per kilobase of transcript per million mapped reads)值被用来来衡量基因的表达量。使用edgeR进行差异表达分析;使用T-test检验评估差异基因的显著性;差异变动基因(differentially expression gene,DEG)的筛选阈值为p<0.05且|log2(fold change)|大于1。组间基因表达模式差异使用UpSetR[7]进行可视化展示。
1.3.4 差异表达基因的功能分析
将所有表达基因与NCBI非冗余蛋白数据库(Nr)进行Blastx比对,E值筛选阈值设置为10-5。使用GO(Gene Ontology)数据库[8]对所有表达基因进行GO注释;计算每个term的基因数量,根据超几何分布检验结果对各term进行富集分析。使用KEGG(Kyoto Encyclopedia of Genes and Genomes)[9]数据库对所有基因进行注释,根据超几何分布检验结果对各pathway进行富集分析。使用STRING数据库分析DEG编码蛋白间的互作关系[10]。
2 结果与分析
2.1 盐处理对甜菜表型的影响
在甜菜受到胁迫后通常会出现一段生长滞后,在其适应胁迫后生长逐渐恢复,但是这种恢复的生长活力往往低于正常条件[11]。甜菜幼苗在300 mM NaCl处理后其表型如图1所示:盐处理下叶片首先失水萎蔫(12 h);而处理时间的延长至(24 h)时幼叶(第三对真叶)逐渐从失水状态恢复正常;当处理时间的继续延长至(72 h)时,第一对真叶表现出部分失绿(图1a)。盐处理下根系随着处理时间的延长颜色逐渐加深,24 h时可以明显观察到根尖变黑,并且黑色区域随处理时间的延长而逐步扩大(图1b)。

(a)叶片;(b)根图1 盐胁迫对甜菜幼苗表型的影响Fig.1 Effects of salt stress on the phenotype of sugar beet seedlings
2.2 甜菜盐胁迫应答基因的鉴定
2.2.1 甜菜盐胁迫下的基因表达图谱
实验对28天苗龄的甜菜幼苗进行300 mM NaCl处理, 5个取样时间点分别为0 h、12 h、24 h、48 h和72 h。每个时间点设计3个生物学重复,最终构建了30个转录组测序文库。RNA-seq测序获得了超过13.5亿的raw reads,平均每个样本4500万个reads。经过质量控制筛选后共获得13.2亿,入选率达到97.6%的有效数据。这些Valid Data在基因组上的平均定位率为92.1%约12.2亿,其中66.8%属于Unique Mapped reads。RNA-seq数据信息、有效数据、映射比例见表1。

表1 表达谱数据概览Tab.1 Summary of Expression profile data
本实验共鉴定出27,677条表达基因,将基因表达水平量化成片段,每千碱基转录本每百万映射reads(FPKM)。使用小提琴图展示样本基因表达分布统计如图2。从图中可以明确看出有样本的测序质量较好,且各生物学重复样本间重复性较好。基于Pearson相关系数法对所有样本间的基因表达相关性进行评估见图3,结果表明各生物学重复样本间基因表达相关性极高(>0.99);同一组织内,不同处理时间点的各样本间基因表达相关性相对较高,且盐处理组之间的相关性要明显高于处理组与对照组(ck)之间的相关性。但叶片和根不同的组织间的基因表达相关性较低。
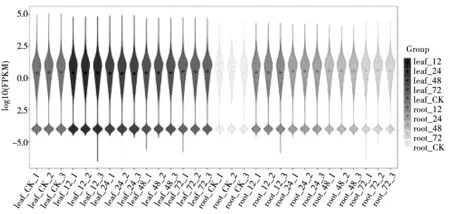
图2 RNA-seq所有样本的基因表达分布统计Fig.2 Distribution of gene expression among RNA-seq samples

图3 样本间相关性分析Fig.3 Correlation analysis among RNA-seq samples
2.2.2 甜菜盐胁迫应答相关基因的鉴定
过滤掉所有样本中低丰度表达基因(FPKM<1)后,对剩余基因在盐胁迫下的表达模式进行评估。最终鉴定到11,992条差异表达基因(differentially expressed gene,DEG),其中叶片和根中的DEG分别有7,391和8,729条。对不同盐处理时间下DEG的统计分析表明,叶片和根中分别含有39.4%(2,909条)和37.1%(3,238条),仅在单一时间点呈现显著差异的DEG,表明某些基因对盐胁迫的应答存在时空特异性。叶片和根中DEG数量最高的处理时间点是12 h,其次是72 h、24 h和48 h(见图4)。DEG的染色体定位显示DEG的分布无明显的染色体偏好(见图5)。
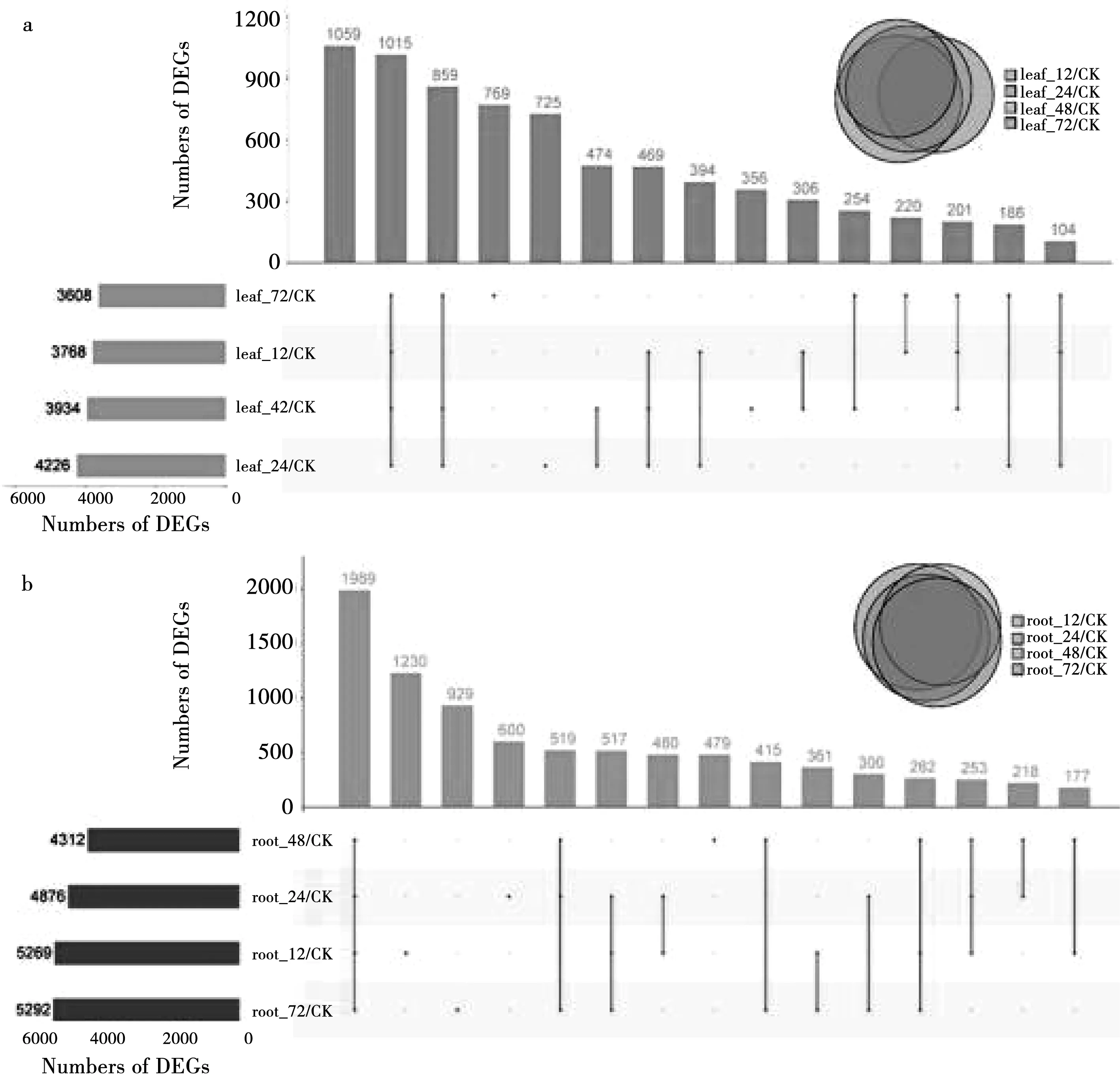
图4 叶片(a)和根(b)各样本间差异基因分布图Fig.4 Distribution of DEGs among samplesin leaves (a) and roots (b)
2.3 甜菜盐胁迫应答基因的功能分析
对DEG的GO分析获得了5,847条GO term注释,其中3,074条为生物过程、719条为细胞成分和2,054条为分子功能。细胞组排名前5的分依次是细胞、细胞区域、膜、细胞器和膜区域;生物进程排名前5的依次是细胞进程、新陈代谢进程、刺激应答、生物调控和生物进程调控;分子功能排名前5的依次是结合、催化活性、转运活性、分子结构活性和转录调控活性(图6)。
本研究利用RNA-seq技术,对盐胁迫后甜菜叶片和根进行了基因表达模式研究,在4个盐处理时间点鉴定出11,992条DEG。通过对这些DEG功能分类表明,其功能可分为三个大类:信号转导相关基因、转录调控基因、功能基因。(Ⅰ)信号转导相关基因:该类基因调控信号转导途径来传递胁迫信号并激活下游应答途径,如蛋白激酶、激素信号相关和ROS信号相关基因等;(Ⅱ)转录调控基因:该类基因通过对转录水平、转录后水平及翻译水平上的调控重构植物在盐胁迫下的基因表达系统,如可变剪接因子、转录因子和一些核酸内切酶等;(Ⅲ)功能基因:它们编码胁迫应答相关物质的合成,直接参与植物胁迫过程中生存和胁迫后恢复的一些特定过程,如光合酶、渗透调节物合成相关基因、和自由基清除相关基因等。
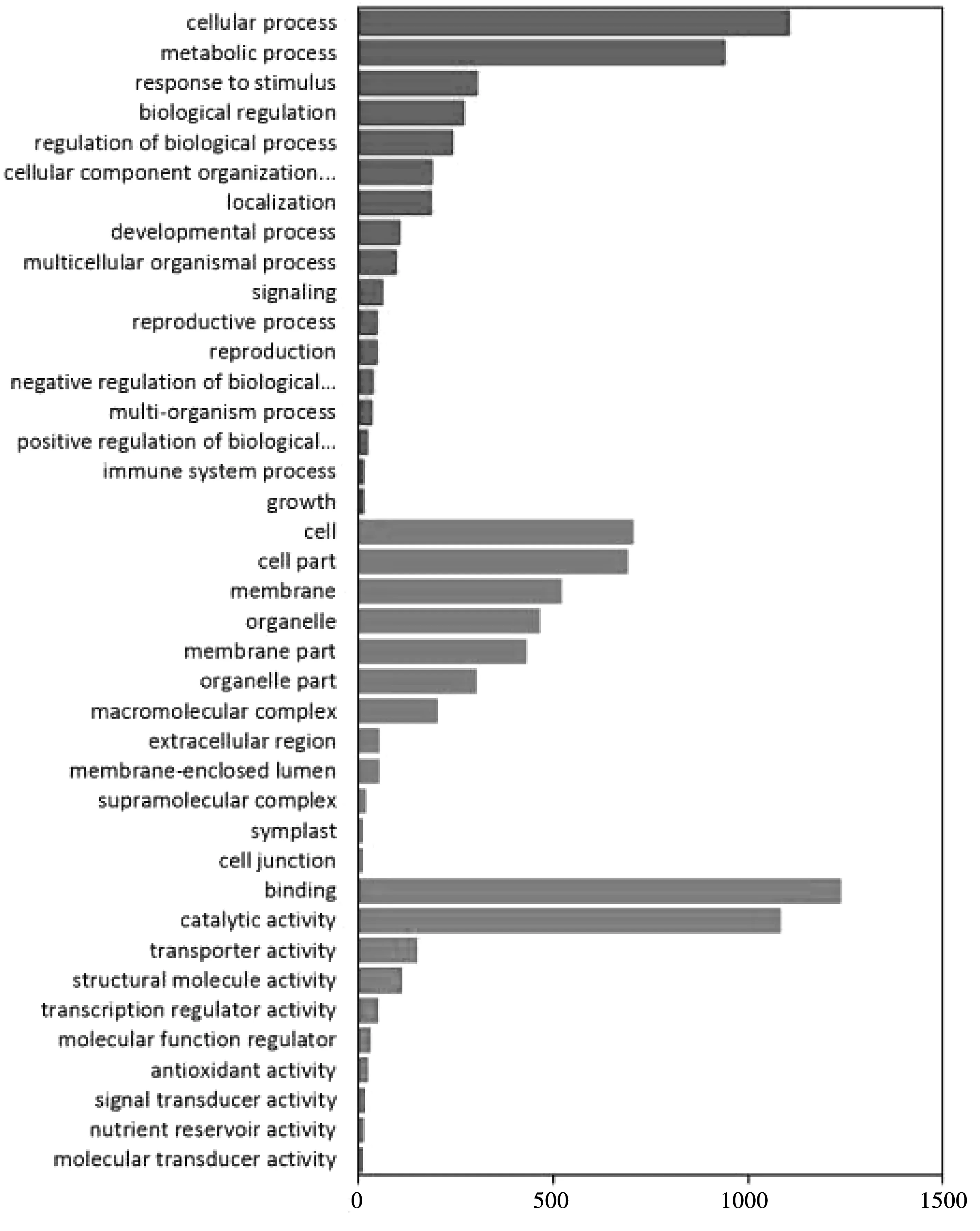
从上到下的顺序为:红色:生物进程;蓝色:细胞组分;绿色:分子功能图6 差异基因的GO注释Fig.6 GO annotation of DEGs
2.3.1 甜菜盐胁迫应答过程中的信号转导
在植物接触NaCl的初始反应中,刺激在数秒内转化为胞质Ca2+浓度变化,构成一种特殊的钙信号[12];随后Ca2+信号依次被钙依赖蛋白激酶(CDPK)、钙调蛋白(calmodulin,CaM)/ calmodulin-like 蛋白、钙调磷酸酶B类蛋白(CBLs)和膜联蛋白传递解码,进而触发下游的细胞反应[13]。
通过本研究鉴定出了300多条差异蛋白激酶编码基因,囊括了前述所有类型蛋白激酶:如编码CDPK 26的Bv9_215750_yqnm、编码calmodulin-like protein 2的Bv2_035110_kyjk、编码CBL相作丝氨酸/苏氨酸蛋白激酶的Bv7_157470_urmk、以及编码膜联蛋白RJ4的Bv2_041310_rnso等。Na+/H+逆转运蛋白(NHX)是植物盐超敏感信号通路(SOS)中的关键组分,它是植物细胞膜上发现的唯一能够把胞质中Na+排除到细胞外的蛋白。本研究中发现了盐胁迫下显著上调的两个NHX基因:Bv1_006770_xkmw和Bv_07430_jkji,它们可能在盐胁迫信号转导过程中起到重要作用。
胞质Ca2+浓度的提高还会刺激ROS和ABA的产生,在细胞水平上可以激活盐过度敏感(SOS)通路,并排除多余的Na+来进而调控离子稳态[14]。其中ROS信号的传播依赖于呼吸爆发氧化酶同源物的激活,该同源物由乙烯、ABA依赖蛋白激酶和磷脂酸(PA)联合控制。本研究中在盐胁迫过程中编码呼吸爆发氧化酶同源蛋白A(Bv5_099740_zpio)显著上调,可能在叶片ROS信号转导过程中发挥至关重要的作用。细胞内ABA的快速累积依赖于双加氧酶对类胡萝卜素前体的裂解,在盐胁迫下甜菜中的Bv_01350_texx和NCED5(Bv_004050_xzzo)两个9-顺式-环氧类胡萝卜素双加氧酶基因NCED1显著上调。我们认为盐胁迫下诱导甜菜ABA合成。Ca2+或ROS均可调节ABA的释放,进而引导下游离子通道的打开或闭合、渗透保护剂的合成以及转录因子的表达等等。此外,一些ABA和乙烯代谢及信号转导相关基因如编码PP2C家族成员的PP2C8(Bv3_052290_rcph)、PP2C24(Bv7_173230_chqm)和PP2C56(Bv8_197440_jnqo);编码玉米黄质环氧酶的Bv_003210_fkxn、以及编码乙烯生物合成途径关键酶1-氨基环丙烷-1-羧酸氧化酶基因(ACO)的Bv8_185300_wndh和Bv1_004040_gcto等均在盐胁迫下显著上调。
2.3.2 甜菜盐胁迫应答过程中转录调控
转录因子可以与靶基因5′ORF区域目标序列特异性性结合,从而调控靶标基因在特定的时间和空间以特定的强度表达。它通过调控一个复杂的网络而改变细胞内的转录组,从而在植物适应环境过程中发挥核心作用。本研究鉴定的500多条转录因子(图7)包含了来自DEG中的53个家族。排名Top5的家族依次是MYB、AP2-EREBP、bHLH、MYB-related和NAC。其中的39个家已经族被植物转录因子数据库Plant Transcription Factor Database 4.0(http://planttfdb.cbi.pku.edu.cn/index.php?sp=Bvu)中的甜菜子数据库收录。此外,本研究鉴定到14个甜菜中存在的转录因子家族还未被该数据库收录如mTERF、ABI3/VP1、LOB、OFP等。
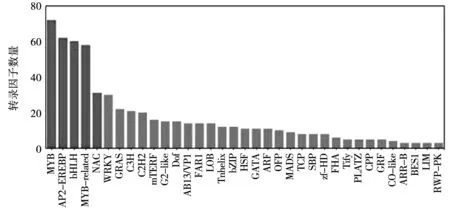
图7 差异基因中的转录因子分布Fig.7 Transcription factor distribution in DEGs
DEG对盐胁迫的应答通常可以分为两种类型:ABA依赖/非ABA依赖型。非ABA依赖基因启动子中通常包含脱水反应元件/C-重复序列(DRE/CRTs),并受AP2-EREBP转录因子的调控。本研究共鉴定出73条AP2-EREBP家族转录因子,其中绝大部分在盐胁迫下差异表达,对该家族成员的染色体定位分析发现它们广泛分布于甜菜的9条染色体上(图8),可能与甜菜非ABA依赖型盐胁迫应答基因的激活密切相关。例如,AP2-EREBP家族的两个脱水反应元件结合蛋白DREB 1(Bv3_066590_ignp)和DREB 2(Bv2_044600_drzk)在盐胁迫下显著上调。另一类依赖于ABA介导的盐胁迫应答基因,通常在其启动子区域包含一个保守的ABA应答顺式作用元件(ABRE)。ABRE可以被含有基础亮氨酸拉链结构(basic leucine zipper structure, bZIP)的转录因子识别。图7是本研究中发现的编码bZIP家族成员的DEG。如:Bv3_050990_kqjn、Bv9_214630_fnpa、Bv1_013750_smoy等。
Dicer-like(DCL)蛋白是双链RNA(dsRNA)特异性的核糖核酸内切酶,在双链RNA或带茎环结构的单链RNA(如miRNA前体)切割过程中发挥关键作用[15]。本研究发现了7个盐胁迫下差异表达的甜菜DCL基因,如Dicer同源蛋白1(Bv1_001950_imjw)。Argonaute蛋白直接与小RNA结合,并在所有小RNA介导的基因沉默过程中发挥重要作用,此外有研究表明Argonaute蛋白还与染色质修饰和可变剪接相关[16]。本研究发现了6个盐胁迫下差异表达的甜菜Argonaute基因。上述结果表明,在多种转录调控途径之间存在相互作用,构成了盐胁迫下复杂的转录调控网络。

图8 甜菜差异表达AP2/ERF家族成员染色体分布Fig.8 Chromosome distribution of DE AP2/ERF family members in sugar beet
2.3.3 甜菜盐胁迫应答过程中的功能基因
植物适应盐胁迫的第一步是解决环境中高盐度引起的渗透胁迫,甜菜碱作为细胞内的渗透保护剂在这一过程中起着至关重要的作用。编码甜菜碱合成的两个关键酶基因胆碱单加氧酶(Bv6_146100_wdro)和甜菜碱醛脱氢酶(Bv5_116230_ntjn)在盐胁迫下显著上调。水通道蛋白也是一种响应盐胁迫的功能蛋白。编码甜菜水通道蛋白PIP2-1的基因Bv7_162410_cqnr在盐胁迫下表现为叶片和根中均显著上调。超氧化物歧化酶(SOD)和过氧化物酶(POD)经常用来评估植株的抗逆性,编码甜菜Gu/Zn-SOD基因Bv_08010_smke、编码Fe-SOD基因Bv5_097450_sxsu和编码Mn-SOD 基因Bv2_045060_jxsu在盐胁迫下表达上调。根中编码POD 66的Hub基因Bv3_066080_qmgn也在盐胁迫下显著上调。硫氧还蛋白(thioredoxin)和谷氧还蛋白(glutaredoxin)在维持植物细胞的氧化还原状态中也起着重要作用[17]。本研究鉴定了16条编码硫氧还蛋白的DEG和10条编码谷氧还蛋白的DEG。包括叶中编码F型硫氧还蛋白的Hub基因Bv4_075150_andd。另外,本研究发现了25条盐胁迫下差异表达的甜菜GST基因,其中甜菜叶片中的Hub基因Bv4_095830_aore。ASA-GSH循环是植物清除ROS的一个重要的途径。谷胱甘肽过氧化物酶(GPX)是该系统的关键酶。本研究中编码GPX的基因Bv5u_121470_tcou的表达受盐胁迫的显著诱导。
叶绿体是对盐胁迫最敏感的细胞器。参与光系统Ⅰ光合电子传递的质子梯度调节蛋白5编码基因Bv3_048340_odeme、参与光反应调控的STN7蛋白激酶编码基因Bv6_155590_ugjw等直接参与光合作用的基因在盐胁迫下显著上调。
2.4 甜菜盐胁迫应答core基因的筛选与功能分析
部分DEG在甜菜叶片或根的表达明显不同具有时空特异性,而有部分DEG在叶片和根中的所有盐处理时间点下均呈现显著差异表达。即这些盐胁迫应答基因的表达不具有明显的时空特异性,这类DEG可能构成了甜菜对盐胁的基础应答,本研究将中将这类DEG归为core基因。我们通过交叉比较了叶片和根中各处理时间点之间的DEG,最终鉴定出244条core基因,其中206条在叶片和根之间的表达趋势相同,38条表达趋势相反(图9)。

图9 基础应答基因表达模式分析Fig.9 Expression pattern analysis of core DEGs
对core基因的GO分析显示:超过90%的core基因都可以注释到氧化还原进程(GO:0055114);超过80%的core基因都可以注释到转录调控,DNA-模板(GO:0006355);此外许多core基因被注释到与胁迫应答直接相关的生物进程如缺水响应(GO:0009414)、脱落酸应答(GO:0009737)、氧化应激响应(GO:0006979)、盐胁迫响应(GO:0009651)和渗透胁迫响应(GO:0006970)等(图10a)。分子功能注释显示core基因主要执行蛋白结合(GO:0005515)和金属离子结合(GO:0046872)等功能。KEGG分析显示core基因主要富集在淀粉和蔗糖代谢与植物激素信号转导通路中(图10b)。
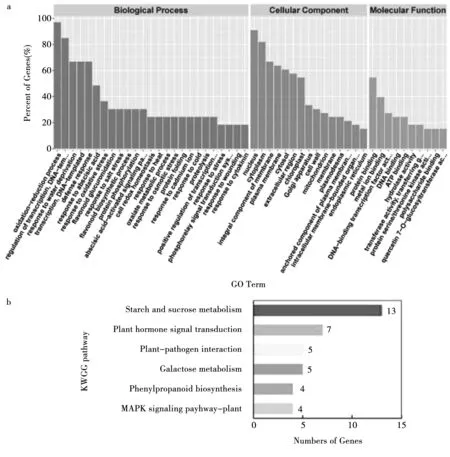
图10 基础应答基因的GO注释(a)和KEGG分析(b)Fig.10 GO and KEGG analysis of core DEGs
对所有core DEG的功能进行分析后发现,盐胁迫下叶片和根中的蔗糖合成酶(Bv7_163460_jmqz)和β-淀粉酶1(Bv_005770_atmm)显著上调。研究显示许多植物在非生物胁迫下会积累蔗糖合成酶和β-淀粉酶1,糖代谢产物不仅在渗透调节中发挥重要作用,而且在光合作用受限时也发挥着供应能量和碳源的作用。植物在感知环境胁迫后,通常会牺牲生长并激活保护性胁迫反应。ABA在胁迫信号整合和下游反应激活过程中发挥重要作用。3个ABA相关core DEG(Bv3_052290_rcph、Bv4_087620_atdx和Bv7_173230_chqm)在盐胁迫下显著上调。Bv3_052290_rcph和Bv7_173230_chqm编码两个蛋白磷酸酶PP2C(protein phosphatase 2C)家族成员,可以作为负调控因子在ABA信号转导中发挥重要作用[18];另外一项研究显示盐胁迫下拟南芥PP2C也可以与AtHKT1相互作用,调节Na+的分布[19]。此外,本研究还发现苯丙素和黄酮醇相关core DEG在叶片和根中表达均上调。非生物胁迫条件下可能激活苯丙素和黄酮醇生物合成途径,通过累积各种酚类化合物清除有害ROS[20]。总之,这些core DEG通过调控甜菜的蔗糖-淀粉代谢、调节植物激素信号通路、激活苯丙素和黄酮醇代谢通路来响应盐胁迫。我们认为对这些代谢途径的调控构成了甜菜对盐胁迫的基础应答。
2.5 甜菜响应盐胁迫的hub基因鉴定与生物信息学分析
关键基因(hub gene)指在某些生物学进程中起至关重要作用的基因,在一些相关通路中,其他基因的调控往往会受到该基因的影响[21]。因此,hub gene 通常可以作为重要的作用靶点。目前可以通过共表达或蛋白相互作用构建互作网络,然后根据网络拓扑结构筛选关键基因。
2.5.1 甜菜DEG共表达网络构建
本研究依据WGCNA算法分别利用叶片和根中DEG的表达值(FPKM值)构建共表达模块。最终分别将叶片和根中的DEG划归为11个共表达模块。无法划归任何模块的基因均统一划归为灰色模块,本研究中叶片的11个模块不包含灰色模块,而根中的11个模块包含灰色模块,该模块拥有28个DEG,占根中DEG的0.32%。继续使用WGCNA算法构建模块与处理时间的对应关系(图11)。

(a)叶片;(b)根图11 模块-样本相关性分析Fig.11 Module-sample correlation analysis
通常与样本具有较高Spearman相关系数的模块也是与处理时间最相关的模块。依据P值最终在叶片和根中分别筛选了5个和4个符合标准的模块作为关键模块进行进一步分析。其中叶片中的5个模块分别是:对应0 h的包含2181条DEG的蓝绿色模块 (Module Turquoise)、对应12 h的包含1115条DEG的棕色模块(Module Brown)、对应24 h的包含58条DEG的黄绿色模块(Module Greenyellow)、对应48 h的包含121条DEG的紫色模块(Module Purple)以及对应72 h的包含1638条DEG的蓝色模块(Module Blue)(图11a)。根中的4个模块分别是:对应0 h的包含2501条DEG的蓝绿色模块(Module Turquoise)、对应12 h包含1038条DEG的黄色模块(Module Yellow)、对应24 h的包含980条DEG的绿色模块(Module Green) 以及对应于72 h的包含2052条DEG的蓝色模块(Module Blue)(图11b)。根中48 h无对应的显著相关(P<0.1)模块。
2.5.2 关键模块基因表达模式分析
表达模式分析显示关键模块间的基因表达存在显著差异,各模块内基因均在模块关联的时间点处呈现显著差异表达(图12)。例如叶片Brown模块内基因在盐处理12 h时呈现显著差异表达(图12a),根 Yellow模块内基因在盐处理12 h时显著差异表达(图12b)。总的来看,对照组关联模块(Turquoise)的大部分基因表达被盐胁迫抑制,处理组关联模块的大部分基因表达受盐胁迫诱导。
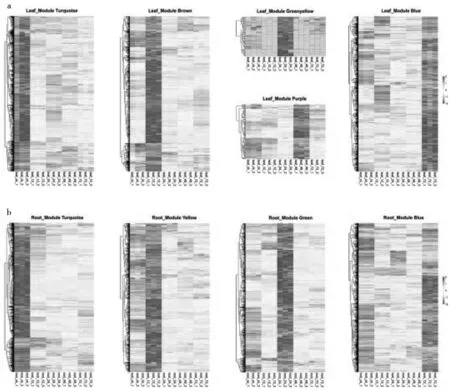
(a)叶片中的5个关键模块;(b)根中的4个关键模块图12 每个关键模块的基因表达模式Fig.12 The expression profiles of genes in each critical module
2.5.3 关键模块基因功能分析
叶片中关键模块基因的GO富集分析显示(图13):盐胁迫下Turquoise模块内富集度较高的生物进程是翻译(GO:0006412)和核糖体大亚基组合(GO:0000027);富集度较高的的细胞组分是叶绿体基质(GO:0009570)和胞质大核糖体亚基(GO:0022625);富集度较高的分子功能是核糖体的结构组成部分(GO:0003735)。Brown模块内基因富集度较高的生物进程是乙烯激活信号通路负调控(GO:0010105);富集度较高的细胞组分是细胞核(GO:0005634);富集度较高的分子功能是钙离子结合(GO:0005509)。Greenyellow模块内基因主富集度较高的生物进程是RNA聚合酶Ⅱ对转录的调控(GO:0006357)和磷酸化(GO:0016310);富集度较高的细胞组分是核仁(GO:0005730);富集度较高的分子功能是RNA聚合酶Ⅱ调控的序列特异性DNA结合(GO:0000977)。Purple模块内基因富集度较高的细胞组分是中央叶泡(GO:0042807);富集度较高的分子功能是蛋白二聚活动(GO:0046983)。Blue模块内基因富集度较高的生物进程是蛋白酶体介导的泛素依赖的蛋白降解过程(GO:0043161)和蛋白水解相关细胞蛋白分解代谢过程(GO:0051603);富集度较高的细胞组分是细胞外空间(GO:0005615)和溶酶体(GO:0005764);富集度较高的分子功能是半胱氨酸型肽链内切酶活性(GO:0004197)。
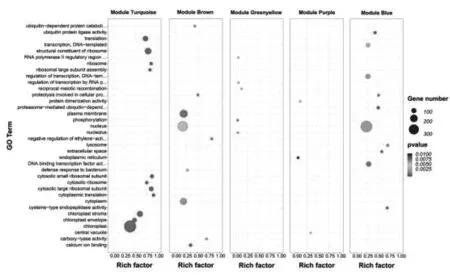
图13 叶片关键模块基因的GO富集分析Fig.13 Go enrichment analysis of critical module genes in leaves
根中关键模块基因的GO富集分析显示(图14):盐胁迫下Turquoise模块内富集度较高的生物进程是草酸代谢过程(GO:0033609)、细胞表面受体信号通路(GO:0007166)和防御应答(GO:0006952);富集度较高的细胞组分是膜的整体构件(GO:0016021)、细胞壁(GO:0005618)和液泡膜(GO:0005774);富集度较高的分子功能是ATP结合(GO:0005524)、蛋白丝氨酸/苏氨酸激酶活性(GO:0004674)和草酸脱羧酶活性(GO:0046564)。Yellow模块内富集度较高的生物进程是盐胁迫应答(GO:0009651)和类黄酮生物合成(GO:0009813);富集度较高的细胞组分是胞质(GO:0005829);富集度较高的分子功能是槲皮素3-O-葡糖基转移酶活性(GO:0080043)和槲皮素7-O-葡糖基转移酶活性(GO:0080044)。Green模块内富集度较高的生物进程是植物型次生细胞壁发生(GO:0009834)、木质素生物合成(GO:0009809)和脂肪酸生物合成(GO:0006633);富集度较高的细胞组分是膜的整体构件(GO:0016021)和细胞外区域(GO:0005576);富集度较高的分子功能是过氧化物酶活性(GO:0004601)、血红素结合(GO:0020037)和作用于酯键的水解酶活性(GO:0016788)。Blue模块内富集度较高的生物进程是DNA模板转录负调控(GO:0045892),DNA模板转录正调控(GO:0045893)和脱落酸应答(GO:0009737);富集度较高的细胞组分是细胞核(GO:0005634);富集度较高的分子功能是核酸结合(GO:0003676)。
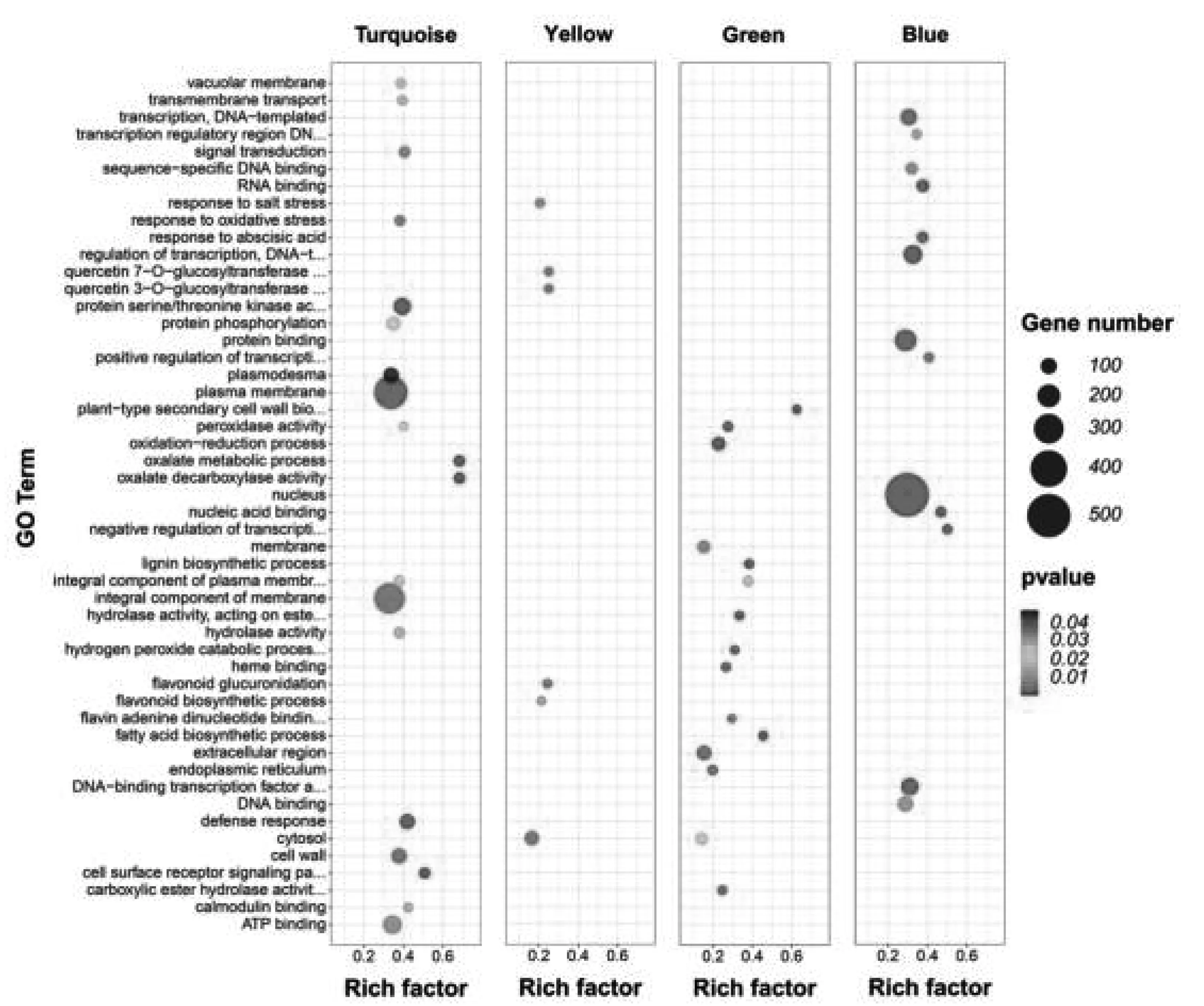
图14 根关键模块基因的GO富集分析Fig.14 Go enrichment analysis of critical module genes in roots
叶片中关键模块的基因KEGG富集分析显示(图15a):Turquoise模块基因富集度较高的通路是核糖体(ko03010)和脂肪酸生物合成(ko00061);Brown模块基因富集度较高的通路是精氨酸和脯氨酸的代谢(ko00330)和内吞作用(ko04144);Greenyellow模块基因富集度较高的通路是光合作用生物中的碳固定(ko00710)和植物-病原互作(ko04626);Blue模块基因富集度较高的通路是植物激素信号转导(ko04075)和其他类型的邻聚糖生物合成(ko00514)。根中关键模块的基因KEGG富集分析显示(图15b):Turquoise模块基因富集度较高的通路是植物-病原互作(ko04626)和RNA聚合酶(ko03020);Yellow模块基因富集度较高的通路是谷胱甘肽代谢(ko00480)和花生四烯酸代谢(ko00590);Green模块基因富集度较高的通路是苯丙素的生物合成(ko00940)和丙酮酸代谢(ko00620);Blue模块基因富集度较高的通路是RNA运输(ko03013)和mRNA监测通路(ko03015)。
总的来看,使用WGCNA法对DEG进行归类后,各关键模块基因的表达模式和功能均存在高度的特异性,符合我们的预期目标。有助于从大量DEG之间挖掘关键耐盐基因,探寻甜菜应对盐胁迫的分子机理。
2.5.4 盐胁迫应答Hub基因的鉴定
本研究首先利用加权基因共表达网络计算模块内基因的连通度,通常连通度值越高的基因越可能是潜在的Hub基因。然而当连通度值极度相近的情况下(如叶片Turquoise模块中Bv5_105240_scsk和Bv1_016890_iiqe的连通度值分别为1140.9和1140.7),以连通度值作为唯一判定标准是不够科学的。因此我们选择模块内连通度值前20 %的基因作为候选Hub基因,利用STRING构建这些基因编码蛋白的互作网络;然后利用CytoHubba中的11种算法分别评估各模块内的top10 Hub基因;最后以候选基因在11种算法top10 Hub基因中的出现频次联合基因的连通度值为每个模块筛选出6个最可能的Hub基因。
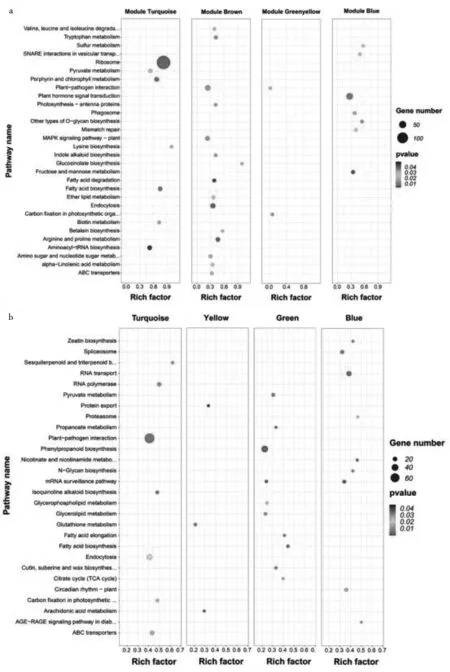
图15 叶片(a)和根(b)中关键模块基因的KEGG富集分析Fig.15 KEGG enrichment analysis of critical module genes inleaves (a) and roots (b)
2.5.4.1 Hub基因在叶片中的鉴定与功能分析
叶片中筛选出的Hub基因见表3-2。盐胁迫下,Turquoise模块内大部分基因的表达呈现下调趋势。Hub基因Bv2_041930_qfdi编码的胆色素原脱氨酶是植物叶绿素生物合成过程中的关键酶,甜菜叶片遭受盐胁迫后叶绿素含量显著降低可能与受到该Hub基因网络的调控。Hub基因Bv1_000340_ezio编码叶绿体30S 核糖体蛋白、Hub基因Bv2_036440_xjzx编码40S核糖体蛋白、Hub基因Bv5_110020_fucg编码mRNA结合伴侣ACD11,这些基因参与mRNA的结合及后续加工过程,它们的下调与盐胁迫后生长发育受阻相符,这些Hub基网络可能参与调控植株从生长向应激状态的转换。Hub基因Bv2_029170_pxtq编码环阿屯醇-C-24-甲基转移酶(SMT),它是植物甾醇代谢途径中的关键限速酶,从图16可以看出,抑制该基因的表达可以提高植物胆甾醇的累积,阻碍了环阿屯醇前体流向豆甾醇合成分支,促进植物甾体类化合物流向其他合成分支方向(如油菜素甾醇),对拟南芥SMT基因的早期研究支持了这种推断[22]。
Brown模块Hub基因Bv2_023810_guyf编码DExH-box ATP依赖RNA解旋酶DExH 17,最新研究显示该基因调控核酸-蛋白质相分离为细胞提供了各种RNA处理步骤的时空控制,通过引导RNA分子进入相分离的区室,调节RNA成熟状态、核糖核蛋白复合体(RNP)的组成并最终调节RNA命运[23]。

表2 叶片关键模块中鉴定的Hub基因Tab.2 The Hub genes detected in critical modules in leaves
本研究中该基因在盐胁迫后显著上调并在12 h时达到峰值,说明以它为核心的基因网络可能通过参与早期盐胁迫应答基因的加工处理为甜菜盐胁迫应激过程做出贡献。Hub基因Bv6_130450_umcu编码叶绿体早期脱水响应蛋白。Hub基因Bv5_099740_zpio编码呼吸爆发氧化酶同源蛋白A参与ROS信号的转导。Hub基因Bv6_149930_qmfq编码莽草酸激酶,莽草酸是芳香氨基酸、生物碱和吲哚衍生物等的前体,该酶通过催化莽草酸的磷酸化参与调控莽草酸代谢,已有研究显示莽草酸途径与水稻的盐胁迫应答过程相关[24]。

绿色:甜菜中存在组件;黄色:DEG;红色边框:植物甾醇代谢图16 盐胁迫下甜菜类固醇生物合成通路分析Fig.16 Pathway analysis of steroid biosynthesis in sugar beet under salt stress
Greenyellow模块受样本总量限制只筛选3个Hub基因。Bv4_074970_andd编码叶绿体中F型硫氧还蛋白,Bv4_095830_aore编码GST蛋白,以他们为核心的Hub基因网络可能为细胞的抗氧化过程做出巨大贡献。
叶片Purple模块Hub基因Bv1_016400_jjoj编码叶绿体中的伴侣蛋白dnaJ 11;Hub基因Bv4_086990_pgms编码泛素连接酶E2 26;以它们为核心的基因网络通过调控蛋白质的加工参与盐胁迫应答。两个Hub基因编码2种转录因子bHLH 35和锌指蛋白22。Hub基因Bv_005490_qmph编码磷酸代谢蛋白8;Hub基因Bv7_172340_kzsr编码非特异性磷脂酶C1(NPC),该酶可以催化磷脂酰胆碱(PC)水解产生甘油二酯(DAG),DAG再次代谢后生成磷脂酸(PA),PA是细胞内重要的第二信使,可以激活多种胁迫应答途径。以它们为核心的基因网络可能通过PLC-DGK/PA途径[25]参与盐胁迫信号的转导与应答。
叶片Blue模块Hub基因Bv5_118050_deeh编码一种E3泛素蛋白U-box结构域的蛋白52。Hub基因Bv6_139660_wjii编码亮氨酸拉链蛋白ATHB-40;hub基因Bv6_133290_wkjd编码翻译起始因子IF-2;以他们为核心的基因网络可能通过调控转录和翻译过程响应盐胁迫应答。Hub基因Bv2_042060_ccft编码胆碱激酶2,以该基因为核心的网络最终也可能通过PLC-DGK/PA途径参与盐胁迫信号的转导与应答。胁迫72 h后的甜菜老叶呈出萎蔫与失绿的性状,说明长时间的盐胁迫已经对甜菜幼苗的存活造成一定影响,Bv2_033120_umqq编码液泡加工酶(VPE);以VPE为核心的基因网络可能与细胞衰老相关。
2.5.4.2 Hub基因在根中的鉴定与功能分析
根中Hub基因见表3。磺化反应是机体对内源性和外源性化合物进行生物转化的关键反应。Turquoise模块Hub基因Bv2_042180_yfwq编码胞质磺基转移酶,它可以催化生物体内黄酮类化合物转化成硫酸盐而降低利用率[26],因此以该基因为核心的网络可能通过调控激素和黄酮类化合物的利用来影响甜菜的发育与抗逆。此外,一些次生代谢相关的Hub基因如编码重金属相关异戊烯基类黄酮植物蛋白的Bv_016430_xjdf和编码7-去氧番木鳖酸葡萄糖转移酶的Bv4_077620_xkxs也在受到胁迫后显著下调。这些结果说明甜菜根细胞在遭受盐胁迫后会调整次生代谢方向,调控植株从生长状态向应激状态转换以适应环境改变。另外编码包含L型凝集素结构的受体激酶、LRR受体类丝氨酸/苏氨酸蛋白激酶和包含重复的锚蛋白NPR4的3个Hub基因都与信号转导相关,这些Hub基因网络可能通过调控甜菜根系在盐胁迫下的信号转导来参与盐胁迫应答过程。

表3 根中从WGCNA核心模块中鉴定的Hub基因Tab.3 The Hub genes detected in critical WGCNA modules in roots
Yellow模块中,Hub基因Bv5_124870_ydpo编码解毒蛋白 48,以该基因为核心的网络可能与金属离子解毒相关。此外,以Bv6_129310_ndoa为核心的Hub基因网络可能通过调控依赖于ATP的离子运输辅助解毒过程。
以Hub基因Bv1_009510_hfps为核心的网络与蛋白泛素化过程相关。编码液泡氨基酸转运蛋白的Hub基因Bv2_046760_tquf在盐胁迫12时表达上调了80倍,液泡占据植物细胞的大部分体积,它参与调节细胞代谢,控制细胞内物质累积和运输,提高植物适应环境变化和生存的能力,该Hub基因网络可能通过调控氨基酸的调配过程在盐胁迫过程中发挥重要作用。Hub基因Bv3_067190_wskz编码过氧化物酶体中脂肪酸β氧化多功能蛋白MFP 2,以该基因为核心的网络可能通过调控能量代谢为甜菜根细胞盐胁迫应答供能。编码谷氨酸脱羧酶4的Hub基因Bv6_131780_uxzh在盐胁迫12时表达上调了7倍,该酶催化谷氨酸盐生成γ-氨基丁酸(GABA),许多研究显示植物在逆境胁迫下会大量累积GABA,首先GABA的合成过程会消耗大量氢离子从而维护细胞内的pH环境;其次生成的GABA可以作为小分子渗透调节物质;过量的GABA会抑制植物的生长,GABA含量的升高可以提高植物对氧化胁迫的耐受性;此外,迅速累积的GABA可能是一种内源性逆境信号分子[27-28]。因此该Hub基因网络通过调控GABA相关代谢参与甜菜根细胞对盐胁迫的应答。
Green模块Hub基因Bv2_044780_rzxx编码3-酮脂酰-CoA合酶,与内质网中的脂肪酸链的伸长相关,该Hub基因网络可能影响膜脂的不饱和度以及非双分子层脂质的比例,从而改变细胞膜的流动性来调控甜菜根系对盐胁迫的应答。Hub基因Bv1_016480_prnw编码IN2-1同源蛋白,该蛋白属于GST蛋白,以该Hub基因为核心的网络可能在根细胞非酶促抗氧化防御过程中发挥重要作用。Hub基因Bv2_043550_pccg编码ABC转运蛋白G超家族成员32。木质素是对植物结构支撑和水分运输十分重要的生物大分子,它也可以提高作物的抗逆性,编码过氧化物酶66的Hub基因Bv3_066080_qmgn可以催化木质素的生物合成。有研究显示盐胁迫会抑制根系的生长并诱导根系木质化[28-30]。以该基因为核心的网络可能通过调控根系木质素的合成响应盐胁迫。
总的来看,我们筛选出的Hub基因在生物学上具有重要意义,有助于解析甜菜盐胁迫应答的分子机理。
2.5.5 甜菜转录水平的盐胁迫应答网络构建
分别为叶片和根构建了由core基因、Hub基因和重要盐胁迫应答基因组成的转录水平盐胁迫应答网络。同时利用STRING数据库对这些基因间编码蛋白间的互作关系进行注释。在叶片中(图17),盐胁迫造成甜菜叶片生长迟滞,并导致叶绿素合成受阻,因此叶片细胞下调了大量核糖体编码基因的表达,以削减mRNA后续加工过程;同时迅速调整次生代谢物的合成流向于有利于胁迫应答的分支。在受到盐胁迫的早期(12 h和24 h),ATP依赖RNA解旋酶基因表达显著上调,调控胁迫应答相关mRNA的紧急加工;同时一些ROS信号和ROS清除相关基因的表达显著上调。在盐胁迫的中期(48 h),一些转录因子如bHLH35和锌指蛋白22编码基因显著上调,同时NPC等基因的上调可能激活PLC-DGK/PA途径调控盐胁迫应答过程。在盐胁迫的晚期(72 h),VPE等基因的上调可能与叶片细胞的衰老相关。值得注意的是,叶片中许多Hub基因均定位于叶绿体,反映出该细胞器在盐胁迫应答过程中的重要作用。此外,盐胁迫后多种泛素蛋白编码基因在各时间点特异性高表达,说明蛋白修饰过程在盐胁迫应答过程中发挥重要作用。
在根中(图18),遭受盐胁迫后根系生长受到抑制,许多受体激酶基因表达显著下调,次生代谢方向迅速发生改变。在受到盐胁迫的早期(12 h和24 h),GABA等渗透调节物质迅速累积,多种转运蛋白基因表达显著上调促进根细胞内物质的重新调配,次生代谢流向木质素生物合成代谢,诱导根系木质化;脂肪酸分解代谢加剧为细胞提供能量;此外,谷胱甘肽相关基因也在这一时期显著上调,有助于根细胞的抗氧化防御。在盐胁迫的晚期(72 h),一些转录因子如RAP2-4和ERF54、转录延伸因子AFF4和钙调蛋白编码基因显著上调。这些基因通过调控转录过程参与盐胁迫应答。
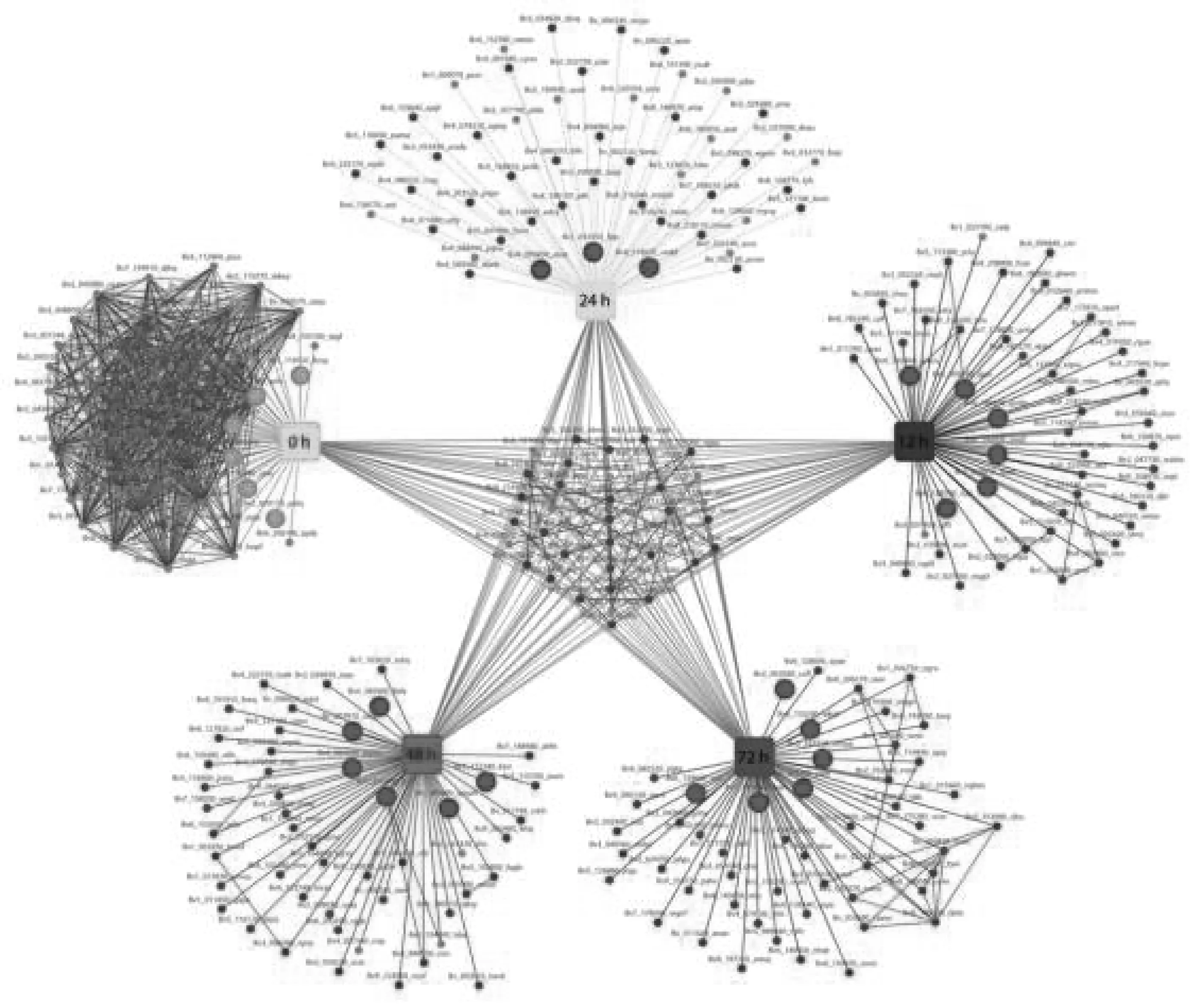
正方形代表与时间点对应的叶中关键模块;大圆点代表hub基因,core基因聚集在中心区域,红色:表达上调,绿色:表达下调;红线代表STRING数据库注释的互作关系图17 叶片转录水平盐胁迫应答网络Fig.17 Transcriptional network of leaves in responses to salt stress

菱形代表与时间点对应的叶中关键模块;大圆点代表hub基因,core基因聚集在中心区域,红色:表达上调,绿色:表达下调;红线代表STRING数据库注释的互作关系图18 根转录水平盐胁迫应答网络Fig.18 Transcriptional network of roots in responses to salt stress
2.6 qPCR检验Hub基因在盐胁迫下的表达模式
采用实时荧光定量PCR(qRT-PCR)法在叶片和根中分别随机抽选15条Hub基因进行表达模式验证。结果显示,叶片中有11条Hub基因在两种检测方法间的R2值(回归方程决定系数)大于0.9;有1条Hub基因在两种检测方法间的R2值在0.8~0.9之间;有2条Hub基因在两种检测方法间的R2值在0.7~0.8之间,仅有1条Hub基因在两种检测方法间的R2值小于0.7(图19a)。根中Hub基因的qRT-PCR定量结果与叶片相似,有9条Hub基因在两种检测方法间的R2值大于0.9;又3条Hub基因在两种检测方法间的R2值在0.8~0.9之间;有1条Hub基因在两种检测方法间的R2值在0.7~0.8之间,仅有2条Hub基因在两种检测方法间的R2值小于0.7(图19b)。
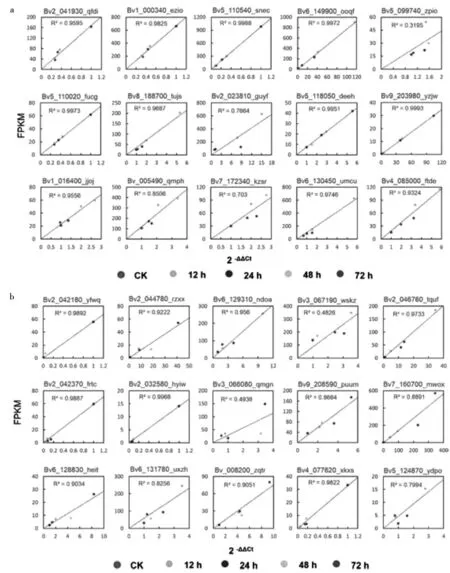
(a)叶片;(b)根;x轴为qRT-PCR结果,y轴为RNA-SEQ结果图19 Hub基因qRT-PCR和高通量测序数据比对Fig.19 Comparison betweenqRT-PCR and sequencing of Hub genes
总的来看,高通量测序检测的Hub基因的表达与和qPCR验证结果在趋势上高度一致,说明我们的测序结果高度可信。个别Hub基因在两种检测方法间存在一定差异(R2值较低)可能是由于检测原理间存在差异或目的基因具有特殊结构(如茎环)等因素造成的。
3 结论
(1)RNA-seq在甜菜中共鉴定出了11,992条盐胁迫应答基因,其中叶片和根中分别包含7391条和8729条。样本间DEG比对分析,鉴定出244条core应答基因,我们认为这些基因通过调控甜菜的蔗糖-淀粉代谢、调节植物激素信号通路、激活苯丙素和黄酮醇代谢通路来响应盐胁迫进而构成了甜菜对盐胁迫的基础应答。
(2)通过对Hub基因的表达模式进行分验证表明及功能分析显示这些Hub基因具有重要的生物学意义,有助于解析甜菜对盐胁迫应答的分子机理。
(3)本研究利用core基因、Hub基因和重要盐胁迫应答相关DEG构建了甜菜转录水平盐胁迫应答网络,通过对该网络中DEG的功能分析,从转录层面论述了甜菜对盐胁迫应答的分子机理。总之,本研究首次为甜菜构建了一个较为全面的转录水平盐胁迫应答网络。
