基于加权基因共表达网络分析探讨黄芩抗肝细胞癌的相关分子机制*
2022-03-28徐玉平蒋向辉
徐玉平,谭 荣,蒋向辉
(凯里学院大健康学院 凯里 556011)
肝细胞癌(HCC)是一种死亡率很高的原发性肝癌,发病率居世界第六位,死亡率居世界第四位[1],其在40岁前发生率较少,而后随着年龄逐渐升高,70岁达到顶峰,男性发病率是女性的2-4倍。酒精性肝硬化、肝炎病毒感染、吸烟等是其重要的致病原因。HCC早期不易察觉,病情发展至中晚期后,常出现食欲减退、乏力、腹胀、肝区疼痛等症状,还可兼发上消化道出血、肝昏迷、肝癌破裂出血、肝肾衰竭等严重并发症。HCC恶性程度高、易转移、预后转归较差,因此,有“癌中之王”之称[2]。目前,肝细胞癌的治疗手段有手术切除、局部消融、肝脏移植、放化疗、介入栓塞术等,但存在供体有限、费用高昂、毒副作用多、易产生耐药性及肿瘤异质性强[3-5]等问题。近年来很多研究表明中药可通过多通路抗肝癌组织血管生成,抑制癌细胞增殖和转移,对肝细胞癌具有一定治疗作用[6],而且还能增强机体抵抗力、改善临床症状、缓解放化疗带来的不良反应,提高患者生活质量。
黄芩(Scutellaria baicalensisGeorgi),性寒,味苦,具有清热燥湿、泻火解毒、止血、安胎等功效,主治湿温、暑湿,湿热痞满,泻痢,黄疸,痈肿疮毒,胎动不安。研究发现,黄芩具有保肝、利胆、抗炎、抗菌、增强免疫、抗肿瘤、神经系统保护和心脑血管保护等多种药理活性[7],在荣震主任医师治疗HCC的学术经验整理研究中[8],无论是复方还是单味药治疗使用频次最高的前10味中药都包括黄芩。文献报道,黄芩汤辅助序贯肝动脉化疗栓塞术对原发性肝癌患者的治疗总有效率为67.86%,显著高于不加黄芩汤对照组的48.21%,治疗后患者的NF-κB、HIF-1α水平、AFP、肿瘤体积显著低于对照组(P<0.01),抑制了肝癌细胞的生长和扩散[9]。然而关于黄芩治疗HCC的临床研究和相关分子机制却少见报道。因此,从基因水平深入探讨黄芩抗肝细胞癌的核心靶点及其相关分子机制,对HCC的防治具有重要意义,也为黄芩及其有效成分在防治肝癌方面的深入研究、开发和临床应用提供参考。
加权基因共表达网络分析(WGCNA)是一种系统生物学方法,通过引入加权的相关系数,构建共表达的基因模块,能从高通量数据中挖掘出模块信息,描述不同样品之间的基因关联模式,根据模块的内联性和模块与表型之间的关联性鉴定候补生物标记物或治疗标靶。曾祎凡等[10]利用WGCNA技术挖掘促进乳腺癌循环肿瘤细胞转移的关键基因,发现DDX5和ACTB在循环肿瘤细胞内高表达,其中DDX5很可能是促进乳腺癌循环肿瘤细胞转移的关键基因,在临床上,DDX5可作为乳腺癌转移早期鉴别的循环肿瘤细胞标志物,也能为改善乳腺癌预后提供新的治疗靶点。由此可见,WGCNA技术广泛应用于基因靶点或疾病生物标志物的筛选之中,适用于中药生物活性成分结合高通量数据联合阐述疾病潜在的作用基因及多成分、多靶点、多通路的分子机制。
本研究基于网络药理学的方法筛选黄芩的有效活性成分及基因靶点,通过WGCNA分析TCGA和GEO中的HCC相关数据,挖掘HCC的hub基因,并对这些基因进行总生存期、无病生存期分析以及免疫组化验证,同时联合GO生物学功能富集分析、KEGG信号通路富集分析进一步探索黄芩抗HCC的相关分子机制,为后期开展黄芩防治HCC的实验和临床研究提供理论参考。
1 材料与方法
1.1 黄芩活性成分及其靶点的筛选与PPI网络构建
中药系统药理学(TCMSP)数据库收纳了在中国药典中注册的499种中草药,包含29 384种成分,3311种靶标和837种相关疾病,提供十二种与ADME(Absorption、Distribution、Metabolism、Excretion)相关的重要特性,如口服生物利用度、类药性、半衰期、Caco-2渗透性、血脑屏障等,用于药物筛选和评估[11],其中口服生物利用度(OB)、肠上皮细胞通透性(Caco-2)和类药性(DL)等被认为是中草药有效性的关键指标[12]。本研究通过TCMSP数据库(https://tcmspw.com/tcmsp.php)检索黄芩的全部活性成分及其作用靶点,以口服生物利用度(OB)≥40%、类药性(DL)≥0.18为筛选条件[11],得到黄芩主要的有效活性成分及其对应的作用靶点。再通过检索UniProt数据库(https://www.uniprot.org/)获得去重复项后的靶点蛋白的基因symbol,将靶点上传到STRING数据库(https://string-db.org/),设置“highest confidence(≥0.900)”,获得靶点蛋白的相互作用信息及其相互作用(Protein-protein interaction,PPI)网络图,接着导入Cytoscape3.6.1,利用cytoHubba插件计算MCC(中心性),筛选连接程度排名前15的基因为黄芩抗HCC候选基因靶点。
1.2 HCC数据下载
从TCGA数据库(https://portal.gdc.cancer.gov/)下载与HCC相关的424个转录组数据,其中包括正常样本50个,肿瘤样本374个,相关参数设置为:数据类别勾选转录组数据、表达类别勾选基因表达量、Workflow类别勾选HTSeq-Counts、组织类别勾选肝和肝内胆管、数据库勾选TCGA,数据集勾选样本量最多的一个数据库(即TCGA-LIHC)。
从TCGA数据库下载与HCC相关的377个临床数据,其中包括病人的生存时间、生存状态、性别、年龄、肝癌分期等数据,相关参数设置为:数据类别勾选临床数据、数据格式勾选bcr xml,组织类别、数据库、数据集与下载转录组数据时的参数一致。
从NCBI的GEO数据库下载GSE41804数据集的探针矩阵文件,该数据集含有20个HCC肿瘤样品和20个非肿瘤样品,通过基因芯片平台GPL570 [HGU133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array下载数据集GSE41804的相关注释信息文件。
1.3 HCC差异基因筛选
利用R语言(4.0.3版)对TCGA转录组数据进行整理,使用edgeR数据包筛选差异表达基因,以log|FC|>1、FDR<0.05为筛选条件,通过limma数据包获得差异基因(DEGs),再使用ggplot2、pheatmap数据包得到差异基因的聚类热图和火山图,并输出差异基因的RPKM值用于后续分析。
GEO数据同样利用R语言(4.0.3版)进行处理,通过使用limma、ggplot2、pheatmap数据包得到差异基因及其聚类热图和火山图。
1.4 WGCNA分析
利用R语言(4.0.3版)的WGCNA包构建HCC加权基因共表达网络,对基因和模块、基因与临床性状进行相关性分析。先通过样本聚类树算法过滤掉离群样本,以保证共表达网络构建的稳定性,同时使共表达网络中的基因之间的连接服从无尺度网络分布,然后利用Pick Soft Threshold函数选择最合适的软阈值β。随后,将邻接矩阵转换为拓扑重叠矩阵(TOM)以降低虚假相关性和噪声,计算出1-TOM作为共表达网络互连的重要生物学指标和基因聚类的距离指标,再采用动态树切割算法识别基因模块,每个模块至少包含50个基因。最后,计算基因模块的特征值(ME),对模块进行层次化聚类,以0.25为剪切高度合并相似模块,并用不同颜色加以标记,根据模块特征关系(ME与临床特征之间成对的皮尔逊相关性,相关系数>0.80)确定具体的模块。绘制出聚类模块特征热图,并计算MM值(基因显著性与模块显著性的相关系数)和GS值(基因与临床特征的相关系数)。基因显著性(GS)定义为基于临床特征与基因表达之间的线性回归P值(GS=logP)的log10变换,模块显著性(MS)是指模块中所有基因的GS的平均值。
1.5 HCC交集基因筛选
一般认为hub基因需要满足以下条件:GS>0.2,MM>0.8,P≤0.05[13]。MS与GS的绝对值越大,说明与疾病越相关。根据WGCNA分析所获得的聚类模块特征热图,选择与正常样本、肿瘤样本正相关性最高的两个基因模块进行数据合并,对TCGA、GEO数据进行相同处理后,利用R语言(4.0.3版)的VennDiagram数据包对上述合并后的两个模块基因与TCGA、GEO的差异基因取交集,输出文本文件并绘制Venn图,得到最终的HCC相关基因,并将得到的交集基因与黄芩的15个候选基因靶点进行比对,获得黄芩抗HCC的潜在基因靶点。
1.6 GO与KEGG富集分析
利用R语言(4.0.3版)的org.Hs.eg.db数据包对上述交集基因进行ID转换,转换后获得相应的entrezID,再利用 R 语言(4.0.3版)的 clusterProfiler、enrichplot、ggplot2等数据包对交集基因进行GO、KEGG富集分析。
1.7 hub基因识别
将WGCNA构建的hub基因模块中的全部基因取交集后,导入STRING数据库,基因间相关性打分设置为0.900,从而获得交集基因网络关系文件,通过Cytoscape3.6.1对网络文件进行可视化分析,下载插件CytoHubba计算每个节点的最大团中心性分数(MCC值)并排序,将得分排名前10的基因作为hub基因,并将hub基因与黄芩抗HCC的潜在基因靶点整合,用于下一步分析。
1.8 OS与DFS分析
利用GEPIA数据库对hub基因进行OS、DFS分析,根据HCC患者hub基因表达量的中位数进行分组,将HCC患者分为低表达组和高表达组,置信区间设置为95%并计算P值,癌症类型选择LIHC(肝细胞癌),绘制生存曲线用于可视化分析,筛选与HCC相关的核心基因。
1.9 免疫组化验证
进入HPA数据库(https://www.proteinatlas.org/),分别在搜索拦中输入上述筛选到的核心基因名点击搜索,选择“pathology”进入病理界面,看该基因在肿瘤样品中的免疫组化图谱,找到“protein expression”,找到肝癌,点击进入肝癌页面,向下拉可以找到免疫组化的图片。抗体HPA代码必须与正常样品保持一致。如若要看基因在正常样品中的免疫组化图片,可以在搜索栏输入基因名后,点击“Tissue”进入组织页面,余下操作同肿瘤样品。收集免疫组化图谱信息,根据蛋白质在组织细胞中的染色程度以及染色细胞百分比,比较正常组织和HCC组织中蛋白表达的差异,并下载保存相应的免疫组化图片。
2 结果
2.1 黄芩活性成分及靶点与PPI网络
通过TCMSP数据库共检索到黄芩化学成分143种,以OB≥40%、DL≥0.18为筛选条件,得到19种主要活性成分,分别为黄芩新素、榄核莲黄酮、甲氧基黄酮、黄芩黄酮II、甲氧基查尔酮、二氢木蝴蝶素、苯基苯并吡喃酮、鼠尾草素、表儿茶素、二甲氧基黄酮、苏荠苎黄酮、豆甾醇、二羧酸苯酯、邻苯二甲酸二异辛酯、表小檗碱、千层纸素、黄烷酮、红花素、二氢黄芩苷,见表1,其中黄芩新素的OB值、DL值最高,可能是黄芩抗HCC最主要的生物活性成分。
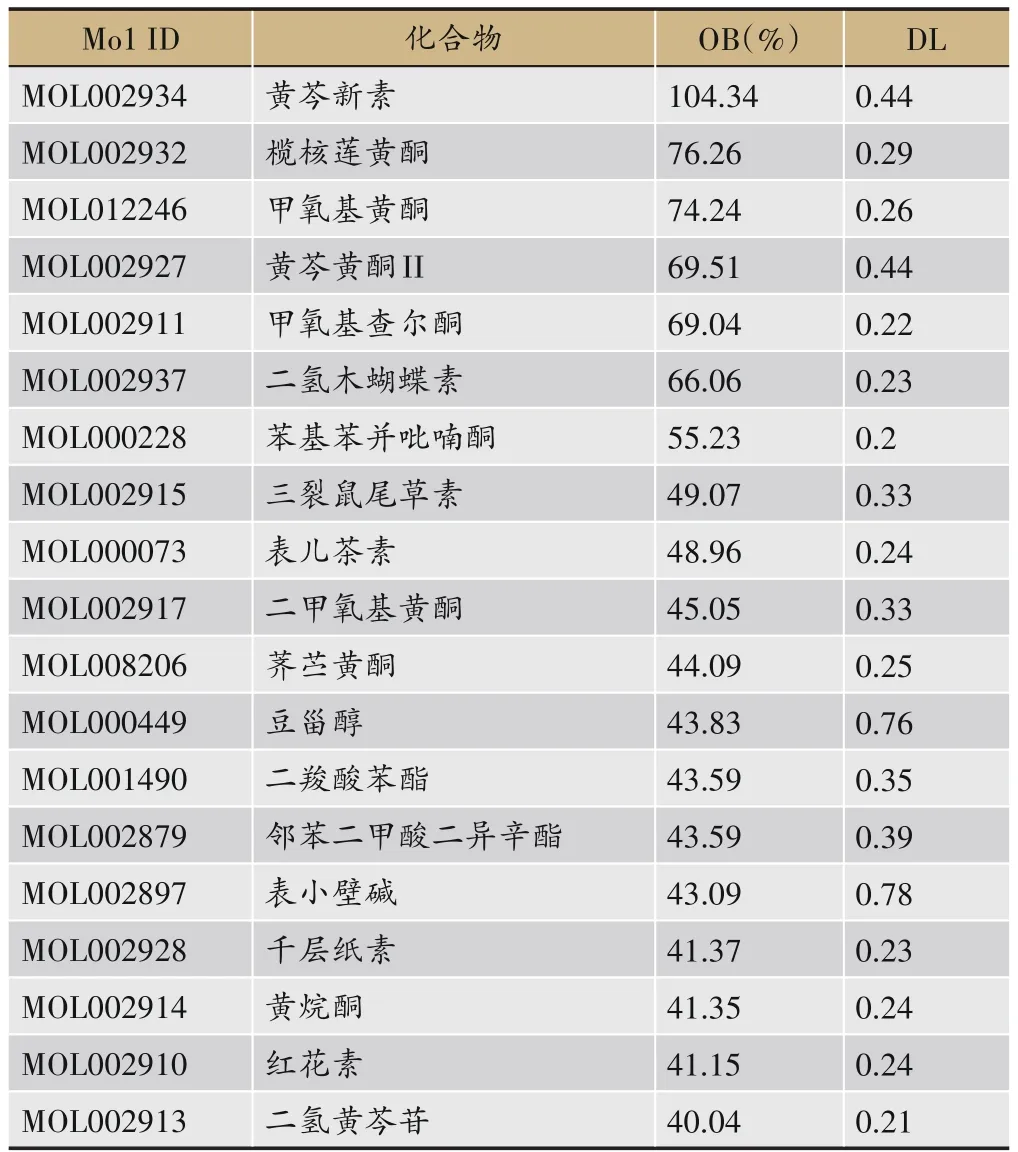
表1 黄芩主要活性成分及其OB、DL值表
19种主要活性成分的靶点去重复项后,导入STRING数据库构建PPI网络,见图1。将靶点蛋白的相互作用信息文件导入Cytoscape3.6.1,利用cytoHubba插件提取中心子网络,以MCC进行排序获得的前15个候选基因靶点依次为 NCOA1、NCOA2、ESR1、AR、RXRA、PPARG、PGR、ESR2、MAPK14、ADRA1B、ADRB2、CYP2C9、PTGS2、ADRA1A、CHRM3,见图2。
2.2 HCC差异基因
从TCGA数据库下载与HCC相关的424个转录组数据样本,共包括13913个基因,筛选后获得2703个差异基因,其中下调基因1680个(左侧),上调基因1023个(右侧),火山图见图3A。
从GEO数据库下载的与HCC相关的20个肿瘤样品和20个非肿瘤样品,共包括21 655个基因,筛选后获得1050个差异基因,其中下调基因621个(左侧),上调基因429个(右侧),火山图见图3B。
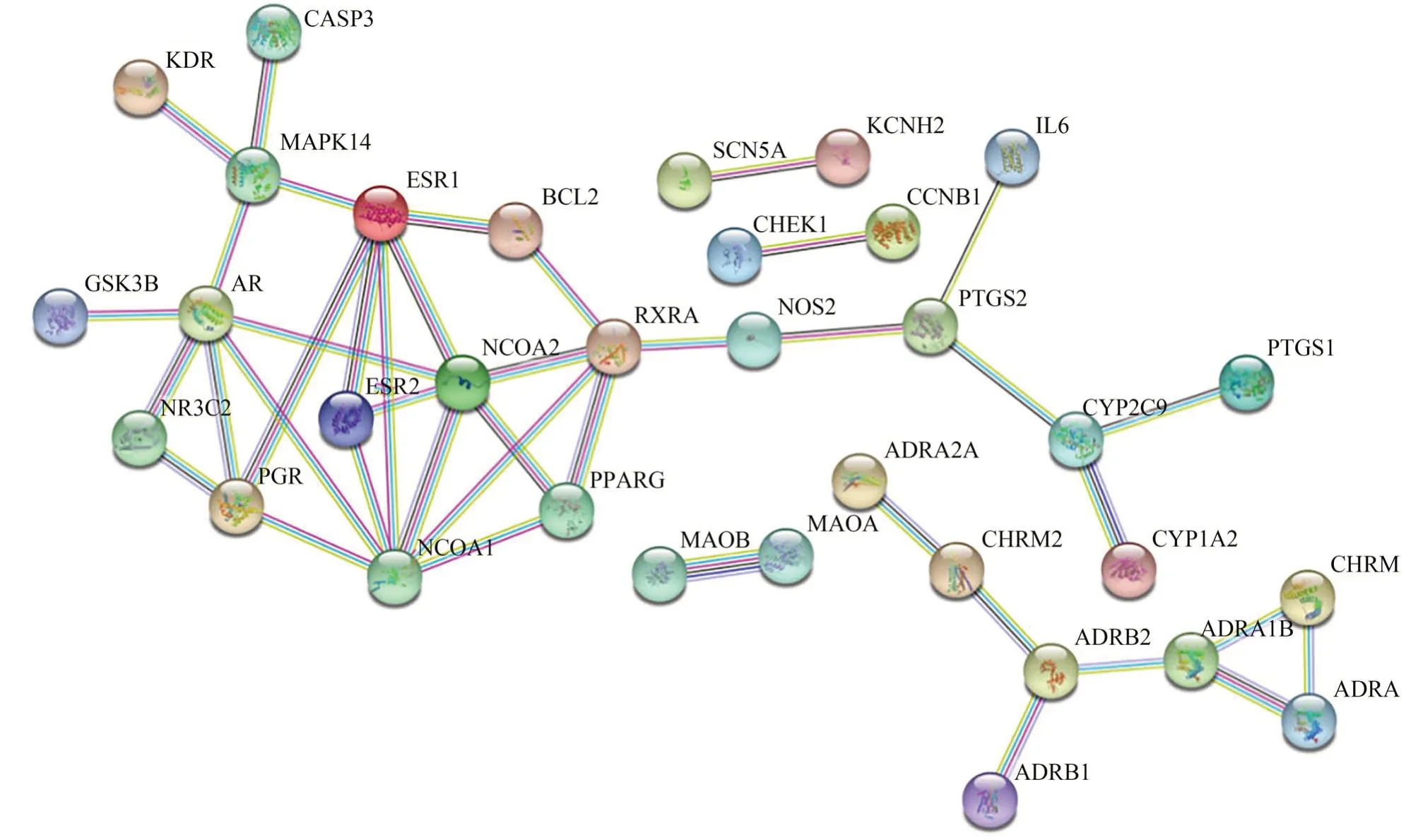
图1 黄芩主要活性成分靶点的蛋白-蛋白相互作用网络图

图2 以MCC进行排序获得的前15个候选基因靶点图(黄芩)
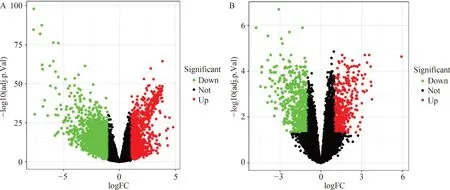
图3 HCC差异基因火山图
2.3 WGCNA分析
本研究中,TCGA数据选择软阈值β=3(无尺度R2=0.9),动态树切割共识别出9个基因模块,见图4A;GEO数据则选择软阈值β=2(无尺度R2=0.9),动态树切割共识别出11个基因模块,见图4B。基因模块与临床性状之间的相关性如下图所示,可知TCGA数据中与正常样本、肿瘤样本正相关性最高的模块分别是黄绿色模块(MEgreenyellow,R=0.64,P=2e-50)、蓝色模块(MEblue,R=0.69,P=8e-62),见图4C;GEO数据中与正常样本、肿瘤样本正相关性最高的模块分别是蓝色模块(MEblue,R=0.44,P=0.005)、青绿色模块(MEturquoise,R=0.75,P=2e-08),见图4D。因此,我们选择这四个模块中的差异基因进行下一步分析。
2.4 HCC交集基因
如图5所示,TCGA数据中黄绿色模块和蓝色模块合并后共4701个基因,GEO数据中蓝色模块和青绿色模块合并后共5928个基因,这些基因与TCGA数据中的2703个差异基因、GEO数据中的1050个差异基因取交集,共得到115个基因,而后将这115个交集基因与黄芩的15个候选基因靶点进行一一比对,获得黄芩抗HCC的潜在基因靶点ADRA1A。
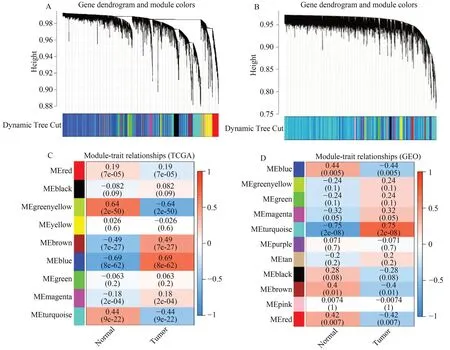
图4 WGCNA聚类树状图及聚类模块特征热图
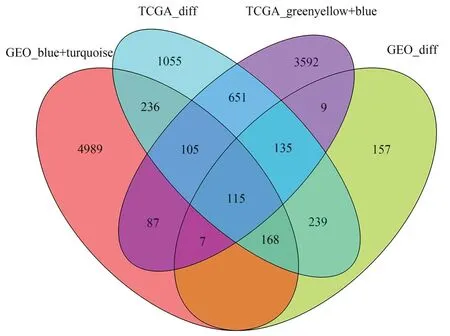
图5 交集基因与差异基因Venn图
2.5 GO与KEGG富集分析

图6 基因富集分析图
GO分析结果如图6A所示,上述115个交集基因富集在体液免疫反应、细胞粘附、补体激活、呼吸系统发育、细胞过渡金属离子稳态、BMP信号通路的调节、反射等生物学过程(BP);KEGG分析结果如图6B所示,上述115个交集基因富集在胆固醇代谢、矿物质吸收、基底细胞癌、泛醌和其他萜类醌的生物合成、视黄醇代谢、细胞粘附、吞噬体等通路。如图7所示,交集基因在HCC中主要涉及Wnt信号通路,即Wnt与细胞膜上的受体结合,激活第二信使Dvl,从而抑制由GSK-3β、Axin、APC组成的三元复合物的活性,使β-catenin不能被GSK-3β正常磷酸化,造成β-catenin在胞浆内大量积聚并移向细胞核内,进而与细胞核内的转录因子TCF/LEF结合,激活TCF转录活性,调节靶基因的表达。
2.6 hub基因识别
通过Cytoscape3.6.1的cytoHubba插件提取上述115个交集基因的中心子网络,使用CytoHubba插件中的MCC算法计算网络中每个节点的最大团中心性分数(MCC值),按MCC值的大小进行排序,MCC得分排名前10的关键基因依次为FCN2、COLEC11、CFP、COLEC10、FCN3、C7、LCN2、HAMP、B3GAT1、LYVE1。将这10个基因与黄芩抗HCC的潜在基因靶点ADRA1A整合,用于下一步分析。
2.7 OS与DFS分析
根据总生存期曲线和无病生存期曲线,观察整合后的11个基因在总生存期与无病生存期的基因表达差异是否具有统计学意义(P<0.05),从而进一步筛选出与HCC密切相关的核心基因。OS分析结果显示,具有统计学意义的基因有FCN3(P=0.033)、ADRA1A(P=0.012)、C7(P=0.03)、COLEC10(P=0.015),且这些基因高表达的患者前80个月生存率更高;DFS分析结果显示,具有统计学意义的基因有CFP(P=0.029)、FCN3(P=0.0086),且这些基因高表达的患者同样生存率更高,见图8。因此,这5个基因可能是黄芩治疗HCC的核心基因,分别为纤维胶凝蛋白3(FCN3)、肾上腺素能α1A受体(ADRA1A)、补体C7(C7)、凝集蛋白家族 10(COLEC10)、补体因子备解素(CFP)。其中,纤维胶凝蛋白3在OS、DFS分析中均具有统计学意义,可能是与HCC关系最密切的基因。

图7 交集基因生成的HCC相关信号通路

图8 HCC的5个密切相关基因OS、DFS分析
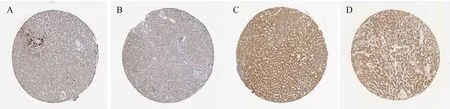
图9 肝正常组织与HCC组织的免疫组化图
2.8 免疫组化验证
通过HPA数据库分别检索COLEC10、CFP、ADRA1A,免疫组化结果为空;FCN3、C7免疫组化结果如图9所示,抗体CAB025945在正常肝组织中FCN3呈现中等强度染色,在HCC组织中则呈现低强度染色,染色细胞百分比为25%-75%;抗体HPA001465在正常肝组织中C7呈现高强度染色,在HCC组织中则呈现中等强度染色,染色细胞百分比均超过75%。这表明与正常肝组织相比,HCC组织中FCN3、C7基因表达量降低,该结果与OS、DFS分析所得结果一致。
3 结论与讨论
本研究基于网络药理学的方法,发现黄芩可能是通过黄芩新素、榄核莲黄酮、二氢黄芩苷等活性成分,降低ADRA1A甲基化频率从而干预HCC的发生;通过对TCGA、GEO数据库中HCC相关数据进行差异基因筛选、WGCNA分析、GO与KEGG富集分析,并经过OS、DFS、HPA 分析验证,发现 FCN3、C7、COLEC10、CFP可能是HCC的核心基因,其作用机制可能涉及体液免疫、补体激活、吞噬作用调节和细胞凋亡等过程。
黄芩化学成分众多,目前为止已被发现的约有40多种,包括黄酮类化合物、萜类、氨基酸、甾醇、挥发油和其他成分,其中黄酮类化合物是黄芩主要的有效成分和特征性成分。研究表明,黄酮类化合物中发挥抗肝癌作用的主要是黄芩素、黄芩苷、汉黄芩素和汉黄芩苷4种活性物质。杜忠良等[14]发现黄芩素能抑制肝癌移植瘤的生长、诱导肿瘤细胞凋亡,其作用机制可能与调控PI3K-AKT通路有关。鲁兴梅等[15]认为黄芩苷可抑制肝癌荷瘤小鼠瘤组织的增长,其作用机制可能与下调瘤组织PI3K、AKT和m TOR基因及蛋白表达有关。王晓东[16]证明汉黄芩素能通过线粒体信号通路促进人肝癌SMMC-7721细胞的凋亡、抑制人肝癌SMMC-7721细胞增殖。本研究综合网络药理学、癌症基因组图谱和基因芯片技术,得到黄芩新素、榄核莲黄酮、二氢木蝴蝶素、二氢黄芩苷等19种黄芩主要活性成分,其中黄芩新素的口服利用度高达104.34%,类药性也达到了0.44,具有很高的研究价值。而且目前国内外对黄芩有效活性成分的研究多集中在黄芩苷、汉黄芩苷、黄芩素、汉黄芩素等含量较高的黄酮类成分,对于黄芩新素的研究非常少,而且多集中在提取及制备工艺方面。近年来研究发现,黄芩新素是诱导白血病细胞HL-60和K562凋亡的主要活性成分,其通过上调PP2A表达,下调miR-21表达,使PI3K/AKT/mTOR信号通路失活,从而降低肺癌发生率[17]。Chen等[18]研究发现,虽然黄芩新素在黄芩中的含量较低,但它能特异性结合在免疫抑制和肿瘤生长中起重要作用的靶蛋白STAT3。因此,我们猜测黄芩新素是否也能作为一个重要的活性物质发挥抗肝癌作用呢?这也将为黄芩有效成分及药理作用的进一步挖掘和HCC的防治提供一个新思路。
GO功能富集分析和KEGG通路富集分析结果显示,黄芩主要涉及与肝癌相关的信号通路是Wnt信号通路,这与文献[19]报道一致,Wnt/β-catenin信号通路参与肝脏疾病进展的所有阶段,该信号通路的异常将导致肝细胞癌、肝母细胞瘤和胆管癌等多种肝癌的发生,Wnt/β-catenin信号通路受到多种因子的调控,当细胞质中β-catenin不能正常降解时,Wnt/β-catenin信号通路活化增强,多种肿瘤疾病由此引发。目前已经有多项研究报道了调控该信号通路可以抑制肝癌的潜在价值[20-21],因此我们有理由相信黄芩的活性物质黄芩新素可能通过调控Wnt/β-catenin信号通路上下游相关基因的表达,作为防治肝细胞癌潜在药物的出现是极有可能的。
本研究发现黄芩抗HCC的潜在基因靶点ADRA1A,筛选出的HCC核心基因为FCN3、C7、COLEC10、CFP。肾上腺素能α1A受体(ADRA1A)是一类通过结合儿茶酚胺刺激交感神经系统(SNS)的G蛋白偶联受体,Chen等[22]的研究表明,与癌旁组织相比,在HCC组织中,ADRA1A mRNA和蛋白的表达水平显著降低,其启动子区的平均甲基化水平显著增加(17.0% vs.25.2%,P<0.0001),ADRA1A基因高甲基化可能导致HCC的发生,这与本研究中ADRA1A的OS分析结果一致(ADRA1A高表达的患者前80个月生存率更高),因此,ADRA1A是黄芩治疗HCC的有前景的基因靶点。纤维胶凝蛋白3(FCN3)是机体天然免疫的关键性分子,主要参与补体激活、吞噬作用调节和细胞凋亡过程[23],研究发现,FCN3功能障碍或表达异常可能造成人类某些感染性疾病、慢性疾病及自身免疫性疾病的发生[24],与KEGG富集分析结果一致。补体C7是补体级联反应的下游,通过补体经典途径或者凝集素途径激活,补体途径还可激活天然免疫系统,在抵抗细菌等病原体感染中起重要作用。张鑫等[25]报道,通过供肝组织SNP检测分型发现,供肝C7 rs6876739基因型与肝移植术后的早期细菌感染显著相关,这可能与移植肝中C7合成减少有关;Würzner等[26]报道,移植肝合成高达50%以上的C7,说明C7可作为HCC的预后因子。凝集蛋白家族10(COLEC10)是HCC中miR-452-5p的直接靶标,miR-452-5p过表达能够显著加速细胞增殖,诱导细胞周期从G1转变,抑制癌细胞的凋亡[27-28],说明COLEC10是HCC的核心基因之一。OS与DFS分析、以及免疫组化验证也已证实,FCN3、C7、COLEC10、CFP的异常表达与HCC的发生发展密切相关。
综上所述,本研究初步揭示了黄芩抗HCC的潜在基因靶点和HCC核心基因,探讨了其相关分子机制,为后期开展黄芩治疗HCC的实验和临床研究提供理论参考。但本研究仍然存在不足和局限性:
(1)数据来源比较单一
网络药理学的重要基础是对不同来源数据进行整合分析,不同数据库如常用的中药成分数据库(ETCM、TCMSP、TCMID、TCM)数据收录差异大、且数据信息之间无有效关联相对比较独立,因此数据收集的完整性和整合分析对研究结果的可靠性、可重现性有较大影响。本研究黄芩活性成分及其靶点的筛选选用的是TCMSP,来源比较单一,一定程度上可能影响研究结果的重复性。以后我们可以尽量通过整合多个来源的数据、同时结合临床或实验方法进行数据采集、检测和补充[29]。
(2)缺乏更加有效、科学的验证
本研究遵守2021年世界中医药学会联合会发布的《网络药理学评价方法指南》[30-31],并按要求对其内容进行可靠性评价、规范性评价和合理性评价。但在结果验证方面,本研究目前还停留在文献验证和计算机实验验证阶段,缺乏更加科学有效和更具说服力的验证方式,如体外细胞模型、动物模型实验验证,甚至临床试验验证等方式。
