冠状病毒感染动物模型比较转录组学数据库的建立
2022-03-24吴玥向志光高苒孔琪
吴玥向志光高苒孔琪
(中国医学科学院医学实验动物研究所,北京协和医学院比较医学中心,国家卫生健康委员会人类疾病比较医学重点实验室,新发再发传染病动物模型研究北京市重点实验室,北京市人类重大疾病实验动物模型工程技术研究中心,国家中医药管理局人类疾病动物模型三级实验室,北京 100021)
冠状病毒是套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒属(Coronavirus)的一类线性单股正链RNA病毒,其中对人类致病性较强的包括SARS-CoV(severe acute respiratory syndrome coronavirus)、MERS-CoV(middle east respiratory syndrome coronavirus)和SARS-CoV-2(severe acute respiratory syndrome coronavirus 2),其病原致病性和动物模型方面有一定的相似性[1]。
SARS-CoV-2引起的新型冠状病毒肺炎(Corona Virus Disease 2019,COVID-19)在世界各国爆发流行,截止2021年11月已造成2.49亿人感染和504万人死亡[2-3]。新型冠状病毒感染危害极强,需要动物模型研究其发病机理、病毒溯源和传播机制、疫苗药物评价等[4]。
冠状病毒感染动物模型大致分为3类:啮齿类、灵长类和其他哺乳动物,主要包括转基因小鼠、叙利亚仓鼠、雪貂、非人灵长类、猫、犬等。不同动物对冠状病毒的易感性不同,能不同层面的反应人类疾病,有助于疫苗药物评价和发病机理研究等[5-8]。基于动物模型从基因层面开展比较转录组学研究有助于理解不同物种、不同毒株、不同滴度、不同时间等条件下基因差异性表达情况[9]。
随着高通量测序技术的不断发展,转录组学数据迅速累积,但目前还没有一个专题数据库将已有的数据资源进行整合,深入分析形成跨种并且多维度比较人与实验动物在感染冠状病毒后的基因表达情况,筛选差异基因。因此,我们首次建立了冠状病毒感染动物模型比较转录组学数据库,通过采集整合基因表达数据库(gene expression omnibus,GEO)、芯片表达数据库(ArrayExpress)等公共数据库中冠状病毒感染动物模型基因表达谱数据,进行不同冠状病毒感染动物模型表达数据的挖掘和分析[10-11]。可提供不同冠状病毒感染不同物种/细胞/时间的基因表达变化情况。根据差异表达基因,分析其涉及的信号通路和生物调控网络。冠状病毒比较转录组学数据库为世界首个冠状病毒基因表达谱专题数据库,在研究人与实验动物感染冠状病毒后表达模式及差异基因挖掘具有重要意义。
1 方法
1.1 数据收集和处理
通过NCBI GEO(http://www.ncbi.nlm.nih.gov/geo/)、EBI ArrayExpress数据库(https://www.ebi.ac.uk/arrayexpress/)等数据库,收集SARS-CoV、SARS-CoV-2、MERS-CoV等冠状病毒感染不同人/动物/细胞的基因芯片、转录组数据。收集背景数据,包括动物模型、病毒株、感染时间、滴度、感染器官或细胞等。将不同物种背景数据与基因表达数据进行对应、整合、质控,标准化、注释,去除批次效应,用于可视化展示和差异基因分析。
1.2 数据库建设
本数据库使用Django(v 2.2.6)软件(https://djangoproject.com)开发,部署在CentOS Linux server(v 7.7)服务器。网站服务使用nginx(v 1.10.1)。使用Plotly(v 1.5.10)实现表达数据在线交互式可视化展示。所有在线分析生成的图片具有交互功能。可在线调整图片属性,例如大小、样式、配色、显示的内容等。分析结果可导出矢量图(SVG格式)。用户也可下载生成的数据。
1.3 访问方式
数据库网址:https://covid.com-med.org.cn。
2 结果
2.1 数据库结构和内容
本数据库由简单检索、专业检索、分析工具、数据统计、帮助信息五个版块组成(图1)。分析工具中包括种间比较、种内比较和多基因基因表达谱3种工具。数据库包括43个数据集,3种病毒(SARS-CoV、SARS-CoV-2、MERS-CoV),22个病毒株,4个物种(人、小鼠、猴、雪貂),14种组织/细胞,29个感染时间,15种病毒滴度,共涉及2373个样本(表1)。

表1 数据库样本信息Table 1 Database sample information

图1 数据库首页Figure 1 Homepage of the database
2.2 检索方式
本数据库主要有2种检索方式,一种是简单检索(simple search),另一种是专业检索(advanced search)。在使用简单检索时,用户可使用基因名称、别名、注释、功能描述和通路信息等进行搜索。数据库将展示所有与搜索内容相关的基因与对应描述。点击基因名称可进入详细页。详情页包括基因信息以及与数据库外链,单基因表达谱信息。
对于专业检索,用户可自由组合多种检索条件,包括物种名称、病毒名称、病毒毒株、细胞/组织类型、项目编号等对数据库中样品进行专业检索。展示详细检索结果,帮助用户找到合适的样本数据,并通过交互式联动饼图进行对应统计展示。可对历史检索信息进行记录,方便查询历史记录。
2.3 基因表达谱
2.3.1 单基因表达谱
通过简单检索,输入基因、蛋白、通路名称后,可从不同层面对所采集数据集进行整合分析。实现不同层面的比较,直观比较冠状病毒感染人类和动物模型后基因表达差异。使用箱形图显示比较结果并显示P值。用户可以查看目标基因在不同物种、病毒株、不同细胞、不同感染时间、不同滴度中的差异性表达情况(图2)。

图2 单基因表达谱Figure 2 Single gene expression profile
2.3.2 多基因表达谱
进入多基因表达谱(GEM profile)页面,通过输入多个基因名称,并选择组合参数。例如可以选择不同物种、不同病毒、不同病毒株、不同感染时间、不同滴度、感染组织/器官、不同测序数据类型。也可更详细的选择感染时间、组别、GSE编号等。最后选择数据归一化方式(raw或Z值转换),通过折线图、柱形图、热图、箱形图、相关性图的形式进行可视化展示(图3)。同时提供内置的工具包对生成的图片进行颜色、大小和样式个性化更改。所有图片均可以通过SVG格式下载使用。可跨物种比较和展示,采用不同物种并排放置的方式展示。可检测多个基因在多个物种中的表达变化趋势。

图3 多基因表达谱绘制Figure 3 Multiple gene expression profile
2.4 差异基因比较分析
差异基因分析是寻找关键marker基因的最主要手段。用户可对感兴趣的物种、实验条件下的基因表达差异分析。通过物种内、物种间差异基因分析这两种工具,在线组合不同指标设计实验,寻找易感基因,并对易感基因进行功能富集分析。值得注意的是,种内差异基因分析工具仅限一种物种。跨种差异基因分析工具允许用户选择两种物种进行比较分析。
确定物种选项后,用户可进一步选择研究的细胞类型、病毒类型、时间、滴度等信息。提交分析后系统将对样品进行自动检测。用户可对样本属性差别较大、不适合用于后续整合比较分析的异常样品进行过滤(图4)。通过聚类热图与PCA聚类图的形式进行可视化展示样品相关性(图5)。用户可根据热图与PCA图选择用于后续分析的样本。

图4 异常样品检测Figure 4 Abnormal sample detection

图5 样品相关性聚类热图Figure 5 Heatmap of correlation results for samples
用户可自由选择比较组,通常做差异基因只能进行24 h感染组(Treat 24 h)与0 h感染组(Treat 0 h)两组比较。感染性实验比较特殊,两个时间点会有4组样品(Treat 24 h,mock 24 h,Treat 0 h,mock 0 h)。为此我们引入复杂设计比较模式(Treat 24 hmock 24 h)-(Treat 0 h-mock 0 h),可在让处理组与各自对照比较完成后,再进行不同时间点比较。这个分析模式也适用于不同物种的比较,例如人的肺细胞和小鼠的肺细胞不能直接比,但是(Human lung treat-Human lung control)-(Mouse lung treat-Mouse lung control)与各自对照比较后再进行比较,便可以实现物种各自组织处理后的变化之间的比较,可分析哪些基因在不同物种中是一致表达的或特异表达的。
用户可对选定样本进行差异基因分析的阈值FDR(默认小于0.05)与Log2FC(默认大于1)进行设置,生成差异分析结果报告。报告目录可索引各部分结果,预测的差异基因以火山图和热图的形式展示。图6为跨种比较分析人类-小鼠感染SARS病毒后基因表达种间聚类热图,用于展示不同物种感染SARS病毒后基因表达聚类情况。还可对差异基因进行功能富集分析,对基因所参与的生物学功能、分子功能和细胞成分进行聚类,以气泡图的形式进行展示(图7)。
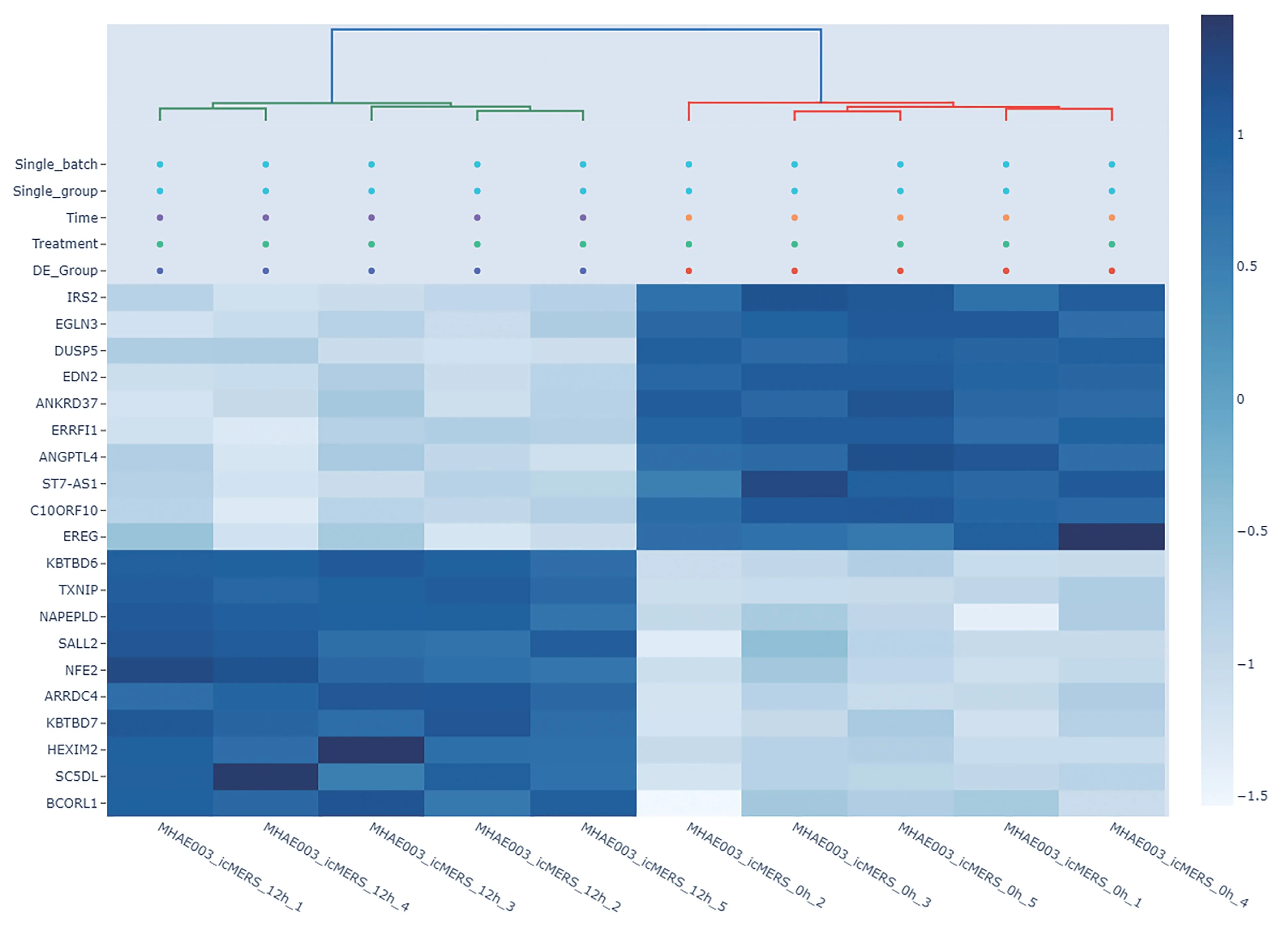
图6 人类-小鼠感染病毒后差异基因热图Figure 6 Differential gene heatmap for human-mouse infected virus

图7 差异表达基因的生物学过程富集结果Figure 7 Biological process enrichment result for differentially expressed genes
3 讨论
为了比较分析冠状病毒感染动物模型后基因表达谱变化,我们采集公共数据库中冠状病毒感染动物模型相关开放数据集,建立了冠状病毒感染动物模型比较转录组学数据库,并持续更新。用户可在基因表达水平进行不同物种、病毒株、滴度、时间等变量的比较分析,提供冠状病毒感染动物模型基因表达层面的数据挖掘及关联分析。通过比较不同基因不同物种在不同感染条件下的表达水平变化,从基因表达水平研究冠状病毒感染不同动物模型的致病机制,并进一步开展聚类分析和功能富集等生物信息分析。
世界卫生组织(WHO)及各国都强调了新冠疫情数据共享和开放的重要性。新的数字平台和开放科学实践的发展可能对加强全球研究和创新合作作出巨大贡献[12-15]。已有多项研究通过现有的开放数据库发表[16-17]。已建立并且在公开期刊发表论文的与冠状病毒研究相关的数据库有GISAID、CoVDB、ViPR、2019nCoVR、Drugvirus、CARD、D3Targets-2019-nCoV、CoV2-ID等,主要储存病毒的基因序列,流行病学信息,药物靶点信息,并且主要基于病毒基因序列进行分析,无基因表达信息的整合分析[18-25]。
冠状病毒感染动物模型比较转录组学数据库通过对不同平台的测序数据进行质控、标准化、去除批次效应,获得校正后的基因表达数据,实现跨平台的数据分析。可展示冠状病毒感染动物模型后的基因表达图谱,筛选差异表达基因。可比较分析不同物种、不同病毒株、不同剂量及不同时间点的基因表达。为发现和预测可能用于新冠致病相关研究的靶基因或治疗靶点提供数据依据。本数据库将继续更新冠状病毒表达谱数据,纳入更多的数据类型,比如自有数据和类器官数据、单细胞测序数据,研究基因在细胞水平的表达情况。本数据库是在中国医学科学院医学实验动物研究所秦川教授的指导下建立的,在此表示感谢。也感谢魏强、薛婧等专家提出的宝贵意见。
