云计算下的基因测序数据并行化生成方法
2022-03-15刘志明
刘志明,冉 昊
(1.吉林建筑大学电气与计算机学院,吉林 长春 130118;2.吉林建筑大学,吉林 长春 130118)
1 引言
基因测序生成的数据能够体现出个体行为特性,对疾病的早期筛查起到关键作用。基因作为最基础的遗传单位,体现出各类遗传信息特征,是一段功能性较强的DNA序列。测序技术的发展促使生命科学进一步发生巨大改变。基因测序就是利用有效的测序手段采集DNA序列,并将化学信号变换为可令计算机识别的数字信号的程序,在生物学多个领域被广泛应用。此外,基因测序还与人们生活密切相关,利用此技术不但可以划分人种,还能辅助医学诊断,为细胞移植提供准确配型数据。但随基因组数的飞速增长,每半年左右就会提高一倍,增加测序数据生成的负担,其中会生成大量冗余数据,降低数据生成速度与精度。
为解决上述问题,相关领域学者提出一些解决方案。例如,郭茂祖等人[1]提出基于RNA-Seq的转录组分析方案。通过对初始基因数据的质控与定量计算,完成数据预处理;分析其差异表达,实现基因筛选;使用统计学与机器学习两种方式对高层差异基因做进一步处理,采用富集分析形式明确基因功能与调控网络,输出最终生成的测序数据。肖颖等人[2]提出基于贝叶斯分析的基因测序数据生成方法。结合基因表达信息,建立基因均值差序列,构建贝叶斯分层混合模型,同时为模型参数赋予先验信息;通过马尔科夫链算法完成模型参数估计,生成测序数据。但基因数据的惊人增长速度对测序生成化方法的运算速度与成本要求逐渐提高。
为此本文将云计算引入到生物信息领域,利用虚拟技术将云端服务器与网络相连,不需要大量的人工对其管理,降低成本,扩大储存空间,在云计算架构下完成初始基因数据预处理,并结合聚类算法生成测序数据,优化生成速度和质量。
2 基于主成分分析的初始数据预处理
基因芯片的发展使基因数据可以被迅速测序,生成基因阵列[3]。这些初始基因数据维数较高,具有一定噪声,导致生成的测序数据无法将生物学的有效信息直观地传达给研究人员。要想实现测序数据的快速、精确生成,必须使用特定方式对这些数据进行预处理,降低维数,使生成的测序数据更能体现基因特征。
本文利用主成分分析法完成初始数据预处理,该方法的核心为将多个变量变换为少数综合性评价指标[4],通过获取的指标数据实现数据处理。这些指标是基于初始数据,经特殊数据处理后,获得体现整体特征的指标。因此,此种方法的本质也属于一种分类降维手段。
主成分分析是将数据原有的相关性指标利用线性组合[5]方式,转换为一组不具备关联性的综合指标,来体现初始数据集合的整体特性。
设定共存在n个样本,任意一个样本具备p个特征向量,样本相对的特征值表示为X1,X2,…Xp,初始特征子集的表达式如下
(1)
利用上述特征集合的列向量X1,X2,…,Xp进行线性组合,获得不同组合形式,得出多个综合数据指标
(2)
式中,ai代表单位向量,且满足如下条件
(3)
全部线性组合形成的指标向量之间存在的协方差等于0,彼此互不关联。此外,基于上述约束条件,将所有指标向量中具有最大方差的线性组合当作首要主成分,并以此类推即可获得预先设置的前K个主成分F1,F2,…,FK。这些主成分根据表示的初始特征数量逐次递减,而特征数量需利用主成分方差评估,也就是取决于初始相关系数矩阵中表示特征的值λi,该值越大,表明主成分体现信息的性能越强。
综上,获取的主成分数据必须存在特征值λi,通过λi确定最终被选出的综合向量。计算过程如下:
步骤一:获取与特征集合相对的相关系数矩阵
(4)
式中,rij可通过下述公式获取
(5)
步骤二:对于上述矩阵,利用雅可比法计算出特征值[6]与向量,同时将特征值根据大小排序λ1≥λ2…≥λp>0,则与其相对的特征向量表示为
(6)
如果事先从初始特征集合内挑选m个主成分完成分为操作,当计算出矩阵的特征值与向量后,通过下述公式获取每种线性组合方法的方差贡献值
(7)
再通过下述公式获取前m个主成分方差贡献率的累计值
(8)
通常情况下,式(8)的值高于85%,即可较好表示整体初始基因数据的基础信息。
步骤三:采用式(9)对获得的主成分数据做标准化处理
(9)
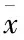
3 基于云计算的基因测序数据并行化生成
3.1 云平台架构设计
云计算下的集群框架[7]通常包括集中式与对等式[8]。本文设计的云架构将虚拟化服务当作核心,因此选取集群式架构。要求网络中所有节点必须具备完整服务,当有消息产生时,响应节点需立即将数据传输到其它节点中,保证网络数据具有高度一致性。本文设计的云架构如图1所示,包括应用层、服务层与资源层。其中服务层可构建通用服务接口,实现本地通信;资源层为平台提供处理器等资源;应用层则利用开发工具接口调试相应服务。
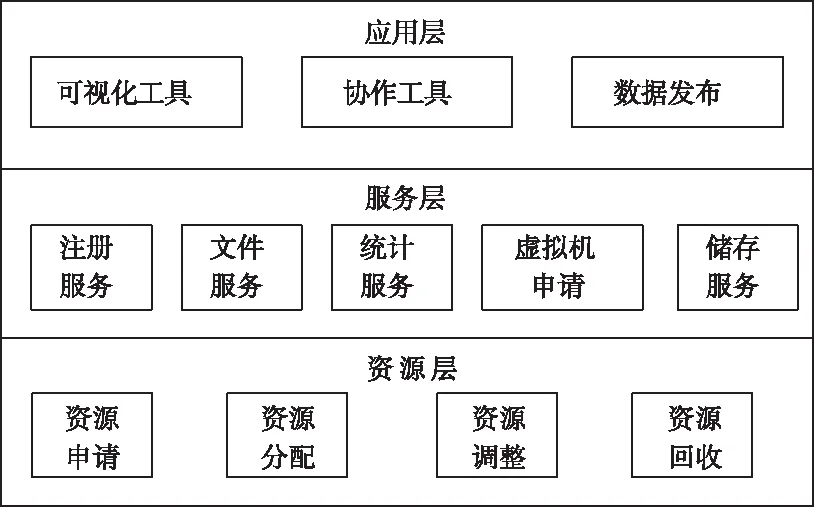
图1 云平台架构示意图
在上述云架构中,云平台共包括七个组件,各组件功能如下:
1)访问接口:为用户提供访问的方式,可实现平台运行状态监测、数据生成进度跟踪、文件访问等功能。
2)信息服务:监测云平台是否稳定运行,采集每个节点的运行状况;
3)注册服务:云平台构建过程、申请虚拟机;
4)任务提交:获取用户提交的请求,分析任务目标,并将任务转移到虚拟机节点;
5)文件服务:在执行任务过程中将用户相关文件发送到执行节点;
6)虚拟机部署[9]:研究用户提交的虚拟机要求,评估资源信息,确定最佳主机,完成虚拟机部署;
7)任务操作:操作用户提交的任务,监控执行状态,再将结果返回到用户端。
以上七个功能不是独立存在的,必须紧密配合才能完成云平台各种服务。
3.2 基因测序数据质量控制策略
要使生成数据更加精准地描述基因特征信息,在数据生成之前需对其进行质量控制。本文结合基因数据发展特征,按照相关规则与标准提出了基因测序数据生成质量控制策略。
利用多模型方法给出序列质量测评报告供审批人员参考。从可能污染物[10]测算、假基因测算、相似度计算等方面共同实现测序数据质量控制。
1)验证是否存在终止密码子与污染物
在经过预处理后的DNA数据中,终止密码子包括TAG、TAA与TGA三种片段。若其中包括这些片段则表明含有终止密码子,该基因可能属于假基因。
污染物通常指某序列对分为前后两段,如果某序列的首端与末端不一致,则判断其中含有一定污染物。
2)结合序列的Trace Files评估序列质量
利用Phred程序即可读出Trace Files,并将待生成数据应有的质量分数保存到文档中。
3.3 并行化生成
在云计算架构中,将总的生成任务划分成多个子块,在并行能力较强的节点上,将任务分配给处理器,最终达到提高生成速度的目的。
结合聚类方法的反单调性,将最小子矩阵(2*2子阵)作为出发点,使用阈值δ依次判别能否形成聚类,若可以实现聚类,则输出生成数据;反之结合反单调性,无法继续形成聚类,将其去除或不做任何处理。
针对某聚类R,若对其加入一行或一列,可以形成更大聚类,将此过程称作对R的扩展行为。对于无法扩展的聚类,其本身就属于最大聚类,将其保存;对于可进一步扩展的聚类,在扩展完成后,对其删除,同时对扩展形成的聚类做进一步处理。
对于某构成聚类的子矩阵〈I,J〉,其层号level(I,J)表示为
(10)
在对〈I,J〉扩展过程中,为防止可能生成的聚类丢失,对其行与列的扩展操作不能同时进行。例如在矩阵A中
(11)
假设δ=1,I={1,2,3},J={1,2,3},div(I,J)=0<δ,〈I,J〉形成聚类,在对其扩展过程中,同时将I扩展到I′={1,2,3,4},J′={1,2,3,4},因为div(I′,J′)=3>δ无法生成聚类,但若单独对〈I,J〉进行扩展,获得〈I′,J〉,此时div(I′,J)=0<δ,〈I′,J〉即为一个聚类。同理对〈I,J〉的列进行单独扩展,获得〈I,J′〉,由于div(I,J′)=0<δ,〈I,J′〉也会生成聚类。但对〈I,J〉进行同步扩展,这两个聚类便会丢失。
为解决上述问题,同时引入两个表R′与C′来记录满足扩展要求的聚类。在此方法中,当第i′层聚类〈I,J〉扩展为i′+1层聚类〈I′,J′〉时,不需考虑〈I′,J′〉是否由〈I,J〉同时扩展得出的,〈I′,J′〉均需被保存到扩展表R′与C′表中。
当处理第i′层扩展聚类时,对R′与C′表中全部第i′层的待扩展聚类执行所有扩展操作,获得新的第i′+1层聚类,将其保存到R′与C′中。当第i′层完成操作时,若第i′层内某聚类〈I,J〉被扩展生成新聚类,表明〈I,J〉并不是最大聚类,可被去除。


(12)
结合每层扩展操作获取的新聚类〈I,J〉,对ME(I,j),j∈J与ME(I,i′),i′∈J进行计算,生成并行化测序数据
(13)
(14)
4 仿真分析
为证明基于云计算的基因测序数据并行生成方法性能,利用Hadoop集群进行性能测试仿真。Hadoop集群的硬件信息如表1所示,仿真总部署如图2所示。

表1 实验环境配置信息表
首先对三种方法预处理后的CPU占用率进行对比,结果如图2~图4所示。

图2 文献[1]方法的CPU利用情况监测
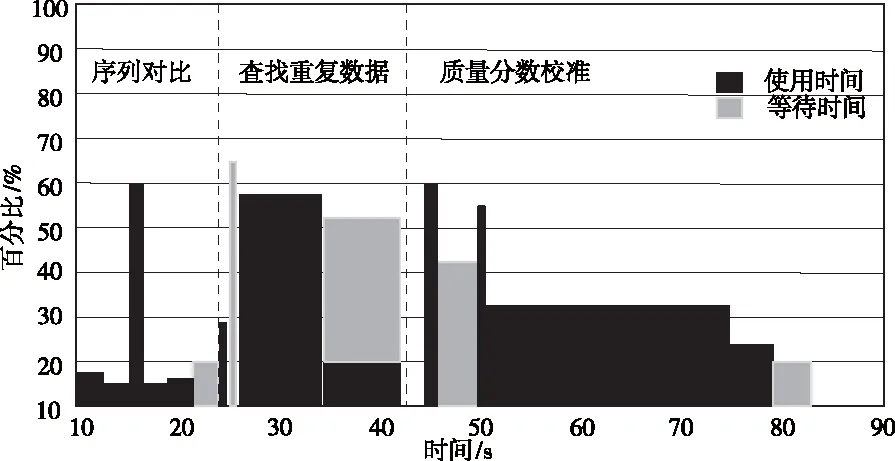
图3 文献[2]方法的CPU利用情况监测

图4 本文方法数据预处理CPU利用情况监测
由三种方法进行数据预处理时CPU的利用情况能够得出:所提方法的CPU利用率更高,尤其在筛选重复数据时,其它算法都出现较长的等待时间。因此能够体现出主成分分析法对重复数据的过滤效果更好,大大降低初始数据维度。
本文在云计算基础上利用了均值聚类算法,设置信度均为85%。测试三种方法生成的基因测序数据与某种疾病的关联程度。
由图5得出,随着生成数据量的增多,与某疾病相关的基因测序关联规则也逐渐增多,更能显现出该疾病的基因。在三种算法中,所提方法在生成相同数据情况下,关联规则最多,因此生成的数据与此种疾病存在较强的关联性。可通过测序数据准确判断出人体是否存在病变基因。

图5 不同算法生成数据性能对比图
5 结论
现阶段,云计算技术在科学、医学等领域得到广泛应用,其具有的优势可以更好地服务于用户。随着基因数据的增长,如何快速、准确生成测序数据是生物信息领域提出的新要求。本文设计一种分层云架构。在此环境下,引入聚类算法生成测序数据。仿真结果表明,该方法生成的数据与基因特征的关联性较强,更加精准体现出基因特性,为海量大规模并行计算提供便捷方式。此外,通过该方法能够揭示出生命本质与规律。因此,云计算已经成为生物信息领域发展趋势。
