基于机器学习的科学数据正式引用识别方法研究*
2022-03-07张志强
杨 宁 张志强
(1.中国科学院成都文献情报中心 成都 610041;2.中国科学院大学经济与管理学院图书情报与档案管理系 北京 100190)
0 引 言
科学数据是指科研活动中产生或经过再加工得到的数据,主要类型包括观察数据、实验数据、统计数据等[1]。一般来说,任意单位的数据都可以称为科学数据,但是有一定范围、可形成完整描述的科学数据集合或科学数据产品才能构成科学数据集[2]。科学数据本身是科研活动过程的投入与产出要素,是科研活动过程的“石油”[3]。科研活动过程就是一个科学数据积累的过程,没有科学数据,就没有科学技术。在数据密集型科研范式的大背景下,科学数据已经成为知识发现和科研创新的重要基础和驱动力,被全球各国视为科技发展的新型战略性资源和科研产出的又一类重要成果。随着长期保存、数据出版、开放共享等工作取得的实质性进展,科学数据的共享和重用行为变得日益普遍和重要,而作为数据共享和重用的关键前提,科学数据引用问题开始得到科研人员的广泛关注[4]。
科学数据引用指科研工作者将科学数据作为论文研究结果的支撑要素,通过参考文献、正文注、脚注、尾注或致谢等形式提供数据来源出处的做法[5]。通常,通过参考文献方式列出的引用被称为科学数据的正式引用,而通过其他方式列出的引用方法,被称为科学数据的非正式引用。数据引用主要具有两个方面的重要作用:一是数据溯源,通过数据引用追溯并获取科学研究的原始数据,重现并验证研究结果,促进数据共享与重用,加快科学研究进程;二是学术评价,数据共享和使用情况,可以作为数据生产者学术产出及数据存储机构服务能力的评价指标[6],丰富并完善科研评价机制。自2011年起,一些国际组织如OECD、ESIP、DCC、DataCite等开始致力于数据规范引用的实践,纷纷制定了科学数据引用规范。2012年底,汤森路透推出了数据引文索引(Data Citation Index,DCI)数据库[7],提供科学数据的引用信息及原文关联地址。
尽管众多科研工作者和国际数据组织对数据引用研究已经做了大量工作,但是由于目前仍然缺乏统一的科学数据引用标准规范、科研人员共享和重用数据缺乏积极性、数据隐私及版权保护机制不健全等因素,造成数据的引用溯源和统计数据存在一定缺失和滞后。格林纳达大学EC3文献计量小组的研究发现,尽管自 2007 年以来 DCI 数据库每年发布的数据集数量和被引次数有所增加,但仍然有约88%的研究数据为零引用[8]。另外,当前科研工作者大多仍旧采用人工方式来识别和抽取科学数据引用,很难适用于大规模文献集的数据引用识别和全学科数据引用研究。
因此,本文引入机器学习技术结合内容分析方法,以生物信息学领域学术论文全文信息作为分析内容,以生物信息学领域科学数据集作为识别对象,对论文参考文献中科学数据集的正式引用项进行抽取和分类,研究基于机器学习方法的科学数据正式引用自动识别方法并进行识别效果评价。
1 相关研究
科学数据引用识别是数据引用研究和影响力评价的基础,研究早期主要通过人工阅读并标注的方法来识别和抽取文献中的数据引用。近年来,随着机器学习、自然语言处理等技术的快速发展,利用文本挖掘来识别文献中的数据引用也开始得到越来越多的关注。当前,科学数据引用识别抽取方法可以归纳为四种主要类型,包括:术语搜索、人工标注、基于规则的识别和基于监督学习的识别[9]。
1.1基于术语搜索的识别方法术语搜索是识别和抽取数据引用最直接的方法,主要利用数据集的名称、描述信息、DOI、URL等信息,通过字符串匹配来实现数据引用识别。Major等[10]通过NASA地球观测系统(EOS)的仪器名称为关键词搜索文献中的数据引用,定量化地揭示了EOS数据产生的学术影响力。Henderson等[11]通过数据集名称、作者、下载地址等信息,搜索并研究了达特茅斯大学CRAWDAD无线数据集在论文中的被引情况。研究发现1 281篇使用CRAWDAD数据集的论文中,大部分都通过较为规范的方式引用数据集,存在的主要问题包括:引用了论文而非直接引用数据集、使用不清晰的标识符来描述数据集以及未提供指向数据集的URL地址等。Li等[12]提出了通过论文全文分析来识别科学项目产生数据被引用情况的工作流,并将工作流应用于癌症基因组图谱(TCGA)项目提供的癌症基因组数据集研究,通过全文文本挖掘识别并分析了TCCA数据集的被引情况。刘小宇等[13]通过平台名称、数据引用声明中的关键词等信息检索文献,调查与分析我国科学数据共享平台所提供科学数据的被引用情况,研究了数据的可回溯性、被引时间规律、被引位置、被引作用与被引句式特点等特征规律。
术语搜索方法具备直接、高效等特点,是当前科学数据引用识别应用较为广泛的一种方法。但术语搜索方法的缺点也较为明显,如需要提前制定检索词、词汇搜索范围较为有限、误检率较高等。因此,术语搜索方法通常用于分析已知数据集的引用识别和影响力研究。
1.2基于人工标注的识别方法人工标注是通过人工阅读文本的方式来识别数据引用的方法,通常需要构建任务语料库来规范识别范围和术语特征,如有多名标注人员参与,还需对标注结果进行一致性检验。Zenk-Möltgen等[14]选取140种社会学期刊的数据政策进行研究,并选取其中5种期刊的论文进行数据引用标注。研究发现社会学领域共享和引用数据的文章较少,大多集中在具有较高影响因子和数据政策的期刊上。Yan等[15]通过人工标注方法在文献中挖掘开放政府数据的引用情况,并分析不同地区研究人员对开放政府数据的使用特点及目的。Zhao等[16]对PLoS One上600份出版物的内容进行分析,对多个学科论文中数据集提及和引用进行抽取和分析。研究发现不同学科的数据集提及和引用差异很大,数据正式引用和数据重用的比例都很低,研究人员更倾向于在正文中引用自己创建的数据集。王雪等[17]对生物信息学领域的中英文文献数据引用行为特征进行分析, 并构建了基于文献计量和网络计量的数据引用行为评价模型。研究认为英文文献的数据引用更为规范且重用率较高, 文献质量与数据集质量之间存在显著的相关关系。丁楠等[18]利用内容分析和人工标注方法,对我国图书情报领域权威期刊数据引用行为进行分析,研究发现我国图情领域期刊的数据引用仍然存在数据引用频次少、数据公开程度低、缺乏统一的数据引用规范等问题。
人工标注的优点是可以通过人工方式产生可靠性较高的结果,标注过程也可以融入更多个性化的需求。然而,由于人工标注方法的效率较低,无法适用于大规模文献集的数据引用识别,对数据进行抽样又会损失大量样本信息。因此,人工标注通常用于小文献集或特定领域文献的数据引用识别。
1.3基于规则的识别方法基于规则的方法主要通过一组静态或动态的规则来识别抽取数据引用语句,这些规则通过领域专家或机器自动推断方式构建,再通过正则表达式等将符合词法、句法规则的数据引用语句进行识别并抽取出来。Ghavimi等[19]从社会科学数据集DOI注册库dalra中检索数据集并通过标题挖掘其规则特征,再利用规则抽取和分析社会科学论文中数据集的引用情况。Grechkin等[20]利用正则表达式在全文中识别和抽取数据集引用,并利用数据集序列号检索数据集状态是公共还是私有,从而自动检测已被发表论文引用但仍然保持私有的过期数据集,加快数据集的公开进度。
基于规则的识别方法精确度高、针对性强,但建立规则库需要领域专家参与。并且该方法具有一定的局限性,普适性规则会造成错误率较高,而针对性规则的灵活性和覆盖性较差。
1.4基于监督学习的识别方法基于监督学习的方法主要采用机器学习分类器,通过标记语料库的部分数据进行训练,然后通过语料库的另一部分数据进行应用和评估。Névéol等[21]提出了一种自动识别生物医学论文中科学数据引用的方法,该方法综合利用条件随机场(CRF)、朴素贝叶斯(NB)和支持向量机(SVM)等机器学习模型和方法,挖掘出PubMed数据库中52 932篇文章的数据引用信息。
基于监督学习的识别方法效率高、泛化能力较强,适用于当前大规模文献集的科学数据引用识别和抽取。但是,由于该方法存在实现技术门槛较高、缺乏通用的数据训练集和测试集等问题,基于监督学习的识别方法的研究和应用还较少。
2 数据与方法
2.1研究思路生物信息学是一门应用计算机科学的方法技术对生命科学大数据进行数据处理分析和知识发现的专门领域学科信息学,是具有代表性的以数据驱动为核心的典型学科信息学研究领域,学科研究文献中涉及到大量的科学数据的共享和引用。根据这一特点,本文以生物信息学领域为例,利用自定义规则和人工方式将参考文献分类标注为数据引用和非数据引用,最后比较各类机器学习方法在科学数据引用分类识别任务中的表现,从而分析判别机器学习方法用于科学数据正式引用自动分类和识别抽取的效果。研究框架如图1所示。
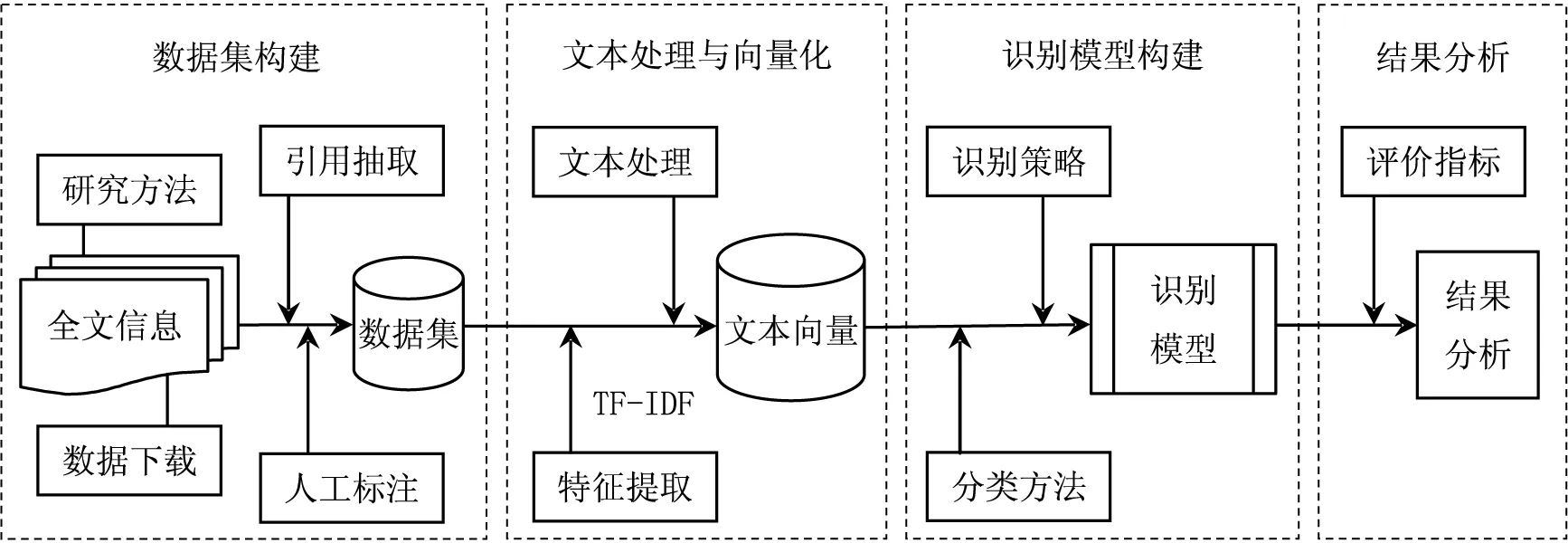
图1 研究框架
2.2数据下载与抽取为使研究具有较好的学科代表性,本研究通过多渠道综合确定生物信息学领域的关键期刊,其中包括期刊引用报告(JCR)、国际计算生物学学会、维基百科和相关的研究论文等[22]。全文文本数据来自PubMed Central(PMC),通过检索期刊名称获取期刊全文条数。为使研究数据具备一定代表性,数据经过去重并筛选掉全文存储篇数小于100篇的期刊,最终确定35种期刊作为研究对象。
数据下载采用生物信息学领域常用的开源工具BioPython[23],该工具包含许多用于生物信息学数据下载和分析的模块,本研究使用其中的Entrez模块进行全文下载,数据下载时间为2021年1月12日,共获取全文数据38 931篇。
PMC数据库的全文数据基于XML格式存储,标准采用美国国立医学图书馆(NLM)的文件类型定义(DTD)标准[24]。XML全文文本由3个部分组成,包括文献基本信息、正文信息和参考文献信息。各部分的标签及主要内容如表1所示。

表1 PMC数据库全文XML标签结构与主要内容
2.3数据预处理经过对数据进行分析得知,参考文献的类型以journal、book和other三种类型为主。其中,标识为other的引用包括了对科学数据、网页、专利、报告等多种数据类型的引用。因此,数据预处理的第一步就是将标识类型为other的参考文献条目抽取出来。另外,由于生物信息学论文涉及到算法和公式较多,文本中存在着大量的LaTeX标记,对数据抽取结果产生较大影响,需要通过正则表达式将无用的LaTeX标记和空格清除。
对于生物信息学领域,科学数据要具备可访问和可重用性才有价值。因此本研究涉及的科学数据要求符合FAIR原则[25],即可发现(Findable)、可访问(Accessible)、可交互(Interoperable)和可重用(Reusable)。在这一原则的规范下,数据预处理的第二步是通过规则将参考文献内容中包含网址或数字对象唯一标识符(DOI)的参考文献条目抽取出来,并分别将引文内容、引文内容与正文中引文所在语句组合保存为最终要分析的文本,经过数据预处理最终共得到15 936条引用文本数据。
2.4数据标注通过对预处理后的数据进行分析发现,引用文本仍然包含各种类型的数据,因此需要通过人工方式进行数据标注。该工作由标注人员完成,其一是具有领域科学数据管理及分析背景的博士;其二是情报学专业博士研究生。在对数据进行分类标注之前,先对二人进行了系统培训,包括解读引用目的、类目归属方法、标注注意事项等。标注规范主要结合引文对象、数据格式、关键词汇及正文中对数据的使用行为进行标注。如果引文对象为数据集,且使用行为包括下载、共享、检索、获取等则标注为数据引用;对于具有使用行为但引文对象不明确的条目,标注人员通过访问URL地址来确定是否为数据引用;其他条目,如明确为其他类型引用、URL地址无法访问、仅为统计数字或作为举例列出等引用条目则不予标注。
在正式进行分类之前随机选择1 000条样本进行练习,对其中分类较为模糊的条目进行讨论并统一,并总结生物信息学常用的各类数据库。由于引用文本类型较多,本文将引用文本数据分为“科学数据引用”及“非科学数据引用”两类,从而将科学数据引用识别转换为一种文本数据向量的二分类问题。部分标引数据如表2所示。

表2 部分参考文献分类标引数据
标引结果的一致性检验采用Carletta的Kappa系数[26],其计算公式为:
(1)
其中,Po代表一致性检验的观察值,而Pe代表一致性检验的期望值,Kappa系数的取值范围是[-1,1]。一般情况下,K≥0.61表明分类结果具有可靠一致性,K≥0.81表明分类结果具有高度一致性。经过对标注结果进行统计计算后,一致性检验的观察值Po和期望值Pe的值分别为0.95和0.74,代入公式后得到Kappa系数的值为0.81,表明标注结果具有高度一致性。最终,经过对不一致结果的分析和讨论,确定了本文的实验数据集。
2.5实验数据集经过预处理和数据标注,本文最终得到由15 936条引用文本构成的实验数据集。其中,数据引用3 067条,非数据引用12 869条。经过统计发现,2001年到2020年间发表的38 931篇论文中有1 570篇存在数据正式引用,其中最多的一篇论文参考文献中包含33项数据集的引用[27]。对数据进行正式引用的论文数量从2009年到2010年间有了一次跨越式增长,一举从36篇增长到125篇,并在此后一直保持着稳定增长的趋势。论文发表总数及具有数据正式引用的论文数量年度分布如图2所示。
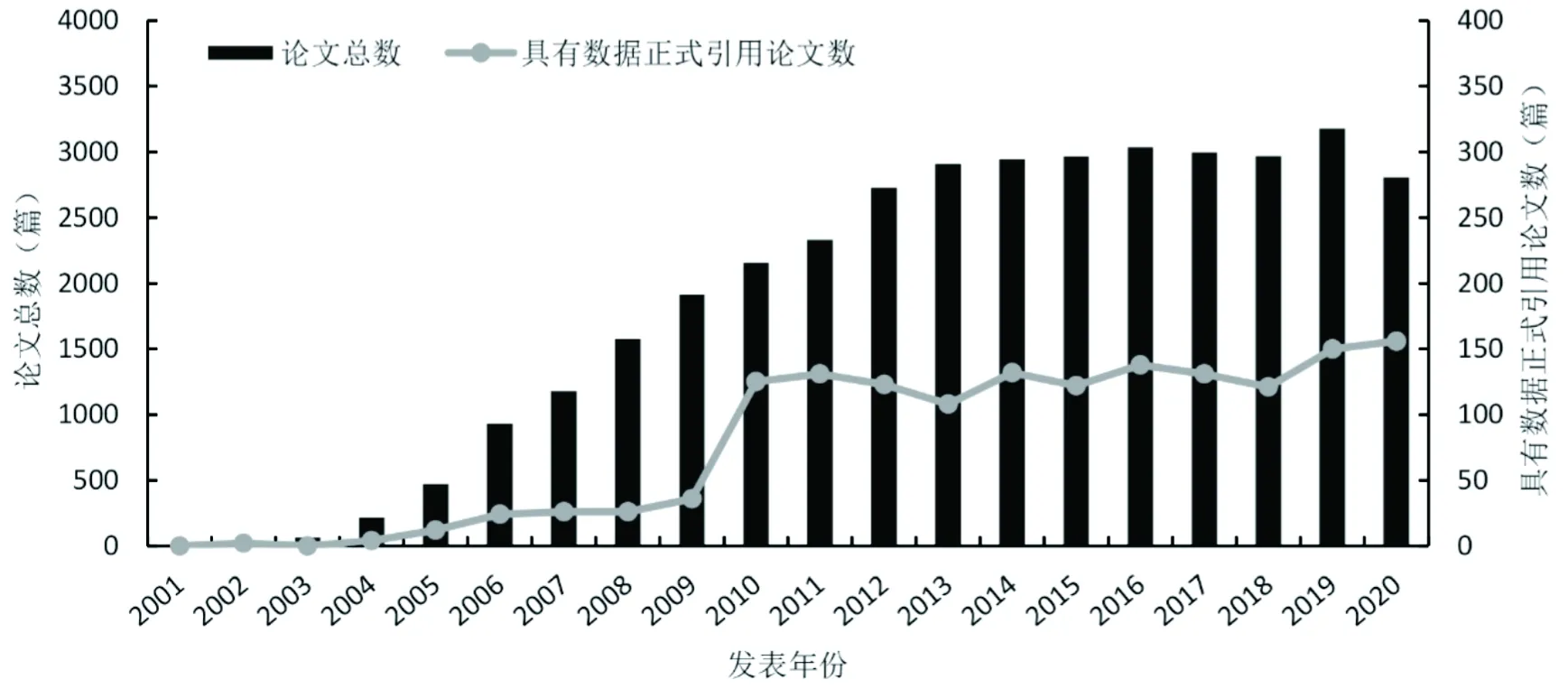
图2 论文发表总数及具有数据正式引用的论文数量年度分布图
2.6文本分类模型本研究采用文本分类任务常用的8种机器学习模型进行实验,以判别机器学习模型在科学数据正式引用识别任务中的应用效果。
2.6.1 朴素贝叶斯 朴素贝叶斯是一种基于概率统计的分类模型,该模型主要利用贝叶斯定理和特征条件独立性假设进行分类。首先分别计算文本中各单词属于某一类别的概率,再根据条件独立性假设最终得到文本属于某一类别的概率。
2.6.2 决策树 决策树是一种经典的用于分类的机器学习模型,该模型首先建立一个树形图,再利用一系列的分类规则对样本点进行逐层判断和剪枝,从而最终实现分类。决策树具有简单快速、计算过程透明等优点,在分类领域应用较为广泛。
2.6.3 随机森林 随机森林是一种集成学习模型,它的基本思想是结合Bagging算法和随机子空间方法,以决策树为基分类器,最终通过组合多个决策树来构建一个集成分类器。随机森林解决了决策树的过拟合问题,并且对噪声和异常值不敏感,能够有效解决数据不平衡问题。
2.6.4 逻辑回归 逻辑回归属于判别式分类模型,它的基本思想是将训练数据转换成对应的结构化数值,将数据拟合进一个逻辑函数来估计文本属于某个类别的概率。逻辑回归的优点是计算消耗资源少,计算结果便于直接观测样本概率分布。
2.6.5 K-近邻 K-近邻属于一种非参数的分类模型,它的基本原理是对于一个给定样本,学习模型会在训练数据中找到与其最相近的k个样本,最后将k个近邻样本中的大多数所属的类别作为该样本的类别,K-近邻算法既可以用于二分类问题也可以应用于多分类问题。
2.6.6 随机梯度下降 随机梯度下降是一种迭代分类模型,它主要用于凸损失函数下线性分类器的判别式学习。该模型可以很好地解决大规模稀疏数据的计算学习问题,在大数据文本分类和自然语言处理中应用较为广泛。
2.6.7 支持向量机 支持向量机是一种建立在统计学习理论基础上的模型,它可以针对有限样本,基于结构风险最小化原理,将实际问题通过非线性变换转换到高维特征空间,学习并最终得到分类决策函数,支持向量机可以在小样本训练集上取得较好的分类效果。
2.6.8 自适应增强 自适应增强是一种迭代提升模型,它的核心思想是利用集成学习技术,针对同一个训练集训练不同的弱分类器,再通过融合这些弱分类器构成一个增强的分类器,并将其作为最终的决策分类器。
3 实验结果与分析
3.1实验方法及评价指标本实验采用五折交叉验证,按照4:1的比例通过类型抽样法将数据分为训练集和测试集,二者不含重复样本。方法模型分别采用引文文本和引文文本结合所在句子两类信息作为输入,分析比较全文信息对分类结果的影响。在文本特征表示过程中,采用基于TF-IDF的向量空间模型进行文本向量化,通过参数调优保留在测试集上效果最好的模型结果。
评价指标采用文本分类任务最常用的精确率(Precision,P)、召回率(Recall,R)和调和平均值(F1-Meature,F1)作为评价指标,并分别计算各指标的宏平均值(算数平均值),对各类机器学习模型在文本实验数据集上的计算结果进行评价。评价指标的计算公式为:
(2)
(3)
(4)
其中,TP表示识别为某类样本中正确的样本数,FP表示识别为某类样本中错误的样本数,FN表示属于某类样本中被识别为其他类别的样本数。
3.2实验结果及分析
3.2.1 不同模型在引文文本分类识别上的效果比较 实验首先利用引文文本进行文本分类并进行结果评价,经过分词后得到文本长度大部分分布在10到60个词区间,分类结果如表3所示。
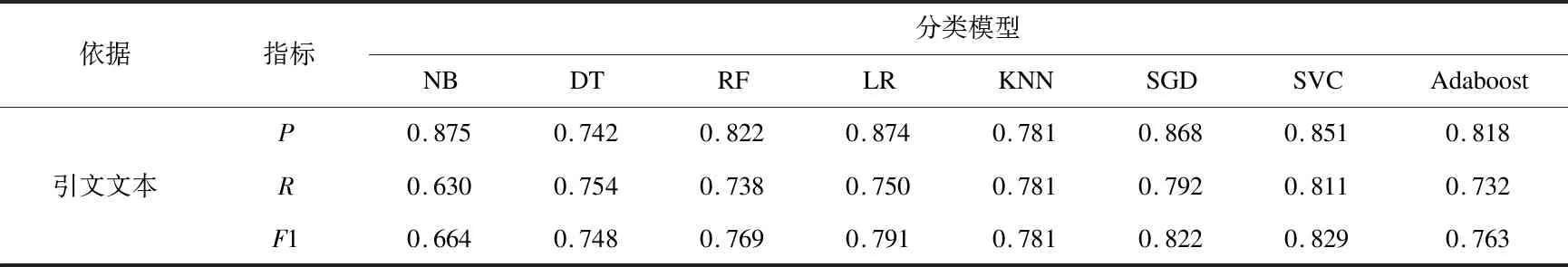
表3 引文文本在不同分类模型中的计算结果
由表3可见,在引文文本自动分类任务中,SVC模型的分类表现最优,精确率和召回率都能达到0.8以上,F1值达到0.829,在所有分类模型中排名最高;SGD模型的表现也较好,F1值达到0.822,仅次于SVC;LR模型的分类效果不及SVC和SGD模型,但是其取得了较高的精确率,分类效果也较好;NB模型的精确率最高达到0.875,但召回率也最差,在此类任务中的效果不够理想;其余DT、RF、KNN和Adaboost模型在引文文本分类任务中的效果都较为一般。
通过结合错误分类实例对分类结果进行更为深入的分析发现,科学数据引用格式不规范问题是导致引用分类效果不佳的主要原因。由于目前还缺乏统一的数据引用标准规范,论文中的数据引用方式仍然较为随意,大部分的引用以列出数据集的URL地址为主。而对于数据集的元数据信息,如作者、出版时间、规范名称、版本号等信息均未列出,从引文文本中能够提取到的特征信息较少。此外,由于训练集和测试集样本规模较小,SVC在小样本分类任务中的优势体现的较为明显,而SGD模型的普适性较强,在各种规模数据集上都可以取得较好的分类效果。由于数据集引用的特征词如“download”“obtain”等都在正文中才会得到体现,因此下一步的实验将利用全文信息对数据集引用的特征进行增强。
3.2.2 全文信息对文本分类结果的影响 由于引文文本所能提供的信息十分有限,导致各分类模型中的表现较为一般。为进一步提高分类效果,本研究尝试利用全文信息丰富输入文本,将引文文本结合其所在语句作为模型输入进行分类计算。例如,引文文本为“NCBI dataset of human mRNA genes. ftp://ftp.ncbi.nlm.nih.gov/refseq/H_sapiens/mRNA_Prot/.”,其所在句为“For simulations we use the dataset of human mRNA genes downloaded from NCBI.”,最终的分类模型输入文本为“For simulations we use the dataset of human mRNA genes downloaded from NCBI [NCBI dataset of human mRNA genes. ftp://ftp.ncbi.nlm.nih.gov/refseq/H_sapiens/mRNA_Prot/].”。经过分词后得到文本长度大部分分布在10到100个词区间,结合全文信息的引文文本在不同分类模型的计算结果如表4所示。
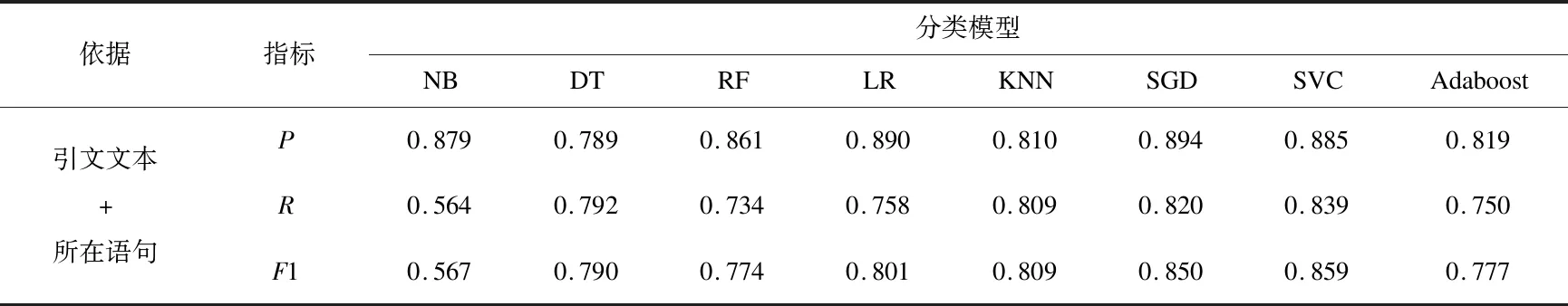
表4 结合全文信息的引文文本在不同分类模型的计算结果
由表4可见,经过输入文本优化后的分类结果中,依然以SVC模型的分类表现为最优,精确率、召回率和F1值都得到了一定的提高,在所有分类模型中仍然排名最高;SGD、KNN、LR模型的分类效果也得到了较为明显的提升,F1值均超过了0.8;其余分类模型的分类效果也都不同程度的提高,只有NB模型的F1值进一步降低。通过深入分析发现该模型受输入数据的表达形式和均衡性影响较大,在样本较少的分类训练结果中表现较差。按照模型F1值进行排序,得到图3所示的8种模型在利用全文信息优化前后的F1值变化对比结果。
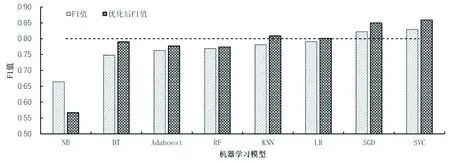
图3 利用全文信息优化前后F1值变化对比图
3.2.3 样本集数量对文本分类结果的影响 由于目前还缺乏可用的数据引用标注数据集,引文文本自动分类首先需要利用人工标注的数据进行训练。因此,方法模型在小样本数据下的分类效果也是科学数据引用识别方法选取的关键。实验计算了8种模型从零样本到全样本的F1值变化情况,得到了样本集数量对文本分类结果影响变化对比结果,如图4所示。
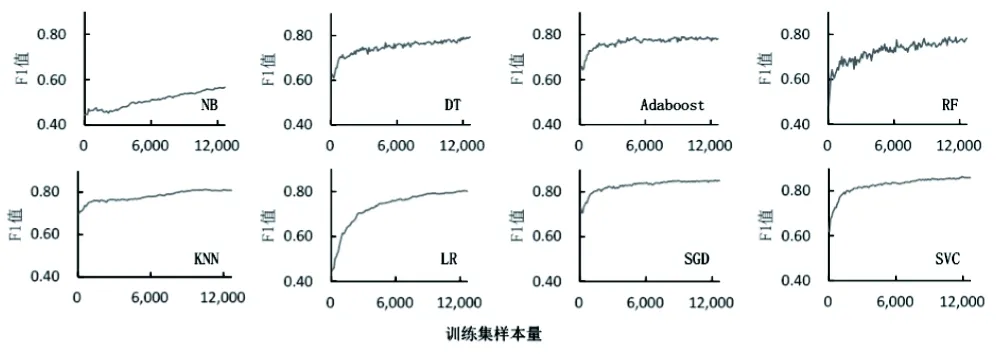
图4 样本集数量对文本分类结果影响变化对比图
由图4可见,8种模型在样本量增加的情况下分类结果都得到了提升。在样本量小于3 000的情况下,SGD模型的F1值达到0.825,超过SVC模型的0.815,并且这种优势一直保持到7 000样本量的时候被SVC模型超越,说明SGD模型在小样本量的情况下就可以得到更好的分类效果。此外, KNN和Adaboost模型在样本量3 000的时候F1值也分别达到0.764和0.757,后续增长则较为平缓,说明二者在小样本量的情况也能取得不错的分类效果,但随样本量增长得到的分类效果提升较为缓慢。而DT、RF和LR模型受样本量增长的影响较大,比较适合用于较大规模样本的数据分类。
4 结果讨论及展望
本文针对科学数据正式引用的识别问题,以生物信息学领域核心期刊学术论文全文信息作为研究对象,利用文本抽取和人工标注形成了生物信息学科学数据正式引用数据集。并通过将科学数据引用识别转化为文本分类问题,对比评估了机器学习领域8种经典分类方法模型在数据集上的分类效果。研究结果表明,SVC和SGD模型在生物信息学领域科学数据正式引用的识别效果最优、全文信息尤其是引文所在句子对数据引用文本分类的提升效果较为明显、SGD模型在小样本数据上的表现要优于SVC模型。
就全文来看,本研究在科学数据识别方法上进行了有效的尝试,但也存在一定的局限性。首先,相关研究领域目前还缺乏可用的权威数据集,本研究虽然通过两人标注并进行一致性检验的方式确定了实验数据集,但由于专业领域背景和认知局限,标注结果难免存在一定的主观性,还需进一步咨询领域专家完善数据集。其次,本研究只涉及了机器学习领域的方法和模型,而近几年深度学习技术发展迅速,BERT、XLNet等模型层出不穷并取得了巨大的进步,下一步工作中将研究基于深度学习的科学数据正式引用识别方法,以期获得更优的识别效果。最后,本研究的结果再次证明科学数据在论文中仍然以提及等非正式引用方式进行标注,科学数据的正式规范引用亟待发展与完善,而这需要首先加大对数据引用状况的识别研究,提高科技界对数据规范引用的重视,并不断推动科学数据规范引用规则的研究与制定,以促进科学数据的规范引用、提高科学数据的价值、提高科研人员共享和重用数据的积极性,这需要科技界的共同努力。
