基于家蚕中肠、脂肪体转录组数据的SNP/Indels位点分析
2022-03-03高妍夏孙志超李季生贾漫丽
王 晖, 高妍夏, 孙志超, 王 敬, 李季生, 李 娜, 黄 露, 贾漫丽, 谢 岩
(承德医学院蚕业研究所/河北省高校特产蚕桑应用技术研发中心,河北承德 067000)
家蚕是一种重要的经济昆虫,同时也是鳞翅目的模式物种。许多古籍记载、现代分子生物学研究均表明,家蚕起源于我国,并逐渐扩散到亚洲、欧洲等地,经过不断地驯化、育种,形成许多品种、品系。不同品种的家蚕体色、个体大小、蚕茧颜色等均存在一定的差异。
分子标记在DNA水平表现为多态性,在动物遗传方面发挥着重要作用。常用的分子标记有扩增片段长度多态性 (amplified fragment length polymorphism,AFLP)、随机扩增多态性DNA标记(random amplified polymorphic DNA,RAPD)、简单重复序列(simple sequence repeat,SSR)、单核苷酸多态性(single nucleotide polymorphism,SNP)、插入缺失(insertion-deletion,Indel)等。SNP是DNA水平的单个核苷酸的改变而产生的多态性;Indel则是DNA水平插入或删除从1 bp至数百bp长度的片段而形成的基因多态性。刘伟等挖掘梯棱羊肚菌全基因组的SNP/Indel位点,选择单胞菌株群体,初步构建Indel标记的遗传连锁图谱。SNP和Indel位点可以鉴定国内不同优良地方鸡种基因的同源性。对于家蚕品种分子水平的鉴定,前期已经有了一定的研究报道。通过RAPD、SSR分子标记初步判定一些家蚕品种之间的多态性和亲缘关系。针对家蚕抗血液型脓病新品种混乱的情况,钱荷英等开发了50个SNP分子标记,初步判断这些SNP分子标记可作为鉴定抗病品种的分子标记。本研究对河北省常用家蚕品种的中肠、脂肪体组织进行转录组测序,挖掘基因中的SNP/Indel位点,并对其分布规律进行分析,以期进一步丰富家蚕的SNP/Indel位点数据库,为家蚕优良品种选育、亲缘关系鉴定等提供参考。
1 材料与方法
1.1 试验材料
家蚕白色茧品种东肥(DF,下同)、米色茧品种彩4(C4,下同)幼虫于2019年6月饲养于承德医学院蚕业所养蚕室,环境条件:温度(25±2) ℃,湿度60%~70%,自然光周期。7月份时,解剖5龄成熟期生长一致家蚕蚕体,分离收集DF、C4的中肠(MG,下同)与脂肪体组织(FB,下同),每个样本3个生物学重复,液氮速冻,转录组测序工作由北京诺禾致源生物科技有限公司完成。
1.2 试验方法
使用RNA提取试剂盒提取总RNA,琼脂糖凝胶电泳、Nano Photometer 分光光度计检测RNA的纯度,Agilent 2100 生物分析仪检测RNA的完整性。高通量测序仪测得的图像数据经 CASAVA 碱基识别转化为序列数据(reads),去除低质量reads后获得clean data。使用 GATK(3.7)软件对样本数据进行变异位点分析,并用 SnpEff(4.3q)软件对变异位点进行注释。通过 clusterProfiler(3.4.4)软件实现差异表达基因的 GO富集分析,分析KEGG 通路中差异表达基因的统计富集。使用Origin 2021b软件作图。
2 结果与分析
2.1 转录组测序结果
由表1可知,2个品种家蚕的脂肪体、中肠经转录组测序后共组装得到17 915条unigene序列,总长度为20 545 285 bp,C4的中肠GC含量范围为50.81%~51.29%,其他组织样品的GC含量范围为47.39%~49.81%;Q20均大于97%,Q30均大于92%,转录组数据可以用于后续分析。

表1 2个家蚕品种脂肪体、中肠转录组测序质量统计
2.2 SNP位点检测
在2个品种家蚕中,脂肪体组织的SNP位点数目都小于中肠。C4脂肪体平均检索到69 756个SNP位点,中肠平均检索到99 490个SNP位点;DF脂肪体平均检索到64 676个SNP位点,中肠平均检索到99 910个SNP位点(图1-A)。C4脂肪体、中肠每个unigene上的平均SNP数量分别为5.84、7.69个;DF脂肪体、中肠每个unigene上的平均SNP数量分别为5.31、7.31个。

C4脂肪体SNP位点数量高于DF脂肪体;但是C4中肠SNP位点数量低于DF中肠。所有组织样品的SNP位点类型,转换平均数目均高于颠换。C4脂肪体转换、颠换平均数目分别为45 302、24 454个;中肠转换、颠换平均数目分别为64 003、35 486个。DF脂肪体转换、颠换平均数目分别为41 494、23 182个;中肠转换、颠换平均数目分别为62 920、36 990个。A/G、C/T 2种转换类型在所有SNP类型中所占比例最高,颠换类型中则是A/T占比最高(图1-B)。
2.3 Indel位点分布特征
在2个品种家蚕中,脂肪体组织的Indel位点数目都小于中肠。C4脂肪体、中肠每个unigene上的平均Indel数量分别为0.34、0.50个;DF脂肪体、中肠每个unigene上的平均Indel数量分别为0.33、0.56个(图2)。在C4脂肪体平均检测到4 081个Indel位点,包括2 373个插入突变和1 708个缺失突变。碱基插入和缺失突变的范围分别为1~24、1~67 bp,其中单核苷酸插入、缺失分别占所有Indel位点数目的35.78%、21.95%。C4中肠平均检测到6 452个Indel位点,包括3 797个插入突变和2 655个缺失突变。碱基插入和缺失突变的范围分别为 1~60、1~179 bp,其中单核苷酸插入、缺失分别占所有Indel位点数目的36.66%、22.41%。在DF脂肪体平均检测到 4 082个Indel位点,包括2 427个插入突变和1 655个缺失突变。碱基插入和缺失突变的范围分别为1~21、1~108 bp,其中单核苷酸插入、缺失分别占所有Indel位点数目的38.99%、21.64%。DF中肠平均检测到7 601个Indel位点,包括4 566个插入突变和3 035个缺失突变。碱基插入和缺失突变的范围分别为1~33、1~129 bp,其中单核苷酸插入、缺失分别占所有Indel位点数目的39.71%、21.43%(图3-A、图3-B)。


2.4 SNP/Indel位点在基因组上的分布
SNP/Indel位点在家蚕基因组上分布于8个区域,在下游区分布的位点数最多,占比为28.15%~30.11%;其次是外显子、基因间隔区、上游区,占比依次分别为25.80%~31.62%、23.18%~27.29%、12.11%~13.60%;占比最少的是供体剪接位点、受体剪接位点,几乎可忽略不计(图4)。

2.5 GO功能注释
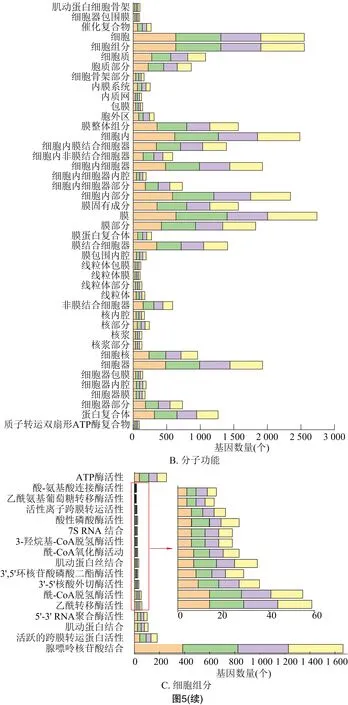
通过对含有SNP/Indel位点的基因进行GO功能注释,可分为三大类,即生物学过程、分子功能、细胞组分。富集在生物学过程的通路主要有代谢过程、细胞过程、有机物代谢过程、主要代谢过程等(图5-A)。富集在分子功能的通路主要有膜、细胞、细胞组分、细胞内等(图5-B)。富集在细胞组分的通路主要有腺嘌呤核苷酸结合、活跃的跨膜转运蛋白活性、肌动蛋白结合等(图5-C)。

2.6 KEGG 功能注释
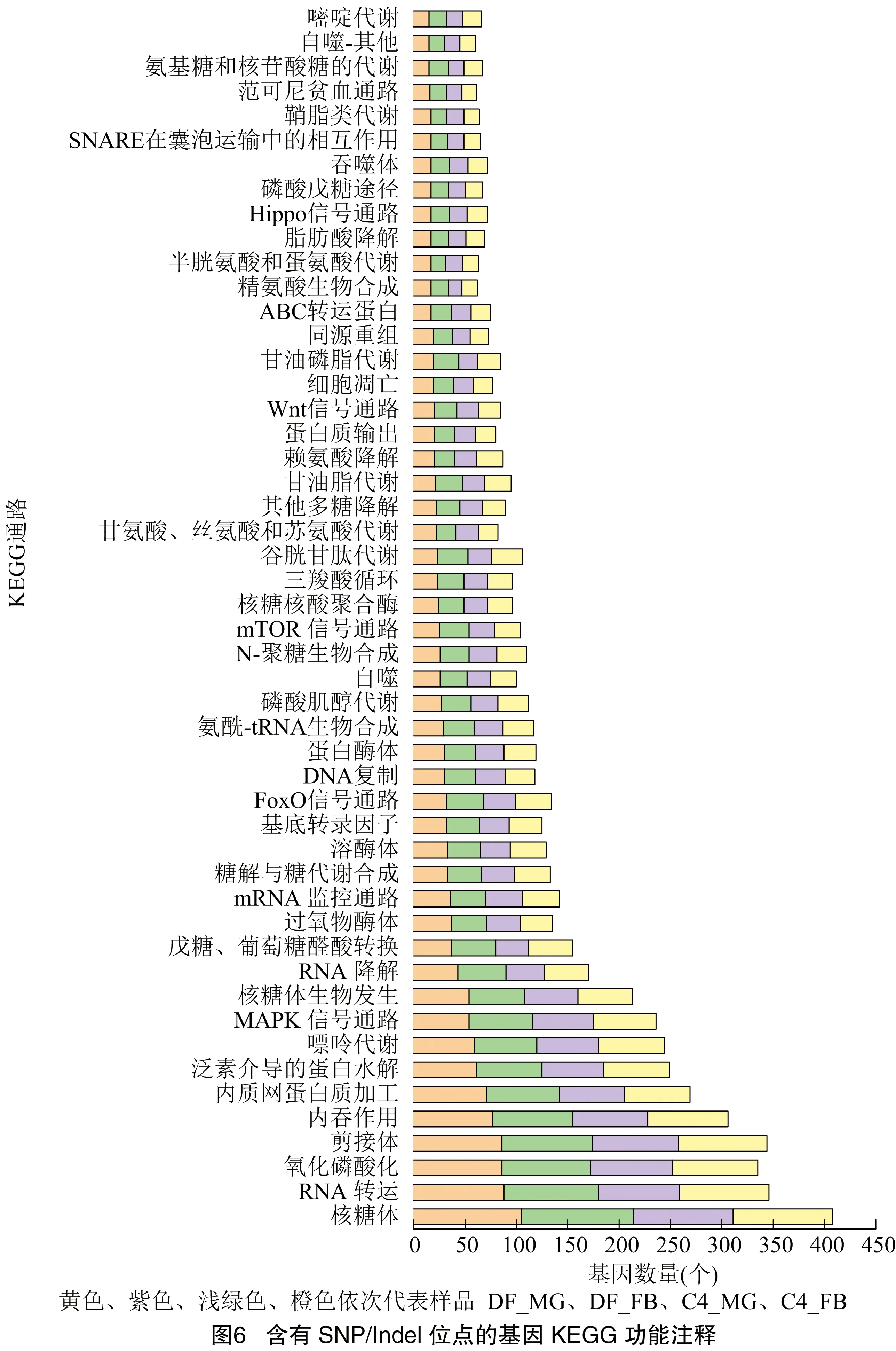
含有SNP/Indel位点的基因进行KEGG功能注释后,发现大多数基因主要富集在核糖体、RNA转运、氧化磷酸化、剪接体、内吞作用、内质网蛋白质加工等与物质代谢、能量代谢紧密相关的代谢通路(图6),这也与上述GO注释的结果相一致。
3 讨论与结论
本研究通过2个品种家蚕的中肠和脂肪体的转录组测序发现,2个家蚕品种的脂肪体均检索到6万多个SNP位点,4 000多个Indel位点;中肠则存在9万多个SNP位点,6 000多个Indel位点。余东亮等比较家蚕品种P50与C108后部丝腺的SNP/Indel位点,共发现1 584个SNP位点,2 776个Indel位点,结合本研究结果,推测SNP/Indel位点的多少主要与组织类型、品种有关。C4中肠SNP、Indel的出现频率分别为1/207、1/3 184 bp,脂肪体SNP、Indel出现频率分别为1/295、1/5 034 bp;DF中肠SNP、Indel的出现频率分别为1/206、1/2 703 bp,脂肪体SNP、Indel出现频率分别为1/318、1/5 033 bp。东海带鱼肝脏转录组序列平均每76.8 bp出现1个SNP;人参果则是约103 bp出现1个SNP位点。波纹唇鱼肝胰脏、食道、前肠、后肠和直肠转录组unigene中SNP的发生频率为1/490 bp;椰心叶甲啮小峰转录组数据中平均每1 000 bp出现1个SNP位点;可见SNP位点的出现频率在不同物种之间差异较大。家蚕中肠和脂肪体SNP位点则以C/T、A/G等2种类型为主,其余4种类型数量相近,这与其他物种的研究报道一致。SNP的转换与颠换类型之比为1.69~1.89之间,远大于理论值0.5,这种现象被称为转换偏差,其在许多物种中广泛存在,这可能与物种适应进化有关。
从家蚕中肠、脂肪体转录组数据中筛选到了SNP/Indel位点信息,通过对包含有SNP/Indel位点的uningene进行GO、KEGG功能注释,可以初步分析家蚕品种、个体、组织之间的差异代谢途径和通路,从而可能将SNP/Indel位点与表型进行关联,开发出特定的分子标记,将来进一步为开展分子标记辅助家蚕育种研究、品种鉴定、亲缘关系分析等奠定基础。
