基于16S rRNA基因对濒危脊椎动物鉴定的可行性
2022-02-24蒋晓霞徐博文李伟刚邵建宏周傲白雪唐泰山赵福振黄海超罗宝正
蒋晓霞 徐博文 李伟刚 邵建宏 周傲白雪 唐泰山 赵福振 苗 丽 黄海超 罗宝正*
(1.拱北海关技术中心,珠海,519001;2.南京海关动植物与食品检测中心,南京,210019;3.郑州海关技术中心,郑州,450003)
在世界范围内,野生动物及其制品在食用、药用、收藏、宠物饲养等领域普遍存在[1],旺盛的市场需求导致针对野生动物及其制品的非法贸易行为频发[2]。多年以来,我国海关公布的野生动物走私案件涉及兽类(Mammalia)、鸟类(Aves)、两栖类(Amphibia)、爬行类(Reptilia)等109种,象牙及其制品、穿山甲(Manisspp.)及其制品的非法贸易多有发生[3]。在预防和处置走私濒危野生动物及其制品行为的过程中,对物种的识别和鉴定是核心要素。脊椎动物是地球上结构最复杂、进化地位最高的生物类型,目前,针对脊椎动物鉴定的国家标准和行业标准均以既定物种鉴定为目标,采用单一物种鉴定方法,如果样品背景不明,难以选择恰当方法,鉴定费时费力。因此,迫切需要建立一种对濒危脊椎动物鉴定的通用物种鉴定方法。
DNA条形码鉴定方法于2003年由Hebert等[4]提出,其应用领域涵盖濒危野生动物保护、入侵物种识别、食品和中药材鉴定等。目前,已有研究人员利用DNA条形码对穿山甲[5]、羚羊角[6]等既定物种及其制品进行鉴定,但根据研究对象生物学特性以及不同的研究目的建立的条形码,较难实现对背景不明的濒危脊椎动物的快速鉴定。本研究以濒危脊椎动物样本为研究对象,通过扩增16S rRNA中短的基因片段并测序,将GenBank中BLAST序列比对结果、遗传距离计算结果相结合,探讨该基因片段用于濒危脊椎动物鉴定的可行性,以期解决濒危脊椎动物鉴定时间长、成本高的问题,为海关、森林公安、海警、市场监督管理等部门执法提供快速、准确的物种鉴定方法。
1 材料与方法
1.1 样本的采集
研究材料均来源于海关走私罚没的已知物种的肌肉、角、牙、毛皮、骨骼和鳞片,共计71个(表1)。
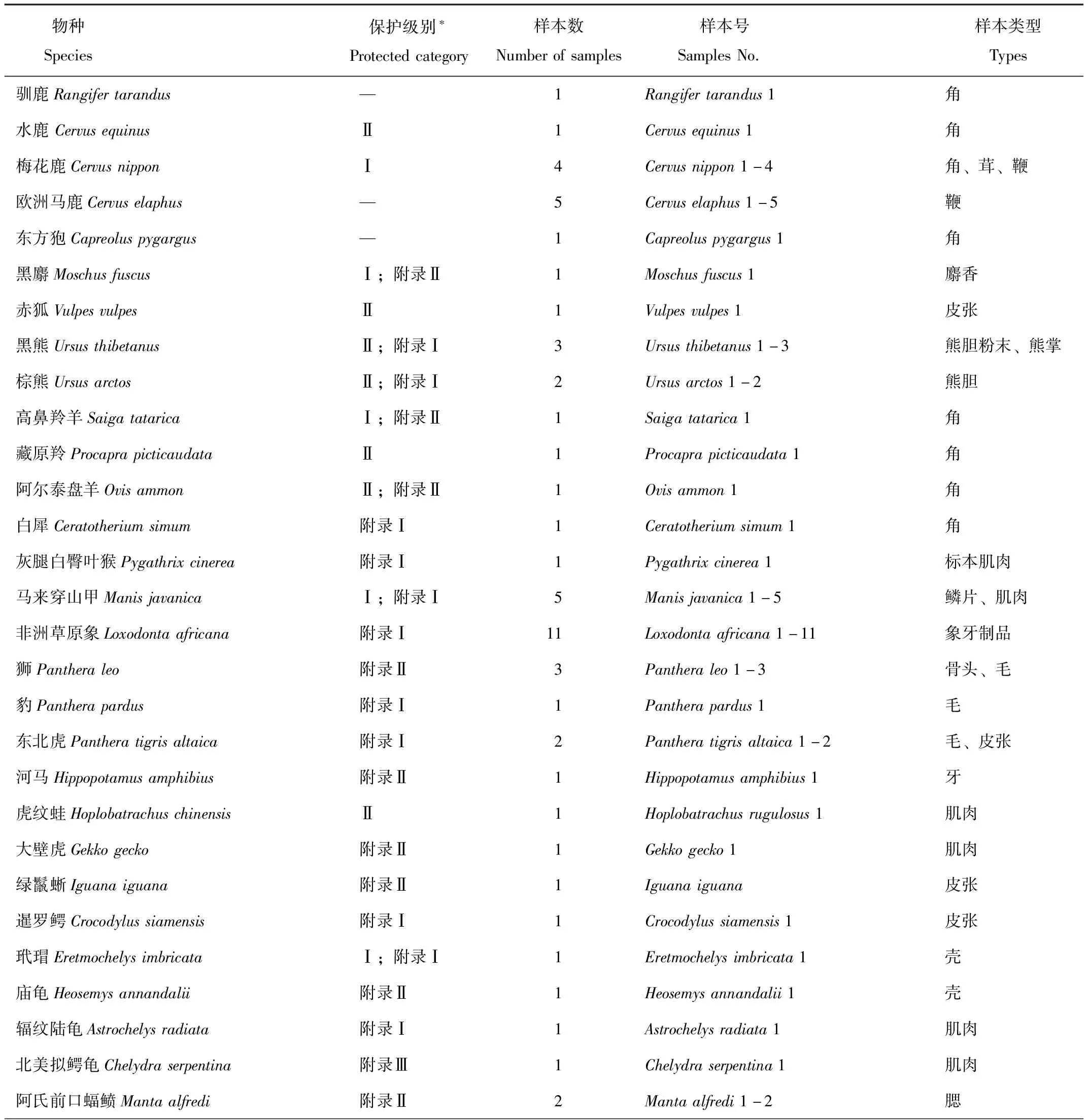
表1 研究样本信息
1.2 样本预处理
用酒精棉擦拭各待检样本表面,室温干燥后使用紫外灯照射30 min,清除其上可能附着的影响检验结果的异物,以提高检验结果的准确性。针对不同状态的样本,采用不同的取样方法。肌肉、毛和皮张使用手术剪取样约30 mg,置于1.5 mL离心管中;动物角、动物牙、动物骨骼或鳞片使用无菌手术刀或电钻取粉末约30 mg,置于1.5 mL离心管中,加入1.0 mL 0.5 mol/L乙二胺四乙酸(ethylene diaminete traacetic acid,EDTA)充分混匀,37 ℃孵育(或室温放置)12~24 h后进行脱钙处理,12 000g离心5 min,丢弃上清液,保留沉淀物。
1.3 PCR引物的合成
根据GenBank中公布的脊椎动物线粒体全基因序列,应用生物学软件DNAMAN进行多序列比对,使用Oligo 7.0在高度保守区域设计1对濒危脊椎动物通用PCR引物,兼顾扩增子序列体现物种多样性信息。上游序列:5′-ACAAATAAGACGAGAAGACCCT-3′,下游序列:5′-TGATCCAACATCGAGGTCGTAA-3′,熔解温度55 ℃,由上海辉睿生物科技有限公司合成。
1.4 样本的DNA提取及PCR扩增
所有样本的基因组DNA提取均采用商品化DNA提取试剂盒(E.Z.N.A.Tissue DNA Kit,Omega,美国),具体方法参考试剂盒说明书。将皮毛、骨、角、牙和鳞片的消化时间均延长至12 h以上,提取后的DNA置于冰箱中-20 ℃保存备用。将样本DNA作为模板进行PCR扩增,每个PCR反应体系25 μL,体系组成为:2×TaqPCR Master mix 12.5 μL(天根生化科技(北京)有限公司),10 μmol/L上下游引物各0.5 μL,补双蒸水至23 μL,目标扩增模板2.0 μL。反应程序为:94 ℃预变性4 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸7 min。扩增产物于1.5%琼脂糖凝胶电泳检测,条带清晰的产物采用Sanger法进行双向测序,由上海生工和拱北海关技术中心动物检疫实验室完成。
1.5 序列比对和分析
将所测得的序列使用美国国家生物技术中心(NCBI)GenBank数据库搜索工具BLAST[9]进行同源性比较,以确定所测序列是否正确。通过MEGA 7.0软件,对测序所得基因序列(剔除经BLAST比对无法确定的物种)和从GenBank数据库下载与扩增引物对应的扩增子16S rRNA基因序列进行比对,基于Kimura-2-parameter(K2P)模型,选择Transition substitution(Ts)替换类型计算各物种的种内和种间遗传距离[10-11]。
1.6 PCR检测灵敏度试验
以新鲜牛肉为代表样本,测试所建立PCR方法的扩增灵敏度。按上述方法提取DNA后,使用NanoDrop®DN-1000分析仪(NanoDrop Technologies,美国)测定新鲜牛肉的DNA质量浓度为266 ng/μL(A260/A280=1.91)。将其稀释为100 ng/μL,再进行10倍梯度稀释,共稀释7个质量浓度,分别为:10、1、0.1、0.01、0.001、0.000 1、0.000 01 ng/μL,用上述模板进行PCR扩增和电泳。
2 结果与分析
2.1 PCR扩增产物琼脂糖凝胶电泳结果
71个脊椎动物样本提取的DNA,40种(62个样本)脊椎动物PCR扩增成功,产物大小约286 bp(图1)。9个未扩增成功,分别为象牙样本8个、绿鬣蜥(Iguanaiguana)皮张样本1个。
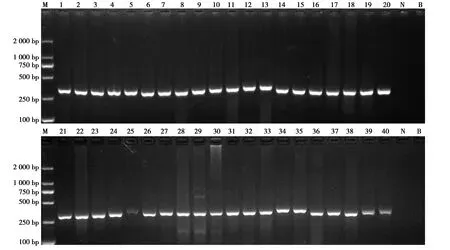
图1 40种脊椎动物样本的PCR扩增电泳结果
2.2 基于BLAST比对的物种鉴定
将测序所得62条基因片段在GenBank数据库进行BLAST同源性比对分析。结果表明,57个样本基因测序比对结果与GenBank数据库中的参考序列具有较高相似度(98.21%~100.00%),测得的序列为16S rRNA基因真实的序列。其中,东方狍(Capreoluspygargus)和西方狍(Capreoluscapreolus)、辐纹陆龟(Astrochelysradiata)和马达加斯加陆龟(Astrochelysyniphora)、阿氏前口蝠鲼(Mantaalfredi)和双吻前口蝠鲼(Mantabirostris)、直齿真鲨(Carcharhinusbrevipinna)和灰色真鲨(Carcharhinusobscurus)、吻海马(Hippocampusreidi)和管海马(Hippocampuskuda)的16S rRNA基因中所测片段序列相似性相同,5个样本基因测序结果在属内种间高度一致,只能鉴定到属的水平。
2.3 遗传距离分析
在两两个体遗传距离比较中,共141条286 bp的16S rRNA基因序列参与遗传距离分析,其中从GenBank数据库下载与所测序列相似性相同的16S rRNA基因序列84条,与测序所得基因序列57条。结果表明,种内遗传距离0~0.5%,种间遗传距离1.0%~39.9%,最小种间遗传距离大于最大种内遗传距离,存在明显的条形码间隔。鹿属(Cervus)的种间遗传距离为1.1%~4.4%,最小种间遗传距离为梅花鹿(C.nippon)与欧洲马鹿(C.elaphus),为1.1%;棕熊属(Ursus)的种间遗传距离为1.6%~2.7%;鹿科(Cervidae)的属间遗传距离为2.7%~6.8%;牛科(Bovidae)的属间遗传距离为2.7%~5.6%。可见,分类阶元高的种间遗传距离平均值较分类阶元低的大(表2)。
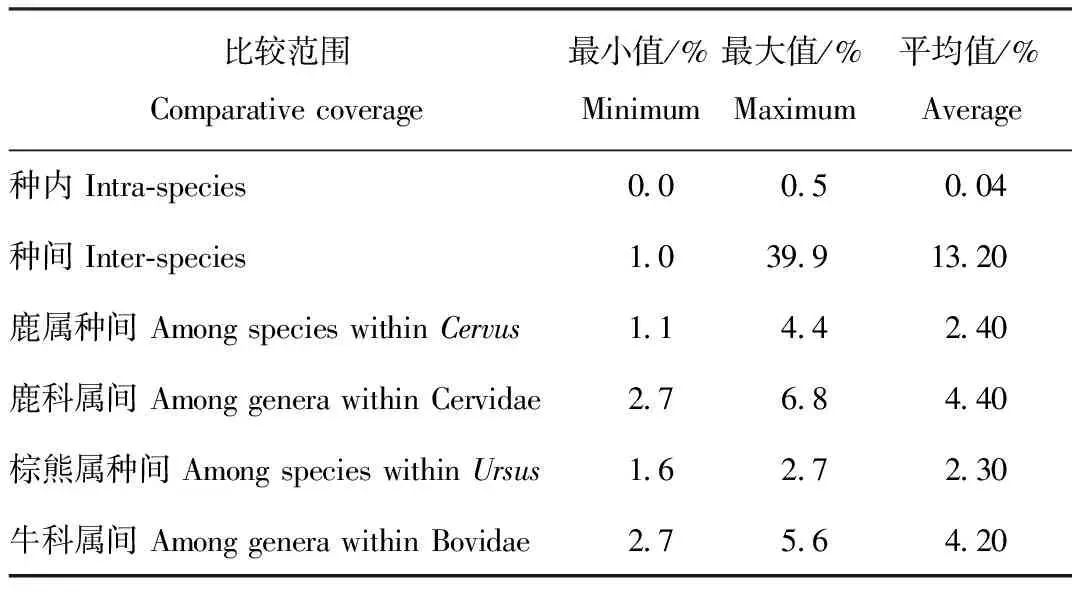
表2 不同分类阶元遗传距离(K2P)统计
2.4 PCR扩增方法的灵敏度测试结果
PCR扩增产物电泳条带的亮度随DNA模板质量浓度的降低逐渐下降,质量浓度为0.001 ng/μL以上的DNA均可见清晰的286 bp条带(图2)。

图2 牛源性成分DNA质量浓度灵敏度的PCR扩增结果
3 讨论
DNA是分子生物学检测的基础,对样本表面进行清洁或直接选取深层组织是试验必需的步骤,针对骨类或与之相似的较坚硬的样本应进行细化以便于水解消化[12],经过脱钙处理的样本能够扩增得到目的条带,而未经过脱钙的样本无法正常扩增[13]。本研究所涉样本种类繁杂,针对角、牙和骨类制品,如梅花鹿角、象牙和龟壳等均进行12 h以上的脱钙处理;针对动物角、毛、牙、鳞片及其制品等,均使用无水乙醇擦拭消毒,充分干燥后再经紫外灯照射,最大限度复原本质,提取高纯度DNA。要在相对独立的房间内进行DNA提取和PCR扩增,同时加强室内通风,以防止DNA交叉污染。由于通用引物扩增时极易发生DNA交叉污染,若在研究过程中未采取有效的防交叉污染措施,将可能导致错误的鉴定结论。
根据本研究,71个样本中有62个样本成功扩增,表明本研究设计的引物通用性较强、扩增效果较好,适用于不同组织、不同状态的样本鉴定,包括已经风化的动物标本。然而,部分工艺品特别是一些深加工品,在加工过程中受高温、高压、辐射等物理因素及酸碱、添加剂等化学因素的影响,其DNA已发生不同程度、不可逆转的变性、断裂或降解[14],这不可避免地制约了本方法的适用范围。有的样品中DNA含量极低,如本研究中的部分象牙工艺品和蜥蜴皮张,提取DNA后达不到PCR扩增需要的核酸浓度。
GenBank数据库中DNA信息储备量庞大、内容丰富,能够为相关研究提供必要支撑。对经测序获得的DNA序列进行BLAST同源搜索比对,是动物种属鉴定的必要步骤[15]。本研究建立的DNA条形码技术能够区分近缘脊椎动物的物种,如梅花鹿和马鹿,但其中5个样本的测序结果经BLAST比对后分别得出2种可能结果,表明建立的方法对于部分物种近缘种鉴定还有一定的局限性。已有的研究结果[16-17]显示,东方狍和西方狍为近缘种,由于分子标记序列长度有限,加之序列相似度很高,使用BLAST比对法无法进行区分;刘大伟等[17]认为对于近缘物种的鉴定,需结合其他分析方法以确保鉴定结论的可靠性,可采用基于COⅠ和Cytb两个基因片段的不同方法进行鉴定。
近年来,种间的最小差异和种内的最大差异被用作DNA条形码间隔,比平均种内和种间差异更加有效[18]。本研究中,种内的最大K2P遗传距离为0.5%,种间的最小K2P遗传距离为1.0%,两者存在间隔区域,且分类阶元高的种间遗传距离平均值较分类阶元低的大,初步表明该序列可用于濒危脊椎动物的物种鉴定。此外,由于同科和同属的样本数量较少,后续研究中有必要扩大采样量以进一步验证该条形码的适用性和可行性。同时,为更好地体现适用性和灵敏度,可与基于COⅠ和Cytb基因序列的DNA条形码进行对比研究。
