浮游动物DNA宏条形码多样性监测采样方法研究
2022-02-22张靖雯杨江华张效伟
张靖雯,杨江华*,张效伟,2
(1.南京大学环境学院,污染控制与资源化研究国家重点实验室,江苏 南京 210023;2.江苏省环境保护化学品安全与健康风险研究重点实验室,江苏 南京 210023)
0 前言
人类活动产生的环境污染、栖息地破坏、土地类型的改变等对生态系统的物种组成产生巨大影响[1],进而导致整个生态系统稳定性下降,生态功能和服务受损。环境管理中,高效准确地监测物种多样性对及时了解水生生态系统的健康状况至关重要[2-3]。目前,对生态系统中生物组成的了解主要依靠传统的形态学物种调查方法,即先将物种从环境中分离,再根据其特定的分类学特征鉴别物种,整个过程极其耗时、耗力,并严重依赖鉴定专家的物种鉴定经验。DNA宏条形码技术作为一种准确、高效的新型生物监测手段,主要利用环境中残存的生物DNA序列识别物种,在河流、湖泊、海洋等生态系统的生物监测中被广泛应用[4-7]。尽管DNA宏条形码技术被越来越多的学者所接受,但是由于缺乏统一的规范化操作流程,导致跨实验室的结果难以横向对比,严重阻碍了该技术在环境管理中的应用,因此亟须针对不同生物类群建立统一规范的DNA宏条形码分析方法。
浮游动物是水生生态系统的重要组成部分,是物质和能量从初级生产者(藻类)到更高营养级(鱼类)物种传递的纽带,对维持水生生态系统稳定具有重要意义[8]。浮游动物生活周期短,对环境因子(如物化参数、污染胁迫等)较为敏感,是水生生态健康状况的重要指示生物[9-10]。目前,依赖形态学物种鉴定的生物调查是淡水生态系统生态健康评估的重要依据。但传统调查难以辨别处于发育早期,分类特征尚未形成的桡足类浮游动物,无法准确反映浮游动物群落的物种组成。而DNA宏条形码技术主要基于物种间特定DNA序列差异来区分物种,采样便捷、准确性高,被广泛应用在生态学领域的诸多研究中[11],如新物种发现、生物多样性评估[12]、生物捕食关系分析[13]及食物网的结构特征分析等[14],也为浮游动物群落组成的精准调查提供了新的机遇。
虽然DNA宏条形码基本检测单位是DNA分子,这与传统调查以生物体为基本检测单位明显不同,但是 DNA宏条形码监测中的样品采集方法主要参照传统形态学调查的采样方法[15-18],即先用浮游生物网富集,之后提取富集获得的混合组织的DNA,进而进行高通量测序和物种组成分析。该过程中究竟富集多大体积的水样才能较准确地反映浮游动物群落的多样性与完整性还存在争议[19-22]。也有学者直接采用过滤水样的方式开展浮游动物多样性研究[23-24],但这种简单的采样方式获得的浮游动物多样性的准确性和稳定性不明确。因此,本研究重点探讨不同的采样方法对浮游动物DNA宏条形码监测结果的影响,为规范化的流域水生生物DNA宏条形码监测提供支撑。
1 材料与方法
1.1 调查点位及样本采集
调查点位位于南京市栖霞区羊山湖(118°57′E, 32°6′N)。样品采集设置4~5个平行样,分别采用直接过滤法和网富集法采样。
(1) 直接过滤法:直接利用过滤器过滤(滤膜孔径为5 μm)不同体积(100,200,500和1 000 m L)的水样,过滤后收集滤膜用于后续DNA提取。
(2) 网富集法:先用标准25#浮游生物网过滤一定体积(5,10,20和1 000 L)的水样,对浮游动物进行初步富集浓缩,浓缩后的样本再用孔径为5 μm的滤膜进行过滤,最后收集滤膜用于后续DNA提取。过滤体积基于水泵的流速和过滤时间换算。
1.2 核酸提取与扩增
采用E.Z.N.A. water DNA kit 水样提取试剂盒(美国Omega公司)提取滤膜DNA。具体操作为:将滤膜装入5 mL无菌管中,加入0.2 mg的无菌玻璃珠,加入1 mL裂解液,用MoBio Vortex-Genie2涡旋振荡仪(美国MoBio公司)均质10 min,之后按照试剂盒操作说明提取DNA。聚合酶链式反应(PCR)扩增体系:总反应体系为50 μL,由37.8 μL 双蒸水, 2 μL DNA,1 μL扩增引物,5 μL 10倍PCR高保真PCR缓冲溶液(High Fidelity PCR buffer),2 μL 50 mmol/L的硫酸镁,1 μL 10 mmol/L的脱氧核糖核苷三磷酸混合物(dNTP mix),0.2 μL Platinum Taq DNA 聚合酶组成(美国Invitrogen公司)[13]。
PCR扩增程序为: 95 °C 预变性30 s,扩增35个循环,95 °C变性10 s, 48 °C退火30 s,72 °C延伸30 s,72°C延伸5 min。PCR结束后用2%的琼脂糖凝胶电泳对PCR扩增效果进行检测,用核酸纯化试剂盒进行产物纯化,用Qubit(美国Invitrogen公司)进行核酸定量。
1.3 高通量测序与生物信息学分析
不同样本扩增后的PCR产物被稀释到10-2g/L 并等体积混合。用Ion Torrent PGM测序专用试剂盒(美国Lifetech公司)构建测序文库。测序文库测序前,用磁珠纯化去除文库中的杂质和短片段(< 100 bp);纯化后的测序文库用2100生物分析仪(美国Agilent公司)检测合格后稀释到100 pmol/L的浓度,用Ion Torrent PGM测序仪进行高通量测序。
测序结束后利用Mothur fastq.info命令将测序输出的原始FASTQ文件转为FASTA文件和Qual质量值文件[25]。利用QIIME软件[26]删除测序质量低于Q20和引物中出现3个碱基以上错配的序列。利用UCHIME[27]识别并去除PCR过程中产生的嵌和子序列[28]。利用USEARCH软件[29]对序列进行聚类,并形成可操作分类单元(OTUs)。利用SAP和BLAST+软件的默认参数进行OTUs序列[30]的比对和注释,其中序列间的相似性>90%,且后验概率>60%的序列被注释到种水平。序列注释数据库包括自建的本土物种数据库和NCBI GenBank中公开的条形码数据库[31-33]。
2 实验结果
2.1 采样方法和体积对浮游动物宏条形码OTUs/序列注释的影响
不同采样方法和体积对浮游动物宏条形码测序结果的影响见图1(a)—(e),采样体积以L为单位, 0.1~1 L为直接过滤水样体积,5~1 000 L为浮游生物网富集过程中的过水体积。由图1(a)和(b)可见,直接过滤法中,DNA宏条形码测序获得的总OTUs数量和注释到浮游动物OTUs数量均随过滤体积的增加而增加;网富集法中,总OTUs随着采样体积的增加而下降,在采样体积为5和1 000 L时,浮游动物OTUs的数量较大。由图1(c)可见,直接过滤法中,随着过滤体积的提高,不能被注释的未知DNA序列占比也逐渐增多,当过滤水样体积为1 L时,有超过40%的DNA序列无法被数据库准确注释;网富集法中,随着浮游动物网过水体积的增加,不能够被注释的序列占比下降,在采样体积为1 000 L时,仅有不到5%的未知序列。由图1(d)可见,直接过滤法中,随着采样体积的不断加大,浮游动物序列占总序列的比例也逐渐下降,当过滤水样体积达到1 L时,浮游动物仅占总序列的10%左右;网富集法中,浮游动物所占总序列的比例随着过水体积的增加而逐渐增加,在采样体积为1 000 L时,有超过90%的序列都为浮游动物。由图1(e)可见,与直接过滤法相比,网富集法能发现更多的浮游动物种类,尤其是能检出更多枝角类和桡足类,而对于部分小型浮游动物轮虫来说,直接过滤水样对其检出效果更好。

图1 不同采样方法和体积对浮游动物宏条形码测序结果的影响
2.2 采样体积对浮游动物多样性的影响
采样体积对浮游动物多样性的影响见图2(a)—(d)。由图2可见,直接过滤法中,大型浮游动物(主要为枝角类和桡足类)的多样性随着过滤体积的增加而增加,其中大型浮游动物多样性在过滤体积为1 L时达到最大值;随着过滤体积的增加,枝角类的多样性有所增加,而桡足类的多样性变化不明显;小型浮游动物轮虫的多样性随着过滤体积的增加而有所下降。网富集法中,大型浮游动物的多样性在富集5 L水样时达到最大,10和20 L时小型浮游动物多样性则大幅降低,而在过滤体积达到1 000 L时多样性再次上升,并与5 L富集体积下的多样性基本保持一致;枝角类多样性与中大型浮游动物的多样性变化趋势基本一致;桡足类的多样性受采样体积的影响较小,但在采样体积达到1 000 L时监测的重复性更好;小型浮游动物轮虫在富集体积达到1 000 L时,其多样性达到最大值。

图2 采样体积对浮游动物多样性的影响
2.3 采样体积对重复样本中OTUs检出率的影响
重复样本的检出率是反映采样方法稳定性的重要指标。浮游动物OTUs在重复采样时的检出率见图3。由图3可见,直接过滤法中,随着过滤体积的增加,检出率>75%的浮游动物OTUs的数量逐渐增加,在总OTUs中的占比也呈上升趋势,这说明实验结果的稳定性和可靠性在不断提高。网富集法中,富集体积为5 L时,4个重复样本中检出率>75%的OTUs占比为82%,在过水体积为10 和20 L时,这一比例分别为73%和69%,当富集水样体积达到1 000 L时,该比例增长到 87%。由此说明选择1 000 L的富集体积得到的浮游动物多样性监测结果稳定性最高。
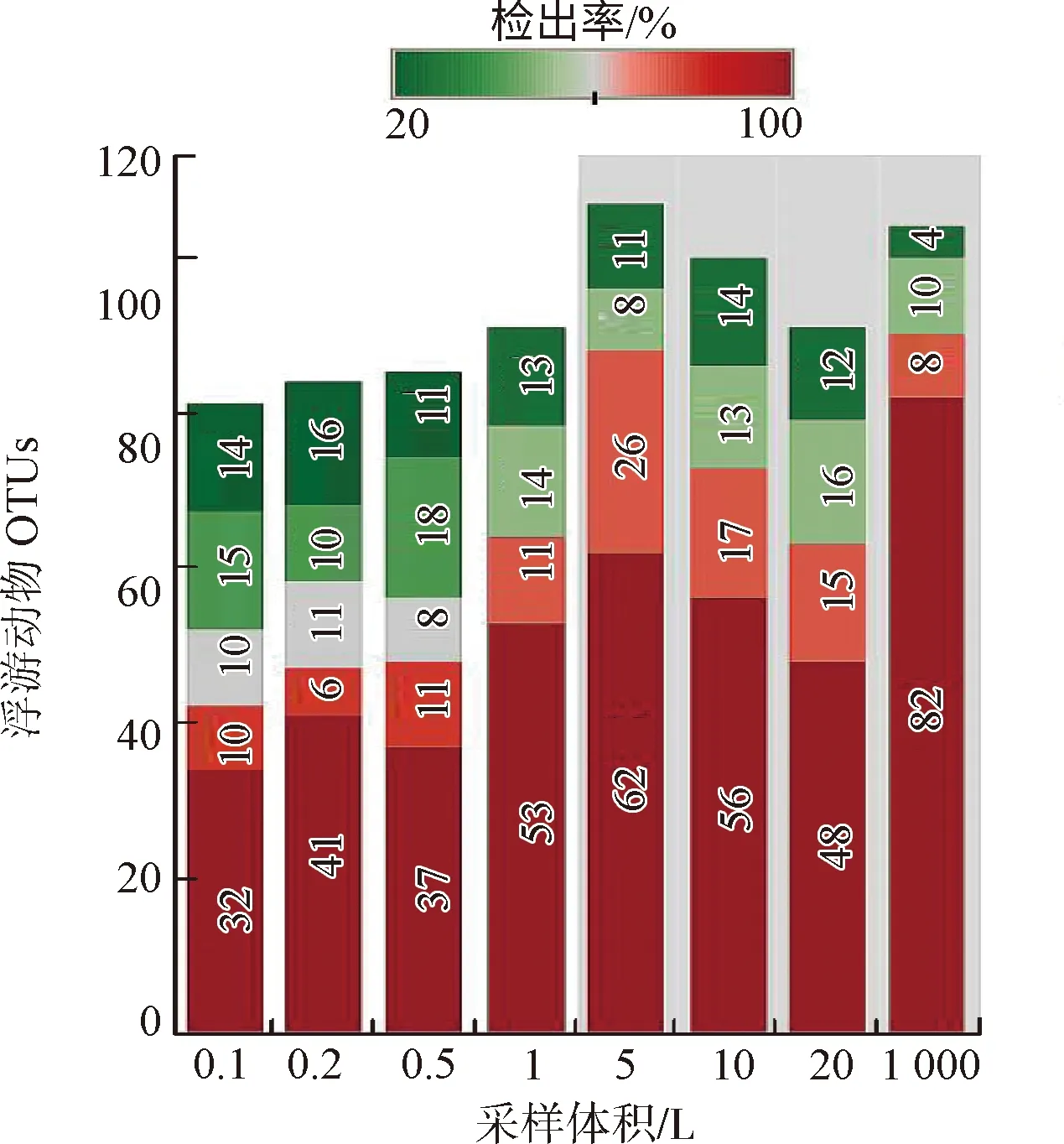
图3 浮游动物OTUs在重复采样时的检出率
3 讨论
传统形态学生物监测中,结合浮游动物的体型大小与分布特点,通常用直接过滤法获取小微型浮游动物(如轮虫),用网富集法获取大型浮游动物(如桡足类和枝角类)[34]。传统调查中采样的目的是要捕获足够量的生物个体,以便后续依据生物的形态学特征进行分类鉴定[35]。而DNA宏条形码技术是通过检测DNA分子来判断生物存在与否,可利用有机体的组织、残片或游离DNA,不需要获取到生物个体[36],因此,DNA宏条形码监测并不能完全遵循传统的采样方式,需要结合环境DNA的自身特点进行采样。
3.1 小型浮游动物
直接过滤水样对小型浮游动物的监测效果更好。在直接过滤小体积水样时,能捕获到分布相对均匀且数量较多的小型浮游动物(如轮虫),DNA的检出效果更好,而对于数量较少的大型浮游动物来说,如桡足类,往往并不能直接富集到[34],DNA的检出效果也较差。而在采用网富集法时,由于25#浮游动物网的孔径会遗漏一些个体较小的轮虫,在富集体积不足时导致检出的轮虫数量要低于直接过滤法。但是,对小型轮虫的遗漏可以通过增大过水体积来弥补不足,当富集体积达到1 000 L时,检出的轮虫多样性与直接过滤1 L水样时所检测出的轮虫多样性基本一致。但是,综合考虑采样效率和经济性时,采用直接过滤1 L水样的方法更适于小型浮游动物的检测。
3.2 大型浮游动物
浮游生物网富集对大型浮游动物的监测效果更好。对于大型浮游动物而言,由于其在水体中数量少、密度低,须先用浮游动物网进行富集浓缩再进行DNA宏条形码的检测。实际上,由于自然水体中“自由DNA”的存在,使得在直接过滤水样中也能检测出某些大型浮游动物的OTUs。其中枝角类的多样性随浮游动物网富集体积增加而显著变化,富集体积为5 L时所检测出的枝角类种数最多,该过滤体积比较适合枝角类多样性的检测。对于桡足类,网富集法所得到的物种多样性高于直接过滤法,该结论与以往的研究结果相一致。Djurhuus等[17]比较了不同的环境DNA样品来源对浮游动物多样性的影响发现,网富集法相较于直接过滤法获得的大型浮游动物桡足类的物种数目更多。本研究中,当富集体积达到1 000 L时,检测出的桡足类多样性最高,平行样间的重复性也较好。然而,在现实采样中,定量富集1 000 L的做法可操作性差,且大体积富集会降低枝角类的检测效率,因此建议浮游动物网的最佳富集体积为5~10 L。
4 展望
本研究建立了适合于浮游动物DNA宏条形码监测技术的采样方法,初步实现了该采样方法的标准化,同时也为DNA宏条形码技术在淡水水体生物多样性监测方面的灵活运用提供了技术支撑。但是,浮游动物在水中的分布受光、热、营养物质的影响,不同水体类型(河流、湖泊、水库)中的浮游动物分布也不同[37],如海水中浮游动物存在明显分层[38],河流的流速、沿岸排污口会对浮游动物产生巨大影响等[39],尤其是在开展物种定量监测时,采样方法则显得尤为重要[40]。因此在未来的浮游动物DNA宏条形码监测中,要考虑上述各方面影响对浮游动物分布的综合作用,针对不同水体类型确定合理的采样深度、采水方式、采样体积等。作为一种新型监测方法,规范化和标准化是DNA宏条形码技术业务化应用的基础。除采样方法需要标准化外,标志基因和扩增引物[41]、评价指标的筛选和建立[42]、生物信息学分析方法等都需要统一的标准[43],以提高物种监测的准确性及监测结果的可比性。
