粳稻糖基转移酶Ⅰ基因家族全基因组鉴定及功能分析
2022-02-15周镇中汪启明饶力群
周镇中,罗 彪,骆 鹰,夏 敏,汪启明,饶力群
(湖南农业大学 生物科学技术学院,道地药用植物规范化栽培与综合利用湖南省工程实验室,湖南 长沙 410128)
水稻(OryzasativaL.)作为重要粮食作物,稳产是世界粮食安全和社会经济稳定的关键因素。全球气候变暖,高温导致水稻花粉育性降低、退颖、籽粒品质降低等,高温成为影响水稻生长和产量的主要限制因素[1-3]。水稻中UDP-葡萄糖基转移酶(GSA1)可通过催化单糖和黄酮类化合物的糖基转移,改变黄酮类糖基的组成和苯丙素的代谢途径,来调节和适应高温胁迫[4]。糖基的转移在糖基转移酶催化完成,从反应数量上讲,糖基的转移是植物体内最多的一类生化反应[5]。非生物胁迫会导致植物产生大量的活性氧(ROS),过量的活性氧导致生物大分子过度氧化而造成生理生化损伤[6]。植物在应对ROS的机制中除了常见的抗氧化酶类,还有许多抗氧化剂。如谷胱甘肽(GSH)、黄酮类化合物和类胡萝卜素等[7]。黄酮类化合物是植物中最具活性的代谢物之一。在黄酮类化合物参与清除ROS的反应中都有UDP-糖基转移酶参与催化,生成具有抗氧化活性的各种衍生物[8]。如花青素3-O-半乳糖基转移酶、花青素3-O-葡萄糖基的转移酶和UDP-葡萄糖醛糖基转移酶的功能主要是涉及黄酮类化合物糖基的转移[9-10]。研究表明,通过黄酮类次生代谢物的产生能有效提升植物对逆境的抗性[11],糖基转移酶参与非生物胁迫和生物胁迫应激产物的糖基化反应[12]。在拟南芥中过表达糖基转移酶AtDGD1提高了拟南芥对高温的耐受性,且拟南芥中AtDGD1的沉默导致拟南芥耐高温能力下降[13-14]。因此,糖基转移酶在植物应对非生物胁迫时起到了重要作用。
随着全基因组测序和生物信息学的发展,在水稻中鉴定出769个转录本是糖基转移酶,可细分为41个家族[15]。水稻中作为脂质、蔗糖等合成酶类的糖基转移酶Ⅰ(GT Ⅰ)基因家族全基因组鉴定及功能分析尚未有研究报道。
本研究通过生物信息学分析在粳稻中鉴定出30个GT Ⅰ基因家族成员。通过进化关系、基因结构、保守结构域、高温胁迫转录组数据挖掘等分析,了解了粳稻GT Ⅰ基因家族成员的结构特征和响应高温的表达情况,最后对GT Ⅰ 基因家族成员的蛋白质三级结构进行建模,旨在为了解粳稻GT Ⅰ家族成员的功能研究奠定基础,为进一步了解粳稻中GT Ⅰ基因家族成员在水稻抗高温方面的研究提供方向,以及在水稻生产中对高温的耐受性具有重要的意义。
1 材料和方法
1.1 GT Ⅰ基因家族的全基因组鉴定
EnsemblPlants(http://plants.ensembl.org/index.html)下载粳稻(OryzasativaJaponica)、籼稻(OryzasativaIndica)、高粱(Sorghumbicolor)、雷蒙德氏棉(Gossypiumraimondii)和拟南芥(Arabidopsisthaliana)全基因组序列、CDS 序列、蛋白质序列以及全基因组注释文件。Pfam 数据库(http://pfam.xfam.org)下载 GT Ⅰ 基因家族(PF 00534)保守结构域(HMM)配置文件。HMMER 3.0 和 BlastP 检索本地数据库水稻及其他物种的基因组中GT Ⅰ 基因家族候选成员。候选成员的蛋白质序列放入NCBI-CDD(Https://www.ncbi.nlm.nih.gov/cdd/)、EMBL-EBI(http://pfam.xfam.org/search#tabview=tab1)和SMART(http://smart.embl.de/smart/set_mode.cgi?GENOMIC=1)确认GTⅠ基因家族成员保守结构域,确定GTⅠ基因家族成员[16]。ExPASy(Http://web.expasy.org/protparam/)对家族成员进行理化性质分析[17],CELLO v2.5(http://cello.life.nctu.edu.tw/)在线分析家族成员亚细胞定位。
1.2 粳稻GT Ⅰ基因家族成员系统发育关系、基因结构及保守结构域分析
各物种GT Ⅰ 基因家族成员蛋白序列在 MEGA 7.0 软件进行遗传进化分析,用NJ法构建进化树(自检值为1 000)[17]。根据基因组注释文件将粳稻GT Ⅰ 基因家族成员进行外显子-内含子结构可视化[18]。GT Ⅰ 基因家族成员蛋白序列放入在线网站MEME(http://meme.sdsc.edu/meme/)进行保守结构域分析[19]。
1.3 GT Ⅰ基因家族成员染色体定位、启动子分析和miRNA-GT Ⅰ基因家族成员预测
水稻基因组注释文件获得GT Ⅰ基因家族成员的位置及结构信息。MapChart 2.2 软件可视化水稻GT Ⅰ 基因家族成员的位置信息。GT Ⅰ 基因家族成员的启动子序列(基因起始转录位点开始选取上游1 500 bp的序列)在PlantCARE(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)进行分析,软件TBtools 启动子分析结果可视化。预测miRNA在GT Ⅰ 基因家族成员潜在的靶位点,GTⅠ基因家族成员cDNA在psRNATarget(http://plantgrn.noble.org/psRNATarget/)预测miRNA前体[20],软件Cytoscape可视化[21]。
1.4 粳稻GT Ⅰ基因家族成员组织表达分析和高温胁迫处理以及转录组测序分析
RiceXPro网站(https://ricexpro.dna.affrc.go.jp/)下载GTⅠ家族成员组织表达公共RNA-Seq样本[22],TBtools数据可视化[23]。粳稻GTⅠ基因家族成员响应非生物胁迫表达情况,日本晴幼苗在28 ℃人工物候箱中生长,光照14 h,黑暗10 h。14 d后选取长势一致的幼苗进行高温处理。在45 ℃人工物候箱进行高温孵育。处理取样时间为0,1,3,6,12,24,48 h,采集茎和叶组织转录组测序。RNA-Seq数据访问号为SRP190858[24]。TBtools软件数据可视化。
1.5 GT Ⅰ基因家族成员高温表达的实时荧光定量验证
用上述非生物胁迫处理方法对日本晴处理,处理取样时间为0,3,6,12,24,36 h。TRIzol试剂按说明提取总 RNA,反转录试剂盒将 2 μg 总 RNA 逆转录为 cDNA,稀释备用。SYBR 预混Ex Taq在BIO-RAD品牌的CFX Connect进行 Real-time PCR。体系 20 μL,10 μL SYBR Green PCR mix,上下游引物 各0.5 μL,2 μL cDNA,7 μL ddH2O。qRT-PCR 的反应程序:94 ℃ 30 s;循环阶段:94 ℃ 5 s,55 ℃ 15 s,72 ℃ 10 s,45个循环;溶解曲线阶段:94 ℃ 15 s,60 ℃ 15 s,95 ℃ 15 s,4 ℃保存。以Actin作为内参,采用 2-ΔΔCt法计算GT Ⅰ家族基因的相对表达量[25],每个时期,每个基因的表达反应有 3 个生物重复和 3 个技术重复。基因引物见表1。

表1 实时荧光定量引物Tab.1 Real-time fluorescence quantitative primers
1.6 粳稻GT Ⅰ基因家族蛋白质三维结构同源建模
蛋白质的三维结构对了解其功能具有重要意义,将GTⅠ基因家族成员蛋白质进行蛋白质建模分析[26],使用网站SWISS-MODEL(https://swissmodel.expasy.org/interactive/)同源建模[27-28],其中模板建模评分(TM-score)的值均大于0.5,说明三维建模可信[29]。PROCHECK(https://www.rcsb.org/alignment)检验三维结构的可靠性[30],其三维结构均方根差(RMSD)小于1,说明结果可靠[31]。三维建模在软件VMD可视化[32]。
2 结果与分析
2.1 粳稻GT Ⅰ基因家族成员及其染色体分布
粳稻全基因组鉴定出30个GT Ⅰ家族成员(表2),由水稻GT Ⅰ基因家族成员蛋白质理化分析结果可知30个GTⅠ基因家族成员的氨基酸组成存在一定的差异,其蛋白质长度为256~1 066 aa,蛋白质的分子质量为29.06~118.72 ku,预测的等电点(pI)变化为5.14~9.91。

表2 粳稻GT Ⅰ基因家族编码蛋白质理化性质及其亚细胞定位Tab.2 Physicochemical properties and subcellular localization of proteins encoded by the GT Ⅰ gene family in japonica rice

表2(续)
由亚细胞定位预测可知,GT Ⅰ基因家族成员的蛋白质主要在胞质和叶绿体,部分成员定位在质膜和线粒体。由基因染色体定位得知,GT Ⅰ基因家族成员在水稻全基因组12条染色体上均有分布(图1),30个家族成员中,在3号染色体分布有5个,在1,6,7号染色体各有4个,在2,4号染色体均有3个,在11号染色体有2个。在5,8,9,10,12号染色体均只存在1个家族成员。

图中编号代表染色体编号;绿色长条长度根据基因组染色体长度显示;染色体上左边数字是基因在染色体上的位置,右边的数字是成员名称。The numbers in the diagram represent chromosome numbers and the length of the long green bar is shown based on the length of the genomic chromosome;the number on the left of the chromosome is the position of the gene on the chromosome,and the number on the right is the member name.
2.2 粳稻、籼稻及其他物种的GT Ⅰ基因家族成员的系统进化关系
为得知水稻GT Ⅰ基因家族与其他物种的GT Ⅰ基因家族的系统发育关系和进化模式,粳稻30个GT Ⅰ基因家族成员、籼稻21个GT Ⅰ基因家族成员、高粱7个GT Ⅰ 基因家族成员、雷蒙德氏棉12个GT Ⅰ基因家族成员和拟南芥15个GT Ⅰ基因家族成员构建进化树(图2)。根据遗传距离将所有GT Ⅰ家族成员分为3个亚家族,其中,亚家族Ⅰ包含了10个基因(4个粳稻GT Ⅰ成员、2个籼稻GT Ⅰ成员、1个高粱GT Ⅰ成员和3个拟南芥GT Ⅰ成员),亚家族Ⅱ包含了32个基因(16个粳稻GT Ⅰ成员、13个籼稻GT Ⅰ成员、3个拟南芥GT Ⅰ成员),亚家族Ⅲ包含了43个基因(10个粳稻GT Ⅰ成员、6个籼稻GT Ⅰ成员、6个高粱GT Ⅰ成员、9个拟南芥GT Ⅰ成员和12个雷蒙德氏棉GT Ⅰ成员)。这3个亚家族中都有拟南芥的GT Ⅰ家族成员。雷蒙德氏棉GT Ⅰ家族成员只存在第Ⅲ亚家族中。粳稻30个GT Ⅰ家族成员和籼稻21个GT Ⅰ家族成员主要分部在第Ⅰ亚家族和第Ⅱ亚家族中,与其他物种相比,它们具有更近的亲缘关系。

★.粳稻; ●.籼稻;■.高粱;▲.拟南芥;√.雷蒙德氏棉。进化树分支的颜色代表不同的亚家族(红色代表亚家族Ⅰ、绿色代表亚家族Ⅱ和蓝色代表亚家族 Ⅲ)。★.Japonica rice;●.Indica rice;■.Sorghum bicolor;▲.Arabidopsis thaliana;√.Gossypium raimondii. The colors of the evolutionary tree branches represent the different subfamilies(red for subfamily I,green for subfamily Ⅱ and blue for subfamily Ⅲ).
根据序列的相似性和系统发育分析,在粳稻中获得了GT Ⅰ基因的同源对。粳稻中总共发现了6对同源组,5个同源基因。利用ClustalW分析计算粳稻GT Ⅰ基因家族的同源基因Ka/Ks值,探究基因进化选择压力(表3)。基因OS-GT28和OS-GT29与OS-GT30、OS-GT24和OS-GT25存在着基因进化选择压力。OS-GT29和OS-GT30的Ka/Ks值为0.919(Ka/Ks<1),说明它们之间存在着基因受纯化选择。OS-GT29和OS-GT24的Ka/Ks值为1.088(Ka/Ks>1),OS-GT29和OS-GT25的Ka/Ks值为1.087(Ka/Ks>1),说明OS-GT29与它们之间存在基因受正向选择。另外OS-GT-28和OS-GT30的Ka/Ks值为0.937(Ka/Ks<1),说明它们之间存在着基因受纯化选择。OS-GT28和OS-GT24的Ka/Ks值为1.075(Ka/Ks>1),OS-GT28和OS-GT25的Ka/Ks值为1.078(Ka/Ks>1),说明OS-GT-28与它们之间存在基因受正向选择。
2.3 粳稻GT Ⅰ基因家族成员结构与保守序列特征
外显子-内含子结构是基因进化的重要特征,也是基因功能多样化的重要因素。进一步分析GT Ⅰ 基因家族成员的外显子-内含子结构和保守结构域(图3)。GT Ⅰ基因家族成员经过聚类分析,把具有保守基序的成员聚类到一块。图3-A显示, GT Ⅰ基因家族成员通过遗传距离分析可以把30个基因家族成员分成六大类。图3-B显示,GT Ⅰ 基因家族成员通过MEME软件分析出保守基序得到了10个Motif,所有基因家族成员都含有Motif 2这个保守的基序。聚类分析中,第Ⅰ类家族成员OS-GT24、OS-GT25、OS-GT26、OS-GT27、OS-GT28、OS-GT29和OS-GT30含有的Motif是相对保守的,含有7类Motif;家族的保守结构域建树,通过保守基序分析得到10个Motif,不同颜色代表不同的Motif类别。图3-C蓝色代表UTR,黄色代表CDS;间隔区代表内含子。每个GT Ⅰ 家族成员的CDS、UTR和内含子的分布都按基因上的比例分布。

表3 粳稻中GT Ⅰ基因家族同源基因进化压力选择Tab.3 Evolutionary pressure selection of GT Ⅰgene family homologous genes in japonica rice

Conservative structural domain building tree of the family,10 Motifs were obtained by conserved motif analysis,different colors represent different Motif categories.In Fig.3-C blue represents UTR,yellow represents CDS;spacer region represents intron.The distribution of CDSs,UTRs and introns of each GT I family member is scaled genetically.
第 Ⅱ 类家族成员中OS-GT21和OS-GT23均含有Motif 2和Motif 1;第Ⅲ和Ⅳ类家族成员中大多只含有Motif 2;第Ⅴ和Ⅵ 类家族成员中大多数含有3个Motif。图3-C显示,除OS-GT04和OS-GT13没有内含子结构外,其余基因家族成员都含有较多的内含子,基因结构较为复杂。
2.4 粳稻GT Ⅰ家族基因启动子和相关miRNA预测
进一步阐明粳稻GT Ⅰ家族基因在非生物胁迫中可能存在的调控机制,对启动子中序列进行了分析,结果共获得38类顺式作用元件(图4),其中TATA-box、CAT-box等核心启动子元件在30个GT Ⅰ 家族成员中均有发现。这些启动子区域发现了大量与激素相关的作用元件,30个GT Ⅰ 家族成员都预测到脱落酸响应元件(ABRE),在7个家族成员中预测到赤霉素响应元件(CARE),在18个家族成员中预测到水杨酸响应元件(TCA),在10个家族成员中预测到茉莉酸甲酯响应敏感基序(CGTCA和TGACG)。30个家族成员中没有出现4种激素都响应的成员,但30个家族成员均预测出响应光的作用元件,如ACE、G-box、I-box等。也有部分成员预测出参与低温反应的顺式作用元件(LTR)和MYB结合位点参与干旱反应的顺式作用元件(MBS)。30个家族成员中有少数几个成员没有直接参与植物苯丙烷类次生代谢途径调节的MYB转录因子结合位点。

图4 GT Ⅰ基因家族成员启动子顺式作用元件Fig.4 Cis-acting elements of promoters of GT Ⅰ gene family members
靶向GT Ⅰ 家族成员miRNA预测(图5)。miRNA可以通过剪切或者抑制翻译来调控靶基因的表达,依据其与靶基因序列的互补程度,miRNA靶基因的数目可能是一个或者多个,有些靶基因受到多个miRNA的调控。在数据库中获得了49个不同类型的相关miRNA,靶向29个GT Ⅰ 基因家族成员,只有OS-GT04没有匹配到对应的miRNA,说明OS-GT04从某种程度可以说不会被miRNA通过剪切或者抑制翻译。osa-miR1857-5p这个miRNA靶向OS-GT26、OS-GT19和OS-GT10,说明这3个家族成员可以被其调控。osa-miR1858a的靶标是OS-GT13和OS-GT22。osa-miR5075、osa-miR1853-3p、osa-miR5809、osa-miR444a-3p.1和osa-miR810b.1也对应2个家族成员,预测出的miRNA是大多数靶向1个GT Ⅰ 基因家族成员,靶向GT Ⅰ 基因家族成员的miRNA预测网络见图5。miR1857是参与果实成熟和非生物逆境调控的miRNA,其对应的靶基因主要是花青素合成和非生物逆境应答等。这说明miRNA与GT Ⅰ 基因家族成员之间的调控网络可能为它们的功能提供了重要的线索,同时也有利于选择候选基因进行未来的研究。
2.5 GT Ⅰ 基因家族成员组织特异性表达和高温胁迫响应
利用网络公共数据对GT Ⅰ 基因家族成员在不同组织部位表达进行了分析(图6),结果显示,GT Ⅰ 基因家族成员在组织中表达具有较强的组织特异性,其中OS-GT21、OS-GT06、OS-GT18、OS-GT29、OS-GT03、OS-GT24、OS-GT11、OS-GT15和OS-GT25在茎部位高表达,在叶片、叶鞘和根的表达表现为低表达;OS-GT17和OS-GT02在叶片中呈现高表达,OS-GT07、OS-GT30、OS-GT28和OS-GT26在叶片中呈现低表达;在叶鞘中,OS-GT27、OS-GT16和OS-GT01呈现高表达,在根中低表达的成员有OS-GT08、OS-GT05和OS-GT09。
通过日本晴高温胁迫下转录组数据分析,GT Ⅰ 家族成员的表达模式如图7所示,大部分家族成员在高温胁迫第3小时开始响应高温胁迫,在高温处理6 h开始表达量变化明显。OS-GT14、OS-GT09和OS-GT22高温胁迫开始前3 h表达量高,随着胁迫时间延长,表达量下降。这3个成员高温胁迫呈负调控。OS-GT24高温胁迫第6小时开始表达。OS-GT07和OS-GT28高温胁迫没有发生表达量变化,这2个成员不响应高温胁迫。

◆.GT Ⅰ 家族成员;●.miRNA。◆.GT Ⅰ family members;●.miRNA.

纵坐标是GT Ⅰ 家族成员。红色表示高表达、蓝色表示低表达。Vertical coordinates are GT Ⅰ family members.Red indicates high expression,blue indicates low expression.
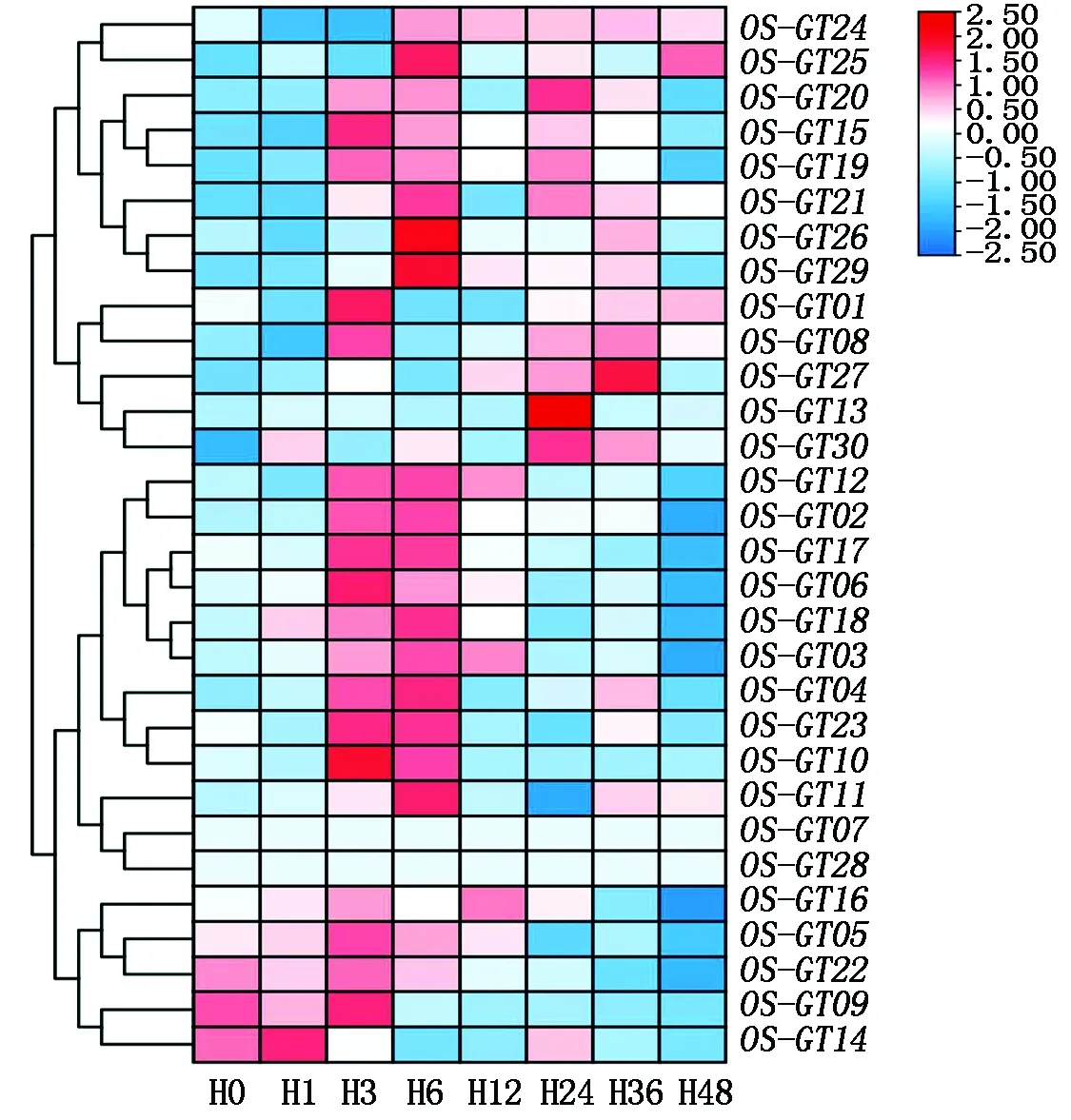
横坐标代表高温胁迫时间节点,分别代表0,1,3,6,12,24,36,48 h;纵坐标是GT Ⅰ家族成员。红色表示表达上调,蓝色表示表达下调。
2.6 粳稻GT Ⅰ基因家族成员高温胁迫表达分析
经高温处理后的材料中10个GT Ⅰ 基因家族成员的差异表达如图8所示,GT Ⅰ 基因家族成员的表达量随着高温处理时间的变化而改变。高温处理3 hOS-GT09的表达量达到最高,随后其表达量随着处理时间的延长逐渐降低;高温处理0~6 hOS-GT01和OS-GT03基因的表达量不断升高,发现6 h表达量达到最高;处理6 h后随着处理时间的增加迅速减少。高温处理3 hOS-GT19的表达量无明显升高,在处理6 h开始表达量迅速上升,在处理24 h达到最高。高温处理前6 hOS-GT29的表达量变化不大,随着时间的延长表达量开始上升。OS-GT07和OS-GT11的表达量没有因为高温处理出现表达量明显变化。有趣的是OS-GT16的表达量从高温处理开始便开始逐步上升,处理12 h后趋于稳定。OS-GT14和OS-GT22随着高温处理时间的延长,其相对表达量下降,其中OS-GT22在高温处理第3小时开始,其表达被抑制。

图8 GT Ⅰ基因家族成员在高温处理的表达水平Fig.8 Expression levels of GT Ⅰ gene family members under high temperature treatment
2.7 粳稻GT Ⅰ 基因家族成员蛋白保守性分析
结构决定功能,GT Ⅰ基因家族成员进行三维结构预测和同源建模(图9)。从图9可以看出,GT Ⅰ基因家族成员蛋白质具有一个保守的序列,相似的三维结构。保守结构的中心是6个β-折叠,其周围分别由7个α-螺旋环绕。OS-GT24、OS-GT25、OS-GT26、OS-GT27、OS-GT28、OS-GT29和OS-GT30的蛋白质作为蔗糖合成酶类,它们的三维结构高度保守。其中OS-GT10、OS-GT11和OS-GT12建模之后的评估值反映出建模是成功的,结合三维结构建模来看,它们之间的结构是相当保守且含有保守蛋白结构。GT Ⅰ基因家族成员在进行三维结构预测时,参与建模的GT Ⅰ 基因家族成员与对应的同源模板重叠后参数见表4,其三维结构的均方根差(RMSD)小于1,说明GT Ⅰ建模结果可靠。其中模板建模评分(TM-score)的值均大于0.5,说明GT Ⅰ 基因家族成员的三维建模是可信的。OS-GT11和OS-GT12的TM-score值为1,说明蛋白与模型完美匹配。这些蛋白质的三维结构模型为它们的生物学功能奠定了基础。

图9 GT Ⅰ家族成员蛋白三维结构Fig.9 Three-dimensional structure of GT Ⅰ family members proteins

表4 粳稻GT Ⅰ 基因家族建模预测比对参数Tab.4 Predicted alignment parameters by modeling the GT Ⅰ gene family in japonica rice
3 结论与讨论
糖基转移酶作为生物体内催化糖基化关键酶,在植物体的能量转移、物质转化,以及非生物胁迫响应等方面发挥了重要作用[43]。鉴定出粳稻GT Ⅰ 基因家族的成员在生长发育中起到了重要作用。OS-GT18在编码的硫代异鼠李糖基转移酶参与类黄酮代谢的糖基化反应且影响水稻分蘖和结实。OS-GT26、OS-GT27、OS-GT28、OS-GT29和OS-GT30编码的酶参与纤维素的合成,在水稻伸长组织中起重要作用,如根的伸长、叶和节间的发育等。且这些成员在逆境胁迫中起作用,在胁迫中表达量上升。另外OS-GT27和OS-GT28水稻灌浆过程中在颖果里强势表达,这2个成员在灌浆时起重要作用。与水稻产量提升相关的GT Ⅰ 基因家族成员是OS-GT19,功能涉及水稻光系统Ⅱ、株高和分蘖等,在一定程度上影响了过氧化物积累,在叶片衰老过程中响应光照的调控。GT Ⅰ基因家族成员大部分发挥的功能是UDP-糖基的转移和糖原的磷酸化,为各种代谢途径提供UDP-葡萄糖和果糖。OS-GT10是控制直链淀粉合成的主要基因,直接影响花粉和水稻种子胚乳的淀粉含量。由以上家族成员功能可知,GT Ⅰ基因家族在水稻发育和抗逆上有重要作用。在今后面对全球变暖所带来的挑战,GT Ⅰ基因家族其他成员在今后水稻改良农艺性状和提高水稻抗逆性方面成为可能。
GT Ⅰ基因家族成员在水稻12条染色体上均有分布,其蛋白产物长度虽然相差较大,但家族成员蛋白质的定位主要在叶绿体、线粒体和胞质,而这些部位为能量转化以及物质运输的主要场所,由此推测,GT Ⅰ基因家族在能量物质转化中起到了作用。如在拟南芥中有与OS-GT14同源的β-二半乳糖二酰基甘油合酶的缺失导致叶绿体膜脂成分改变,和光合复合体不稳定导致光合作用减弱[44]。在GT Ⅰ 基因家族遗传距离分析中,水稻GT Ⅰ 基因家族中存在5个同源基因,它们在进化过程中均发生点突变,OS-GT28、OS-GT29和OS-GT30基本上还是保持稳定的进化。因此,它们之间的功能也是相似的,均属于水稻蔗糖合成酶。同时这几个家族成员的基因结构和保守结构域也极为相似,有趣的是整个家族中保守基序都比较少(除去隶属于水稻蔗糖合成酶类的成员)。不同物种的GT Ⅰ 基因家族成员系统进化关系把它们分为3个亚家族。亚家族 Ⅰ 中,AT-GT01、AT-GT02和AT-GT03属于UDP-甘油转移酶基因,这3个基因在拟南芥生长发育过程中淀粉代谢、花粉发育和抗逆上发挥了作用[45-46]。第 Ⅰ 亚家族中粳稻GT Ⅰ 基因家族成员OS-GT01、OS-GT02、OS-GT03与拟南芥GT Ⅰ 基因家族成员的遗传距离最近,且OS-GT01、OS-GT02、OS-GT03也是属于UDP-葡萄糖基转移酶。相关研究也表明,OS-GT03 在水稻花粉内壁的形成和花粉成熟上发挥了作用[45-46]。在第 Ⅲ 亚家族中,AT-GT11和OS-GT26的遗传关系很近,且均属于蔗糖合成酶类,在植物体中都起到了纤维素合成的作用[47]。由此可以推测,GT Ⅰ 基因家族在各个植物中存在一定的保守性,这为植物GT Ⅰ 基因家族成员的功能研究提供了方向。
经过启动子分析得知,家族成员均响应调节植物生长发育和响应胁迫脱落酸(ABA)[48],这进一步表明,GT Ⅰ 基因家族成员在水稻生长发育和抵抗胁迫方面起到了重要作用。本研究通过水稻不同组织表达分析得知,GT Ⅰ 基因家族在水稻的各个部位的表达具有特异性。OS-GT27、OS-GT16和OS-GT01在叶鞘中呈现高表达,在拟南芥中与OS-GT01同源的基因LEW3的功能体现在缺失这一基因导致植株的育性降低、纤维素的合成受损、另外次生细胞壁出现缺陷,木质部塌陷[49]。根据OS-GT27蔗糖合成酶参与纤维素的合成推测,OS-GT01、OS-GT16和OS-GT27在水稻拔节期提高表达让水稻在生长过程中满足纤维素的合成,维持次生细胞壁的稳定。在高温处理中,OS-GT14和OS-GT22在高温胁迫下呈现负调控,其表达量是随着高温处理的时间下降,说明这2个成员的表达是受高温抑制的。与OS-GT14同源的拟南芥AtDGD1基因主要参与叶绿体内囊体薄膜成分合成,过表达AtDGD1基因提升拟南芥耐高温能力[50-51]。OS-GT26、OS-GT29在高温胁迫处理第6小时开始高表达,这2个成员作为蔗糖合成酶确实在抵抗逆境胁迫上起作用。通过对日本晴GT Ⅰ基因家族成员在高温胁迫下的表达模式探究,发现GT Ⅰ基因家族的成员中OS-GT07和OS-GT11不响应高温胁迫。OS-GT29和OS-GT22作为蔗糖合成酶基因在高温处理6 h后持续高表达,OS-GT14随着处理时间的延长表达量下降。可知GT Ⅰ基因家族的大部分成员在水稻应对高温胁迫的分子机制中发挥了作用,一定程度上为GT Ⅰ基因家族成员在水稻耐高温机制研究上提供了方向。
本研究通过GT Ⅰ基因家族成员蛋白质建模可知,家族成员的蛋白质结构是保守的,与系统发育、保守基序和基因结构的分析结果较一致。结构决定功能,这30个家族成员中蔗糖合成酶的蛋白质结构高度相似,其建模的评分反映着蛋白与模型之间完美重合,说明这种建模的方法是可行的。通过已知蛋白的结构来匹配笔者研究的蛋白,为更好地研究GT Ⅰ 基因家族成员的生物学功能提供了重要线索。
