以2b-RAD技术构建凡纳滨对虾遗传连锁图谱及生长性状QTL定位
2022-02-03王怡悦刘红徐姚
王怡悦,刘红*,徐姚
(1上海水产养殖工程技术研究中心(上海海洋大学),上海 201306;2上海市水产动物良种创制与绿色养殖协同创新中心(上海海洋大学),上海 201306;3水产科学国家级实验教学示范中心(上海海洋大学),上海 201306)
0 引言
【研究意义】凡纳滨对虾(Litopenaeus vannamei)是世界上最重要的养殖虾种之一,也是目前选择育种最广泛的虾种。凡纳滨对虾因其广盐性特性已在全球海水、淡水等水域大规模养殖,其中淡水养殖具有集约化程度高、便于管理等优点,在凡纳滨对虾养殖生产中的应用越来越广泛,因此耐低盐群体经济性状的遗传改良工作显得十分重要。传统的选育方法易受环境因素影响,且时间成本高;而分子标记辅助育种(Molecular marker-assisted breeding,MAS)可通过分子标记对目标性状基因型进行选择,更准确地定位目的基因,加速遗传育种进度(熊建华等,2011)。生长相关基因参与调节生物体的生长、发育等过程,通过现代分子生物技术手段对生长性状相关数量性状位点(Quantitative trait locus,QTL)进行定位,挖掘与QTL连锁的生长相关功能基因,对促进性状改良具有重要意义。遗传连锁图谱在动植物经济价值相关性状的QTL和功能性状鉴定中扮演着重要角色,与大西洋鲑(Salmo salar)、罗非鱼(Oreochromis niloticus)等其他水产品种相比,凡纳滨对虾的生长相关标记和基因信息非常有限(Yu et al.,2019)。因此,亟待构建遗传连锁图谱为凡纳滨对虾生长相关基因的筛选及分子标记辅助选育等研究提供理论参考。【前人研究进展】目前,遗传连锁图谱构建及鉴定性状相关QTL的方法已广泛应用于各种水生生物育种研究。戴欢等(2017)在半滑舌鳎(Cynoglossus semilaevi)遗传图谱定位到3个与抗哈维氏弧菌病性状相关的QTL区间;周童(2018)构建了合浦珠母贝(Pinctada fucata)高密度遗传连锁图谱,并根据表型数据定位获得与7个生长性状相关的13个主效QTL区间;Liu等(2020a)从乌鳢(Channa argus)遗传连锁图谱的5个连锁群(Linkage group,LG)中鉴定出14个与体质量和体长相关QTL及1个性别决定QTL。生长相关基因与生物体的生长、发育紧密相关,具体是指与体型大小、体质量、生长速度等性状调控相关的基因。在现有的凡纳滨对虾遗传连锁图谱研究中,已定位出部分生长相关QTL(Andriantahina et al.,2013;Yu et al.,2015,2019),但有关生长相关基因发掘的研究报道较少。随着高通量测序技术的快速发展,传统的扩增片段长度多态性(Amplified fragment length polymorphism,AFLP)、随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)及简单重复序列(Simple sequence repeat,SSR)等分子标记已逐渐被快捷、高效的SNP(Single nucleotide polymorphisms,SNP)分子标记取代(Liu et al.,2020b)。简化基因组技术2b-RAD是基于IIB型限制性内切酶的大规模SNP分子标记开发和分型手段,具有更加快速、便捷的优点,可低成本获得目标区域内的SNP标记信息,已广泛应用于群体遗传学、遗传连锁图谱及QTL定位等研究领域(胡景杰和任红艳,2018)。王伟峰等(2019)通过2b-RAD技术对遗传雌性半滑舌鳎样本开发大量SNP分子标记,以了解半滑舌鳎当前的群体遗传结构,结果表明有效群体大小随其连锁不平衡程度衰减而呈下降趋势;Liu等(2020b)以花鲈(Lateolabrax maculatus)全同胞F1家系为作图群体,基于2b-RAD技术构建了首张花鲈高密度遗传连锁图谱,并鉴定出24个生长相关QTL区间;常雪晴等(2022)利用2b-RAD技术对三门湾海域7种代表性无脊椎动物进行遗传结构和遗传多样性分析,结果发现该海域7个物种的遗传多样性水平较低,可能是受人工增殖放流等因素的影响。【本研究切入点】2b-RAD作为一种改进的测序技术,具有标签数目可控、分型准确率高的优点(Wang et al.,2012),但至今鲜见基于2b-RAD技术构建凡纳滨对虾遗传连锁图谱的研究报道。相对于选育家系,野生群体具有更丰富的遗传多样性及更高的杂合度和等位基因数,因此,构建凡纳滨对虾野生群体遗传连锁图谱,挖掘生长相关基因,对促进凡纳滨对虾经济性状改良及其分子标记辅助育种具有重要意义。【拟解决的关键问题】以世代选育耐低盐家系的凡纳滨对虾与厄瓜多尔野生凡纳滨对虾杂交获得的子一代及亲本为作图群体,基于2b-RAD技术构建高密度遗传连锁图谱,结合生长性状表型数据鉴定生长相关QTL区间并对QTL附近基因进行功能注释,筛选生长相关候选基因;通过测定耐低盐群体和一般群体差异表达基因的相对表达量,进一步验证可能与凡纳滨对虾生长性状相关的候选基因,为后续开展凡纳滨对虾分子标记辅助育种、生长相关功能基因精细定位研究等提供理论依据。
1 材料与方法
1.1 构建作图群体及DNA提取
选择上海海洋大学洋山基地耐低盐选育凡纳滨对虾(EB)×(CE)为父本、厄瓜多尔野生凡纳滨对虾(Y)为母本,单尾交配,以亲本及F1代为作图群体用于构建遗传连锁图谱。2020年11月获得的F1代虾苗在上海海洋大学洋山基地进行养殖,并测量记录亲本的体长及体质量,剪取其附肢置于95%乙醇中,提取DNA,-80℃保存备用。F1代4月龄时随机选取150尾分别测量并记录其体长、体质量等信息,用于生长性状相关QTL分析。剪取肌肉组织分装于冻存管中,液氮速冻保存,运回实验室提取基因组DNA,采用1%琼脂糖凝胶电泳和NanoDrop 2000检测DNA质量和浓度,检测合格的DNA样品用于下一步测序。
1.2 2b-RAD文库构建及测序
采用2b-RAD测序方法:以2b-RAD五标签串联技术对2个亲本及150个F1子代个体的DNA进行测序并构建文库,所有样品均采用标准型5'-NNN-3'接头与酶切标签连接。基因组DNA采用IIB型限制性内切酶进行酶切,酶切产物分别加入5组不同接头,T4 DNA连接酶连接;通过PCR扩增连接产物,最后根据接头信息将5个标签按顺序进行串联;连接产物添加barcode序列,混合文库。文库制备完成后进行质量检测,通过Illumina Hiseq×Ten平台以Illumina nova为策略进行Paired-end测序。
1.3 数据筛选及基因分型
对原始数据进行质量控制,利用Pear v0.9.6将成对的Raw reads进行拼接,过滤删除含N碱基比例大于8%和低质量的Reads。依据建库时各样品所在位置,提取出各样品对应的Reads,过滤删除不含酶切识别位点的Reads,得到各样品的高质量测序Reads,即为Enzyme reads。利用SOAP将各样品的Enzyme reads比对至参考基因组(GCF_003789085.1)上进行数据分析,最后各样品数据经过滤和校正后,利用最大似然法进行SNP标记分型。原始SNP数据按以下标准进行筛选:(1)剔除等位基因只含有1种或大于2的位点;(2)剔除基因组碱基为N和同一位置存在2种分型的位点;(3)筛选掉样品中基因分型率低于80%的位点;(4)排除包含多个SNP的标签;(5)选择出等位基因频率大于0.05,缺失率小于0.2;(6)保留nn×np(母本杂合子)、lm×ll(父本杂合子)和hk×hk(双亲杂合子)的SNP类型。最后使用SnpEff对获得的SNP进行注释分析。
1.4 图谱构建
筛选父本杂合、母本纯合或杂合的位点构建父本图谱,筛选母本杂合、父本纯合或杂合的位点构建母本 图 谱,JoinMap 5.0设LOD(Logarithm of the odd)为2~15得到父母本图谱。经连锁群划分后对每个连锁群采用回归算法进行排序,运用Kosambi函数将重组率转换为遗传距离(cM);最终以MergeMap(http://138.23.178.42/mgmap/)将父母本图谱合并得到整合图谱。
1.5 QTL定位
对作图群体杂交F1代取样的同时,测量其体质量、全长、体长、头胸甲长、头胸甲宽、头胸甲高、第一腹节长、第一腹节宽、第一腹节高、第二腹节长、第二腹节宽、第二腹节高及尾节长共13个生长相关性状对应的表型数据。在SPSS 20.0中完成每个性状的极值、平均值、标准差及性状间的相关分析。其中体质量表型极端值较多,为使表型分布更接近正态分布,故对该性状去除极端值前后的表型分别进行分析,最终以14个性状为数据进行后续分析。结合得到的凡纳滨对虾高密度遗传图谱,利用MapQTL 6.0对生长性状进行QTL定位。数据导入后利用置换检验1000次重复,估算单个连锁群及基因组范围内α=0.05水平上的LOD阈值,然后以区间作图法进行QTL分析。
1.6 生长相关候选基因筛选
对获得的生长性状相关QTL区间标记上、下游1 kb内基因进行GO功能注释分析和KEGG信号通路富集分析,筛选出与凡纳滨对虾生长相关的候选基因。根据前期研究已知,在低盐条件下洋山耐低盐世代选育群体较对照群体生长更快。为了验证候选基因在凡纳滨对虾中的表达情况,以洋山耐低盐世代选育凡纳滨对虾群体为试验组,选择与其遗传关系较远的凡纳滨对虾群体为对照组,2个群体虾苗在上海海洋大学金山基地进行生长对比养殖试验。2个群体仔虾进行为期7 d的降盐试验后,各设3组平行放入盐度约为1‰的水泥池继续养殖,养殖期间各池条件保持一致。养殖100 d试验结束后,每池随机捞取50尾对虾测量其体长、体质量,并采集眼柄、肝胰腺、肌肉、鳃、肠道、胃组织等样品,液氮速冻后分别提取总RNA。以18S为内参基因,进行实时荧光定量PCR检测,扩增引物见表1,并根据2-ΔΔCt法换算凡纳滨对虾生长相关候选基因的相对表达量。对候选基因在凡纳滨对虾肝胰腺、眼柄、肌肉、鳃、肠道、胃组织中的相对表达量进行分析,筛选相对表达量最高的组织为材料,进一步研究候选基因在低盐条件生长速度不同群体对虾中的表达量情况。

表1 4个候选基因及内参基因的实时荧光定量PCR扩增引物序列信息Table 1 Primer sequence information of 4 candidate genes and reference gene in real-time quantitative PCR
2 结果与分析
2.1 2b-RAD测序结果
经2b-RAD测序共产生23.74亿条Raw reads,包括母本的1391万条Raw reads、父本的1391万条Raw reads及150个F1代的23.46亿条Raw reeds(平均每个子代1564万条Raw reads)。过滤后,选用20亿条Clean reads进一步分析,结果表明,亲本平均测序深度为17.64倍,F1代平均测序深度为13.60倍。所有样本的比对率均超过24.00%(表2)。
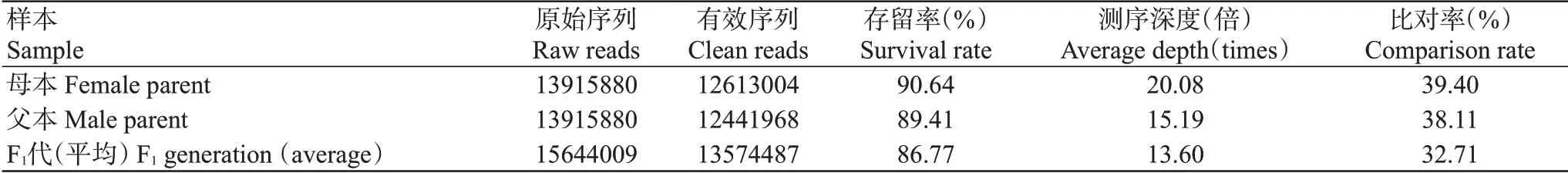
表2 凡纳滨对虾的2b-RAD测序结果Table 2 2b-RAD sequencing results of L.vannamei
2.2 SNP筛选及遗传连锁图谱构建情况
从2个亲本和150个F1代中初步筛选获得28836个SNPs标记,为增加分型准确率,进一步筛选后获得6577个SNPs标记。在这些SNPs标记中,划分出nn×np、lm×ll和hk×hk 3种分离类型。首先分别构建母本和父本各自的遗传连锁图谱,然后整合二者共有的SNPs标记以获得中性图谱(图1),最终构建的凡纳滨对虾遗传连锁图谱包括3136个SNPs标记,可划分为44个连锁群。总图谱长度为5430.54 cM,平均图距为1.73 cM(表3)。
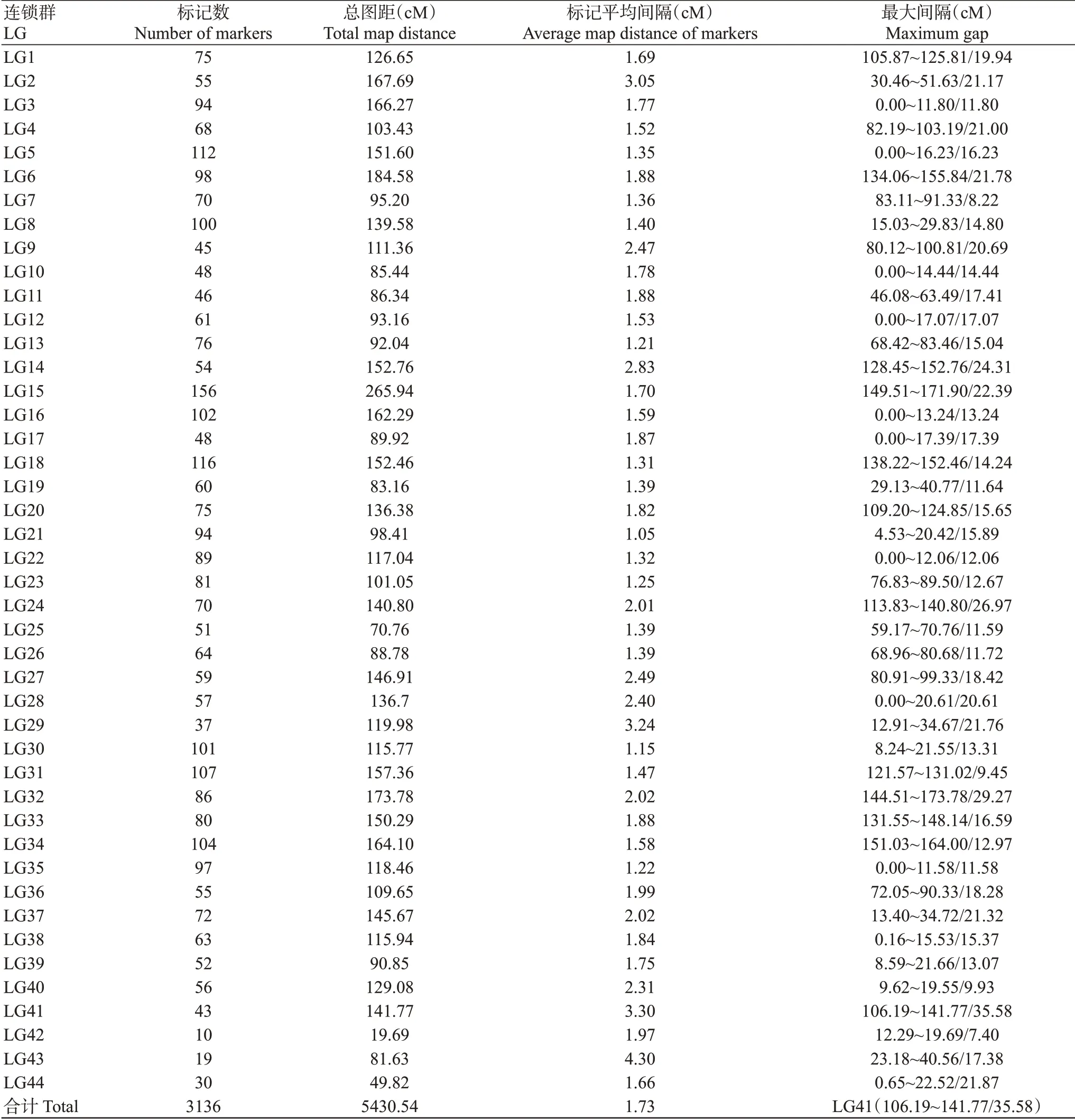
表3 凡纳滨对虾遗传连锁图谱信息统计结果Table 3 Statistics of L.vannamei genetic linkage map

图1 凡纳滨对虾遗传连锁图谱Fig.1 L.vannamei genetic linkage map
2.3 生长性状QTL定位结果
结合14个生长性状的表型数据,对所有作图群体个体进行QTL分析,最终在LG1、LG3、LG4、LG5、LG6、LG8、LG9、LG11、LG13等23个连锁群上共鉴定出79个QTLs(表4)。LOD范围为3.00~11.04,可解释的表型变异值范围为9.0%~28.8%,其中最主效的QTL为TZ_original.8。采用R语言LinkageMapView包,将QTL区间标注于遗传连锁图谱上,不同颜色代表不同性状,同颜色的标记分别为QTL区间的左右标记,图2为TZ.1、TZ.2、TZ.3和TZ.4共4个QTLs在遗传连锁图谱中的位置。

图2 体质量性状QTL在遗传连锁图谱中的位置Fig.2 Position of body mass trait-related QTL on the genetic linkage map
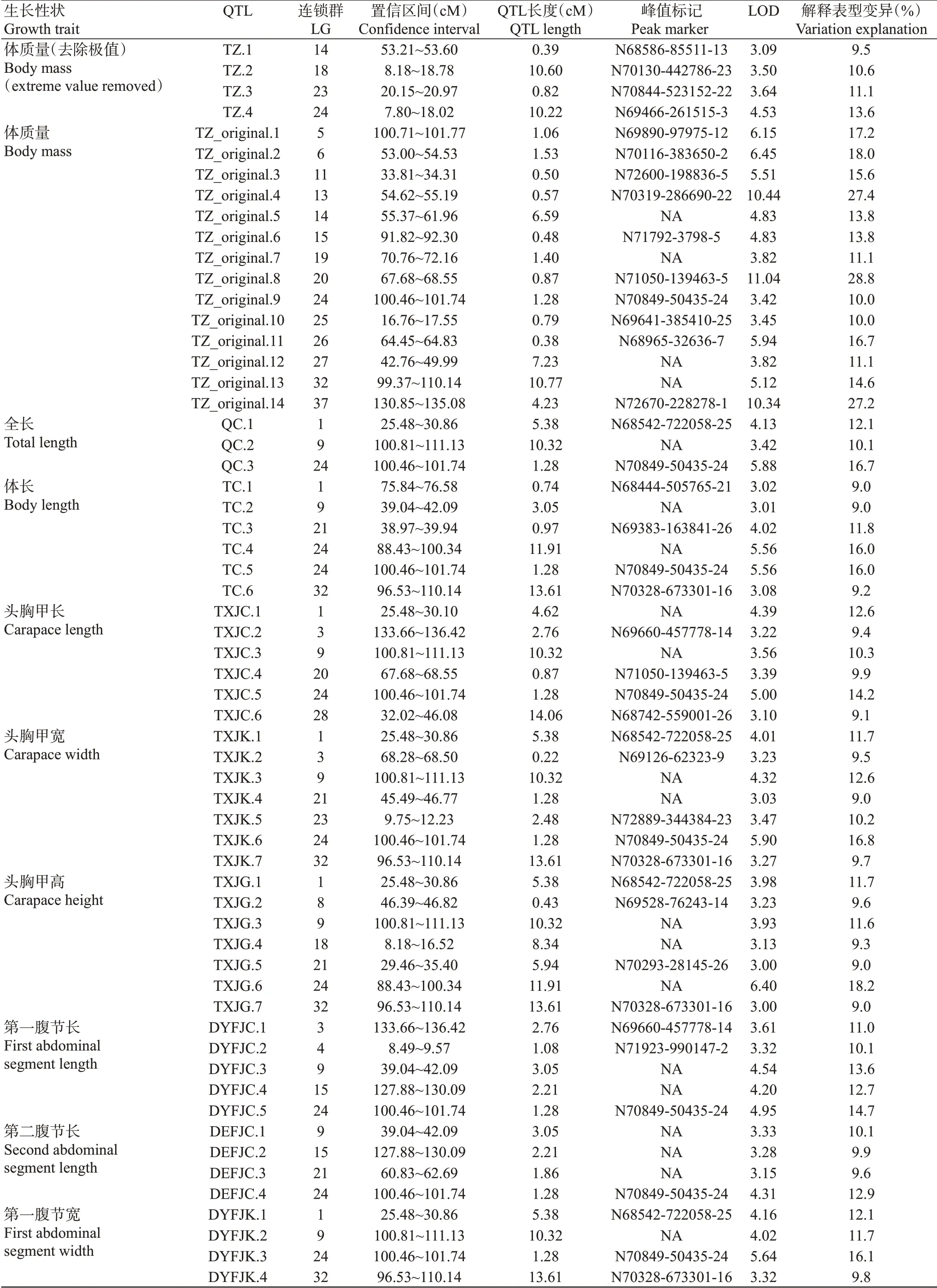
表4 凡纳滨对虾生长性状相关QTL定位信息Table 4 QTL mapping information of growth-related traits of L.vannamei
2.4 候选基因功能分析结果
从52个QTLs定位获得26个基因,GO功能注释分析发现这些基因主要注释到细胞组成、细胞过程、生长、代谢过程及生物调节等功能条目上,而KEGG信息通路富集分析结果表明主要富集在过氧化物酶、RNA降解、轴突指导、神经活性配体—受体相互作用等通路上。根据GO功能注释及KEGG信号通路富集分析结果,最终选定TOB2、CRAT、CCT6、KLF4共4个基因为凡纳滨对虾生长相关候选基因,具体基因信息见表5。通过为期100 d的凡纳滨对虾养殖试验,结果(表6)发现试验组凡纳滨对虾的终末体质量、终末体长、日增重率、体长日增长率及特定生长率均显著高于对照组凡纳滨对虾(P<0.05,下同),表明耐低盐世代选育凡纳滨对虾群体的生长情况优于常规的凡纳滨对虾群体。
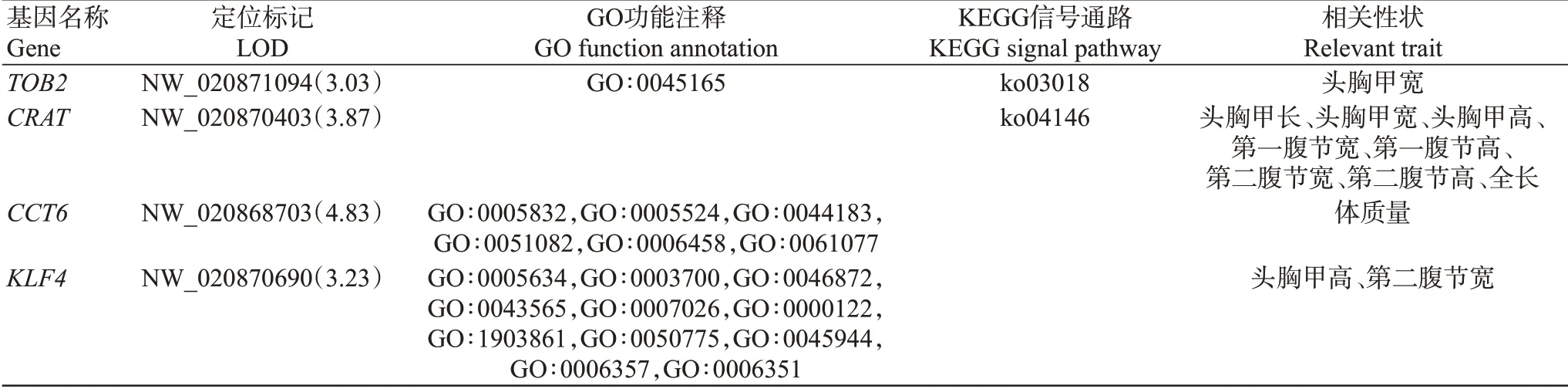
表5 凡纳滨对虾生长相关候选基因信息Table 5 Information of growth-related candidate genes of L.vannamei

表6 不同群体凡纳滨对虾养殖100 d后的生长指标Table 6 Growth indexes of L.vannamei of different populations at 100 d of culture
以18S为内参基因,采用实时荧光定量PCR检测4个候选基因在凡纳滨对虾眼柄、鳃、肠道、胃、肌肉和肝胰腺组织中的相对表达量。结果表明,4个候选基因在6种组织中均普遍表达(图3),其中,CRAT和TOB2基因在肝胰腺组织中的相对表达量最高,KLF4和CCT6基因则以肠道组织中的相对表达量最高。图4为4个候选基因在低盐条件不同生长速度凡纳滨对虾群体中的表达情况,CCT6、KLF4和TOB2基因在试验组凡纳滨对虾中的相对表达量高于对照组凡纳滨对虾,其中CCT6基因表达差异达显著水平;对照组凡纳滨对虾中的CRAT基因相对表达量略高于试验组凡纳滨对虾,但差异不显著(P<0.05)。

图3 4个候选基因在凡纳滨对虾眼柄、鳃、肠道、胃、肌肉及肝胰腺组织中的表达情况Fig.3 Expression of 4 candidate genes in eyestalk,gill,intestine,stomach,muscle,and hepatopancreas of L.vannamei

图4 4个候选基因在不同凡纳滨对虾群体中的表达情况Fig.4 Expression of 4 candidate genes in different L.vannamei populations
3 讨论
3.1 遗传连锁图谱
遗传连锁图谱已成为遗传学研究的强有力工具,广泛应用于目的基因定位、比较基因组及分子标记辅助育种等研究领域。与传统的遗传图谱构建方法(RAPD、AFLP和SSR)相比,2b-RAD技术在大规模SNP挖掘和基因分型方面具有高密度、高通量、高效率和低成本等优点;且2b-RAD测序中所有酶切片段均用于测序,从而减少片段选择导致的部分信息缺失,因其简单灵活的优点,已广泛应用于遗传图谱构建、群体遗传学及系统进化研究等。目前,应用2b-RAD技术已成功构建栉孔扇贝(Chlamys farreri)(Jiao et al.,2014)、白鲢(Hypophthalmichthys molitrix)(Wang et al.,2019)、红尾鲶(Hemibagrus wyckioides)(Zhou et al.,2021)等水生生物的高密度遗传连锁图谱。王星火等(2021)采用2b-RAD技术对三门湾海域3种优势鱼种进行群体分析,结果表明3种鱼类群体的遗传多样性总体上处于较低水平,应当加强资源保护,也进一步证实了2b-RAD技术用于群体多样性分析的可行性。
已构建的凡纳滨遗传连锁图谱作图群体均选用选育家系为亲本(表7),而本研究选择耐低盐家系与野生虾为亲本构建的F1代群体。野生群体较选育群体的杂合度和等位基因数更高,具有更丰富的多态信息含量。以往的研究主要利用RAPD、AFLP和SSR等方法建立凡纳滨对虾的遗传图谱,在图谱相邻标记间的平均距离为7.6~17.1 cM(Pérez et al.,2004;张留所,2006;Andriantahina et al.,2013)。本研究采用2个亲本及150个F1代共152个凡纳滨对虾个体,基于2b-RAD技术构建遗传连锁图谱,结果发现共有3136个SNPs标记定位在44个连锁群上,连锁群数目与已构建凡纳滨对虾图谱(Yu et al.,2015;Zeng et al.,2020)的连锁群数目相同,且与凡纳滨对虾单倍体染色体数目(Campos-Ramos,1997)相符合。本研究中的凡纳滨对虾遗传连锁图谱全长5430.54 cM,优于Yu等(2015)、Huang等(2020)所构建的遗传连锁图谱长度(4271.43和4161.55 cM),但精细程度不及这2张遗传连锁图谱,标记平均遗传距离(1.73 cM)相对更大。图谱精细程度存在差异的原因可能是构图群体不同导致遗传变异情况不同,另一方面可能是构图群体规模相对偏小。

表7 近十年凡纳滨对虾遗传连锁图谱研究进展Table 7 Advancements in L.vannamei genetic linkage maps in the recent decade
3.2 QTL定位
QTL定位分析可有效识别与动植物重要经济性状相关的分子标记,挖掘QTL,利用与目标性状紧密连锁的分子标记进行分子标记辅助选择育种,从而促进对性状的遗传改良。近年来,随着动物基因组研究的快速进展,遗传连锁图谱和QTL定位分析已广泛应用于水生生物研究领域。Song等(2012)利用94个半滑舌鳎F1子代定位获得4个生长相关QTLs;Xiao等(2015)选用大黄鱼72个F1子代,分别对体质量、全长和体高进行QTL定位,结果得到11个生长性状相关QTLs;Wang等(2016)利用106个牡蛎F1子代定位到5个生长性状的27个QTLs,分布于整合图谱的8个连锁群上。遗传标记可直接用于优良品种选育,QTL定位则为这些分子标记的识别提供了有效途径。本研究结合已构建的遗传连锁图谱,对与凡纳滨对虾生长相关性状(体长、体质量、头胸甲长、头胸甲宽、头胸甲高、第一腹节长、第一腹节宽、第一腹节高、第二腹节长、第二腹节宽、第二腹节高和尾节长等重要指标)进行QTL分析,探究性状间的相互关系,确定每个性状在连锁群上的位置及效应,以期找到与性状紧密连锁的分子标记,为下一步分子标记辅助选择育种奠定基础。结果表明,共有79个生长性状QTLs分散于44个连锁群上,其中3个QTLs(TZ_original.4、TZ_original.8和TZ_original.14)解释的表型变异率大于20.0%。Huang等(2020)对凡纳滨对虾体质量性状进行QTL定位,发现LOD平均值为2.74,PVE平均值为9.65。本研究中的凡纳滨对虾体质量相关QTL的LOD平均值(5.55)和PVE平均值(15.56),均高于Huang等(2020)的研究结果。在连锁群LG24上定位到所有生长性状相关QTL,其中体长性状有2个QTLs位于连锁群LG24上,表明在凡纳滨对虾育种中其体长、体质量、头胸甲长、头胸甲宽、头胸甲高、第一腹节长、第一腹节宽、第一腹节高等生长性状间存在较高的表型相关性,某个性状的选育均会影响另一性状。此外,本研究中的LOD值范围为3.00~11.04,体现出较高的定位水平,所定位的QTL均为主效QTL;可解释的表型变异率范围为9.0%~28.8%,则表明本研究定位的QTL较准确。
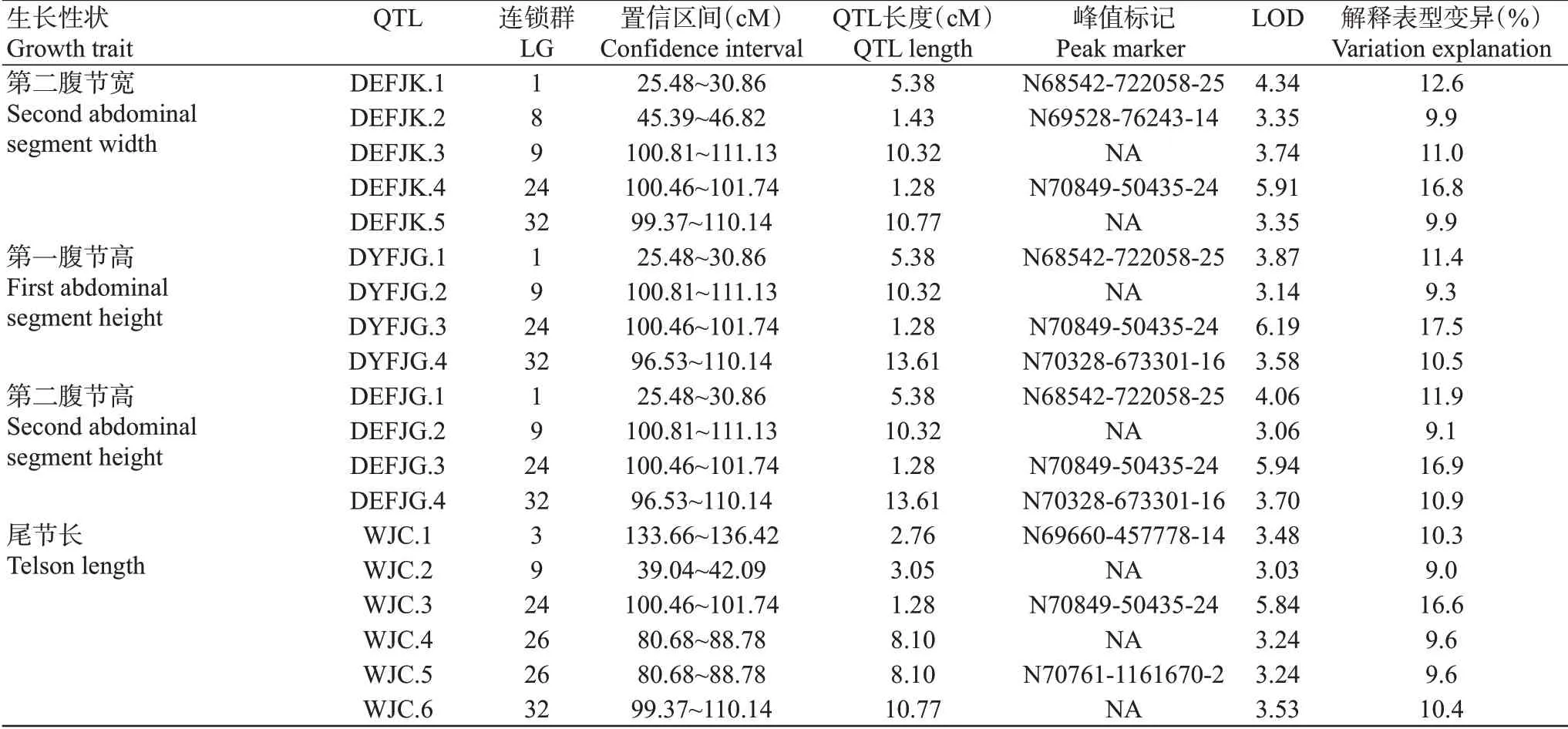
续表4凡纳滨对虾生长性状相关QTL定位信息Continued Table 4 QTL mapping information of growth-related traits of L.vannamei
3.3 候选基因筛选
数量性状相关候选基因可通过QTL定位进行挖掘。Huang等(2020)通过dd-RAD测序与QTL标记的对比,结合高pH转录组数据挖掘出HIF1B、TAF3、ENPEP、DDR2、SGMS1、H2AFV共6个凡纳滨对虾高pH耐受相关候选基因;Zeng等(2020)使用QTL分析及耐受氨氮能力最高和最低的凡纳滨对虾群体转录组进行分析,确定了与氨氮耐受相关的候选基因ATP5M;曹景龙和王卫民(2021)对团头鲂遗传图谱进行性别QTL定位,通过对QTL区间内SNP分子标记上、下游基因的筛选和功能注释,挖掘到DCTN2基因为团头鲂性别决定的重要候选基因;刘峰等(2022)以罗非鱼全同胞家系为对象,采用SSR分子标记鉴定出15个罗非鱼耐盐QTLs,发现NKCC2基因位于可解释19.1%雄性耐盐差异的QTL区间峰值处,即该基因可作为罗非鱼耐盐选育的重要候选基因。本研究通过GO功能注释和KEGG信号通路富集分析,分别从QTL区间TZ_original.6、QC.1、TXJK.4和TXJG.2中注释到CCT6、KLF4、TOB2和CRAT共4个候选基因。
T-复合多肽1ζ亚基(T-complex protein 1 subunit zeta,CCT6)基因编码含TCP1复合物的伴侣蛋白(Chaperonin containing TCP1 complex,CCT)。CCT是伴侣素家族的重要成员之一,发生异常时会影响细胞骨架的形成和降解。CCT6是一种参与肌动蛋白、微管蛋白和转导蛋白等各类蛋白结合及介导的伴侣蛋白(段芳蕾等,2011;杨春红等,2015)。在哺乳动物中,关于CCT6基因的研究已有较多报道,且推测该基因与肿瘤细胞的增殖相关(王廷峰等,2009),但在水产动物研究中鲜见报道。本研究结果表明,从体质量相关QTL(TZ_original.6)中定位到CCT6基因,该QTL可解释的表型变异为13.8%,LOD为4.83;在凡纳滨对虾眼柄、肝胰腺、鳃、胃、肠道、肌肉等组织中均能检测到CCT6基因表达,且以肠道中的相对表达量最高,其次是肝胰腺。肠道和肝胰腺作为消化吸收的主要场所,对凡纳滨对虾生长发育至关重要(姜永华等,2003;田娟等,2018)。本研究选择耐低盐世代选育凡纳滨对虾群体为试验组,与常规的凡纳滨对虾群体(对照组)在同一条件进行低盐养殖试验,养殖100 d后采集肠道组织检测CCT6基因表达情况,结果发现耐低盐世代选育凡纳滨对虾群体的CCT6基因相对表达量显著高于常规的凡纳滨对虾群体,且对虾生长指标也优于常规的凡纳滨对虾群体,故推测CCT6基因在凡纳滨对虾生长过程中发挥重要作用,但CCT6基因参与的具体功能和作用机理还需进一步探究。
Krüppel样 因 子4(Krueppel-like factor luna,KLF4)是转录因子Krüppel家族(KLFs)的一员,参与多种细胞增殖、分化和凋亡的调控,在哺乳动物的多种肿瘤细胞转移中发挥调节作用(龚海英等,2022)。KLF4基因在肠道中高表达,主要调节细胞的增殖与分化,可通过抑制凋亡来促进细胞生存,还参与细胞DNA损伤修复(杨帆和冯心河,2020)。本研究中,KLF4基因在凡纳滨对虾肠道组织中的相对表达量最高,提示KLF4基因可能对肠道组织细胞的增殖、分化产生影响。ErbB2转录因子2(Transducer of ErbB2,2,TOB2)编码的蛋白属于BTG/TOB增殖蛋白家族,该家族成员基因表达与细胞增殖和细胞分裂周期有关(何洪杰和于景翠,2015)。Suzuki等(2012)研究发现TOB基因有助于维持细胞的存活,降解时会导致DNA损伤,从而引发细胞凋亡;还有研究表明TOB2基因在斑马鱼(Shi et al.,2004;Xiong et al.,2006)、小鼠(Chen et al.,2015)的胚胎发育过程中发挥重要作用。本研究结果表明,TOB2基因在凡纳滨对虾6个不同组织中均有表达,其中以肝胰腺组织中的相对表达量最高,推测TOB2基因对肝胰腺组织细胞的增殖、分化产生影响。肉碱乙酰转移酶(Carnitine acetyltransferases,CRAT)是能量稳态和脂肪代谢的重要酶类,对于脂肪酸的β-氧化过程必不可少,因此在真核生物的能量代谢中发挥重要作用(Cordente et al.,2004;王会征和兰玉彬,2020)。CRAT基因功能可解释本研究中该基因在凡纳滨对虾肝胰腺中的相对表达量最高,与中华绒螯蟹肝胰腺组织中crat基因表达量最高的结果(刘丽等,2018)一致。本研究中,CRAT基因在不同凡纳滨对虾群体中的相对表达量差异不显著,可能是CRAT基因对低盐环境的响应不敏感。
4 结论
基于2b-RAD技术构建的凡纳滨对虾遗传连锁图谱鉴定出79个与生长性状相关的QTLs,并筛选出4个与凡纳滨对虾生长性状相关的候选基因(CCT6、KLF4、TOB2和CRAT)。可见,以2b-RAD技术结合QTL定位能高效、快捷挖掘出凡纳滨对虾生长性状相关候选基因,为开展分子标记辅助育种、生长相关功能基因精细定位研究等提供技术支持。
