半糯粳稻品种核心标记的筛选及DNA指纹图谱的构建
2022-02-02赵春芳赵庆勇吕远大陈涛姚姝赵凌周丽慧梁文化朱镇王才林张亚东
赵春芳,赵庆勇,吕远大,陈涛,姚姝,赵凌,周丽慧,梁文化,朱镇,王才林,张亚东
半糯粳稻品种核心标记的筛选及DNA指纹图谱的构建
赵春芳1,赵庆勇1,吕远大2,陈涛1,姚姝1,赵凌1,周丽慧1,梁文化1,朱镇1,王才林1,张亚东1
1江苏省农业科学院粮食作物研究所/国家耐盐碱水稻技术创新中心华东中心/江苏省优质水稻工程技术研究中心,南京 210014;2江苏省农业科学院种质资源与生物技术研究所,南京 210014
【目的】构建一套基于水稻重要性状基因标记的品种DNA指纹鉴定体系,为加强主栽优良食味半糯粳稻品种的种质管理与品种保护奠定基础。【方法】以34个江浙沪主栽优良食味半糯粳稻品种为供试材料,通过已知标记多态性鉴定、公共数据库基因序列比对、基因组重测序等多种方法对水稻重要性状调控基因的关键差异位点进行筛选,开发核心SNP或InDel标记;利用As-PCR技术将SNP标记发展为依据电泳条带鉴别的简单PCR标记,通过电泳条带特征化和类型分析获得基因型信息,构建半糯粳稻品种的DNA指纹图谱库。【结果】筛选出来源于40个水稻重要性状调控基因的54个核心标记,包括18个SNP标记和36个InDel标记;54个标记在20个品种中共检测到155个有效特征化条带,转化为155个0/1数据位点,建立各品种的DNA指纹数据库,可区分全部供试品种。遗传多样性分析表明,品种间遗传相似性变异范围为0.47—0.90,其中,南粳7718与苏香粳100的相似性系数最小,而南粳9308与南粳9036的相似性系数最大,二者存在8个数据位点差异。亲缘关系分析可将供试品种划分为6个分支,其中,南粳7718为独立分支,表明其与其他品种间的亲缘关系较远。核心标记鉴定效果的进一步验证表明,该套标记可以对14个半糯粳稻新品种进行有效区分,聚类图显示其分布于Ⅱ、Ⅲ和Ⅳ 3个类群,证实各品种间基因型信息的差异性;并利用该套标记鉴定了一个未知半糯水稻的品种真实性,根据基因型和聚类分析,将其判定为南粳9108。【结论】经优化筛选,获得可准确区分当前主推半糯粳稻品种的54个核心标记,并发展为可电泳检测的简单PCR标记,利用该套标记组合构建了34个江浙沪地区主栽优良食味半糯粳稻品种的DNA指纹图谱。
水稻;半糯粳稻;DNA指纹图谱;分子标记
0 引言
【研究意义】近年来,中国南方优质粳稻育种取得一定进展,培育出一系列低直链淀粉含量的优良食味粳稻品种,其直链淀粉含量介于糯稻与黏稻之间,一般含有低直链淀粉含量的等位基因(如Wx、Wx等),也称为“半糯粳稻品种”[1-2]。典型的代表性品种有:江苏省的南粳46、南粳9108、宁香粳9号、苏香粳100等,上海市的沪软1212、松香粳1018等,浙江省的嘉58、嘉农早香等[3-4]。这类半糯粳稻的米饭具有“软、香、糯、弹”的食味特点,符合长三角地区人们喜食偏软米饭的饮食习惯。半糯粳稻品种审定以来,在苏、沪、浙、皖等省/市得到迅速推广应用,已成为地产优质高档粳米的核心品种[5]。然而,在稻米产业发展过程中,种子套牌假冒、稻谷掺杂事件层出不穷,严重扰乱种子和稻米市场,影响种业创新、品牌创建,给种植者和经营者带来经济损失,同时,育种家们发现品种存在种性退化、食味品质下降等问题,进而影响了稻米品质和品牌的市场竞争力。因此,构建半糯粳稻品种的分子鉴定体系、建立各品种的DNA指纹图谱具有重要意义。【前人研究进展】DNA指纹图谱构建的关键在于获得核心分子标记并建立相应的检测标准,国际植物品种权保护联盟(International Union for the Protection of New Varieties of Plants,UPOV)在BMT测试指南草案中,将SSR和SNP确定为构建DNA指纹数据库的标记方法[6-7],其中,SSR标记因具有多态性好、技术简便快捷、共显性等特点,在水稻种质资源鉴定、遗传群体构建、基因定位及标记辅助育种等研究领域发挥了重要作用[8-9]。目前,中国水稻品种的审定监管和真伪鉴定主要依据行业标准《NY/T1433-2014 水稻品种鉴定技术规程SSR标记法》的48个SSR标记和《NY/T2745-2021 水稻品种真实性鉴定SNP标记法》的96个SNP。两套标记均是基于测序籼稻品种93-11和粳稻品种日本晴的序列变异在全基因组范围内进行多态性筛选而建立,对变异丰富的籼稻品种或遗传背景差异大的籼粳稻品种具有较高的鉴别能力[10-11]。特别地,农作物SNP标记因具有针对性强、变异丰富、数量巨大等特点[12],已逐渐取代了传统SSR标记,目前,在几乎所有研究物种中都被用作高效的遗传标记,构建了玉米、水稻、茶树等多种作物的DNA指纹图谱和分子身份证[13-14]。但是,两类标记在对省内栽培粳稻品种进行鉴别时,效果并不理想[15-16],表现出较低的基因多样性指数[17-19],表明栽培水稻品种间基因交流频繁,直接使用公共标记可能难以对其进行准确分子鉴定和指纹图谱构建。【本研究切入点】随着水稻功能基因组的发展,水稻品质、抗性、高产、株型等重要性状调控基因被陆续克隆和功能研究,性状关联的基因组特定差异位点也得以发现[20-21],并开发了相应PCR标记用于品种基因型检测[22-23]。但是,迄今为止,将这些特定变异系统地进行多态性筛选,并应用于近缘栽培水稻品种间分子鉴别上的研究尚少。【拟解决的关键问题】本研究以长江中下游地区主栽优良食味半糯粳稻品种为对象,利用水稻重要农艺性状关联的基因特异性SNP和InDel标记,构建一套快速、精准的品种DNA指纹鉴定体系,以期为半糯粳稻品种的真实性鉴定和品种种性稳定遗传提供技术支撑。
1 材料与方法
1.1 供试材料
供试材料选取34个目前江浙沪地域生产上推广应用的优良食味半糯粳稻品种(电子附表1),其中,江苏省农业科学院粮食作物研究所培育的南粳系列粳稻品种16个,江苏省地区研究所、南京农业大学、种业公司等培育的粳稻品种11个,来源于上海市的粳稻品种6个,浙江省嘉兴市农业科学研究院培育品种1个,D1—D20编号品种用于核心标记的筛选,D21—D34编号品种用于核心标记的验证。
1.2 DNA提取
所有试验材料的种子经浸种、催芽后,在光照培养箱中发芽成苗。采用CTAB法提取每个样品叶片的基因组总DNA[24]。利用超微量紫外可见分光光度计(Colibri)测定DNA浓度和质量,将工作液浓度统一调整为50 ng·μL-1。
1.3 InDel标记设计与筛选
目前,已有日本晴、9311、苏御糯、明恢63、桂朝2号等多个水稻品种全基因组数据公布(https:// www.ncbi.nlm.nih.gov/和http://rice.plantbiology.msu.edu/),通过对品质、产量、抗性、抽穗期等水稻重要性状控制基因编码区域序列的比对搜索,筛选插入缺失序列,在变异位点两侧合适位置分别设计InDel引物对。一些淀粉合成酶、稻瘟病抗性等基因的分子标记直接引自相关文献报道[22-23, 25-28]。合成的InDel标记经PCR扩增、PAGE凝胶电泳检测,以判断其扩增效果和在供试材料中的多态性,筛选获取有效标记。
1.4 重测序和已克隆基因SNP/InDel的筛选
对InDel标记无法区分的南粳46(D1)、南粳9108(D3)、南粳505(D4)、南粳56(D7)、南粳9308(D8)、南粳9036(D9)、沪早香软1号(D12)、金香玉1号(D14)等8个品种进行基因组重测序。按照Illumina的操作方法构建文库,再用Illumina HiSeq 2500 platform以平均每个样本20×测序深度进行重测序,以日本晴为参考基因组对所有Paired-end reads进行定位,并通过质量控制,删除MAF<5%,位点检出率<80%的SNP/InDel,得到196 314个SNP/InDel。根据变异位点在基因组中的分布位置可分为编码区、基因间区和上下游区三类,编码区变异率仅占15%左右,数量相对较少。由于待区分品种主要在抽穗期、株型等表型性状上存在差异,对这些关键性状的调控基因进行编码区SNP/InDel搜索,筛选获取功能位点,开发新的InDel和SNP标记。
1.5 SNP分型的PCR标记设计
当前SNP基因分型方法主要有基于凝胶电泳检测和高通量自动化分型2类[4],其中,等位基因特异性PCR(allele specific-PCR,As-PCR)是常用的基于凝胶电泳的SNP分型技术[29]。As-PCR标记通过在引物设计时将SNP碱基安排在引物序列3′末端,根据PCR扩增电泳条带的有无,判断靶序列的变异类型,直接获得SNP分型结果。通过下载编码区SNP两侧DNA序列,按照As-PCR标记四引物设计原则,在Tetra-Primer ARMS-PCR网站(http://primer1.soton.ac. uk/primer1.html)获取各SNP标记的引物序列,经PCR扩增、凝胶电泳检测四引物标记的效果。
1.6 电泳检测及条带特征化
18个SNP标记引物对的扩增产物在质量比浓度为2.0%琼脂糖凝胶上进行电泳,核酸染料染色。36个InDel标记引物对的扩增产物根据特征谱带的长度及类型选择不同类型凝胶,差异碱基数在10 bp以上,采用2.0%琼脂糖凝胶检测,差异碱基数不足10 bp,选择9.0%聚丙烯酰胺凝胶电泳检测。将电泳图上存在个体间差异的条带记为特征化条带,所有品种中共有条带记为非特征化条带。
1.7 数据分析
根据电泳图上的特征化条带,将每个品种中同一位置上清晰且可重复出现的条带记为1,无带记为0,建立原始数据矩阵。采用SLT_NT-sys_2.10软件中Similarity程序计算34个半糯粳稻品种的遗传相似性,以Clustering程序中SHAN进行UPGMA(非加权组平均法)聚类分析,绘制各品种间的亲缘关系聚类树状图。
2 结果
2.1 分子标记筛选
在多个水稻品种全基因组数据库中,下载品质、产量、抗性、抽穗期等水稻重要性状控制基因编码区域序列,通过序列比对搜索插入缺失位点,开发InDel标记,结合已开发的粒型、品质和抗稻瘟病基因标记[22-23, 25-28],对20个半糯粳稻品种(D1—D20)进行多态性检测,经标记优化,筛选到29个扩增效果好、多态性高的InDel标记。根据29个标记的电泳特征谱带对半糯品种进行类群划分,发现该套标记无法对南粳46、沪早香软1号、南粳9108、金香玉1号、南粳9036、南粳9308、南粳56、南粳505等8个品种有效地辨别。因而,将这8个品种进行基因组重测序和序列差异位点分析,重点对已克隆水稻重要性状调控基因的SNP进行检索,筛选到来源于17个基因的26个SNP或InDel功能位点。通过将编码区SNP转化为As-PCR四引物电泳标记,对半糯品种基因型进行鉴定。以29个InDel标记的品种分类结果为基准,通过逐步增加新SNP或InDel标记对各类群进行进一步区分,20个半糯粳稻品种被划分为更多的类群,直到把各类群分到仅有一个半糯粳稻品种为止,且保证最相似2个半糯粳稻品种间的差异标记数(差异标记数是指该标记在二者间扩增的特征化条带存在差异)占总标记数的5%以上。最终确定增加18个SNP标记和7个新InDel标记(InDel-1/InDel-2/InDel-9/InDel-10/ InDel-12/InDel- 14/InDel-16),可实现对20个半糯粳稻品种的相互区分。54个标记(36个InDel标记和18个SNP标记)来源于40个水稻重要性状调控基因,标记的引物序列及详细信息见表1。
2.2 核心标记的扩增效果及条带特征化
54个标记可以在20个半糯粳稻品种(D1—D20)中均得到良好的扩增效果,通过PCR扩增条带的凝胶成像,获得各品种的DNA特征条带。挑选符合目标产物大小的特征带型进行凝胶回收、序列分析,以确证条带正确性。以DNA Marker为对照,将DNA条带进行有效特征化,并按照SNP-1—SNP-18、InDel-1—InDel-36的顺序依次对特征化条带进行位点编号,图1和图2显示了部分标记的PCR扩增效果和DNA特征化条带。获得每个品种特征化信息即身份编号,说明54个标记可以实现对20个半糯粳稻品种的准确辨别。

M:DNA Marker;D1—D20分别对应20个半糯粳稻品种;*:有效特征化位点;×:无效位点

表1 54个标记的引物序列及详细信息

续表1 Continued table 1

续表1 Continued table 1

续表1 Continued table 1
2.3 DNA指纹图谱库构建
按照SNP-1—SNP-18、InDel-1—InDel-36顺序依次对特征化条带进行位点编号,共检测到155个特征化条带位点,将每个品种中同一位置上清晰且可重复出现的条带记为1,无带记为0,获得20个半糯粳稻品种的DNA特征条带图谱库,建立了各品种包含155个位点的DNA特征指纹数据库(表2)。
2.4 品种间亲缘关系分析
根据54个标记检测到的155个0/1数据矩阵,对20个半糯粳稻品种进行了遗传多样性分析。检测到各半糯粳稻品种间遗传相似性系数变异范围为0.47— 0.90,最小值(0.47)在南粳7718和苏香粳100间检测到,表明二者亲缘关系最远。最大值(0.90)在南粳9308和南粳9036间检测到,二者间8个数据位点存在差异,差异位点数占总位点数的5.2%(大于相似品种判定的临界值5.0%)。由UPGMA聚类图可知(图3),20个半糯粳稻品种(D1—D20)分别处于不同的分支,表明54个核心标记可以对其进行准确鉴别。以遗传距离0.34为阈值,34个品种可划分为4个类群,其中,南粳7718单独为第Ⅰ类群,表明与其他品种间的亲缘关系均较远,苏香粳3号、早香粳1号、嘉58等划分为第Ⅱ类群,南粳5055、南粳505、宁粳8号、徐稻9号等处于第Ⅲ类群,南粳46、沪早香软1号、南粳9108、金香玉1号等划分于第Ⅳ类群,同一单位选育品种(如南粳系列品种)未表现出聚集分布。
2.5 核心标记鉴定效果的验证
为进一步验证核心标记对其他半糯粳稻品种的区分效果,选取近年推广应用的14个半糯粳稻品种(D21—D34),利用54个标记对其进行PCR扩增和凝胶成像。14个品种均可得到良好的扩增效果,其条带大小均符合20个半糯粳稻品种(D1—D20)的DNA条带特征。按照SNP-1—SNP-18、InDel-1—InDel-36的顺序依次对特征化条带进行编号,获得14个半糯粳稻品种(D21—D34)的特征化信息。通过155个特征化条带的0/1数据读取,分别构建了14个品种(D21—D34)的DNA指纹数据库(表3),各品种间及其与D1—D20半糯粳稻品种间均存在多位点差异。从聚类图中可以看出,14个品种(D21—D34)分布于第Ⅱ、Ⅲ和Ⅳ等3个类群中,具有独立分支(图3),表明该套核心标记可以实现对市场主推半糯粳稻品种的准确鉴别。
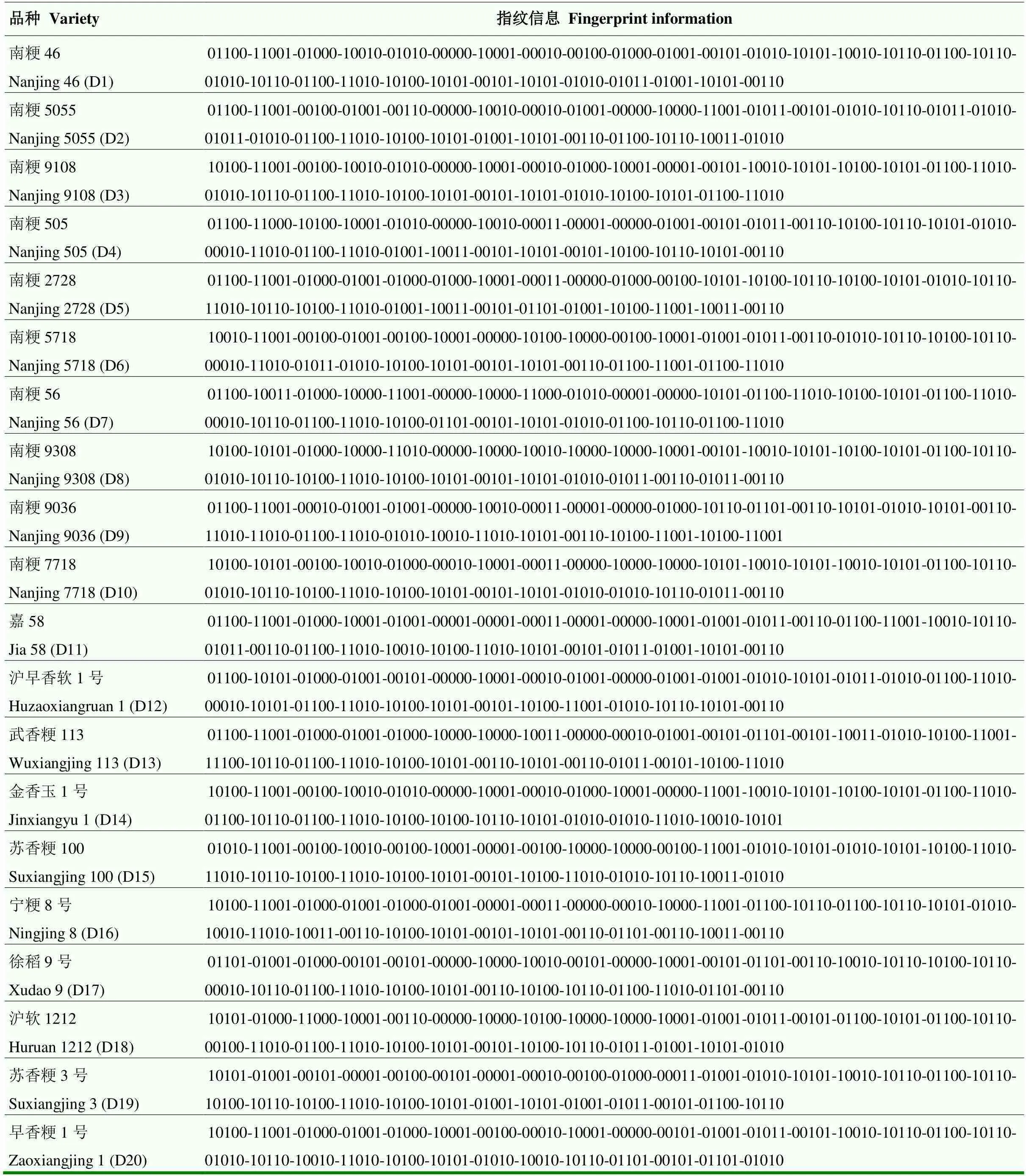
表2 20个半糯粳稻品种的DNA指纹数据库
2.6 品种真实性鉴定的案例分析
待鉴定水稻品种DX为34个品种中的一个疑似品种,以DX品种的叶片DNA为模板,利用54个标记进行PCR扩增、DNA条带特征化和0/1数据矩阵建立,获得DNA指纹数据库。将该指纹数据库导入已知半糯品种指纹数据库进行聚类分析,得到含有DX品种的35个品种的聚类图(图3)。从聚类图可以看出,DX品种与南粳9108位于相同位置,表明二者遗传相似性最高。通过DNA指纹数据比较,DX与南粳9108存在1个位点差异,占总位点数的0.65%,遗传相似度为99.5%。农业行业标准NYT1433-2014中将48个SSR标记中≥2个标记差异的品种认为是不同品种,按照标记比率计算约为5%差异;标准NY/T 2745-2021中将差异位点数≥4(总位点数为96),作为不同品种的判定标准,按照比例计算约为4.2%;在玉米及其他研究领域中将差异标记百分率5%作为品种真实性检测结果的判断标准[13];而在国家标准《GB/T 38551-2020 植物品种鉴定-MNP标记法》中将遗传相似度≥99%的2个品种,判定为相同品种或极近似品种。因此,判定待测水稻品种DX为南粳9108,符合品种真实性的判定标准。
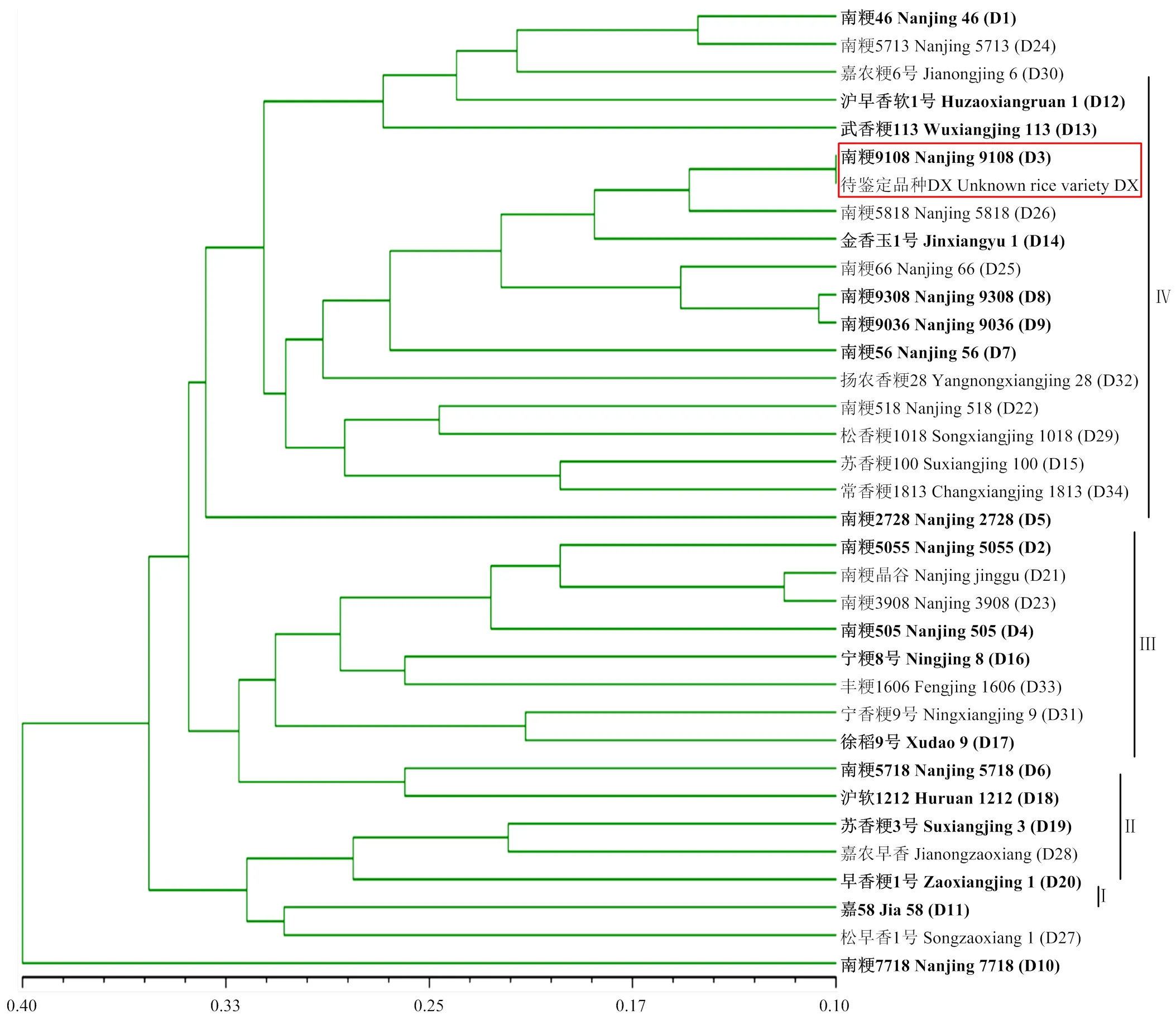
字体加粗的品种(D1—D20)为筛选核心标记所用的20个半糯粳稻品种,其他(D21—34)为14个其他半糯粳稻品种;红框表示对一个未知半糯粳稻品种DX的真实性鉴定

表3 14个其他半糯粳稻品种的DNA指纹数据库
3 讨论
3.1 核心标记的选择
目前,水稻品种真伪的分子鉴定较多研究中依据NYT1433-2014标准中的48个SSR标记[10-11, 30],但是该套标记在鉴定区域内栽培粳稻品种时的效果较差,表现出多态水平低、干扰条带多、区分能力差等问题。其主要原因可能是生产上主栽粳稻品种往往来源于相同的骨干亲本品种或者在同一品种基础上仅改良少数甚至单个性状的派生品种,造成品种间的遗传差异越来越小。本研究34份水稻品种为江浙沪地区的主栽优良食味半糯粳稻品种,其中,江苏省品种居多,仅江苏省农业科学院培育的南粳系列品种有16个,品种间携带相同低直链淀粉含量基因型[1, 31]。系谱分析表明品种间近亲繁殖系数高,遗传信息交流频繁,其中,有些品种是对已有品种某个性状的定向改良,如南粳9108是在南粳46中选育的早抽穗型自然变异株,金香玉1号是对南粳9108稻瘟病抗性的定向回交选育。本研究也尝试使用NY/T 2745-2021标准中96个SNP标记,但是,通过位点检索,多数SNP位点在半糯粳稻品种中表现无差异。因此,基因组随机分布的公共SSR标记或SNP标记均不能实现半糯粳稻品种间的分子鉴别。本研究选择了锚定品种间的差异农艺性状,进行特定性状调控基因的序列变异检索,通过分子标记的多途径开发、PCR优化和多态性筛选,最终鉴定到扩增效果良好的18个SNP标记和36个InDel标记,组成54个核心标记。
该套PCR标记来自40个水稻重要性状控制基因的核心序列,关联了品质(淀粉合成相关基因、分支酶基因、去分支酶基因)、香味、抗胁迫(抗病、抗胁迫)、株型、粒性等多方位的水稻关键性状。因此,在构建DNA指纹数据库的同时,也衔接了水稻重要性状的表型特征,并且标记序列主要来自于基因的编码区,与基因组其他位置序列同源性低,标记表现出错配率低、重复性好、稳定性高等良好的检测效果,完全可以满足区域内水稻品种的真伪和纯度鉴定,以及个性化DNA指纹图谱构建的需要。利用该套标记对34个半糯粳稻品种进行基因分型和统计分析,建立了每个品种唯一的基因型,实现了个体间的精准鉴别和区分,并可对未知品种准确地进行归类和判定。相比较而言,本研究开发的54个核心标记(PCR标记)明显优于NYT1433-2014中的48个SSR标记。另外,该套标记操作简便、使用成本低,特别适用于研究条件一般的实验室进行小样本量材料的鉴别。后续还可结合高效的SNP分型技术,如KASP、芯片定制、多重PCR等方法,进一步实现样本的规模化鉴定。
3.2 浙江沪主栽半糯粳稻品种间遗传差异
作物DNA指纹图谱的作用是区分不同生物个体,除对品种及其变异具有灵敏的鉴别能力以外,还可以根据DNA指纹的差异程度进行遗传聚类分析,掌握不同品种间的亲缘关系和遗传距离,帮助分析品种的特性[32-34]。目前,优良食味半糯粳稻品种在长江中下游地区逐年扩大推广,近三年仅在江苏省年种植面积占全省水稻种植面积的1/3以上,并且是地产优质稻米品牌打造的主力品种。因此,构建的DNA指纹图谱可直接用于指导生产上品种种子和市场上品种品牌稻米的真实性鉴定。基于本研究54个标记检测到155个多态性位点,品种间两两比较的遗传相似性系数为0.47—0.90,其中南粳品种间的相似性系数较高,如南粳9308和南粳9036、南粳晶谷和南粳3908间的遗传相似性系数为0.90和0.88,揭示了南粳品种的遗传基础较狭窄,这与它们拥有共同的育种亲本材料而直接相关。在DNA指纹图谱上,这两对品种间的标记差异位点数均在8个以上,大于差异率为5%“不同品种”的判定标准,说明本研究开发的核心标记用于当前长江中下游地区生产上主栽半糯粳稻品种的真实性鉴定和DNA指纹图谱构建是可行的。通过聚类分析,同一单位培育品种未表现出聚集分布,而是与其他单位培育的品种相互聚集,说明不同单位半糯粳稻品种间存在相互基因渗透,这与亲本材料的相互借鉴利用直接相关,如处于类群Ⅳ中的沪早香软、嘉农粳6号、苏香粳100均与南粳系列品种共用亲本南粳46或原始亲本关东194。值得注意的是,南粳7718为单独一个类群,表明与其他品种间的亲缘关系较远,即使与亲本材料南粳5055(早熟晚粳稻品种)仍有较大遗传距离,可能与选育过程中偏向选择早熟型的熟期类型(南粳7718为中熟中粳稻),而导致其遗传信息偏离;也有可能由另一亲本材料较远源的遗传关系而引起的。在鉴定一个由公司提供的未知半糯样品DX时,指纹数据库比较发现,其与真实品种南粳9108(原种)间仍存在1个位点差异、而非100%相似度,说明品种在公司繁育过程中,可能由于隔离不严格、操作不规范而致使种子发生混杂或天然杂交串粉;当然,如果是多年种质,也可能由于品种自身的分离变异而引起。研究结果进一步表明,该套鉴定体系可对当前使用的半糯粳稻种子的纯度及与原种间的遗传差异做出预判断。
本研究结果还可应用于水稻种质资源类群划分、分子育种及其他相关研究中,为水稻品种管理、种子和稻米市场监测、企业维权等提供检测技术;同时,为水稻分子育种中通过标记辅助选择加快优质高产高抗水稻新品种的选育进程奠定基础。
4 结论
筛选出来源于40个水稻重要性状基因的54个最佳PCR标记(18个SNP标记和36个InDel标记)组合,可精准对34个浙江沪地区主栽优良食味半糯粳稻品种进行区分。54个标记共检测到155个有效特征化条带,组成34个品种的DNA指纹图谱库,并生成每个品种的0/1指纹数据。
[1] 赵春芳, 岳红亮, 田铮, 顾明超, 赵凌, 赵庆勇, 朱镇, 陈涛, 周丽慧, 姚姝, 梁文化, 路凯, 张亚东, 王才林. 江苏和东北粳稻稻米理化特性及和基因序列分析. 作物学报, 2020, 46(6): 75-85.
ZHAO C F, YUE H L, TIAN Z, GU M C, ZHAO L, ZHAO Q Y, ZHU Z, CHEN T, ZHOU L H, YAO S, LIANG W H, LU K, ZHANG Y D, WANG C L. Physicochemical properties and sequence analysis ofandgenes inrice cultivars from Jiangsu province and northeast of China. Acta Agronomica Sinica, 2020, 46(6): 75-85. (in Chinese)
[2] 王才林, 张亚东, 陈涛, 朱镇, 赵庆勇, 姚姝, 赵凌, 赵春芳, 周丽慧, 魏晓东, 路凯, 梁文化. 姊妹系间杂交快速培育优良食味半糯粳稻新品种的育种效果. 中国水稻科学, 2021, 35(5): 455-465.
WANG C L, ZHANG Y D, CHEN T, ZHU Z, ZHAO Q Y, YAO S, ZHAO L, ZHAO C F, ZHOU L H, WEI X D, LU K, LIANG W H. Rapid breeding of new semi-glutinousrice varieties with good eating quality by crossing between sister lines. Chinese Journal of Rice Science, 2021, 35(5): 455-465. (in Chinese)
[3] 王才林, 张亚东, 朱镇, 赵凌, 陈涛, 林静. 优质水稻新品种南粳46的选育与应用. 中国稻米, 2008(3): 38-40.
Wang c l, zhang y d, zhu z, zhao l, chen t, lin j. Breeding and application of a new high quality rice variety Nanjing 46. China Rice, 2008(3): 38-40. (in Chinese)
[4] 张丽霞, 费剑华, 周锋利, 王凯, 胡泽军, 曹黎明, 闫影, 吴书俊. 优良食味粳稻新品种‘沪软1212’的选育及其品质特性. 分子植物育种, 2021, 19(13): 4443-4448.
ZHANG L X, FEI J H, ZHOU F L, WANG K, HU Z J, CAO L M, YAN Y, WU S J. Breeding and quality characteristics of a new high eating qualityrice variety ‘Huruan1212’. Molecular Plant Breeding, 2021, 9(13): 4443-4448. (in Chinese)
[5] 王才林, 张亚东, 赵春芳, 魏晓东, 姚姝, 周丽慧, 朱镇, 陈涛, 赵庆勇, 赵凌. 江苏省优良食味粳稻的遗传与育种研究. 遗传, 2021, 43(5): 442-458.
WANG C L, ZHANG Y D, ZHAO C F, WEI X D, YAO S, ZHOU L H, ZHU Z, CHEN T, ZHAO Q Y, ZHAO L. Inheritance and breeding ofrice with good eating quality in Jiangsu province. Hereditas, 2021, 43(5): 442-458. (in Chinese)
[6] 徐云碧, 王冰冰, 张健, 张嘉楠, 李建生. 应用分子标记技术改进作物品种保护和监管. 作物学报, 2022, 48(8): 1853-1870.
XU Y B, WANG B B, ZHANG J, ZHANG J N, LI J S. Enhancement of plant variety protection and regulation using molecular marker technology. Acta Agronomica Sinica, 2022, 48(8): 1853-1870. (in Chinese)
[7] 滕海涛, 吕波, 赵久然, 徐岩, 王凤格, 堵苑苑, 杨坤, 唐浩, 李祥羽. 利用DNA指纹图谱辅助植物新品种保护的可能性. 生物技术通报, 2009(1): 1-6.
TENG H T, Lü B, ZHAO J R, XU Y, WANG F G, DU Y Y, YANG K, TANG H, LI X Y. DNA fingerprint profile involved in plant variety protection practice. Biotechnology Bulletin, 2009(1): 1-6. (in Chinese)
[8] 李茂柏, 王慧, 白建江, 朴钟泽. 利用SSR分子标记构建水稻品种DNA指纹图谱的研究进展. 中国稻米, 2011, 17(1): 4-6.
LI M B, WANG H, BAI J J, PIAO Z Z. Research progress in constructing DNA fingerprints of rice varieties using SSR molecular markers. China Rice, 2011, 17(1): 4-6. (in Chinese)
[9] 杨梦婷, 黄洲, 干建平, 徐君驰, 庞基良. SSR分子标记的研究进展. 杭州师范大学学报(自然科学版), 2019, 18(4): 429-435.
YANG M T, HUANG Z, GAN J P, XU J C, PANG J L. Research progress of SSR molecular markers. Journal of Hangzhou Normal University (Natural Science Edition), 2019, 18(4): 429-435. (in Chinese)
[10] 李梓榕, 袁雄, 陈叶, 郑兴飞, 胡中立, 李兰芝. 基于全基因组SNP高效鉴定水稻种质资源并构建指纹图谱. 分子植物育种, 2020, 18(18): 6050-6057.
LI Z R, YUAN X, CHEN Y, ZHENG X F, HU Z L, LI L Z. Effective identification for varieties by genome-wide SNPs and establishment of fingerprint for rice germplasm. Molecular Plant Breeding, 2020, 18(18): 6050-6057. (in Chinese)
[11] 郑向华, 叶俊华, 程朝平, 魏兴华, 叶新福, 杨窑龙. 利用SNP标记进行水稻品种籼粳鉴定. 作物学报, 2022, 48(2): 342-352.
ZHENG X H, YE J H, CHENG Z P, WEI X H, YE X F, YANG Y L.identification by SNP markers inL.. Acta Agronomica Sinica, 2022, 48(2): 342-352. (in Chinese)
[12] 王富强, 樊秀彩, 张颖, 刘崇怀, 姜建福. SNP分子标记在作物品种鉴定中的应用和展望. 植物遗传资源学报, 2020, 21(5): 1308-1320.
WANG F Q, FAN X C, ZHANG Y, LIU C H, JIANG J F. Application and prospect of SNP molecular markers in crop variety identification. Journal of Plant Genetic Resources, 2020, 21(5): 1308-1320. (in Chinese)
[13] 田红丽, 杨扬, 王璐, 王蕊, 易红梅, 许理文, 张云龙, 葛建镕, 王凤格, 赵久然. 兼容型maizeSNP384标记筛选与玉米杂交种DNA指纹图谱构建. 作物学报, 2020, 46(7): 1006-1015.
TIAN H L, YANG Y, WANG L, WANG R, YI H M, XU L W, ZHANG Y L, GE J R, WANG F G, ZHAO J R. Screening of compatible maizeSNP384 markers and the construction of DNA fingerprints of maize varieties. Acta Agronomica Sinica, 2020, 46(7): 1006-1015. (in Chinese)
[14] 樊晓静, 于文涛, 蔡春平, 林浥, 王泽涵, 房婉萍, 张见明, 叶乃兴. 利用SNP标记构建茶树品种资源分子身份证. 中国农业科学, 2021, 54(8): 1751-1760.
FAN X J, YU W T, CAI C P, LIN Y, WANG Z H, FANG W P, ZHANG J M, YE N X. Construction of Molecular ID for Tea Cultivars by Using of Single nucleotide Polymorphism (SNP) Markers. Scientia Agricultura Sinica, 2021, 54(8): 1751-1760. (in Chinese)
[15] 朱勇良, 谢裕林, 黄凌哲, 吴建祥, 乔中英, 王建平, 黄萌, 陈培峰. 太湖稻区及国内部分香稻SSR指纹图谱构建及遗传多样性初析. 植物遗传资源学报, 2012, 13(4): 666-671.
ZHU Y L, XIE Y L, HUANG L Z, WU J X, QIAO Z Y, WANG J P, HUANG M, CHEN P F. Establishment of SSR fingerprint map and preliminary analysis of genetic diversity among aromatic rice from Taihu and other areas in China. Journal of Plant Genetic Resources, 2012, 13(4): 666-671. (in Chinese)
[16] LI Z J, CHEN Y H, ZHANG D J, ZHANG G F, LU B X. Genetic diversity analysis and DNA fingerprinting of the main japonica rice varieties in Heilongjiang province. Quality Assurance and Safety of Crops & Foods, 2018, 11(1): 23-29.
[17] 左示敏, 周娜娜, 陈宗祥, 许明, 许学宏, 毛从亚, 汤义华, 张亚芳, 叶凯, 顾世梁, 潘学彪. SSR标记在江苏粳稻品种鉴定中的应用研究. 扬州大学学报(农业与生命科学版), 2014, 35(4): 46-51.
ZUO S M, ZHOU N N, CHEN Z X, XU M, XU X H, MAO C Y, TANG Y H, ZHANG Y F, YE K, GU S L, PAN X B. Identification ofrice varieties released in Jiangsu province by SSR markers. Journal of Yangzhou University (Agricultural and Life Science Edition), 2014, 35(4): 46-51. (in Chinese)
[18] 龚红兵, 余波, 盛生兰, 潘学彪, 曾生元, 李闯, 左世敏, 景德道, 林添资, 陈宗祥. 江苏主栽粳稻品种的遗传与食味结构分析. 作物学报, 2016, 42(7): 1083-1093.
GONG H B, YU B, SHENG S L, PAN X B, ZENG S Y, LI C, ZUO S M, JING D D, LIN T Z, CHEN Z X. Genetic analysis and taste quality of main conventionalrice varieties grown in Jiangsu province. Acta Agronomica Sinica, 2016, 42(7): 1083-1093. (in Chinese)
[19] 贾裕, 王道一, 沈秋萍, 孙海燕, 罗兵. 太湖地区水稻育种亲本遗传多样性分析及指纹图谱构建. 常熟理工学院学报, 2020, 34(5): 80-85.
JIA Y, WANG D Y, SHEN Q P, SUN H Y, LUO B. Genetic diversity analysis and fingerprint construction of rice breeding parents in the Taihu lake region. Journal of Changshu Institute of Technology, 2020, 34(5): 80-85. (in Chinese)
[20] LI Y, XIAO J H, CHEN L L, HUANG X H, CHENG Z K, HAN B, ZHANG Q F, WU C Y. Rice functional genomics research: past decade and future. Molecular Plant, 2018, 11: 359-380.
[21] 张昌泉, 冯琳皓, 顾铭洪, 刘巧泉. 江苏省水稻品质性状遗传和重要基因克隆研究进展. 遗传, 2021, 43(5): 425-441.
ZHANG C Q, FENG L H, GU M H, LIU Q Q. Progress on inheritance and gene cloning for rice grain quality in Jiangsu province. Hereditas, 2021, 43(5): 425-441. (in Chinese)
[22] 田志喜, 刘巧泉, 汤述翥, 李家洋, 顾铭洪, 严长杰, 钱前, 严松, 谢会兰, 王芳. 水稻淀粉合成相关基因分子标记的建立. 科学通报, 2010, 55(26): 2591-2601.
TIAN Z X, LIU Q Q, TANG S Z, LI J Y, GU M H, YAN C J, QIAN Q, YAN S, XIE H L, WANG F. Development of gene-tagged molecular markers for starch synthesis-related genes in rice. Chinese Science Bulletin, 2010, 55(26): 2591-2601. (in Chinese)
[23] YAN C, TIAN Z, FANG Y, YANG Y, LI J, ZENG S, GU S, XU C, TANG S, GU M. Genetic analysis of starch paste viscosity parameters in glutinous rice (L.). Theoretical and Applied Genetics, 2011, 122: 63-76.
[24] MURRAY M G, THOMPSON W F. Rapid isolation of high molecular weight plant DNA. Nuclear Acids Research, 1980, 8: 4321-4326.
[25] WANG Z, JIA Y, RUTGER J N, XIA Y. Rapid survey for presence of a blast resistance genein rice cultivars using the dominant DNA markers derived from portions of thegene.Plant Breeding, 2007, 126: 36-42.
[26] 刘洋, 徐培洲, 张红宇, 徐建第, 吴发强, 吴先军. 水稻抗稻瘟病基因的分子标记辅助选择与应用. 中国农业科学, 2008, 41(1): 9-14.
LIU Y, XU P Z, ZHANG H Y, XU J D, WU F Q, WU X J. Marker-assisted selection and application of blast resistant genein rice. Scientia Agricultura Sinica, 2008, 41(1): 9-14. (in Chinese)
[27] SHOMURA A, IZAWA T, EBANA K, EBITANI T, KANEGAE H, KONISHI S, YANO M. Deletion in a gene associated with grain size increased yields during rice domestication.Nature Genetics, 2008, 40(8): 1023-1028.
[28] 王军, 杨杰, 陈志德, 仲维功. 水稻香米基因标记的开发与应用. 分子植物育种, 2008, 6(6): 1209-1212.
WANG J, YANG J, CHEN Z D, ZHONG W G. Development and application of fragrance gene markers in rice. Molecular Plant Breeding, 2008, 6(6): 1209-1212. (in Chinese)
[29] MYAKISHEV M V, KHRIPIN Y, HU S, HAMER D H. High throughput SNP genotyping by allele-specific PCR with universal energy-transfer-labeled primers. Genome Research, 2001, 11(1): 163-169.
[30] 王明湖, 张孝天, 吴国林, 蒋琪, 施勇烽. SSR标记在宁波地区水稻品种DNA指纹图谱构建及纯度鉴定中的应用. 中国稻米, 2009, 25(6): 50-54.
WANG M H, ZHANG X T, WU G L, JIANG Q, SHI Y F. DNA fingerprints construction and purity identification based on SSR markers for rice varieties in Ningbo city. China Rice, 2009, 25(6): 50-54. (in Chinese)
[31] 田铮, 赵春芳, 张亚东, 赵庆勇, 朱镇, 赵凌, 陈涛, 姚姝, 周丽慧, 梁文化, 路凯, 王才林, 张红生. 江苏省半糯型粳稻蒸煮食味品质性状的差异分析. 中国水稻科学, 2021, 35(3): 249-258.
TIAN Z, ZHAO C F, ZHANG Y D, ZHAO Q Y, ZHU Z, ZHAO L, CHEN T, YAO S, ZHOU L H, LIANG W H, LU K, WANG C L, ZHANG H S. Differences in eating and cooking quality traits of semi-waxyrice cultivars in Jiangsu province. Chinese Journal of Rice Science, 2021, 35(3): 249-258. (in Chinese)
[32] LU B R, CAI X X, XIN J. Efficientandrice identification based on the InDel molecular method: its implication in rice breeding and evolutionary research. Progress in Nature Science, 2009, 19: 1241-1252.
[33] EBANA K, YONEMARU J, FUKUOKA S, IWATA H, KANAMORI H, NAMIKI N, NAGASAKI H, YANO M. Genetic structure revealed by a whole-genome single-nucleotide polymorphism survey of diverse accessions of cultivated Asian rice (L.). Breeding Science, 2010, 60: 390-397.
[34] WANG C H, ZHENG X M, XU Q, YUAN X P, HUANG L, ZHOU H F, WEI X H, GE S. Genetic diversity and classification ofwith emphasis on Chinese rice germplasm. Heredity, 2014, 112: 489-496.
Screening of Core Markers and Construction of DNA Fingerprints of Semi-WaxyRice Varieties
ZHAO ChunFang1, ZHAO QingYong1, LÜ YuanDa2, CHEN Tao1, YAO Shu1, ZHAO Ling1, ZHOU LiHui1, LIANG WenHua1, ZHU Zhen1, WANG CaiLin1, ZHANG YaDong1
1Institute of Food Crops, Jiangsu Academy of Agricultural Sciences/East China Branch of National Center of Technology Innovation for Saline-Alkali Tolerant Rice/Jiangsu High Quality Rice R&D Center, Nanjing 210014;2Institute of Crop Germplasm and Biotechnology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014
【Objective】A set of variety DNA fingerprint identification system based on the core markers of genes regulating rice important traits was constructed, which will establish a foundation for strengthening the germplasm management and protection of the mainly promoted semi-waxyrice varieties with high eating quality.【Method】34 semi-waxyrice varieties mainly cultivated in Jiangsu, Zhejiang and Shanghai were used as the test materials. The key differential sequence sites in genes regulating rice important traits were screened and core SNP or InDel markers were developed through multiple methods such as polymorphism testing of existing markers, gene sequence alignment from public databases and genome resequencing. SNP markers were developed into simple PCR markers based on electrophoretic bands by As-PCR technology. Genotype information was obtained by electrophoretic band characterization and type analysis, and the DNA fingerprint database of the semi-waxyrice varieties was constructed. 【Result】 54 core markers derived from 40 key genes regulating rice important traits were obtained, including 18 SNP and 36 InDel markers; 155 characteristic and effective bands were identified by 54 markers in the tested rice varieties, which were transformed into 155 0/1 data sites. The DNA fingerprint database of each variety was established and could distinguish it from all tested varieties. Genetic diversity analysis showed that the variation range of genetic similarity among varieties was 0.47-0.90, among which the lowest similarity coefficient was detected between Nanjing 7718 and Suxiangjing 100, while the highest similarity coefficient was detected between Nanjing 9308 and Nanjing 9036, among which there were 8 differential data sites. Genetic relationship analysis indicated that 34 varieties were divided into 6 branches, of which Nanjing 7718 was an independent branch, suggesting it has a distant relationship from other varieties. Further verification of the identification effect of core markers showed that the set of markers could effectively distinguish 14 new semi-waxyrice varieties. The cluster diagram showed that they were distributed in three groups of Ⅱ, Ⅲ and Ⅳ, confirming the differences of genotype information among varieties; using this set of markers, the authenticity of an unknown semi-waxy rice variety was also identified. According to genotype and cluster analysis, it was determined as Nanjing 9108.【Conclusion】After optimization and screening, 54 core markers that could accurately distinguish all the tested semi-waxyvarieties were obtained, and developed into simple PCR markers detected by electrophoresis. Using this set of marker combinations, the DNA fingerprints of 34 semi-waxyvarieties in Jiangsu, Zhejiang and Shanghai were constructed.
rice (L.); semi-waxyrice; DNA fingerprints; molecular markers

10.3864/j.issn.0578-1752.2022.23.001
2022-07-25;
2022-09-09
江苏省农业科技自主创新资金(CX[20]2002)、江苏省重点研发计划(BE2021301)、现代农业产业技术体系建设专项资金(CARS-01)
赵春芳,E-mail:czhao@jaas.ac.cn。通信作者张亚东,E-mail:zhangyd@jaas.ac.cn
(责任编辑 李莉)
