低温胁迫下巴西橡胶树三倍体全长转录组测序分析
2022-01-18毛常丽张凤良李小琴
毛常丽,李 玲,张凤良,李小琴,杨 湉,吴 裕
(云南省热带作物科学研究所,云南景洪666100)
低温寒害一直是限制我国橡胶树种植面积扩大和越冬安全的主要因子,抗寒性状也一直是我国橡胶树良种培育的主要选择指标之一。根据《NY/T607-2002橡胶树选育种技术规程》中的育种程序,选出一个优良无性系一般需要25~30年。云南省热带作物科学研究所培育的三倍体无性系“云研77-2”和“云研77-4”具有抗寒、高产特性,在云南省累积推广种植160 000 hm2。这两个品种从1974杂交到2000年良种审定经历了26年(后经细胞学鉴定为三倍体)。一般认为,树木三倍体具有良好的速生、抗逆和丰产性,实际上只能说从三倍体中更有机会获得性状突出的个体。橡胶树自然三倍体的发生率特别低,要想通过传统的育种程序选出优良三倍体就更不容易。
转录组学是从RNA水平研究基因表达的情况,是研究细胞表型和功能的重要手段。转录组测序技术(RNA-Seq)通过深度测序对样品中的转录因子进行分析,能够反映出转录水平上的基因表达情况,其已被广泛应用于植物种质评价、性状筛选等方面。笃斯越橘(Vaccinium uliginosum)、厚朴(Magnolia officinalis)、枇杷(Eriobotrya japonica)、樱桃(Prunus avium)[1-4]等木本物种的转录组学抗逆性研究为橡胶树抗逆性转录组学的研究提供了新的思路。
目前,橡胶树转录组学主要基于第二代高通量测序平台的RNA-Seq技术对病害、产胶、生殖发育等[5-7]方面进行研究,且用于测序的材料为二倍体,而对三倍体材料进行的转录组测序还未见报道。基于田间鉴定[8]、生理指标测定[9-11]等结果已确定三倍体品种云研77-2和云研77-4抗寒性比其母本GT1强,但抗寒性增强是来自于母本的2n配子中抗寒效应基因的加倍或是其他原因则还未见报道。如果能探清其抗寒机制,将有助于三倍体抗寒品种的培育。本研究以云研77-4为材料作全长转录组测序及分析,旨在得到完整的转录组序列,为橡胶树三倍体分子辅助选择及抗寒三倍体育种奠定理论基础。
1 材料和方法
1.1 实验材料
材料选择为橡胶树三倍体无性系“云研77-4”,其亲本为GT1×PR107;苗木以GT1开放授粉种子苗为砧木芽接培育;选择长到第3蓬叶且顶蓬叶稳定,整株无病虫害,长势一致的苗木,准备实施低温胁迫处理。
1.2 方法
在4℃人工气候箱下处理2 h、6 h和20 h,对照为不处理(0 h),每处理设3次重复。处理完后剪取第2个叶蓬中下部复叶的中间小叶,并迅速用液氮冻干后转移至-80℃冰箱保存备用。先对各样品分别提取RNA,再将不同处理的RNA混合,对混合样进行测序。样品提取、文库构建及质控、转录组测序及分析委托北京百迈客生物科技有限公司进行。参考已报道的橡胶树全基因组序列对本研究的转录组序列进行注释。
2 结果与分析
2.1 数据产出及统计
云研77-4冷处理后的样品经PacBio三代转录组测序,共获得41.85 Gb clean data的数据,按照full passes>=3且序列准确性大于0.9的筛选条件,从原始数据中提取到环形一致序列(Circular Consensus Sequencing,CCS)504 032条,平均每条序列的长度为2 157 bp。对CCS序列进行长度分布分析,得到其长度分布范围主要为1 800~2 200 bp(图1)。过滤掉CCS序列中不包含5’引物、3’引物及polyA尾的非全长序列,共得到全长非嵌合序 列(Full-length Non-Chimeric sequence,FLCN)413 878条,占总CCS序列的82.11%。

图1 橡胶树三倍体三代转录组全长测序CCS长度分布
2.2 FLCN的一致序列(consensus isoform)分析
使用SMRTLink软件中的IsoSeq模块对FLNC序列进行相似性序列聚类,得到一致序列157 638条,从其中筛选出准确度大于99%的高质量转录本154 484条,平均每条一致序列长度为2 181 bp,其consensus isoform序列长度分布如图2。用GMAP(Genomic Mapping and Alignment Program)将校正后的一致序列与橡胶树参考基因组进行序列比对,用cDNA_Cupcake软件将比对后的序列进行去冗余处理,过滤identity小于0.9,coverage小于0.85的序列,共得到非冗余转录本序列96 437条。对非冗余的转录本进行可变剪接分析,检测到基因位点23 307个,其中新基因位点4 288个,新发现转录本70 055个。为了探究更多的转录组信息,同时对三倍体橡胶树中的融合转录本进行了分析,共得到融合转录本1 983个。

图2 consensus isoform长度分布
2.3 新转录本分析
2.3.1 SSR分析
从新发现的转录本中筛选500 bp以上的转录本,利用MISA软件进行SSR分析,结果见表1。对检测到的SSR进行类型划分,划分为完美单碱基重复(p1)、完美双碱基重复(p2)、完美三碱基重复(p3)、完美四碱基重复(p4)、完美五碱基重复(p5)、完美六碱基重复(p6)和混合SSR(c,即包含至少两个完美SSR,且之间距离小于100 bp),并对其进行密度统计,统计结果见图3。
由表1、图3可知,橡胶树新转录本中存在6种类型的SSR,每种重复类型的数量差异较大,主要以单碱基重复为主,且在所有的碱基重复中,以完美单碱基重复类型最多。
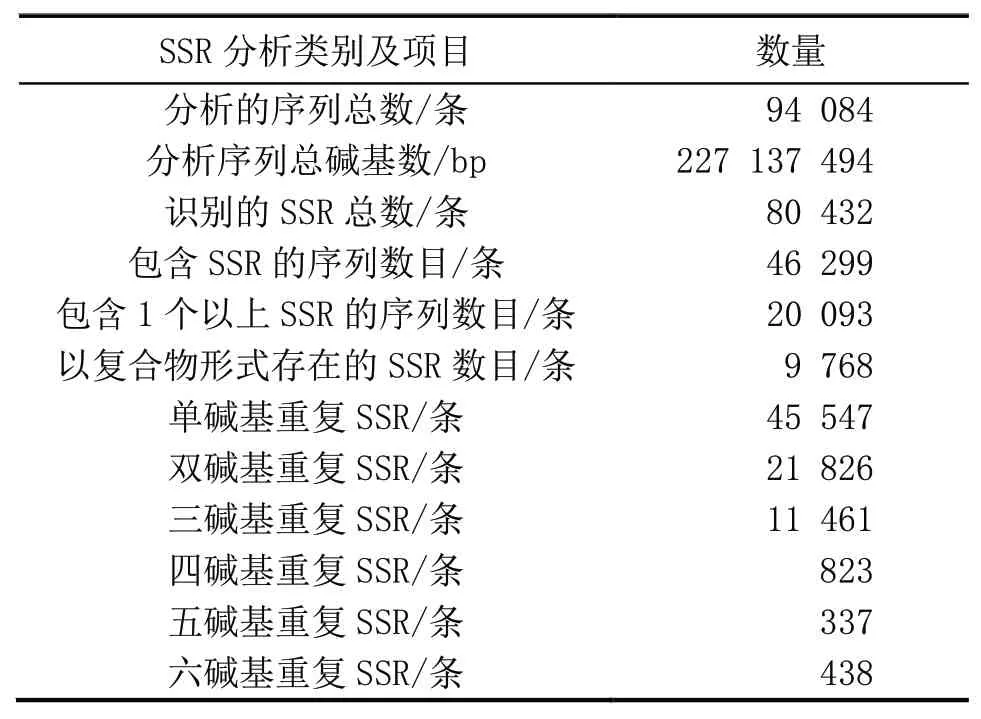
表1 橡胶树新转录本SSR分析

图3 橡胶树三代转录组新转录本SSR类型分布图
2.3.2 新基因编码区序列预测
对可变剪接分析中得到的新转录本使用TransDecoder软件[12]预 测 其编 码 区 序 列(Coding Sequence,CDS)及其对应的氨基酸序列,共获得开放阅读框(Open Reading Frame,ORF)66 182条,其中完整ORF57 516条。在获得的ORF中,0~500 aa的有54 475条,占82.31%,500~1 000 aa的有10 537条,占15.91%,1 000~2 300 aa的有1 167条,占1.76%(图4)。

图4 橡胶树转录组预测的CDS编码蛋白长度分布
2.3.3 长链非编码RNA(Longnon-coding RNA,lncRNA)预测及转录因子分析
通过对转录本中不编码蛋白的lncRNA进行编码潜能筛选,从而判定该转录本是否为lncRNA。利用CPC[13]、CNCI[14]、pfam、CPAT[15]四种分析方法对新发现的转录本进行lncRNA预测,分别预测到5 598、7 164、15 207、17 488个lncRNA,其中4种方法均预测到的lncRNA共3 575个(图5)。同时,根据lncRNA在参考基因组注释信息上的位置对lncRNA进行分类绘图,结果见图6。由图6可知,本研究材料中的lncRNA主要以lincRNA为主,占53.19%,其次为sense_lncRNA,占27.30%。

图5 四种筛选方法维恩图

图6 lncRNA位置分类
LncRNA的存在为下一步研究橡胶树表观遗传、转录水平及转录后水平调控基因的表达提供了新的思路。
转录因子是指能够结合在某基因上游特异核苷酸序列上的蛋白质,这些蛋白质可以调控RNA聚合酶与DNA模板的结合,从而调控基因的转录。使用iTAK软件[16]对三倍体橡胶树三代测序转录因子进行预测,共预测到转录因子12 064个,且预测到的转录因子包括了在其他物种中有报道的RLK-Pelle_DLSV、C3H、NAC、bHLH、MYBrelated及RLK-Pelle_LRR-VIII-1等 类 型。C3H、bHLH、MYB-related等转录因子在植物抗逆、生长发育及有效成分合成方面均有报道,这为今后开展橡胶树相关研究提供了重要信息。同时,为了更直观地呈现全长转录组测序所得到的注释序列在全基因组上的分布密度及全长转录组测序的完整性,我们将注释到的转录本与全基因组进行了Circos可视化绘图(图7)。从图7可知,本研究材料全长转录组序列中的基因密度、转录本密度和全基因组中的基因密度、转录本密度相当,lncRNA在各条染色体上均有分布。
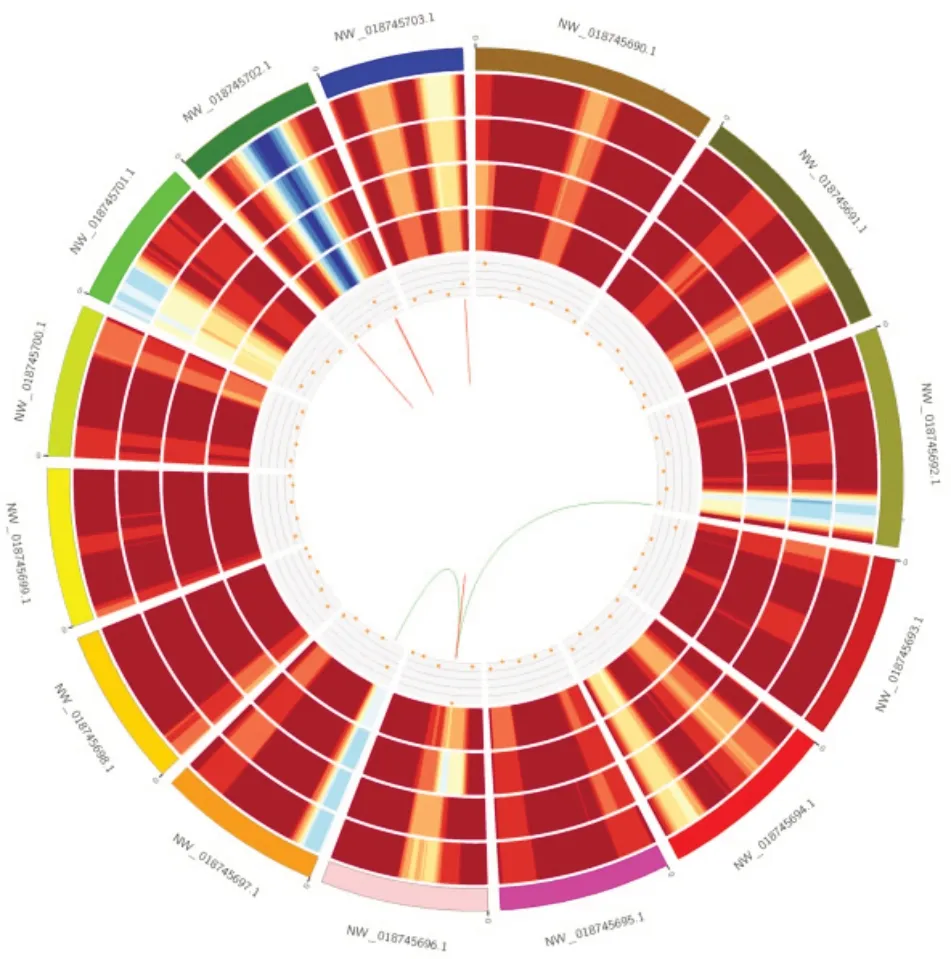
图7 基因组水平Cicros可视化图
2.4 新转录本功能注释
为探清三倍体橡胶树新转录本的功能,用BLAST软件(version 2.2.26)[17]将得到的新转录本与NR[18]、Swissprot[19]、GO[20]、COG[21]、KOG[22]、Pfam[23]、KEGG[24]数据库比对,进行新转录本功能注释,共注释到功能基因66 684个。7个数据库注释到的转录本数量见表2。
从表2可以看出,7个功能数据库中注释到的转录本长度大部分在1 000 bp以上,这也进一步说明全长转录组测序能测得较长的序列。综合7个功能数据库中注释到的转录本,其功能分类主要涉及细胞组分、分子功能及生物学过程三大方面,这为抗寒性相关基因的筛选奠定了基础。

表2 7个数据库注释到的转录本数量
3 讨论
转录组是研究动植物生命过程的有效方法之一,随着测序手段的不断更新,近几年发展起来的三代全长转录组测序因其能读取更长的序列而被广泛应用于植物的生长发育、非生物胁迫等领域。笔者所在单位云南省热带作物科学研究所在国际上首次获得了达到染色体级别的高质量巴西橡胶树优良品种GT1的参考基因组序列[25]这为本研究的转录组注释、组装提供了重要参考。
橡胶树转录组测序已经应用于自根幼态无性系体胚发育[26]、橡胶树丛枝病机理[5]及苗期生长优势[27]等方面,并从中筛选了相关的调控基因,为橡胶树分子辅助育种奠定了重要的理论基础,但是现有的报道均基于Illumina HiSeq的二代转录组测序技术,且测序的样品均为二倍体。本研究通过PacBio三代转录组测序技术对三倍体橡胶树抗寒无性系进行测序,共产生41.85 Gb clean data的数据,分析得到高质量转录本154 484条,平均每条序列长度为2 181 bp,数据得量、转录本总数以及单条转录本序列长度均大于先前报道的橡胶树二代转录组数据[28-29]。将得到的数据分析后得到新转录本70 055条。对94 084条转录本进行分析,发现了潜在的80 432个SSR分子标记,高于Wirulda等[30]用454高 通 量 测 序 技 术 获 得 的24 000个SSRs,这些标记可广泛应用于橡胶树种质资源评价、分子辅助育种、抗逆性鉴定等。利用CPC、CNCI、pfam、CPAT四种方法预测到共有的lncRNA 3 575个,同时对新转录本进行了NR、Swissprot、GO、COG、KOG、Pfam、KEGG功能注释,共注释到功能基因66 684个,高于前人注释到的37 432个功能基因[28]。转录因子可与基因5’端上游特定序列专一性结合,从而保证目的基因在特定的时间与空间进行表达。本研究中共预测到转录因子12 064个,其中包含了与抗逆性相关的MYB、C3H转录因子,下一步将从中筛选出与三倍体橡胶树抗寒性相关的转录因子,为研究三倍体抗寒性提供参考。从全长转录组数据和全基因组数据Circos可视化图可以看出,全长转录组数据在全基因组测序得到的数据覆盖率相当,说明全长转录组序列可靠,可为下一步二代转录组序列的拼接、组装及注释提供参考。
4 结论
本研究基于三代测序技术建立了三倍体橡胶树的转录组数据库,并对序列进行分析,为后续的橡胶树分子辅助育种、二代转录组数据组装,特别是三倍体橡胶树的转录组分析提供重要的基础,下一步将继续从lncRNA、转录因子等方面分析三倍体橡胶树抗寒性标记及强抗寒性的分子机制,为三倍体选育种提供有用标记及参考。
